交互融合特征表示与选择性集成的DNA结合蛋白识别方法与流程
本发明涉及生物学与信息学交叉领域,特别是涉及一种利用机器学习预测dna结合蛋白的方法。
背景技术:
dna结合蛋白在各种细胞过程中发挥着极其重要的作用,在理解和解释蛋白质功能中,识别dna结合蛋白是一个非常重要的任务。从蛋白质序列(一级结构)出发,利用机器学习方法对蛋白质的结构和功能进行预测,是目前生物信息学研究的热点问题,也是一种重要研究手段。
基于机器学习的dna结合蛋白的预测方法有两大类:基于蛋白质结构的预测;基于蛋白质序列的预测。基于蛋白质结构预测dna结合蛋白能得到较高的识别率,然而,由于没有足够的蛋白质结构信息,这类方法无法被广泛应用在高通量序列的诠释中。因此,目前的方法更多的是基于氨基酸序列的蛋白质功能预测。大量实验已经表明,蛋白质一级结构(氨基酸残基排列顺序)或多肽相似,其折叠后的空间构象与其功能也很相似,基于这个事实,利用蛋白质序列预测蛋白质功能,包含两个主要过程:1)提取蛋白质序列中包含的生物信息,把蛋白质序列转化为相应的特征向量;2)利用得到的特征向量,使用机器学习中的算法,进行模型训练并对未知序列做功能识别。
在已过的几十年间,基于蛋白质序列的有效特征表示方法,主要包括有:1)基于氨基酸组成的方法,这类方法考虑了相邻的且连续的氨基酸残基间的信息;2)基于伪氨基酸组成的方法,这类方法考虑了非相邻(不连续)氨基酸残基间的信息;以及3)基于蛋白质频率谱的方法,这类方法考虑了蛋白质的进化信息。基于氨基酸组成方法(aac),使用序列的统计信息,如常用的k-mers方法,这类方法简单,但所生成特征维数较高(20k),存在维灾和过拟合问题。基于伪氨基酸组成方法,由kuo-chenchou提出并命名为pseaac,它考虑了序列的局部顺序和全局顺序,能够较好的表达序列中的顺序与位置信息,该方法能将序列的位置信息映射到所生成特征向量中。基于蛋白质频率谱的方法,使用携带有进化信息的位置特异性得分矩阵(pssm:positionspecificscoringmatrix),该矩阵表达了与其比对序列相关的同源物信息。
研究表明进化信息、物化属性以及序列的结构与位置等信息,对dna结合蛋白的识别均具有一定的作用。如果仅仅采用氨基酸组成信息或者蛋白质频率谱等单个信息的特征表示方法,所生成识别特征都显得过于单一。目前在相关文献中主流的做法是,考虑不同的属性(如不同的蛋白质物化属性)和信息(如进化信息与结构信息等),并对这些方法生成的特征向量进行组合,所生成的高维特征向量作为后继分类器的输入。我们把这类方法称为组合式融合特征表示(cffr:combinedfusionfeaturerepresentation),它将氨基酸的物化属性、进化信息的频率谱以及序列信息(相邻和不相邻残基信息)进行组合,能够取得不错的预测性能。然而,这类方法把物化属性与进化信息等均视为彼此独立的特征进行组合,忽略了它们之间还应该存在着交互效应,并且利用这种交互效应能够更进一步提高dna结合蛋白的预测性能。
技术实现要素:
传统的特征表示把不同方法生成的特征,视为彼此独立的特征进行组合,忽略了这些特征之间还应该存在着交互效应。为了提高对dna结合蛋白的预测能力,针对蛋白质序列数据,本发明提出交互融合的特征表示与选择性集成分类器:交互融合的特征表示,能够考虑物化属性与进化信息之间的交互效应,和非相邻残基间的位置信息,充分挖掘隐藏在蛋白质序列背后的潜在的生物信息,生成具有强判别能力的特征;选择性集成分类器,是通过对特征表示的参数进行扰动,生成不同的输入特征空间,并使用选择(或修剪)策略得到具有差异性的基分类器,投票集成得到具有强泛化能力的整体分类器。本发明能够显著提高对dna结合蛋白的预测能力,同时本发明的特征表示也有利于从交互作用的视角去理解dna结合蛋白在细胞中的功能与作用。
本发明所解决的技术问题是提供交互融合特征表示与选择性集成的dna结合蛋白识别方法。相比于现有的方法,本发明方法具有更加卓越的性能,这也间接表明本发明的交互融合特征表示能够生成携带有强判别信息的特征,同时选择性集成还能进一步提升整体学习器的泛化能力,最终能够保证对dna结合蛋白的准确预测。
附图说明
图1.dna结合蛋白预测模型的框架图
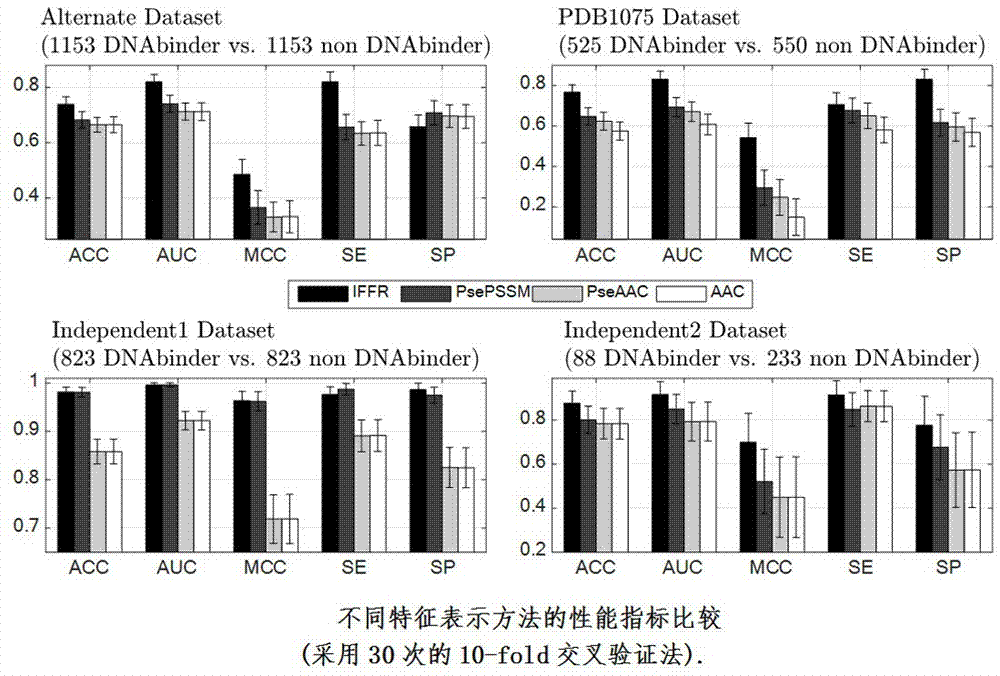
图2.不同特征表示方法的性能指标(acc,auc,mcc,sensitivityandspecificity)比较(采用30次的10-fold交叉验证法).
具体实施方式
在机器学习实际应用中,通常认为“数据和特征决定了机器学习的上限,而模型和算法能够逼近这个上限”。因此,本发明同时从这两方面着手:1)对多种生物信息进行有效融合,生成具有强判别能力的特征;2)对多个分类器进行选择集成,生成具有强泛化能力的分类器。图1给出我们的预测模型框架,包括交互融合特征表示和选择性集成分类器。左边(虚线框)是交互融合特征表示,右边(虚线框)是选择性集成分类器。
1)交互融合特征表示
特征表示,是根据序列中的数学关系以及生物化学属性等指标,将由字符组成的序列,数值化成一个固定维数的特征向量。针对蛋白质序列数据,本发明先给出一种新的交互融合特征表示模型,该模型能够同时考虑不种物化属性和进化信息自身内部的相关性,以及物化属性与进化信息之间的交互效应。
进化信息的得分矩阵pssm,即位置特异性得分矩阵(position-specificscoringmatrix),它是一个行数为l(l为序列长度)列数为20(20类标准氨基酸)的矩阵。蛋白质数据搜索程序psi-blast,能够通过多次迭代寻找最优结果,对于寻找蛋白家族的新成员或者发现远亲物种的相似蛋白非常有效,使用它能够生成一个位置特异得分矩阵pssm:
元素
物化属性得分矩阵pcsm,本发明给出氨基酸的物化属性得分矩阵(physicochemicalscoringmatrix,)。在对dna结合蛋白的识别过程中,我们假设不同氨基酸物化属性对预测结果将产生不同的贡献,因此,在特征表示过程中必须考虑合适的氨基酸物化属性。aaindex是一个包含多个氨基酸物理化学属性的氨基酸指数表,其中aaindex1部分的每一项表示氨基酸的某种物理化学属性量化后的数据,含有20个数值。对于第j种物化属性q(j),任一条蛋白质序列s可表示为
跳空距离为λ的得分矩阵λ-gapsm,考虑到蛋白质序列中不同距离的氨基酸残基之间存在着相互作用,借鉴伪氨基酸组成(非相邻残基)分析思想,给出λ-gap得分矩阵,给定得分矩阵
为λ-gap得分矩阵,其中aλ=(aij)(l-λ)×l为(0-1)矩阵,
aij∈{0,1},即
其中参数λ(1≤λ≤l-1)表示矩阵aλ中任一行向量ai中两个非零元1之间的距离(λ-gap)。特别地,当λ=0时,a0退化为单位矩阵il,也即0-gap得分矩阵
g0=a0p=ip=p
λ-gap得分矩阵间接刻画了序列中不相邻残基之间(跳空距离为λ)的位置信息。
给定长度为l的蛋白质序列,有pssm矩阵p和pcsm矩阵q,水平拼接得到矩阵w=(p,q)=(wij)l×(m+20),由上面λ-gap得分矩阵定义,可得λ-gap得分矩阵
gλ=aλw=aλ(p,q)=aλp,aλq
由协差矩阵和分块矩阵运算,容易得到,
对上式方阵∑按列拉直运算(matrixvecoperator),保留满足i≤j的元素的元素σij,则所得向量为
v=vec(σ)=(σ1,1,σ1,2,σ2,2,…,σ1,m+20,σ2,m+20,…,σm+20,m+20)。
显然该向量的维数仅与m有关,而与l(序列长度)和λ(跳空距离)无关。
本发明的特征表示模型中,分别利用了物化属性q和进化信息p各自本身所蕴含的相关性信息
当λ=0时,特征表示算法λ-gapiffr仅考虑了序列的不同物化属性和进化信息,算法1退化为二信息交互式融合iffr;特别地,当λ=0时,且算法1的第4行w=p时,算法1退化为基于进化信息的特征表示,记为covpssm;当λ=0时,且算法1的第4行w=q时,算法1退化为基于物化属性的特征表示,记为covpcsm;传统的做法是,将这两种特征表示covpssm和covpcsm所生成的特征向量进行组合式串联,记为cffr。
2)选择性集成分类器
给定蛋白质序列集,随机划分训练集strn,验证集sval和测试集stst。假设
理论上,最优基分类器子集t*可通过穷举法得到。然而,当l较大时,穷举法的计算量太大。一种简单直观的选择策略是:对基分类器ci按性能指标m进行排序,选取前k(奇数)个基分类器构成的子集t*做为对集成分类器t的修剪,并对子集
为了验证本发明的有益效果,选取4个dna结合蛋白序列数据进行分析,它们的样本容量相对较充足(≥300),同时它们又都是序列相似性小于40%的数据集,这些能保证实验结果的可信性。表1给出数据的汇总信息与数据来源1。
表1用于实验验证的基准数据集(蛋白质序列数据集)
系统评估本发明的预测性能,分别采用jackknife校验法和k-fold交叉校验法(k-foldcv)对本发明进行比较和评估。其中k-foldcv能够有效降低由于数据不充分而造成的过学习和欠学习状态的发生,在实践中,10-foldcv被认为是标准方法;jackknife校验法被认为是较客观的统计校验方法,它能够避免由于训练和测试数据的随机划分而造成的随机性,保证实验结果的可复制性。
对算法性能的评估指标有:预测准确率(acc:accuracy)、敏感性(se:sensitivity)、特异性(sp:specificity)和综合评价预测结果的相关性系数mathews相关系数(mcc:mathewscorrelationcoefficient),详细定义如下:
其中,tp(真阳性)表示dna结合蛋白被预测为dna结合蛋白的个数,tn(真阴性)表示非dna结合蛋白被预测为非dna结合蛋白的个数,fp(假阳性)表示非dna结合蛋白被错误预测为dna结合蛋白的个数,fn(假阴性)表示dna结合蛋白被错误预测为非dna结合蛋白的个数。
acc表示预测结果中真阳性与真阴性之和在总测试实例中的百分比;se表示真阳性在所有预测为阳性测试数据中的百分比;sp表示真阴性在所有预测为阴性测试数据中的百分比。对于完美的预测系统,这三指标都应该达到100%。然而,对于非平衡数据集,若se增加时,则sp必然下降,反之亦然,这些指标不能很好的评估预测结果,相比较mcc是个更平衡的评估标准,其取值范围在[-1,+1]之间,值为1表示预测结果与真实类别完全相关,值为0表示是完全随机的预测,值为-1表示完全相反的相关性。另外,roc曲线图中曲线下面积(areaunderthecurve,auc)可以作为更加客观的分类性能评估标准。roc曲线图是一个单位平方,两坐标轴(真阳性率和假阳性率)的数值从0到1,auc最大值为1,对应于完美分类器。
必须指出的是,以下用于比较的实验结果均是使用基分类器:线性核svm(参数默认),由于我们更多专注于本发明的特征表示方法,所以未对分类器做任何的优化。事实上,可以通过调整分类器与参数,以及选用更为有效的物化属性,可以得到更高的预测结果。以下分别评估本发明的特征表示算法和选择性集成方法。
首先,在4个独立数据集上利用jackknife验证,比较本发明的特征表示算法iffr和covpcsm,covpssm,cffr三算法的性能,其中covpcsm方法只是单一的考虑物化属性自身信息,生成的特征维数(d=21)较少,识别效果一般;covpssm方法只是单一的考虑进化信息,生成的特征维数(d=210)相对较多,识别效果较好;而cffr方法是对它们二者进行简单串联组合,所生成特征向量同时考虑物化信息和进化信息,识别效果略优于covpssm的结果。本发明的iffr方法不仅考虑了物化属性内部和进化信息内部的相关性,并且更进一步考虑了物化属性和进化信息之间的交互效应项,取得最好的识别性能。详细结果如表2。
表2在4个独立数据集上不同特征表示方法的性能比较(采用jackknife校验法)
acffr=combinedfusionfeaturereresentation;
biffr=interactivefusionfeaturerepresentation.
注意:表格中加粗的数值表示最好的识别结果.
其次,在4个独立数据集上,考查本发明的特征表示算法iffr(λ=0)与三个经典的特征表示算法(psepssm,pseaac和aac)的性能比较,为使比较的结果更加客观可信,实验使用30次的10-foldcv校验结果进行分析。
从图2知,在数据集alternatedataset,pdb1075dataset和independent2dataset中,基于iffr特征表示算法具有卓越的性能,其平均性能均优于其它算法(psepssm,pseaac和aac)。在全部数据集中,iffr特征表示通常有较小的标准误差,这在某种程度上说明iffr特征表示对训练样本集的随机构成不敏感,鲁棒性更好。在数据集independent1dataset中,基于psepssm特征表示算法也有很好的表现,明显优于pseaac和aac的结果。这是因为iffr与psepssm都使用了pssm进化信息,也就是pssm所携带的进化信息比序列自身所包含的信息更为丰富也更加重要,因此,考虑进化信息能够达到提升预测性能的目的。总之,相比较于经典算法(psepssm,pseaac和aac),在4个独立数据集中本发明的iffr特征表示是有效的。
最后,在基准数据集pdb1075上,对本发明的选择性集成算法gapiffr-se和其它预测方法进行比较,其中用于比较的8个卓越方法包括有:idna-prot|dis,psedna-pro,idna-prot,dna-prot,dnabinder,idnapri-pseaac,kmer1+aac和local-dpp。基于jackknife校验的比较结果如表3所示,容易看出,在众多的比较方法中,本发明的选择性集成算法gapiffr-se具有最好的预测性能,也即识别率达到最大值79.91%,mcc指标取得最大值0.61,se指标也取得最大值87.43。因此,相比较于现有的最好方法,本发明方法具有更加卓越的性能,这也间接表明本发明的交互融合特征表示能够生成携带有强判别信息的特征,同时选择性集成还能进一步提升整体学习器的泛化能力,最终能够保证对dna结合蛋白的准确预测。
表3:在数据集pdb1075上本发明识别方法和其它预测方法的性能比较(采用jackknife校验法).
以上为本发明的优选实例,但本发明的实施并不限于上述实例。本领域人员阅读了上述内容后,任何对于本发明的修改和替代,都可被认为处于本发明的权利要求限定范围内。
- 还没有人留言评论。精彩留言会获得点赞!