一种基于玻尔兹曼更新策略的蛋白质结构预测方法与流程
本发明涉及生物信息学、智能信息处理、计算机应用领域、蛋白质结构预测,尤其涉及的是一种基于玻尔兹曼更新策略的蛋白质结构预测方法。
背景技术
蛋白质是生命体的重要组成部分,是生命活动的承担者。蛋白质的基本组成单元是氨基酸,自然界中常见的氨基酸有20多种,蛋白质是由c(碳)、h(氢)、o(氧)、n(氮)组成,一般蛋白质可能还会含有p(磷)、s(硫)、fe(铁)、zn(锌)、cu(铜)、b(硼)、mn(锰)、i(碘)、mo(钼)等,氨基酸是由中心碳原子及其相连的氨基、羧基、氢原子以及氨基酸的侧链组成,氨基酸经过脱水缩合形成肽键,由肽键连接起来的氨基酸形成一条长链,即为蛋白质。
蛋白质分子在生物细胞化学反应过程中起着至关重要的作用。它们的结构模型和生物活性状态对我们理解和治愈多种疾病有重要的意义。蛋白质只有折叠成特定的三维结构才能产生其特有的生物学功能。要了解蛋白质的功能,就必须获得其三维空间结构。因此,获得蛋白质的三维结构对人类来说是至关重要的,1961年,anfinsen提出了氨基酸序列决定蛋白质三维结构这一开创新的理论。而三维结构直接决定了蛋白质的生物性功能,所以人们对蛋白质的三维结构产生了浓厚兴趣并展开研究。国外学者肯德鲁和佩鲁茨对肌血蛋白和血红蛋白进行了结构分析,得到其蛋白质三维结构,是人类第一次测定蛋白质的三维结构,二人借此夺得年诺贝尔化学奖。此外,英国晶体学家bernal与1958年提出了蛋白质四级结构的概念,将其定义为蛋白质一级结构、二级结构以及结构的延伸发展。多维核磁共振方法和射线晶体方法是近些年来发展起来的两个最主要的测定蛋白质结构的实验方法。多维核磁共振方法是将蛋白质放在水中,利用核磁共振直接测定其三维结构的方法。而射线晶体方法是目前为止最有效的蛋白质三维结构测定手段。到前为止,使用这两种方法测定的蛋白质占了已测蛋白质中的绝大比例。由于釆用实验方法的条件有限、时间有限,需要花费大量的人力和物力,而且测定的速度远远跟不上序列的测定速度,所以急需一种既不依赖化学实验,又具有一定准确率的预测方法。这样如何简便、快速、高效地对未知蛋白质进行三维结构预测,成为研究者的棘手问题。在理论探索和应用需求的双重推动下,依据提出的蛋白质一级结构决定蛋白质三维结构的理论,利用计算机设计适当的算法,以序列为起点,三维结构为目标的蛋白质结构预测自20世纪末蓬勃发展。
以序列为起点,利用计算机和优化算法预测蛋白质的三维结构被称之为从头预测。从头预测方法直接基于蛋白质物理或知识能量模型,利用优化算法在构象空间搜索全局最低能量构象解。构象空间优化(或称采样)方法是目前制约蛋白质结构从头预测精度最关键的因素之一。优化算法应用于从头预测采样过程必须首先解决以下三个方面的问题:(1)能量模型的复杂性。蛋白质能量模型考虑分子体系成键作用以及范德华力、静电、氢键、疏水等非成键作用,致使其形成的能量曲面极其粗糙,局部极小解数量随着序列长度的增加呈指数增长;能量模型的漏斗特性也必然会产生局部高能量障碍,导致算法极易陷入局部解。(2)能量模型高维特性。就目前而言,从头预测方法只能应对尺寸较小的目标蛋白,一般不超过100。对尺寸超过150残基以上的目标蛋白,现有优化方法均无能为力。这也就进一步说明了随着尺寸规模的增加,必然造成维数灾问题,完成如此浩瀚的构象搜索过程所涉及的计算量是目前最先进的计算机也难以承受的。(3)能量模型的不精确性。对于蛋白质这类复杂的生物大分子,除了考虑各种物理成键和知识推理的作用之外,还要考虑它与周围溶剂分子的相互作用,目前还无法给出精确的物理描述。考虑到计算代价问题,近十年来研究者陆续提出了一些基于物理的力场简化模型(amber,charmm等)、基于知识的力场简化模型(rosetta,quark等)。然而,我们还远远无法构建起能引导目标序列朝正确方向折叠的足够精确力场,导致数学上的最优解并不一定对应于目标蛋白的天然态结构;此外,模型的不精确性也必然会导致无法对算法性能进行客观地分析,从而阻碍了高性能算法在蛋白质结构从头预测领域中的应用。
因此,现在的蛋白质结构预测方法在预测精度和能量函数方面存在着缺陷,需要改进。
技术实现要素:
为了克服现有的蛋白质结构预测方法能量函数不精确和预测精度较低的缺陷,本发明提供一种预测精度较高的基于玻尔兹曼更新策略的蛋白质结构预测方法。
本发明解决其技术问题所采用的技术方案是:
一种基于玻尔兹曼更新策略的蛋白质结构预测方法,所述方法包括以下步骤:
1)设置初始种群规模np、最大迭代次数gen、交叉概率cr、玻尔兹曼温度因子kt,输入查询序列,片段库,预测的二级结构信息,迭代次数g=0;
2)对种群所有构象进行初始化,对种群中每个构象进行片段组装,利用片段库中相应位置的片段的二面角替换构象中对应位置上的残基二面角,直到所有的残基二面角至少被替换过一次;
3)构象交叉,操作如下:
3.1)选第i,i∈[1,np]个构象ci为目标构象,产生一个随机数r,r∈[0,1],如果r小于cr,则跳到3.2),否则跳至步骤4);
3.2)随机选择一个构象cj,j≠i,利用计算二级结构算法dssp获取构象ci的二级结构信息;
3.3)根据ci残基位置随机选择一个交叉点p,判断交叉点p对应的残基被预测的二级结构的类型;
3.4)针对ci和cj,从交叉点p开始依次互换二面角对直到从交叉点p起预测的二级结构类型和交叉点p处对应的二级结构类型不同为止,产生一个新构象ci′;
4)构象变异,对构象ci′和cj′,变异过程如下:
4.1)对构象ci′和cj′进行9残基片段组装,生成两个构象ci″和cj″;
4.2)分别对构象ci″和cj″求二级结构相似性分值ess:
其中l是查询序列长度,
4.3)从构象ci″和cj″中选择二级结构相似得分es′s最高的构象作为变异成功构象;
5)基于玻尔兹曼概率密度函数进行选择,过程如下:
5.1)对种群中的每个构象求二级结构相似性分值ess,并求出最小的二级结构相似性分值e″ss;
5.2)如果e′ss大于e″ss,则用e′ss对应得构象替换e″ss对应得构象实现种群更新,否则根据二级结构相似性分值求玻尔兹曼接受概率pss:
其中二级结构相似性差值δess=ess″-ess′;
5.3)产生一个随机数r′,r′∈[0,1],如果r′<pss,则用e′ss对应得构象替换e″ss对应得构象实现种群更新,否则保持种群不变;
6)g=g+1,判断是否达到最大迭代次数gen,若不满足条件终止条件,则遍历种
群执行步骤3),否则输出最后预测结果。
本发明的技术构思为:一种基于玻尔兹曼更新策略的蛋白质结构预测方法,包括以下步骤:首先预测查询序列的二级结构信息,构建片段库;其次建立基于二级结构信息的相似性函数,设计交叉变异策略;最后根据二级结构相似性设计玻尔兹曼概率密度函数实现种群更新,利用玻尔兹曼概率密度函数能够有效地提高算法采样能力、允许结构更加合理的构象进入种群和预测精度。
本发明的有益效果为:构象空间采样能力较强、能够有效地使得有潜力的构象被保存下来进而提高预测精度。
附图说明
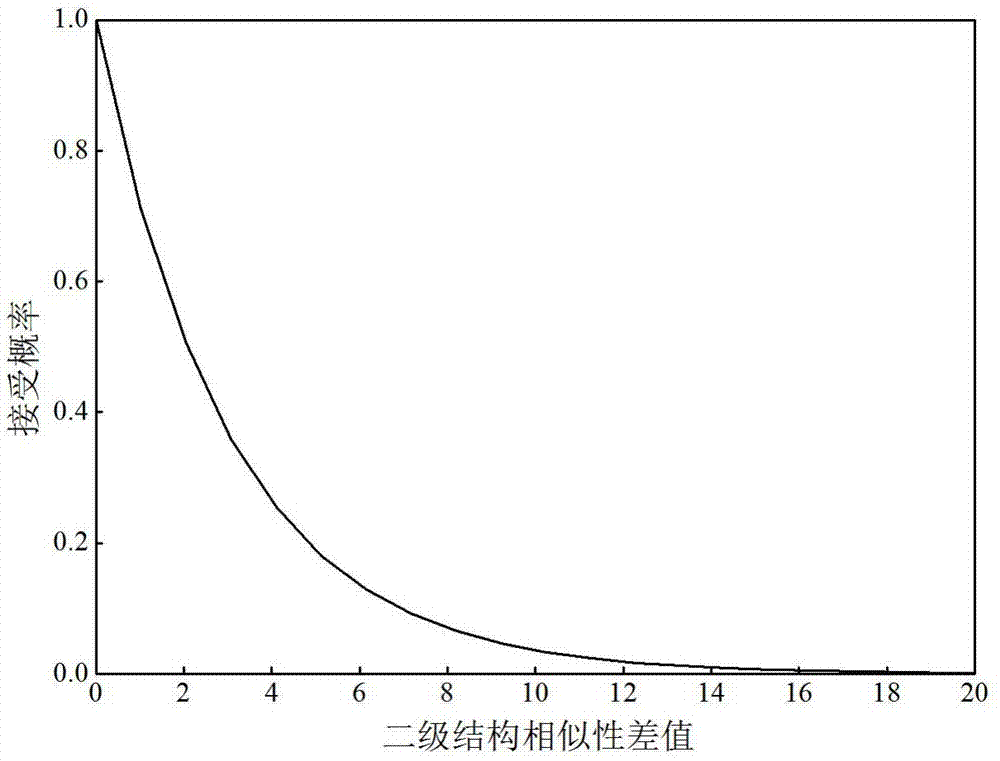
图1是蛋白质1ail概率密度函数分布图。
图2是蛋白质1ail利用基于玻尔兹曼更新策略的蛋白质结构预测方法预测得到的三维结构示意图。
具体实施方式
下面结合附图对本发明做进一步描述。
参照图1和图2,一种基于玻尔兹曼更新策略的蛋白质结构预测方法,包括以下步骤:
1)设置初始种群规模np、最大迭代次数gen、交叉概率cr、玻尔兹曼温度因子kt,输入查询序列,片段库,预测的二级结构信息,迭代次数g=0;
2)对种群所有构象进行初始化,对种群中每个构象进行片段组装,利用片段库中相应位置的片段的二面角替换构象中对应位置上的残基二面角,直到所有的残基二面角至少被替换过一次;
3)构象交叉,操作如下:
3.1)选第i,i∈[1,np]个构象ci为目标构象,产生一个随机数r,r∈[0,1],如果r小于cr,则跳到3.2),否则跳至步骤4);
3.2)随机选择一个构象cj,j≠i,利用计算二级结构算法dssp获取构象ci的二级结构信息;
3.3)根据ci残基位置随机选择一个交叉点p,判断交叉点p对应的残基被预测的二级结构的类型;
3.4)针对ci和cj,从交叉点p开始依次互换二面角对直到从交叉点p起预测的二级结构类型和交叉点p处对应的二级结构类型不同为止,产生一个新构象ci′;
4)构象变异,对构象ci′和cj′,变异过程如下:
4.1)对构象ci′和cj′进行9残基片段组装,生成两个构象ci″和cj″;
4.2)分别对构象ci″和cj″求二级结构相似性分值ess:
其中l是查询序列长度,
4.3)从构象ci″和cj″中选择二级结构相似得分e′ss最高的构象作为变异成功构象;
5)基于玻尔兹曼概率密度函数进行选择,过程如下:
5.1)对种群中的每个构象求二级结构相似性分值ess,并求出最小的二级结构相似性分值e″ss;
5.2)如果e′ss大于e″ss,则用e′ss对应得构象替换e″ss对应得构象实现种群更新,否则根据二级结构相似性分值求玻尔兹曼概率密度函数值pss:
其中δess=ess″-ess′;
5.3)产生一个随机数r′,r′∈[0,1],如果r′<pss,则用e′ss对应得构象替换e″ss对应得构象实现种群更新,否则保持种群不变;
6)g=g+1,判断是否达到最大迭代次数gen,若不满足条件终止条件,则遍历种
群执行步骤3),否则输出最后预测结果。
本实施例以序列长度为73的α折叠蛋白质1ail为实施例,基于玻尔兹曼更新策略的蛋白质结构预测方法,所述方法包括以下步骤:
1)设置初始种群规模100、最大迭代次数1000、交叉概率0.5、玻尔兹曼温度因子3,输入查询序列,片段库,预测的二级结构信息,迭代次数g=0;
2)对种群所有构象进行初始化,对种群中每个构象进行片段组装,利用片段库中相应位置的片段的二面角替换构象中对应位置上的残基二面角,直到所有的残基二面角至少被替换过一次;
3)构象交叉,操作如下:
3.1)选第i,i∈[1,np]个构象ci为目标构象,产生一个随机数r,r∈[0,1],如果r小于0.5,则跳到3.2),否则跳至步骤4);
3.2)随机选择一个构象cj,j≠i,利用计算二级结构算法dssp获取构象ci的二级结构信息;
3.3)根据ci残基位置随机选择一个交叉点p,判断交叉点p对应的残基被预测的二级结构的类型;
3.4)针对ci和cj,从交叉点p开始依次互换二面角对直到从交叉点p起预测的二级结构类型和交叉点p处对应的二级结构类型不同为止,产生一个新构象ci′;
4)构象变异,对构象ci′和cj′,变异过程如下:
4.1)对构象ci′和cj′进行9残基片段组装,生成两个构象ci″和cj″;
4.2)分别对构象ci″和cj″求二级结构相似性分值ess:
其中l是查询序列长度,
4.3)从构象ci″和cj″中选择二级结构相似得分es′s最高的构象作为变异成功构象;
5)基于玻尔兹曼概率密度函数进行选择,过程如下:
5.1)对种群中的每个构象求二级结构相似性分值ess,并求出最小的二级结构相似性分值e″ss;
5.2)如果e′ss大于e″ss,则用e′ss对应得构象替换e″ss对应得构象实现种群更新,
否则根据二级结构相似性分值求玻尔兹曼概率密度函数值pss:
其中δess=ess″-ess′;
5.3)产生一个随机数r′,r′∈[0,1],如果r′<pss,则用e′ss对应得构象替换e″ss对应得构象实现种群更新,否则保持种群不变;
6)g=g+1,判断是否达到最大迭代次数gen,若不满足条件终止条件,则遍历种
群执行步骤3),否则输出最后预测结果。
以序列长度为73的α折叠蛋白质1ail为实施例,运用以上方法得到了该蛋白质的近天然态构象,最小均方根偏差为
以上说明是本发明以1ail蛋白质为实例所得出的优良效果,显然本发明不仅适合上述实施例,在不偏离本发明基本内容所涉及范围的前提下对其做各种变形和改进,不应排除在本发明的保护范围之外。
- 还没有人留言评论。精彩留言会获得点赞!