疾病相关蛋白数据库的制作方法
本发明涉及生物信息学领域。具体地,本发明涉及疾病相关蛋白数据库,其构建方法,以及该数据库应用。
背景技术
蛋白是生物体重要的组成成分,具有重要的生物学功能,如作为生物催化剂、调节代谢、免疫保护、物质的转运和存储、细胞间信号传递等。许多疾病的发生和机体内的蛋白发生变异相关,例如镰刀型细胞贫血病,即是由于基因突变导致亚基n端的第6个氨基酸残基由正常的带负电的极性亲水谷氨酸残基(glu)变成了不带电的非极性疏水缬氨酸(val),即由正常的蛋白变异为疾病相关蛋白,进而使得红细胞扭曲成镰刀状,由于这种僵硬的镰刀状红细胞不能通过毛细血管,加上镰刀状细胞的血红蛋白的凝胶化使血液粘滞度增大,阻塞毛细血管,进而引起局部组织器官缺血缺氧,产生脾肿大、胸腹疼痛等症状。而蛋白的变异多源于基因变异。
人类遗传相关的疾病长期以来一直威胁着人们的健康与生命,随着遗传学与分子生物学的技术和研究进展,许多由于氨基酸序列的改变而导致的人类遗传相关疾病的基因变异己被鉴定。根据已发现的可引起蛋白序列发生变异从而导致疾病发生的基因变异信息,人们建立了多种相应的基因突变数据库,如人类基因突变数据库(hgmd)、基因组数据库(gdb)、在线人类孟德尔遗传信息数据库(omim)、clinvar、refgene等。
总体来说,与人类疾病相关的突变数据库大致可分为两大类,一类是综合的突变数据库,如omim、hgmd等,其中ncbi的omim数据库提供了大概20,000条左右的记录,专门记载着与疾病有关的突变信息。但是信息仅以文本为主方式记录,如基因名称、突变信息、以及专家所提供的注释,而并没有提供蛋白突变的序列。而人类基因突变数据库hgmd,收录了大概60,000个左右的突变信息,但未收录突变的氨基酸序列,并且不提供批量下载,还需要商业授权。同时,omim和hgmd这两个权威库中并没有互相覆盖,所以来自这两个数据库的相同基因所具有的突变数量也不一致,其中有几百个完全大相径庭,也就是说迄今为止,不管是omim还是hgmd都没有完全建立收录到所有己知的与疾病相关的人类突变蛋白质序列。另一类是专门的突变蛋白数据库。以pmd为代表,该数据库是由ddbj的研究小组在上个世纪九十年代收集并开发的突变蛋自质数据库,他们从大量文献中收集了来自不同物种(包括人类在内)的大量突变蛋白质序列,在目前版本中共有来自459个不同条目的218,193条突变蛋白质序列,其中部分突变序列含有疾病信息,然而,该数据库自2007年3月起至今尚未更新,因此其所收集的突变蛋白质序列数量仍然有限,至少不可能涵盖在2007年3月以后发现的任何新的与疾病相关的人类突变蛋白质。另外,swissprot也收集了54,018个人类基因的polymomhism和部分与疾病相关的突变位点数据其中一部分记录具有编号记载的疾病信息。
大量的疾病相关蛋白的信息散布在科学文献和各类生物学数据库中,为了帮助相关研究者方便使用这些数据并发现数据之间的有用的关联,有必要系统构建了一个整合各种信息渠道中的与疾病相关的人类突变蛋白质序列集和蛋白结构数据库。构建这样的数据库是非常有意义的,具体表现在:首先,可以将分散在全球形式不同的疾病蛋白数据统一管理和维护,方便研究人员进行查询;其次,建立针对性更强的疾病相关蛋白数据库,可利用大数据的技术挖掘隐含在疾病蛋白数据背后的规律和意义,帮助研究者针对疾病的发生和发展进行研究。
目前已有相关的研究,如上海交通大学的奚洪建立了蛋白质序列变异与疾病相关性及蛋白质相互作用数据库(蛋白质序列变异与疾病相关性及蛋白质相互作用数据库的构建,奚洪,上海交通大学博士学位论文,2010年;syspimp:theweb-basedsystematicalplatformforidentifyinghumandisease-relatedmutatedsequencesfrommassspectrometry,hongxi,nucleicacidsresearch,2009,vol.37,913-920)等。但目前的数据库在使用时仍然存在一些问题或缺陷:(1)目前数据库的蛋白信息无法及时更新,如奚洪的数据库中包含了omim、pmd、swissprot的34,891条数据,这些数据在收录进入数据库后,无法进行自动更新;(2)目前数据库收录的来源不够广泛;(3)目前数据库为本地存储的信息,使用不便;(4)目前的数据库多为英文信息,对于英文有困难的中国使用者来说存在语言方面的障碍。(5)目前的数据库里面有很多没有结构信息。
因此,如何建立一个数据源广泛、更新及时、使用方便、包含结构信息的疾病相关蛋白数据库,成为本领域亟待解决的技术问题。
技术实现要素:
为解决上述技术问题,本发明提供了一种疾病相关蛋白数据库,该数据库具有现有疾病相关蛋白数据信息收录广泛、更新及时、包含结构信息、无需本地安装且访问速度快的特点。同时提供了该疾病相关蛋白数据库的构建方法,以及在科学研究中的应用。
根据本发明的第一个方面,本发明提供了一种疾病相关蛋白数据库,所述疾病相关蛋白数据库收录了疾病相关蛋白信息,其特征在于所述疾病相关蛋白信息的来源为基因突变数据库、蛋白突变数据库、以及用户上传的疾病相关蛋白数据,所述疾病相关蛋白数据库中收录的信息为疾病-变异基因-疾病蛋白序列-疾病蛋白结构的完成数据。
根据本发明的一个方面,所述疾病相关蛋白数据库提供了疾病相关蛋白的完成数据信息,具体地,提供了包括蛋白名称、发现方法、关键词、蛋白链种类、氨基酸数量、原子数量、提交日期、发布日期、最后修正、结构作者和引用作者、蛋白结构的3d显示模型的信息。
根据本发明的一个方面,所述基因突变数据库中的基因突变信息被整理为对应的疾病相关蛋白数据。所述基因突变数据库为任何记载基因突变信息的数据库,优选为omim、hgmd、swissprot数据库。
根据本发明的一个方面,所述蛋白突变数据库为任何记载疾病相关蛋白信息的数据库,所述数据库优选为pmd数据库。
根据本发明的一个方面,所述用户上传的疾病相关蛋白数据为数据库使用者进行查询时提交的疾病相关蛋白信息,该数据库可保留用户上传的相关蛋白信息,该数据库优选为疾病相关蛋白三维预测系统,所述三维预测系统可以是现有的,例如swiss-model,也可以是自建的。
根据本发明的一个方面,本发明提供的数据库将基因突变数据库、蛋白突变数据库、以及用户上传的疾病相关蛋白数据中的疾病、或变异基因、或疾病蛋白序列,或疾病蛋白结构的不完全数据补充为疾病-变异基因-疾病蛋白序列-疾病蛋白结构的完全数据。
根据本发明的一个方面,本发明的疾病相关蛋白数据库的数据库模块基于mongodb+大型磁盘文件系统。
由于蛋白数据库和分子数据库以及知识库内的数据源的来源复杂,网络爬取数据较多,数据呈现非结构问题,考虑到后期维护成本的因素,数据采用非范式设计方式。另一方面,由于疾病相关蛋白数据本身带有研究性质,数据属性的成熟统一不够,后期数据标准和知识数据标准可能存在较大的变动可能性。目前常用的关系型数据库如mysql等不适合于本系统内的数据存储。为了解决该问题,发明人在选择数据库模块的开发时,采用nosql类型数据库mongodb以应对数据属性改动的情况,可以为系统留下扩展性空间。同时,采用mongodb+大型磁盘文件系统构建疾病相关蛋白数据库,可获得更快速的响应速度。因此,总结来说,该数据库模块的选择对于疾病相关蛋白数据库的构建是最优选的,具有数据易爬取、后期维护成本低、后期可变动数据标准、响应速度快的优秀效果。
根据本发明的一个方面,本发明的疾病相关蛋白数据库可选择性的包含在线可视化平台技术,该技术可采用本领域已知的现有技术,例如中国专利申请cn107798218a中记载的在线可视化平台技术。
根据本发明的一个方面,所述疾病包括在人、动物、植物、微生物中发生的与蛋白变异相关的疾病。
根据本发明的一个方面,所述疾病相关蛋白数据库提供了收藏或下载的功能,所述收藏或下载的方式可选择为单独数据的收藏或下载,或者批量数据的收藏或下载。
根据本发明的一个方面,所述疾病相关蛋白数据库被架设在云端服务器。
根据本发明的一个方面,所述疾病相关蛋白数据库收录的范围包括人、动物、或者植物的疾病相关蛋白。
根据本发明的第二个方面,所述数据库通过如下方法构建:
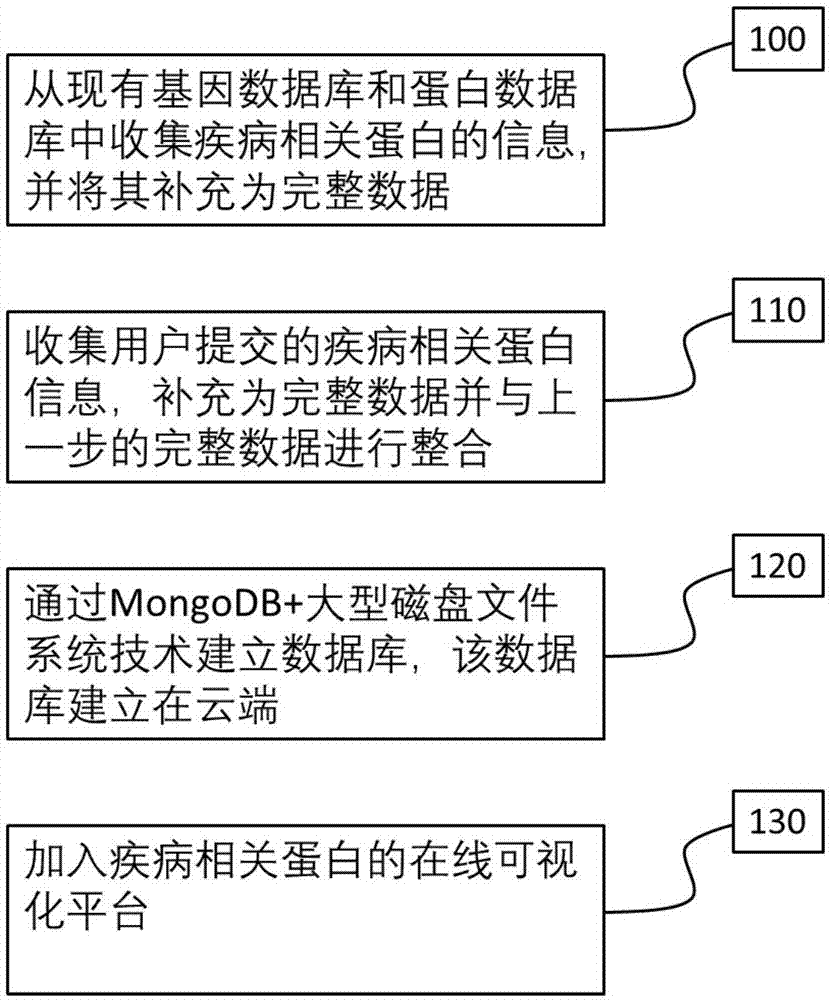
步骤1:数据收集与整理:收集现有数据库的疾病相关蛋白数据或疾病相关的基因变异数据,同时收集使用者提交的通过在线疾病相关蛋白三维预测系统收录的疾病相关蛋白数据,按统一格式整合这两部分数据;其中,当所述的数据为疾病相关的基因变异数据时,现将该基因变异数据在结构数据库中筛选比对,匹配结构后记录其相关疾病蛋白的信息。最终形成的疾病-变异基因-疾病蛋白序列-疾病蛋白结构的完成数据。
步骤2:数据库的架设:利用现有数据库技术对步骤1中的数据进行建库,并将数据库架在云端;所述的数据库技术具体为mongodb+大型磁盘文件系统技术。
根据本发明的一个方面,上述数据库的构建方法还可以选择性地包括步骤3:加入蛋白质的在线可视化平台。该平台是一个搭建在用户端、用于在线可视化数据文件的系统。其目的是为了让用户更直观便捷地理解数据文件以及科学计算的过程和结果,对其有一个感性的认知。
根据本发明的一个方面,所述在线可视化平台支持的数据类型包括3d展示操作蛋白质模型。支持数据类型包括pqr、pdb、mmcif、gro、sdf、mol2、mmtf、mrc/map/ccp4、dx/dxbin、cube、brix/dsn6、xplor/cns,其中优选pqr、pdb、sdf、mol2。
根据本发明的一个方面,所述在线可视化平台的可视化方式包含:axes、backbone、ball+stick、cartoon、licorice、spacefill、surface。
根据本发明的一个方面,上述步骤1中的统一格式包括蛋白名称、发现方法、关键词、蛋白链种类、氨基酸数量、原子数量、提交日期、发布日期、最后修正、结构作者和引用作者、蛋白质的3d显示。
根据本发明的第三个方面,所述数据库可用于疾病相关蛋白数据的收录和查询。进一步地,所述数据库可用于疾病相关蛋白的在线三维可视化。
通过本发明,实现了如下的有益效果:(1)本数据库的数据来源广泛、更新及时;(2)可通过用户的查询行为进行数据的补充;(3)访问方便,界面友好;(4)除人类疾病相关蛋白外,本数据库也包括了动物、植物的疾病相关蛋白;(5)构建了疾病-变异基因-疾病蛋白序列-疾病蛋白结构的完成数据库;(6)数据易爬取;(7)后期维护成本低,且变动数据标准;(8)响应速度快。
附图说明
图1为构建本发明的疾病相关蛋白数据库的流程示意图;
图2为疾病相关蛋白的基因蛋白结构映射表;
图3为疾病相关蛋白的数据示意图;
图4为人类inpp5e蛋白数据下所收录的蛋白数据信息;
图5为疾病相关蛋白的在线三维可视化的示意图。
具体实施方式
实施例1疾病相关蛋白数据库的构建
步骤1:按照如下来源收录疾病相关蛋白数据:(1)收录来自omim、hgmd数据库中记载的疾病相关基因突变数据,并将这些疾病基因的信息和结构数据库进行筛选比对,匹配结构后输出疾病相关蛋白的数据信息;(2)收录来自pmd、swissprot数据库的疾病相关蛋白信息;(3)收集用户上传的疾病相关蛋白数据信息。将上述三类信息按照疾病-变异基因-疾病蛋白序列-疾病蛋白结构补充完整,形成疾病相关蛋白的完成数据。
选择omim的allelicvariants、clinicalsynopsis和genemaplocus,swissprot的polymorphismsanddiseasemutation作为疾病相关基因或者蛋白突变数据库的来源,以网络爬虫技术爬取这些数据库中的基因名称、编号、突变位点以及突变前后的氨基酸残基。由于不同数据库之间的数据在存在重叠的同时,又不完全一致,因而选用数据之间的唯一编码,即基因的名称或编号找到其原始的正常蛋白序列,并根据其提供的突变信息,将其还原为相应的突变蛋白质序列。这些爬取的数据按照较短的周期进行更新,如每周、每两周、每个月进行新一轮的爬取,对新增的基因信息按照相同的方法进行补充。对于swissprot的polymorphismsanddiseasemutation以及msipi数据库,经过去冗余后,恢复其中的人类突变蛋白质数据库。将msipi中的正常蛋白序列以及多条由该正常蛋白变化而来的蛋白质序列的信息进行拆分,将其导入数据源。将这些数据去冗余后,检查数据的完全性,将每条数据补充为包含蛋白名称、发现方法、关键词、蛋白链种类、氨基酸数量、原子数量、结构信息、提交日期、发布日期、最后修正、结构作者和引用作者、蛋白质的3d显示的完整数据。
收集用户上传的疾病相关蛋白数据信息,这些数据通常是不完整的,仅涉及完整数据的一方面,例如蛋白名称、蛋白序列等。分析比对用户提交数据和数据库已有信息的匹配性,并根据比对结果进行数据信息的分类,对于与现有数据不匹配的数据信息,按照上述完全数据的标准通过检索添加而补充完整,并将其与上述的合并。
数据库的最终数据中,所述的疾病相关蛋白的完成数据信息具体包括:蛋白名称、发现方法、关键词、蛋白链种类、氨基酸数量、原子数量、结构信息、提交日期、发布日期、最后修正、结构作者和引用作者、蛋白质的3d显示信息。
步骤2:数据库的架设:
利用现有的mongodb数据库技术对步骤1中的数据进行建库,并将数据库架在云端;所述的数据库技术具体为mongodb+大型磁盘文件系统技术。
步骤3:加入疾病相关蛋白的在线可视化平台
加入蛋白质的在线可视化平台。其中,该可视化平台可支持的数据类型包括pqr、pdb、mmcif、gro、sdf、mol2、mmtf、mrc/map/ccp4、dx/dxbin、cube、brix/dsn6、xplor/cns。可视化方式包含:axes、backbone、ball+stick、cartoon、licorice、spacefill、surface。结果如图3所示。
实施例2疾病相关蛋白数据库的应用
以疾病类型为检索入口,在数据库中检索bart—pumphrey(指节垫合并白甲和耳聋),结果如图4和图5所示。
以人类inpp5e蛋白为例,在数据库中的蛋白名称检索入口检索该蛋白。检索结果如5所示。
- 还没有人留言评论。精彩留言会获得点赞!