基于非支配排序的混合蝙蝠算法的DNA编码序列优化方法与流程
本发明属于dna计算中编码设计领域,涉及群体智能优化算法和dna编码,具体涉及用粒子群算法、蝙蝠算法和非支配排序算法来优化dna序列。
背景技术:
1994年,美国南加州大学的阿德尔曼教授提出用dna分子进行计算的一种新的计算模式。在这种新的计算模式当中dna分子被作为信息存储的单元并且分子之间的生化反应代表计算操作的过程。在这种背景下,沃森-克里克碱基互补配对原则是最重要的反应之一,因为这一反应能够成功地检索存储在dna分子中的信息。然而必须保证在实际的生化反应过程中,编码每一个信息元的dna分子能够被唯一识别,因为不期望的反应通常会导致不正确的计算。因此设计质量较高的dna序列非常重要,这样能够确保集合中的dna序列仅仅只与其互补的序列杂交。
粒子群算法是一种群体智能优化算法。该算法能以较大概率收敛于全局最优解。实践证明,它适合在多目标环境中寻优,具有较快的速度和更好的全局搜索能力。粒子群算法的基本思想是对种群中粒子的适应度进行计算,寻找个体极值和群体极值,然后对粒子的位置和速度进行更新,再计算适应度找到个体极值和群体极值,如此迭代直到满足最大进化代数。
蝙蝠算法是一种新型群智能优化算法。其模拟自然界中蝙蝠的回声定位行为,在搜索过程中不断调整搜索频率,加快了算法的收敛速度。同时,蝙蝠个体通过调节声波脉冲频度和脉冲响度的变化来协调算法的探索和开采,增强了算法的搜索能力和鲁棒性。
技术实现要素:
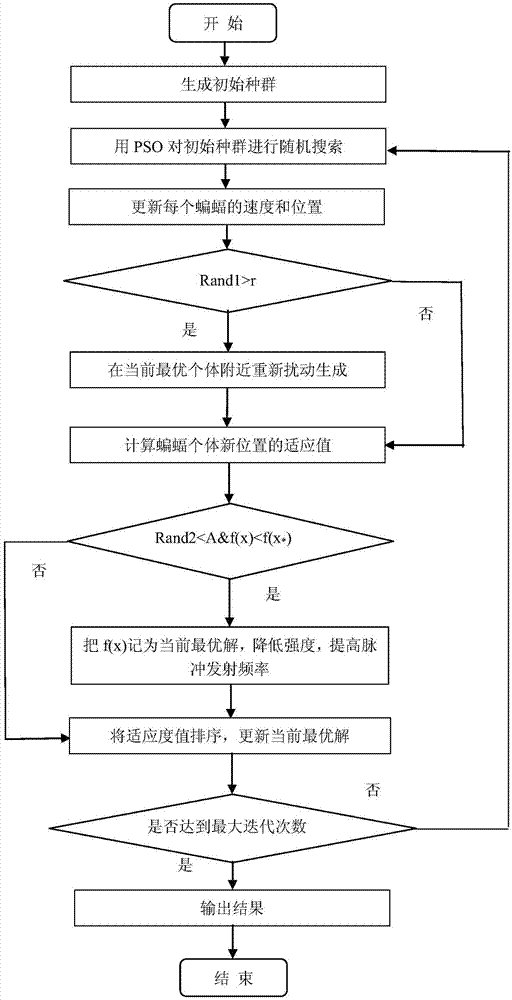
本发明为解决上述技术问题,本发明提供一种基于非支配排序的混合蝙蝠算法的dna编码序列优化方法,该方法包括以下步骤:
步骤1:生成初始种群,初始化算法需要的参数;
步骤2:用粒子群算法对初始种群进行搜索,每个粒子通过不断更新速度和位置搜索最优编码集合;
步骤3:将粒子群搜索到的最优编码集合作为蝙蝠算法的输入,更新蝙蝠算法的速度和位置;
步骤4:对于每个蝙蝠个体,产生一个随机数rand1,判断rand1和脉冲频度r的大小,如果rand1大于脉冲频度r,则执行步骤5,否则执行步骤6;
步骤5:在当前最优个体附近重新扰动生成;
步骤6:计算蝙蝠个体新位置的适应值;
步骤7:判断蝙蝠个体产生随机数rand2和音强a的大小以及每个个体适应度值和全局最优值的大小,若rand2<a并且个体适应度值小于全局最优值,则执行步骤8,否则执行步骤9;
步骤8:把当前个体作为最优解,降低音强,提高脉冲发射频率;
步骤9:将适应度进行排序更新当前最优解;
步骤10:判断是否达到最大迭代次数,若是进行步骤11,否则返回步骤2;
步骤11:对结果进行适应度排序,把排序后的结果输出。
本发明与现有技术相比具有以下创新点:
1、用非支配排序对初始种群进行适应度计算和排序,引入非支配策略不仅可以筛选出距离真实前沿较近的个体,而且可以使得所求个体均匀的分布在真实前沿边缘;
2、粒子群优化算法避免算法陷入局部最优,使得算法有更好的全局搜索能力,使用蝙蝠算法能够避免粒子群算法后期陷入局部最优,并且蝙蝠算法收敛速度快;
3、本发明提出的基于非支配排序的混合蝙蝠算法的dna序列优化算法能够搜索出质量较优的dna编码序列。
附图说明
图1为本发明的实现流程图。
具体实施方式
下面结合附图对本发明作进一步说明。本发明中涉及的约束条件有六条,分别为连续性约束、发夹结构、h-measure、相似性、解链温度、gc含量。本发明将约束条件的前四项作为目标函数,最后两项作为约束条件。用在权利要求的第2步骤中计算每个个体的适应度值。
连续性约束表示在一个dna序列中相同的碱基在给定的阈值内不能连续出现超过阈值的数量。发夹结构表示所要研究的序列可能产生二级结构的概率。h-measure表示两序列间存在的互补碱基(包括滑动移位)的个数。相似性表示用于计算的两条dna序列在相同方向的相似度。解链温度表示dna分子在变形的过程中,一半的dna分子由双链变成单链时的温度。gc含量约束表示dna序列中任意一个标签中胞嘧啶(c)和鸟嘌呤(g)的数量占整个标签碱基数量的百分比,本专利将其约束在50%。
本发明构造满足组合约束条件的最优dna编码序列,首先要构造出全部的dna序列作为初始种群。接着用粒子群算法对初始种群进行搜索寻优。其次,利用已经得出的dna编码序列,用蝙蝠算法进行优化,得到最优的dna编码序列,用非支配排序计算最优序列的适应度并对其进行排序。最后,选出最优dna编码序列。
如图1所示,本发明一种基于非支配排序的混合蝙蝠算法的dna编码序列优化方法,该方法包括以下步骤:
步骤1:生成初始种群,初始化算法需要的参数;
步骤2:用粒子群算法对初始种群进行搜索,每个粒子通过不断更新速度和位置搜索最优编码集合;
步骤3:将粒子群搜索到的最优编码集合作为蝙蝠算法的输入,更新蝙蝠算法的速度和位置;
步骤4:对于每个蝙蝠个体,产生一个随机数rand1,判断rand1和脉冲频度r的大小,如果rand1大于脉冲频度r,则执行步骤5,否则执行步骤6;
步骤5:在当前最优个体附近重新扰动生成;
步骤6:计算蝙蝠个体新位置的适应值;
步骤7:判断蝙蝠个体产生随机数rand2和音强a的大小以及每个个体适应度值和全局最优值的大小,若rand2<a并且个体适应度值小于全局最优值,则执行步骤8,否则执行步骤9;
步骤8:把当前个体作为最优解,降低音强,提高脉冲发射频率;
步骤9:将适应度进行排序更新当前最优解;
步骤10:判断是否达到最大迭代次数,若是进行步骤11,否则返回步骤2;
步骤11:对结果进行适应度排序,把排序后的结果输出。
以下通过实验对本发明的有益技术效果作进一步的说明。
实施例1
本发明的实施例是在以本发明技术方案为前提下进行实施的,给出了详细的实施方式和具体的操作过程,但本发明的保护范围不限于下述实施例。实例中dna编码长度等于20,连续性约束、发夹结构约束、h-measure约束、相似性约束、解链温度约束、gc含量约束如上所述。
步骤1:对种群进行初始化生成20个长度为20维的dna编码序列,如表1所示。初始化算法所需要的相关参数,蝙蝠发射频度r取0.5,响度a取0.25,脉冲频度增加系数0.9,衰减系数0.99。
表1:
步骤2:用粒子群算法对初始种群进行搜索,首先初始化粒子群的个体极值和全局极值,通过对粒子的速度和位置的更新,不断寻找到个体极值和全局极值,最终得到全局的最优解,对全局最优解进行适应度计算并进行排序获得最优种群。就本实例通过matlab进行仿真实验得到粒子群优化算法得到最优种群数为17。本实例步骤2提到的适应度是用非支配排序对所有个体进行等级分配,若i个体在种群中的等级为ri,则可设定1/ri为该个体的适应度值;
步骤3:将步骤2用粒子群算法得到的17条20维的dna序列用蝙蝠算法继续进行寻优操作。本实例用matlab里面的zeros()函数初始化17行20列的全为0的矩阵作为蝙蝠算法的初始速度v,粒子群算法得到的最优种群作为蝙蝠算法的位置x,在20维搜索空间,在t+1时刻蝙蝠i的速度和位置更新方式为:
其中x*代表全局最优位置。频率fi为蝙蝠个体i在搜索时的脉冲频率。
按照上面的两个公式对蝙蝠的速度和位置进行更新操作;
步骤4:对于每一个蝙蝠个体随机生成一个随机数rand1,判断rand1和脉冲频度r=0.5的大小,若rand1>r,则执行步骤5,否则执行步骤6;
步骤5:按照公式(3)在当前最优个体附近重新扰动生成,更新每个蝙蝠的位置;
其中ε是一个介于[-1,1]的随机数,at为蝙蝠在t时刻的平均响度;
步骤6:计算蝙蝠个体新位置的适应值;
步骤7:判断蝙蝠个体产生随机数rand2和音强a=0.25的大小以及每个蝙蝠个体适应度值f(xi)和全局最优值f(x*)的大小,若rand2<a并且f(xi)<f(x*),则执行步骤8,否则执行步骤9;
步骤8:把当前个体xi作为最优解,降低音强a,提高脉冲发射频率r;
步骤9:用非支配排序把当前个体进行适应度值排序获得最优解;
步骤10:判断是否达到最大迭代次数500代,若是进行步骤11,否则返回步骤2;
步骤11:对结果进行适应度排序,选出适应度最高的7个序列作为最优解,最终构造出最优dna序列,最优dna序列集合如表2所示。
表2:
本发明提出基于非支配排序的混合蝙蝠算法的dna序列优化方法,用粒子群算法对初始种群进行搜索。在粒子群算法得到的最优解用蝙蝠算法继续进行优化,将最优解进行非支配排序,最终选出排序后适应度值最大的7个序列作为最优的dna序列集合。本发明在intel(r)cpu3.6ghz、4.0gb内存、windows7运行环境下,借助matlab对该算法进行仿真实验,实验结果表明本实例的方法结果优于其他算法的实验结果
以上所述,仅为本发明较佳的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明披露的技术范围内,根据本发明的技术方案及其构思以等同替换或改变,都应涵盖在本发明的保护范围内。
- 还没有人留言评论。精彩留言会获得点赞!