一种基于深度学习的原代肿瘤细胞分割识别方法及系统与流程
本发明属于生物医学图像处理领域,具体涉及一种适用于多类癌细胞的基于深度学习的原代肿瘤细胞分割识别方法及系统。
背景技术:
随着个体化医疗时代的到来,针对不同癌症病患个体的药物筛选与评价也迎来了新的挑战。抗癌药物筛选的一种做法是原代培养,即利用病患个体的穿刺组织、积液等在96孔板中对癌细胞进行培养,并加入多组药物,一段时间后观察各孔中存活癌细胞的多少。然而这种方式形成的培养环境中包含癌细胞、各种正常细胞与杂质,因此ccd拍照得到的照片中各种形态的细胞都存在。在这种情况下要判断哪些形态是癌细胞需要耗费大量的人力与时间,而且通常只能半定量甚至定性判断,无法准确地评估各组抗癌药物对癌细胞杀伤的效果。而传统图像识别的算法面对这类复杂的照片又难以取得很好的效果。
技术实现要素:
针对上述问题,本发明的目的是提供一种基于深度学习的原代肿瘤细胞分割识别方法及系统,采用深度学习技术,对培养环境ccd照片中的癌细胞区域自动检测与分割,从而能快速而又精确地定量计算存活癌细胞区域的面积大小,以评估不同药物对癌症病患个体的作用。不仅如此,该方法本身还同样适用于其他复杂培养环境中各类细胞或类器官的检测与分割,只要配有相应的训练集即可。
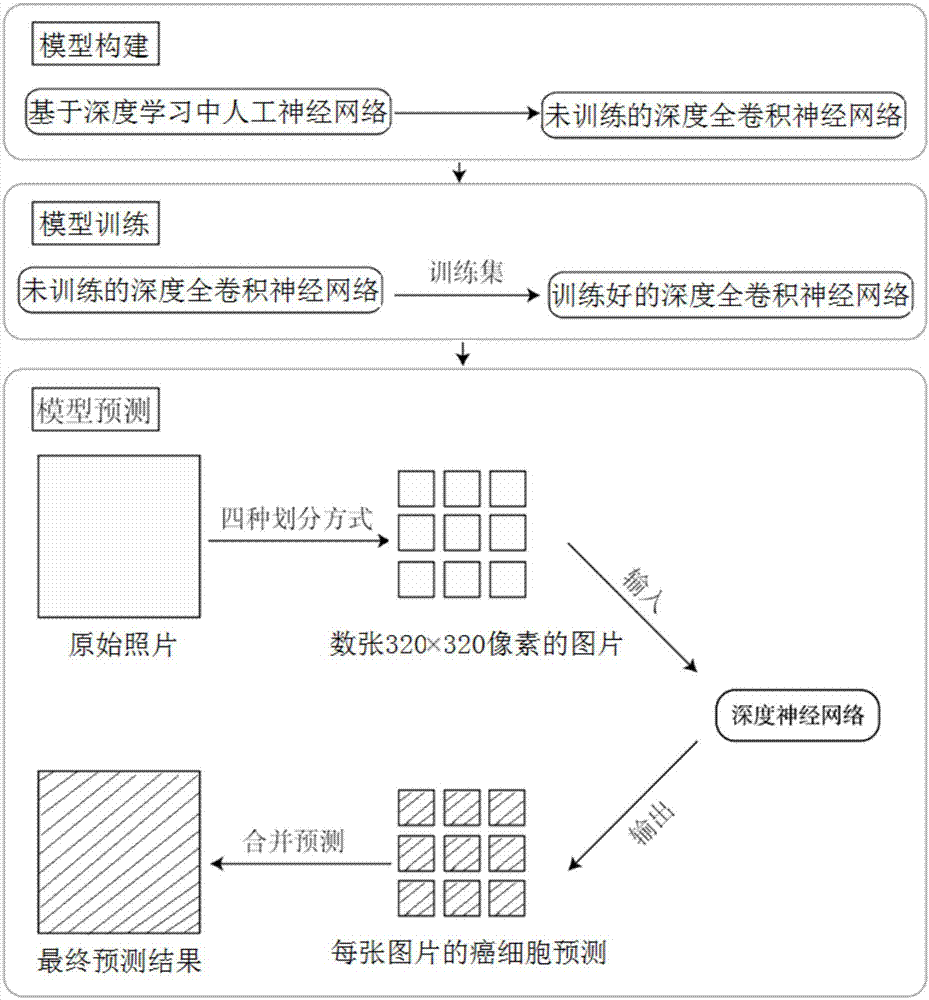
为实现上述目的,本发明采取以下技术方案:一种基于深度学习的原代肿瘤细胞分割识别系统,其包括模型构建模块、模型训练模块和模型预测模块;所述模型构建模块用于基于深度学习中的人工神经网络构建深度全卷积神经网络模型;所述模型训练模块用于根据预先构造的训练集对所述深度全卷积神经网络模型进行训练,得到训练好的深度全卷积神经网络模型;所述模型预测模块用于将原始照片分割后输入到训练好的深度全卷积神经网络模型,并将得到的各分割图片的癌细胞区域分割结果汇总,得到原始照片的癌细胞区域分割结果。
进一步地,所述模型预测模块包括图片分割模块、图片识别模块和结果汇总模块;所述图片分割模块用于采用有重叠区域的滑动窗口将任意大小的原始照片分为数张320×320像素的rgb图片,并发送到所述图片识别模块;所述图片识别模块将各分割图片输入到已训练好的深度全卷积神经网络模型,得到各分割图片的癌细胞区域分割结果,并发送到所述结果汇总模块;所述结果汇总模块用于对所有分割图片的癌细胞区域分割结果进行汇总,得到原始照片的癌细胞区域分割结果。
一种基于深度学习的原代肿瘤细胞分割识别方法,其包括以下步骤:1)基于深度学习中的人工神经网络构建深度全卷积神经网络模型;2)采用训练集对构建的深度全卷积神经网络模型进行训练,得到训练好的全卷积神经网络模型;3)对待识别的原始照片进行分割,并将分割后的图片输入训练好的全卷积神经网络模型中得到各分割图片的癌细胞分割结果,对各分割图片的癌细胞分割结果进行汇总,得到原始照片的癌细胞分割结果。
进一步地,所述步骤1)中,构建的深度全卷积神经网络模型包括输入层、卷积层、反卷积层、池化层、去池化层和输出层;所述卷积层和反卷积层分别设置为五层,两两所述卷积层之间均设置一所述池化层,两两所述反卷积层之间均设置一所述去池化层,输入数据由所述输入层输入到第一卷积层,依次经过卷积和池化处理后,输出到第一反卷积层,并依次经反卷积和去池化处理后,经第五反卷积层输出到所述输出层。
进一步地,所述卷积层和反卷积层中,除最后一层外,其余所有的卷积层与反卷积层均采用3×3的卷积核与相应的填补使得该层的输入与输出具有相同的高度与宽度;模型的最后一层采用1×1卷积核无填补的卷积层,输出为320×320的双通道矩阵。
进一步地,各所述池化层均采用核为2×2步幅为2的最大池化,所述池化层用于对该池化层的输入进行采样,并记录各采样值的原始位置以供所述去池化层使用;所述去池化层均采用2×2的核与2的步幅,利用所述池化层采样的原始位置把相应的值填补上去,并在其余位置均填入0。
进一步地,所述步骤2)中,采用训练集对构建的深度全卷积神经网络模型进行训练,得到训练好的全卷积神经网络模型的方法,包括以下步骤:2.1)对训练集进行数据增强处理,得到能够加强模型泛化性能的训练集;2.2)将得到的训练集输入到构建的深度全卷积神经网络模型中,并计算深度全卷积神经网络模型的损失函数;2.3)对深度全卷积神经网络模型的损失函数进行优化,得到训练好的深度全卷积神经网络模型。
进一步地,所述步骤3)中,对待识别的原始照片进行分割,并将分割后的图片输入训练好的全卷积神经网络模型中得到各分割图片的癌细胞分割结果,对各分割图片的癌细胞分割结果进行汇总,得到原始照片的癌细胞分割结果的方法,包括以下步骤:3.1)采用带有重叠区域的滑动窗口对原始照片进行划分,生成一系列320×320像素的图片;3.2)将得到的各320×320像素的图片输入到训练好的深度神经网络模型进行预测,得到各320×320像素的分割图片的癌细胞识别结果;3.3)对所有分割图片的癌细胞分割结果进行汇总,得到原始图片的癌细胞分割结果。
进一步地,所述步骤3.1)中,采用带有重叠区域的滑动窗口对原始照片进行划分,生成一系列320×320像素的图片的方法为:
第一种:以原始照片的坐标原点(0,0)为起点,采用320×320像素的滑动窗口依次沿原始照片的横轴进行分割,直到原始照片横轴的最末端,得到第一行320×320像素的分割图片,然后以坐标点为(0,320)的点为起点,进行第二行的图片分割,并依次重复,直到原始照片全部分割完毕;
第二种:以原始照片的(160,160)为起点,采用320×320像素的滑动窗口依次沿原始照片的横轴进行分割,直到原始照片横轴的最末端,得到第一行320×320像素的分割图片,然后以坐标点为(0,320)的点为起点,进行第二行的图片分割,依次重复,直到原始照片全部分割完毕;
第三种:以原始照片上坐标点(160,0)为起点,采用320×320像素的滑动窗口依次沿原始照片的横轴进行分割,直到原始照片横轴的最末端,得到第一行320×320像素的分割图片,然后以坐标点(160,320)为起点,进行第二行的图片分割,依次重复,直到原始照片全部分割完毕;
第四种:以原始照片的(0,160)为起点,采用320×320像素的滑动窗口依次沿原始照片的横轴进行分割,直到原始照片横轴的最末端,得到第一行320×320像素的分割图片,然后以坐标点(0,480)为起点,进行第二行的图片分割,依次重复,直到原始照片全部分割完毕。
本发明由于采取以上技术方案,其具有以下优点:1、本发明采用深度学习技术建立深度全卷积神经网络模型,能够对培养环境ccd照片中的癌细胞区域自动检测与分割,从而能快速而又精确地定量计算存活癌细胞区域的面积大小,以评估不同药物对癌症病患个体的作用。2、本发明比起传统方法具有很强的鲁棒性,只要配有相应的训练集,即可对不同类型的癌细胞、培养环境与照明条件下的癌细胞进行检测与分割,且准确率高。3、本发明对训练集进行了数据增强处理,加强了深度全卷积神经网络模型的泛化性能,进一步提高了癌细胞识别的准确性。4、本发明在对原始照片进行分割时,采用带有重叠区域的滑动窗口对原始照片进行划分,并在对各分割照片进行汇总时,对各张照片中的重复区域取并集,保证了癌细胞不会因处于划分图片边缘而未被检测出来,进一步提高了检测结果的准确性。本发明可以广泛应用于生物医学图像处理领域。
附图说明
图1是本发明基于深度学习的原代肿瘤细胞分割识别系统结构图;
图2是本发明深度全卷积神经网络结构示意图;
图3是本发明池化与去池化示意图;
图4是本发明数据增强示意图;
图5(a)~图5(d)是本发明对原始图片的四种划分方式。
具体实施方式
下面结合附图和实施例对本发明进行详细的描述。
如图1所示,本发明提供的一种基于深度学习的原代肿瘤细胞分割识别系统,其包括模型构建模块、模型训练模块以及模型预测模块。其中,模型构建模块用于基于深度学习中的人工神经网络构建深度全卷积神经网络模型;模型训练模块用于根据训练集对深度全卷积神经网络模型进行训练,得到训练好的深度全卷积神经网络模型;模型预测模块用于将原始照片分割后输入到训练好的深度全卷积神经网络模型,并将得到的各分割图片的癌细胞区域分割结果汇总后,得到原始照片的癌细胞区域分割结果。
其中,训练集中包含一定数量的320*320像素的rgb图片与其相应的癌细胞区域分割结果(同样是320*320像素,黑色代表癌细胞区域,白色代表其他区域)。训练集由人工标注得到,训练集越大往往训练后的神经网络泛化性能越好。
进一步地,模型预测模块包括图片分割模块、图片识别模块和结果汇总模块,其中,图片分割模块利用有重叠区域的滑动窗口将任意大小的原始照片分为数张320×320像素的rgb图片,并发送到图片识别模块;图片识别模块将各分割图片输入到已训练好的深度全卷积神经网络,得到各分割图片的癌细胞区域分割结果;结果汇总模块用于对所有分割图片的癌细胞区域分割结果进行汇总,得到原始照片的癌细胞区域分割结果。
进一步地,本系统在windows与linux操作系统中均能运行,需要安装与操作系统对应的python语言与pytorch框架。优选安装显卡gpu以加速该模型的训练与使用,本发明选用的是nvidia的geforcegtx1080ti显卡。
基于上述基于深度学习的原代肿瘤细胞分割识别系统,本发明还提供一种基于深度学习的原代肿瘤细胞分割识别方法,包括以下步骤:
1)基于深度学习中的人工神经网络构建深度全卷积神经网络模型;
本发明构建的深度全卷积神经网络模型基于深度学习中的人工神经网络,整个网络中不存在全连接层,因此被称为全卷积神经网络。模型的输入是像素为320×320的rgb三通道图像,输出是像素为320×320的双通道矩阵,两个通道分别代表该像素属于癌细胞区域或其他区域的分数。最后利用softmax函数将输出分数转化为相应的概率。
如图2所示,为整个深度全卷积神经网络的结构图。本发明构建的深度全卷积神经网络模型包括输入层、卷积层、反卷积层、池化层、去池化层和输出层。卷积层和反卷积层分别设置为五层,两两卷积层之间均设置一池化层,两两反卷积层之间均设置一去池化层,输入数据由输入层输入到第一卷积层,依次经过卷积和池化处理后,输出到第一反卷积层,并依次经反卷积和去池化处理后,经第五反卷积层输出到输出层。其中,每一卷积层均设置为两层,第一反卷积层设置为一层,第二到第五反卷积层均设置为两层。
下面对各卷积层、反卷积层、池化层和去池化层进行介绍。
①卷积层与反卷积层
除了模型最后一层外,所有的卷积层与反卷积层均采用3×3的卷积核与相应的填补使得该层的输入与输出具有相同的高度与宽度。这里每个卷积层与反卷积层后面都会加上批标准化层与使用relu激活函数的激活层。批标准化层使前层输出数据分布趋于统一,能避免个别极端数据在多层计算后误差放大导致失效的情况,起到稳定模型的作用,激活层的作用是去线性化。其中,各卷积层的卷积核的初始值以及权重参数初始值均随机生成。
模型的最后一层是采用1×1卷积核无填补的卷积层,输出为320×320的双通道矩阵。
②池化层与去池化层
池化层均采用了核为2×2步幅为2的最大池化,池化层用于对该层的输入进行采样,使得输出的高度与宽度是输入的一半,并记录了采样值的原始位置以供去池化层使用。
如图3所示,与每个池化层对应的去池化层也采用了2×2的核与2的步幅,输出的高度与宽度是输入的两倍,利用池化层采样的原始位置把相应的值填补上去,在其余位置均填入0。
本发明中,卷积层用conv表示,池化层用pool表示,反卷积层用deconv表示,去池化层用unpool表示,各层的具体参数说明如下表1所示。
表1深度全卷积神经网络结构说明
2)采用训练集对构建的深度全卷积神经网络模型进行训练,得到训练好的全卷积神经网络模型;
采用训练集对构建的深度全卷积神经网络模型进行训练的方法,包括以下步骤:
2.1)对训练集进行数据增强处理,得到能够加强模型泛化性能的训练集。
如图4所示,为了加强模型的泛化性能,模型的训练使用了旋转与镜像的数据增强,因为癌细胞的检测与分割并不依赖于这些操作。训练集中的图片(左上角)及其相应的分割有相等的概率会变换成以下各种方式作为输入。
2.2)将得到的训练集输入到构建的深度全卷积神经网络模型中,并计算深度全卷积神经网络模型的损失函数。
模型的损失函数为交叉熵损失,将每个像素分类正确的概率取负对数然后进行平均得到。损失函数还包含了基于l2范数的正则化项用来防止模型过拟合。其中,交叉熵损失函数是神经网路中常用的损失函数,本发明在此不再赘述。
2.3)采用adam(adaptivemomentestimation,自适应矩估计)优化方法对深度全卷积神经网络模型的损失函数进行优化,得到训练好的深度全卷积神经网络模型。
对损失函数的优化可采用带动量的随机梯度下降法,需要根据实际情况调整的超参数包含批处理大小、学习速率、动量与参数衰减。
3)对待识别的原始照片进行分割,并将分割后的图片输入训练好的全卷积神经网络模型中得到各分割图片的癌细胞分割结果,对各分割图片的癌细胞分割结果进行汇总,得到原始照片的癌细胞分割结果。
3.1)采用带有重叠区域的滑动窗口对原始照片进行划分,生成一系列320×320像素的图片;
如图5(a)~5(d)所示,对于任意大小的原始照片,本发明采用带有重叠区域的滑动窗口对原始照片进行划分,生成一系列320×320像素的图片用来作为模型的输入。假设图5中原始照片大小为3200×3200像素,则它会被四种方式划分为数张320×320像素图片。
四种划分方式分别为:
第一种:以原始照片的坐标原点(0,0)为起点,采用320×320像素的滑动窗口依次沿原始照片的横轴进行分割,直到原始照片横轴的最末端,得到第一行320×320像素的分割图片,然后以坐标点为(0,320)的点为起点,进行第二行的图片分割,并依次重复,直到原始照片全部分割完毕;
第二种:以原始照片的(160,160)为起点,采用320×320像素的滑动窗口依次沿原始照片的横轴进行分割,直到原始照片横轴的最末端,得到第一行320×320像素的分割图片,然后以坐标点为(0,320)的点为起点,进行第二行的图片分割,依次重复,直到原始照片全部分割完毕;
第三种:以原始照片上坐标点(160,0)为起点,采用320×320像素的滑动窗口依次沿原始照片的横轴进行分割,直到原始照片横轴的最末端,得到第一行320×320像素的分割图片,然后以坐标点(160,320)为起点,进行第二行的图片分割,依次重复,直到原始照片全部分割完毕;
第四种:以原始照片的(0,160)为起点,采用320×320像素的滑动窗口依次沿原始照片的横轴进行分割,直到原始照片横轴的最末端,得到第一行320×320像素的分割图片,然后以坐标点(0,480)为起点,进行第二行的图片分割,依次重复,直到原始照片全部分割完毕。
3.2)将得到的各320×320像素的图片输入到训练好的深度神经网络模型进行预测,得到各320×320像素的分割图片的癌细胞识别结果,即对于每个像素分类成神经网络输出概率较大的那类。
3.3)对所有分割图片的癌细胞识别结果进行汇总,得到原始图片的癌细胞识别结果。
对各分割图片的癌细胞预测结果进行汇总时,应对各张图片中的重复区域取癌细胞分割预测结果的并集,以保证癌细胞不会因处于划分图片边缘而未被检测出来。最后得到了一张完整的同样大小的图片,黑色表示癌细胞区域,白色表示其他区域。计算黑色像素的个数即得到了癌细胞区域的面积。
上述各实施例仅用于说明本发明,其中各部件的结构、连接方式和制作工艺等都是可以有所变化的,凡是在本发明技术方案的基础上进行的等同变换和改进,均不应排除在本发明的保护范围之外。
- 还没有人留言评论。精彩留言会获得点赞!