用于生物图像中的联合细胞和区域分类的深度学习系统和方法与流程
优先权
本申请要求于2017年12月7日提交的美国专利申请号62/596036的优先权。
背景技术:
数字病理学涉及将整个组织病理学或细胞病理学切片扫描成可在计算机屏幕上解释的数字图像。这些图像随后将由成像算法处理或由病理学家解释。为了检查组织切片(实际上是透明的),使用选择性结合细胞成分的有色组织化学染色剂制备组织切片。临床医生或计算机辅助诊断(cad)算法使用颜色增强或染色的细胞结构来识别疾病的形态学标记,并相应地进行治疗。观察该测定可以实现多种过程,包括疾病诊断、对治疗反应的评估、以及研发抗击疾病的新药物。
免疫组织化学(ihc)载玻片染色可以用于识别组织载玻片的细胞中的蛋白质,并且因此广泛地用于对诸如生物组织中的癌性细胞和免疫细胞等不同类型的细胞的研究中。因此,可以在研究中使用ihc染色以理解癌组织中免疫细胞(诸如,t细胞或b细胞)的差异表达的生物标记物的分布和位置以用于免疫应答研究。例如,肿瘤经常包含免疫细胞的浸润液,该浸润液可以防止肿瘤的发展或有利于肿瘤的向外生长。
原位杂交(ish)可用于寻找遗传异常或病状的存在,例如在显微镜下观察时在形态学上表现为恶性的细胞中特异性地致癌基因扩增。原位杂交(ish)使用与靶基因序列或转录物反义的标记的dna或rna探针分子来检测或定位细胞或组织样品中的靶核酸靶基因。通过将固定在切片上的细胞或组织样品暴露于标记的核酸探针来进行ish,所述核酸探针能够与细胞或组织样品中的给定靶基因特异性杂交。通过将细胞或组织样品暴露于已经用多个不同核酸标签标记的多个核酸探针,可以同时分析几个靶基因。通过利用具有不同发射波长的标记,可以在单个步骤中对单个靶细胞或组织样品进行同时多色分析。
技术实现要素:
针对对生物样本的数字化组织切片进行图像评分和自动图像分析的问题的传统方法通常涉及对图像(或其区域)中的每个细胞进行细胞检测、细胞分割和特征提取,然后进行细胞分类以及区域分割和分类。本公开提供了一种替代性方法,由此直接使用经训练的多层神经网络直接从所获取的图像对细胞和区域进行联合且同时分类。这样,本公开利用深度学习方法,其中在训练多层神经网络之后自动地检测、分割和分类细胞。在一些实施方案中,使用半自动图像分析方法生成的细胞级数据和区域级数据均被用作训练多层神经网络的基础事实。
在一些实施方案中,现有的基于特征的分割技术可以用于生成细胞轮廓的可靠估计,并且可以用作训练多层神经网络的基础事实。认为的是,手动执行此任务会使样品图像(或其区域)中所有细胞的基础事实测量过程不可行,因为样品图像通常非常大并具有数千个细胞。在一些实施方案中,在基于细胞的分类中有用的特征向量的先验知识被用作附加的输入通道,以进一步辅助多层神经网络提高分类准确性。在一些实施方案中,并非使用用户来对每一个细胞进行基础事实测量,而是首先利用现有的基于特征的解决方案来导出初始细胞标记,并且病理学家或其他医学专业人员浏览初始结果集,并校正已被错误分类的细胞。这种半自动解决方案有助于生成除此之外难以获得的大量细胞基础事实数据和区域基础事实数据。
鉴于前述内容,在本公开的一个方面中,是一种用于训练多层神经网络以检测和分类样品图像(例如,具有一种或多种染色剂的生物样品组织的样品图像)内的不同细胞类型(例如肿瘤细胞、基质细胞、淋巴细胞等)和组织区域(例如肿瘤区域、富含淋巴细胞的区域、基质区域、坏死区域等)的方法,所述方法包括:接收样品图像数据;使用半自动过程来生成组织区域基础事实数据和细胞分类基础事实数据两者;以及使用所生成的基础事实数据来训练多层神经网络;其中,基础事实数据的生成考虑了关于某些类型的组织或区域中是否可能存在某些类型的细胞的先验知识。
在一些实施方案中,所生成的组织区域基础事实数据是由病理学家手动识别的。在一些实施方案中,使用自动图像分析算法来执行细胞分类以提供预期的细胞分类(例如,检测种子中心,然后通过对输入图像内的特征进行分析来对所检测的细胞进行分类)。在一些实施方案中,由病理学家检查由图像算法确定的预期细胞分类,并且使用来自病理学家的输入来校正任何错误分类(例如,通过使用图像和注释检查和编辑平台),使得提供最终的细胞分类数据作为多层神经网络的基础事实数据。在一些实施方案中,将组织区域分类数据和最终细胞分类数据进行组合,使得样品图像内的每个像素被标记有表示组织类型和/或细胞类型的单个标记(即,标记是向量)。在一些实施方案中,组织标记和细胞标记的组合排除了生物学上不可行的那些组织和细胞组合,即在自然中不会出现或出现概率非常低的、特定区域中的细胞的组合。在一些实施方案中,样品图像是单形图像(即,用原色染色或因本文所指出的单个生物标记物的存在而染色的图像)。在一些实施方案中,样品图像用苏木精和曙红来染色(h&e图像)。在一些实施方案中,样品图像是单纯免疫组织化学染色的图像或多重图像,由此在生成基础事实数据之前,首先对多重图像解混(例如利用本文所指出的解混模块)。在一些实施方案中,样品图像源自乳腺组织样品。
在本公开的另一方面中,是一种用于训练多层神经网络以检测和分类样品图像内的不同细胞类型和区域的方法,所述方法包括:为了生成用于训练分类器的基础事实,在生物样本的样品图像内识别出不同的组织区域;基于所述样品图像内的图像特征对细胞进行检测和分类,以提供预期的细胞分类;使用来自病理学家的输入来校正不正确分类的预期的细胞分类,以提供最终的细胞分类;基于所识别的组织区域和最终细胞分类的组合,为所述样品图像内的每个像素指定标记,其中,仅为生物学上可行的那些细胞和区域组合指定标记;以及使用所述样品图像和指定给所述样品图像内的每个像素的所述标记来训练所述多层神经网络(即,利用在像素级建立的基础事实数据来训练所述多层神经网络)。
在一些实施方案中,由病理学家手动地执行对所述样品图像内不同区域的识别(例如,所述病理学家可以对图像的与不同组织类型相对应的部分进行注释)。在一些实施方案中,对所述样品图像内的不同组织区域的识别是自动的或半自动的。
在一些实施方案中,基于所述样品图像内的特征对细胞进行检测和分类包括:(i)检测细胞核;(ii)计算前景分割掩码,(iii)从所检测的细胞核导出特征;以及(iv)使用分类器(例如,支持向量机等)基于所导出的特征对所述核进行分类。在一些实施方案中,不生成分割掩码,而是利用将标记指定给整个像斑而不是为每个像素指定标记的detectnet。在这种情况下,像斑对应于以每个细胞为中心的斑点(参见detectnet:用于digits中的对象检测的深度神经网络,网址为https://devblogs.nvidia.com/parallelforall/detectnet-deep-neural-network-object-detection-digits/)。
在一些实施方案中,利用图像分析算法来自动进行对细胞的检测和分类。在一些实施方案中,自动细胞检测和细胞分类提供了可以由病理学家检查的预期细胞分类。在一些实施方案中,病理学家检查预期细胞分类并判定预期细胞分类是否准确。在一些实施方案中,病理学家校正任何不准确性,从而提供修正的细胞分类,所述修正的细胞分类与正确识别出的预期细胞分类一起提供,以向多层神经网络提供最终的细胞分类集以进行训练。
在一些实施方案中,为每个像素指定的标记是向量,所述向量包括所分类的细胞类型和所述像素在所述样品图像中所处的组织区域或组织类型的标识。在一些实施方案中,组织区域包括肿瘤区域、基质区域、富含淋巴细胞的区域和坏死区域。在一些实施方案中,细胞类型包括肿瘤细胞、基质细胞和淋巴细胞。在一些实施方案中,生物学上可行的组合是通常在特定类型的组织中找到细胞的那些组合。仅作为例子,生物学上可行的组合包括:(i)肿瘤区域中的肿瘤细胞;(ii)肿瘤区域中的基质细胞;(iii)肿瘤区域中的淋巴细胞;(iv)基质区域中的肿瘤细胞;(v)基质区域中的基质细胞;(vi)基质区域中的淋巴细胞;(vii)富含淋巴细胞的区域中的肿瘤细胞;以及(viii)富含淋巴细胞的组织中的淋巴细胞。在一些实施方案中,所述方法进一步包括向经训练的多层神经网络提供所述未标记图像,并接收所述未标记图像内的每个像素的预测标记。
在一些实施方案中,将所指定的标记和样品图像提供给多层神经网络,诸如lenet、alexnet、zfnet、googlenet、vggnetresnet、vggnetvgg16、vgg19和densenet,如本文所述。这些神经网络通常在自然图像的大型且公开可用的图像数据档案库(例如imagenet)上进行训练。鉴于通常用于生物样本(诸如肿瘤样品)的训练图像较少,因此采用迁移学习方法。在一些实施方案中,可以使用“迁移学习”方法,其中,将预训练网络(诸如以上提及的那些)连同其学得的权重一起用于在新的训练图像集上初始化训练过程,并对所述预训练网络和所述权重进行训练以使所述权重适应于目前的特定应用。在一些实施方案中,预训练网络在可能或可能不与感兴趣的应用直接相关、并且可能或可能不是生物学性质的现有公共或私有数据集上进行训练。在一些实施方案中,最终层的权重被随机地初始化以避免落入次优局部最小值,而在其他实施方案中,最终层的权重被不变地导入。
本公开的另一方面是一种系统,所述系统包括:多层神经网络,以及基础事实训练模块,所述基础事实训练模块被配置为:(i)在生物样本的样品图像内识别出不同的区域(例如组织区域);(ii)基于所述样品图像内的图像特征对细胞进行检测和分类,以提供建议的细胞分类;(iii)使用来自病理学家的输入来校正不正确分类的建议的细胞分类,以提供最终的细胞分类;(iv)基于所识别的区域和最终细胞分类的组合,为所述样品图像内的每个像素指定标记,其中,仅为生物学上可行的那些细胞和区域组合指定标记;以及(v)使用所述样品图像和指定给所述样品图像内的每个像素的所述标记来训练所述多层神经网络。在一些实施方案中,样品图像是单形图像(例如,用原色或单个生物标记物染色的图像)。在其他实施方案中,样品图像是多重图像。
在一些实施方案中,基础事实训练模块是半监督式的。在一些实施方案中,对不同组织区域的识别包括用不同区域来注释样品图像。在一些实施方案中,注释由病理学家执行。在一些实施方案中,细胞的检测和分类利用识别细胞特征(例如形态特征、核特征等)的图像分析算法。在一些实施方案中,基于所述样品图像内的特征对细胞进行检测和分类包括:(i)检测细胞核;(ii)计算前景分割掩码,(iii)从所检测的细胞核导出特征;以及(iv)使用分类器(例如,支持向量机等)基于所导出的特征对所述核进行分类。在一些实施方案中,基础事实训练模块适于识别生物学上可行的细胞分类和组织区域识别组合,所述组合包括:(i)肿瘤区域中的肿瘤细胞;(ii)肿瘤区域中的基质细胞;(iii)肿瘤区域中的淋巴细胞;(iv)基质区域中的肿瘤细胞;(v)基质区域中的基质细胞;(vi)基质区域中的淋巴细胞;(vii)富含淋巴细胞的区域中的肿瘤细胞;以及(viii)富含淋巴细胞的区域中的淋巴细胞。在一些实施方案中,多层神经网络选自现有的卷积(或全卷积)神经网络架构,诸如lenet、alexnet、zfnet、googlenet、vggnet、resnet、vgg16和densenet。
本公开的另一方面是一种非暂态计算机可读存储介质,其编码有指令,所述指令可由计算系统的处理资源执行,以使所述计算系统能够进行以下操作:在生物样本的样品图像内识别出不同的组织区域;基于所述样品图像内的特征对细胞进行检测和分类,以提供建议的细胞分类;基于所识别的组织区域和最终细胞分类的组合,为所述样品图像内的每个像素指定标记,其中,仅为生物学上可行的那些细胞和区域组合指定标记,并且其中,所述最终细胞分类包括正确地分类的建议的细胞分类和修正的建议的细胞分类,所述修正的建议的细胞分类从病理学家接收作为输入;以及使用所述样品图像和指定给所述样品图像内的每个像素的所述标记来训练所述多层神经网络。在一些实施方案中,对不同组织区域的识别包括从病理学家接收组织区域注释。
本公开的另一方面是一种用于对生物样品的未标记图像内的细胞进行分类的方法,所述方法包括:计算所述未标记图像的前景分割掩码;通过利用计算出的前景分割掩码对所述未标记图像进行过滤来识别出所述未标记图像中的各个细胞;将多层神经网络应用于所述未标记图像,所述多层神经网络被训练成联合地检测和分类不同的细胞类型和组织区域,其中,所述经训练的多层神经网络的应用为所述未标记图像内的每个像素提供了预测标记,所述预测标记对应于如由所述多层神经网络识别出的不同细胞类型;以及为每个识别出的单个细胞指定细胞标记。在一些实施方案中,通过以下方式来指定细胞标记:(i)量化在所识别出的单个细胞内带有各个预测标记的像素的数量;以及(ii)将具有最大数量的预测标记指定为细胞标记(多数投票)。根据权利要求12所述的方法,进一步包括生成包含每个所标记的单个细胞的描述性标志的覆盖图。在一些实施方案中,所述方法进一步包括量化不同地标记的各个细胞并计算表达分数。在一些实施方案中,量化是在预定视野中执行的。在一些实施方案中,由病理学家基于未标记图像内的形态来识别所述预定视野。在一些实施方案中,对肿瘤区域或基质区域中的淋巴细胞的数量进行量化。
本公开的另一方面是一种系统,所述系统包括:细胞检测模块,所述细胞检测模块适于识别未标记图像内的细胞;经训练的多层神经网络,所述经训练的多层神经网络用于为所述未标记图像内的每个像素提供预测标记,所述预测标记对应于不同的细胞类型;以及细胞标记模块,所述细胞标记模块适于将细胞标记赋予由所述细胞检测模块识别出的细胞。在一些实施方案中,通过以下方式来标记细胞:(i)量化在所识别出的细胞内带有各个预测标记的像素的数量;以及(ii)将具有最大数量的预测标记指定为细胞标记(多数投票)。在一些实施方案中,所述细胞检测模块识别所述未标记图像内的种子。在一些实施方案中,所述系统进一步包括评分模块,所述评分模块被配置为对所述未标记图像中的细胞标记计数并且基于各种计数的细胞标记的比率来输出表达分数。在一些实施方案中,在所述未标记图像内的预定视野内执行评分。在一些实施方案中,所述系统进一步包括覆盖掩码模块,所述覆盖掩码模块被配置为将表示不同细胞类型的标志叠加在所述未标记图像上。
本公开的另一方面是一种非暂态计算机可读存储介质,其编码有指令,所述指令可由计算系统的处理资源执行,以使所述计算系统能够进行以下操作:检测未标记图像内的细胞;将多层神经网络应用于所述未标记图像,所述多层神经网络被训练成联合地检测和分类不同的细胞类型和组织区域,其中,所述经训练的多层神经网络的应用为所述未标记图像内的每个像素提供了预测标记,所述预测标记对应于如由所述多层神经网络识别出的不同细胞类型;以及为每个识别出的单个细胞指定细胞标记。在一些实施方案中,通过以下方式来指定细胞标记:(i)量化在所识别出的单个细胞内带有各个预测标记的像素的数量;以及(ii)将具有最大数量的预测标记指定为细胞标记(多数投票)。在一些实施方案中,对未标记图像内的细胞的检测包括:(i)计算所述未标记图像的前景分割掩码;以及(ii)通过利用计算出的前景分割掩码对所述未标记图像进行过滤来识别出所述未标记图像中的各个细胞。在一些实施方案中,所述系统进一步包括用于基于所指定的细胞标记来对所述未标记图像进行评分的指令。在一些实施方案中,所述系统进一步包括用于生成包括表示不同细胞层的标志的覆盖图、并将所述覆盖图应用于所述未标记图像的指令。
鉴于免疫疗法的作用越来越大,因此越来越需要检测和量化h&e图像中的肿瘤浸润性淋巴细胞(til)。此处要解决的主要问题是在统一的深度学习框架中对h&e乳腺癌图像进行联合细胞检测和分类以及区域分割和分类。尽管此处仅针对两个特定的分类问题(基于细胞的分类和基于区域的分类)和特定域(h&e乳腺癌图像)提出了解决方案,但所提出的解决方案适用于其他域的更广泛的分类问题。联合细胞和区域分类工作的目的是通过定量(例如,百分比)度量和空间度量来表征和量化肿瘤的微环境、识别肿瘤间和肿瘤内和细胞的空间关系、以及表征肿瘤浸润性淋巴细胞(包括基质til和肿瘤内til)。
在同一深度学习框架中组合不同分类问题的优点在于:各种分类问题的结果组合于是可以自动完成并由学习框架进行协调,而无需依赖任何后处理(基于集成学习的方法,或基于参数调整的方法)来组合两组结果(例如,细胞级结果和区域级结果)。例如,当我们考虑在一个域中具有k1个分类类别(集合a)的图像、以及然后对于同一图像但在不同域中具有k2个分类类别(集合b)的问题时,然后我们可以将问题视为(k1×k2)多类问题,并且如果(集合a,集合b)中存在某些不可行的元素生物学组合,则我们可以从可以由网络指定的(k1×k2)个类的可容许集合中丢弃它们。因此,利用对所述问题的先验生物学知识来生成一致的结果。诸位申请人进一步提出,与分别各自且单独地解决细胞和区域分类问题相比,区域和细胞分类两者的总体准确性都可以更好,因为同时学习这两者的迭代过程倾向于相互一致的组合分类标记。
附图说明
为了大致理解本公开文本的特征,参考了附图。在附图中,始终使用相同的附图标记来标识相同的要素。
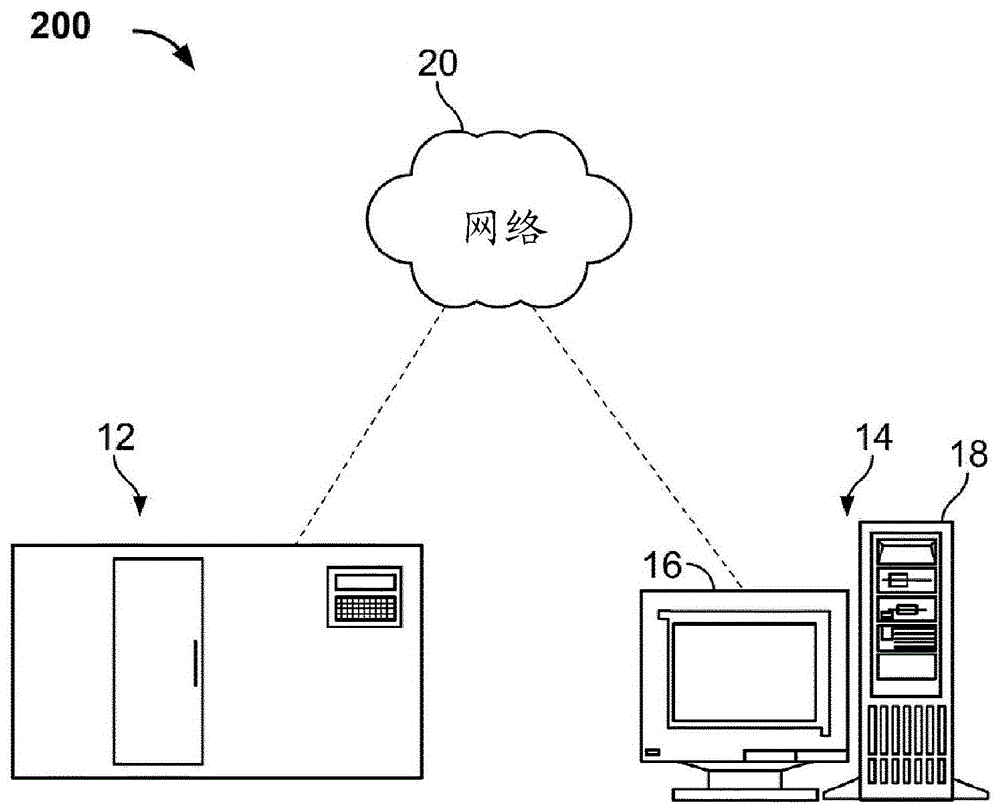
图1展示了包括图像获取装置和计算机系统的代表性数字病理学系统。
图2阐述了可用于数字病理学系统或数字病理学工作流程中的各种模块。
图3a展示了训练多层神经网络以及使用经训练的多层神经网络来提供分类结果。
图3b概述了使用样品图像来训练多层神经网络的步骤。
图3c展示了使用经训练的多层神经网络来对未标记图像中的细胞进行分类的步骤。
图4展示了样品图像(a)、组织区域覆盖图(b)和细胞分类覆盖图(c),其中,肿瘤被描绘为青色,基质被描绘为黄色,淋巴细胞被描绘为红色,并且伪影/排除的区域被描绘成深蓝色。对于图像中的每个像素,指定了区域和细胞标记两者。并且,在像素级为区域和细胞标记指定了颜色代码,以指示其所属的特定分类。
图5展示了样品图像(a)、组织区域覆盖图(b)和细胞分类覆盖图(c),其中,肿瘤被描绘为青色,基质被描绘为黄色,淋巴细胞被描绘为红色,并且伪影/排除的区域被描绘成深蓝色。
图6展示了可以在某些组织类型中找到的各种生物学上可行的细胞组合。
图7展示了样品图像基础事实以及来自组合分类器的分类结果,其中,肿瘤被描绘为青色,基质被描绘为黄色,淋巴细胞被描绘为红色,并且伪影/排除的区域被描绘成深蓝色。上部图像是原始图像,并且细胞基础事实覆盖在原始图像的顶部。
图8展示了样品图像基础事实和分类结果。(a):原始图像,(b):由病理学家生成的细胞级分类基础事实,(c):由算法得到的细胞级分类结果,(d):由算法得到的区域级分类结果,其中,肿瘤被描绘为青色,基质被描绘为黄色,淋巴细胞被描绘为红色,并且伪影/排除的区域被描绘成深蓝色。
具体实施方式
还应当理解,除非明确相反地指示,否则在本文所要求保护的包括多于一个步骤或动作的任何方法中,所述方法的步骤或动作的顺序不一定限于叙述了所述方法的步骤或动作的顺序。
如本文所使用的,除非上下文另外明确地指示,否则单数术语“一个/种(a/an)”和“所述(the)”包括复数指示物。类似地,除非上下文另外明确地指示,否则单词“或”旨在包括“和”。术语“包括(includes)”是包含性地定义的,使得“包括a或b”意指包括a、b或a和b。
如本说明书和权利要求中使用的,“或”应被理解为具有与如上所定义的“和/或”相同的含义。例如,在将所列项目分开时,“或”或“和/或”应解释为包容性的,即包括所列元素中的多个元素或至少一个元素,但也包括一个以上元素,以及(可选地)其他未列出的项目。只有明确相反地指示的术语,诸如“……中的仅一个”或“……中的恰好一个”或者在权利要求中使用时的“由……组成”等将指代恰好包括许多要素或要素列表中的一个要素。一般而言,如本文中所使用的术语“或”之后有诸如“两者之一”、“中的一个”、“中的仅一个”或“中的恰好一个”之类的排他性术语时仅应被解释为指示排他性备选方案(即,“一个或另一个但不是两个”)。“基本上由……组成”在权利要求中使用时它的普通意义如同在专利法领域中使用的那样。
术语“包括(comprising)”、“包括(including)”、“具有(having)”等可互换地使用并且具有相同的含义。类似地,术语“包括(comprises)”、“包括(includes)”、“具有(has)”等可互换地使用并且具有相同的含义。具体而言,每个术语的定义均与美国专利法中常见的“包含”定义相一致,并且因此应解释为开放性术语,意指“以下中的至少一项”,并且还应解释为不排除其他特征、限制、方面等。因此,例如,“具有部件a、b和c的装置”是指所述装置至少包括部件a、b和c。类似地,短语:“一种涉及步骤a、b和c的方法”是指所述方法至少包括步骤a、b和c。此外,尽管本文中可以以特定顺序概述步骤和过程,但是本领域技术人员将认识到排序步骤和过程可以变化。
如本文中在说明书和权利要求中使用的,短语“至少一个”在引用一个或多个要素的列表的情况下,应被理解为意指选自所述要素列表中的这些要素中的任何一个或多个的至少一个要素,但不一定包括在所述要素列表内具体列出的每一个要素中的至少一个,并且不排除要素列表中的要素的任何组合。此定义还允许除了所述要素列表内具体指明的要素之外可以任选地存在短语“至少一个”所指代的要素,而无论与具体指明的那些要素相关还是无关。因此,作为非限制性例子,“a和b中的至少一者”(或等同地,“a或b中的至少一者”,或等效地“a和/或b中的至少一者”)在一个实施方案中可以指代至少一个a,可选地包括一个以上a,而不存在b(并且可选地包括除b之外的要素);在另一个实施方案中,指代至少一个b,可选地包括一个以上b,而不存在a(并且可选地包括除a之外的要素);在又一个实施方案中,指代至少一个a,可选地包括一个以上a和至少一个b,可选地包括一个以上b(和可选地包括其他要素);等等。
如本文所使用的,术语“生物样品”、“样品”或“组织样品”是指从包括病毒在内的任何生物体获得的包括生物分子(诸如蛋白质、肽、核酸、脂质、碳水化合物或其组合)的任何样品。生物的其他例子包括哺乳动物(诸如人类;兽类,诸如猫、狗、马、牛和猪;以及实验动物,诸如小鼠、大鼠和灵长类动物)、昆虫、环节动物、蛛形纲动物、有袋动物、爬行动物、两栖动物、细菌和真菌。生物样品包括组织样品(诸如组织切片和组织穿刺活检)、细胞样品(诸如细胞学涂片(诸如巴氏涂片或血液涂片)或通过显微切割获得的细胞样品)或者细胞组分、片段或细胞器(诸如通过裂解细胞并通过离心或其他方式分离它们的组分获得的)。生物样品的其他例子包括血液、血清、尿液、精液、粪便物、脑脊液、间质液、粘液、泪液、汗液、脓、活检组织(例如,通过外科手术活检或穿刺活检获得的)、乳头抽吸物、耳垢、乳汁、阴道液、唾液、拭子(诸如口腔拭子)或含有源自第一生物样品的生物分子的任何材料。在某些实施方案中,本文使用的术语“生物样品”指从肿瘤制备的样品(例如均质或液化样品)或从受试者获取的肿瘤的一部分。
如本文所使用的,术语“生物标记物”或“标记物”是指某些生物状态或病状的可测量的指标。具体地,生物标记物可以是可以被特定地染色并且指示细胞的生物特征(例如,细胞类型或细胞的生理状态)的蛋白质或肽(诸如,表面蛋白质)。免疫细胞标记物是选择性地指示与哺乳动物的免疫应答相关的特征的生物标记物。生物标记物可以用于确定身体对疾病或病症的治疗反应如何,或者受试者是否易患疾病或病状。在癌症的情况下,生物标记物是指指示体内癌症存在的生物物质。生物标记物可以是肿瘤分泌的分子或机体对癌症存在的特定反应。遗传、表观遗传学、蛋白质组学、糖组学和成像生物标记物可以用于癌症诊断、预后和流行病学。这种生物标记物可以在非侵入性收集的生物流体(如血液或血清)中进行测定。几种基于基因和蛋白质的生物标记物已经用于患者护理,包括但不限于afp(肝癌)、bcr-abl(慢性髓性白血病)、brca1/brca2(乳腺癌/卵巢癌)、brafv600e(黑色素瘤/结肠直肠癌)、ca-125(卵巢癌)、ca19.9(胰腺癌)、cea(结肠直肠癌)、egfr(非小细胞肺癌)、her-2(乳腺癌)、kit(胃肠间质瘤)、psa(前列腺特异性抗原)、s100(黑色素瘤)和许多其他生物标记物。生物标记物可以用作诊断物(用于鉴别早期癌症)和/或预后物(用于预测癌症的侵袭性和/或预测受试者对特定治疗的反应和/或癌症复发的可能性)。
如本文所使用的,术语“视野(fov)”是指具有预定大小和/或形状的图像部分。在一些实施方案中,fov是数字图像中用于进一步手动或自动检查和分析的区域。通过分析数字图像的一些特征,例如通过评估数字图像的像素的强度值,可以自动或手动选择fov。pct/ep2015/062015描述了用于基于图像中的某些预定标准或特性而生成特定fov的方法(例如,对于被多于两种染色剂染色的生物样品,识别图像的仅包括两种染色剂的区域)。
如本文所使用的,术语“图像数据”涵盖从生物组织样品获取(诸如借助于光学传感器或传感器阵列)的原始图像数据或经过预处理的图像数据。具体地,图像数据可以包括像素矩阵。
如本文所使用的,术语“免疫组织化学”是指一种通过检测抗原与特定结合药剂(诸如,抗体)的相互作用来确定样品中抗原的存在或分布的方法。在允许抗体-抗原结合的条件下,样品与抗体接触。抗体-抗原结合可以借助于与抗体结合的可检测标记(直接检测)或借助于与特异性结合第一抗体的第二抗体结合的可检测标记(间接检测)来检测。
如本文所使用的,术语“掩码”是指数字图像的派生物,其中,掩码中的每个像素被表示为二进制值,例如“1”或“0”(或“真”或“假”)。通过使用所述掩码来覆盖数字图像,在对数字图像施加的进一步处理步骤中隐藏、移除或以他方式忽略或过滤掉映射到二进制值中的特定一个值的掩码像素的数字图像的所有像素。例如,可以通过将具有高于阈值的强度值的原始图像的所有像素指定为真并且在其他情况下指定为假来从原始数字图像中生成掩码,从而创建可以过滤掉将被“假”掩码像素覆盖的所有像素的掩码。
如本文所使用的,术语“多通道图像”或“多重图像”涵盖从生物组织样品获取的数字图像,其中,不同的生物结构(诸如细胞核和组织结构)同时用特定的荧光染料、量子点、发色剂等染色,其每一个都发出荧光或者在不同的光谱带中可检测到,从而构成多通道图像的通道之一。
概述
本公开涉及用于训练多层神经网络以在样品图像内对细胞和区域进行联合且同时分类的自动系统和方法。本公开还涉及用于使用经训练的多层神经网络对未标记图像内的细胞进行分类的自动系统和方法。
本公开的至少一些实施方案涉及计算机系统和用于分析从生物样品中捕获的数字图像的方法,所述生物样品包括用一个或多个原色(例如苏木精和曙红(h&e))以及一个或多个检测探针(例如,包含促进对样品内的目标进行标记的特定结合实体的探针)染色的组织样品。虽然本文的例子可以指特定组织和/或用于检测某些标记物(并且因此检测疾病)的特定染色剂或检测探针的应用,但本领域技术人员将认识到的是可以应用不同的组织和不同的染色剂/检测探针来检测不同的标记物和不同的疾病。
图1和图2中展示了用于对样本进行成像和分析的数字病理学系统200。数字病理学系统200可以包括成像设备12(例如,具有用于扫描承载样本的显微镜切片的装置的设备)和计算机14,由此成像设备12和计算机可以通信地耦合在一起(例如,直接地或通过网络20间接地)。计算机系统14可以包括台式计算机、膝上型计算机、平板计算机等、数字电子电路系统、固件、硬件、存储器201、计算机存储介质(240)、计算机程序或指令集(例如,其中,所述程序存储在存储器或存储介质中)、一个或多个处理器(209)(包括经过编程的处理器)、以及任何其他硬件、软件、或固件模块或其组合(诸如本文中进一步描述的)。例如,图1中展示的计算系统14可以包括具有显示装置16和外壳18的计算机。计算机系统可以以二进制形式存储数字图像(本地地诸如存储在存储器、服务器或另一个网络连接装置中)。还可以将数字图像分成像素矩阵。像素可以包括由位深定义的具有一个或多个位的数字值。本领域技术人员将认识到,可以利用其他计算机装置或系统,并且本文所描述的计算机系统可以通信地耦合到附加部件,例如样本分析仪、显微镜、其他成像系统、自动切片制备设备等。本文进一步描述了这些附加部件以及各种计算机、网络等中可以利用的一些。
成像设备12(或包括存储器中存储的预先扫描的图像的其他图像来源)通常可以包括但不限于一个或多个图像捕获装置。图像捕获装置可以包括但不限于相机(例如,模拟相机、数字相机等)、光学器件(例如,一个或多个透镜、传感器聚焦透镜组、显微镜物镜等)、成像传感器(例如,电荷耦合装置(ccd)、互补金属氧化物半导体(cmos)图像传感器等)、胶片等。在数字实施方案中,图像捕获装置可以包括协作以证明即时聚焦的多个透镜。图像传感器(例如,ccd传感器)可以捕获样本的数字图像。在一些实施方案中,成像设备12是明场成像系统、多光谱成像(msi)系统或荧光显微镜系统。数字化的组织数据可以例如由图像扫描系统生成,诸如(亚利桑那州图森市的)ventanamedicalsystems公司的ventanaiscanht扫描仪或其他适合的成像装备。本文进一步描述了附加成像装置和系统。本领域技术人员将认识到,由成像设备12获取的数字彩色图像通常由基本颜色像素构成。每个彩色像素可以在三个数字分量上编码,每个数字分量包括相同数量的位,每个分量对应于原色,通常是红色、绿色或蓝色,也由术语“rgb”分量表示。
图2提供了对在当前公开的数字病理学系统中利用的各种模块的概述。在一些实施方案中,数字病理学系统采用具有一个或多个处理器209和至少一个存储器201的计算机装置或计算机实施的方法,所述至少一个存储器201存储非暂态计算机可读指令以由所述一个或多个处理器执行从而使所述一个或多个处理器执行多层神经网络220以及基础事实训练模块210或测试模块230中的至少一者中的指令(或存储的数据)。
在一些实施方案中,基础事实训练模块210适于从成像模块202接收一个或多个样品图像,所述成像模块通信地耦合到成像设备12或存储模块240。在接收到图像数据之后,基础事实训练模块210可以执行一个或多个模块(或子模块)(即模块203至207),使得可以生成基础事实数据并将其提供给多层神经网络220以进行训练。在一些实施方案中,基础事实训练模块210可以首先执行区域识别模块203以使得可以对样品图像内的区域或组织区域进行分类。在一些实施方案中,区域识别模块203从病理学家或其他经训练的医学专业人员接收输入,所接收的信息包括图像注释。随后,基础事实训练模块210可以执行细胞检测模块204,以识别样品图像内的细胞,并在进行细胞检测之后使用细胞分类模块205对这些细胞进行分类。可以运行分类调整模块206,使得可以调整来自细胞分类模块205的细胞分类结果,诸如通过使用来自病理学家或其他训练医学专业人员的输入。最后,可以使用数据组合模块207来组合来自区域识别模块203的区域分类数据和来自细胞分类模块205和/或分类调整模块的细胞分类,以便为样品图像内的每个像素提供单个标记。然后可以将像素标记数据(即,生成的基础事实信息)与样品图像一起提供给多层神经网络220,使得可以训练多层神经网络。在一些实施方案中,基础事实训练模块210是半监督式的。在一些实施方案中,从图像模块202接收的作为输入的样品图像是单形图像。在其他实施方案中,从成像模块接收的作为输入的样品图像是多重图像。
在一些实施方案中,测试模块230被配置为从成像模块202接收一个或多个未标记图像。在接收到未标记图像数据之后,测试模块230可以执行一个或多个模块(或子模块),即模块204和208。在一些实施方案中,测试模块适于首先执行细胞检测模块204中的指令,使得可以识别出未标记图像内的细胞。在细胞识别之后,将未标记图像提供给经训练的多层神经网络220,使得可以为未标记图像内的所有像素指定预测细胞标记。然后,测试模块210利用细胞标记模块208来为每个识别出的细胞指定标记。在一些实施方案中,附加模块(未描绘)可以由测试模块(例如评分模块)运行,以基于细胞标记提供表达分数,并且可以由覆盖图生成模块运行,以提供标识标志作为未标记图像上的覆盖图。
本领域技术人员还将认识到,可以将附加模块结合到工作流中,并且与基础事实训练模块210或测试模块230一起使用。在一些实施方案中,如本文更详细地描述的,可以运行图像处理或预处理模块以将某些滤波器应用于获取的图像或识别组织样品内的某些组织学和/或形态结构或特征。同样,感兴趣区域选择模块可以用于选择样品图像或测试的特定部分进行分析。另外,可以运行解混模块以在进一步处理之前对多重图像进行解混。
图像获取模块
在一些实施方案中,并且作为初始步骤并参考图2,数字病理学系统200运行图像获取模块202以捕获具有一种或多种染色剂的生物样品的图像(例如,使用成像设备12)或图像数据(即,图像可以是单形图像或多重图像)。在一些实施方案中,接收或获取的图像是rgb图像或多光谱图像。在一些实施方案中,捕获的图像被存储在存储器201中(或存储在存储模块240内)。可以从获取的图像中导出基础事实数据以训练神经网络,即,可以获取或检索样品图像或样品图像数据以生成基础事实数据(步骤300至302)。同样,可以获取或检索未标记图像或未标记图像数据,并且可以使用经训练的多层神经网络对未标记图像或未标记图像数据内的细胞进行分类(步骤303至305)。样品图像或未标记图像中的任何一者都是整个切片图像或其任何部分(例如,预定视野)。
在一些实施方案中,代替捕获的原始图像,可以使用来自捕获的原始图像的任何一组可选的预处理图像,作为独立的输入图像或者与捕获的原始图像相组合。因此,如本文所述,当将经训练的网络应用于未标记图像时,可以使用类似的预处理步骤。
图像或图像数据(在本文中可互换使用)可以使用成像设备12获取(诸如实时地)。在一些实施方案中,图像是从显微镜或能够捕获承载样本的显微镜切片的图像数据的其他仪器获取的,如本文所指出的。在一些实施方案中,图像是使用2d扫描仪获取的,诸如能够扫描图像块的扫描仪,或者能够以逐行方式扫描图像的行扫描仪,诸如ventanadp200扫描仪。可替代地,图像可以是先前已经获取(例如,扫描)并且存储在存储器201中(或者就此而言,经由网络20从服务器中检索到)的图像。
在一些实施方案中,系统接收至少一个图像作为用于导出基础事实数据的输入。一般而言,作为输入而接收的所述图像各自包括与染色剂(包括,显色、荧光团、量子点等)相对应的信号。在一些实施方案中,所述至少一个图像已经用至少一种原色(苏木精或曙红)染色。在其他实施方案中,已经在用于标识特定生物标记物的ihc测定或ish测定中的至少一者中染色了所述至少一个图像(在本文称为“生物标记物”图像)。在一些实施方案中,多个单形图像是作为输入的,并且这些图像可以从连续组织切片(即,从相同的异种移植组织块中导出的连续切片)中导出。在一些实施方案中,输入图像可以是多重图像,即,根据本领域技术人员已知的方法针对多重测定中的多个不同标记物进行染色的图像。
典型的生物样品在向样品施加染色剂的染色/测定平台上进行处理。在一些实施方案中,样本处理设备是自动设备,诸如由ventanamedicalsystems公司出售的benchmarkxt仪器、symphony仪器、benchmarkultra仪器。ventanamedicalsystems公司是许多披露了用于执行自动分析的系统和方法的美国专利的受让人,包括美国专利号5,650,327、5,654,200、6,296,809、6,352,861、6,827,901和6,943,029,以及美国公开专利申请号20030211630和20040052685。可替代地,可以手动处理样本。
市售的h&e染色器的例子包括来自roche的ventanasymphony(单个切片染色器)和ventanahe600(单个切片染色器)系列h&e染色器;来自agilenttechnologies的dakocoverstainer(批量染色器);来自leicabiosystemsnusslochgmbh的leicast4020小型线性染色器(批量染色器)、leicast5020多功能染色器(批量染色器)和leicast5010autostainerxl系列(批量染色器)h&e染色器。市场上有其他适合用作染色/测定平台的商业产品,其中一个例子是ventanamedicalsystems公司(亚利桑那州图森市)的discoverytm。
相机平台还可以包括明场显微镜(一个例子是ventanamedicalsystems公司的ventanaiscanht产品)或具有一个或多个物镜和数字成像器以及一组光谱滤镜的任何显微镜。可以使用用于捕获不同波长的图像的其他技术。适于对染色的生物样本进行成像的另外的相机平台在本领域是已知的并且可从诸如zeiss、canon、appliedspectralimaging和其他公司商购获得,并且这种平台容易适用于本主题公开文本的系统、方法和设备。
如本领域技术人员将认识到的,可以针对不同类型的核和/或细胞膜生物标记物来对生物样品进行染色。例如在“sambrook等人,molecularcloning:alaboratorymanual,coldspringharborlaboratorypress(1989)”和“ausubel等人,currentprotocolsinmolecularbiology,greenepublishingassociatesandwiley-intersciences(1987)”中讨论了用于染色组织结构的方法和选择适于各种目的的染色剂的指南。
作为一个非限制性例子,并且在检测乳腺癌的情况下,在一些实施方案中,在ihc测定中将组织样品染色以存在一个或多个生物标记物,所述一个或多个生物标记包括雌激素受体标记物、孕酮受体标记物、ki-67标记物或her2标记物。由此,在一些实施方案中,用作输入的生物标记物图像是包括与雌激素受体(er)标记物、孕酮受体(pr)标记物、ki-67标记物、或her2标记物中的至少一个的存在相对应的信号(与可能例如为发色的或荧光的染色剂相对应的信号)的ihc图像。在一些实施方案中,可以对样品进行分析以检测或测量样品中er、her2、ki-67和pr蛋白质的存在,例如定性或定量测量。作为另一个非限制性例子,并且在检测非小细胞肺癌的情况下,在一些实施方案中,在ihc测定中将组织样品针对一个或多个生物标记物(包括pd-l1生物标记物)的存在进行染色。由此,在一些实施方案中,用作输入的生物标记物图像是包括与pd-l1标记物、cd3标记物和cd8标记物的存在相对应的信号的ihc图像。
无论将获取的图像用作样品图像以进行基础事实测量还是用作未标记图像以进行分类,图像都可能包含需要掩码的信息。在一些实施方案中,用如本文所述的组织掩码模块来可选地对输入图像进行掩码。在一些实施方案中,输入图像被掩码成使得仅组织区域存在于图像中。在一些实施方案中,生成组织区域掩码以由组织区域来掩码非组织区域。在一些实施方案中,可以通过识别组织区域并自动或半自动(即,具有最小化的用户输入)排除背景区域(例如,对应于没有样品的玻璃的全切片图像的区域,诸如仅存在来自成像源的白光的区域)来创建组织区域掩码。如本领域技术人员将认识到的,除了由组织区域来掩码非组织区域之外,组织掩码模块还可以根据需要掩码其他兴趣区,诸如,被识别为属于某一组织类型或属于疑似肿瘤区域的组织的一部分。在一些实施方案中,使用分割技术通过在输入图像中由非组织区域掩码组织区域来生成组织区域掩码图像。适当的分割技术是如本领域已知的这种技术(参见《数字图像处理》,第三版,rafaelc.gonzalez,richarde.woods,第10章,第689页和医学成像手册,处理与分析,isaacn.bankmanacademicpress,2000,第2章)。在标题为“animageprocessingmethodandsystemforanalyzingamulti-channelimageobtainedfromabiologicaltissuesamplebeingstainedbymultiplestains[用于分析从由多个染色剂染色的生物组织样品中获得多通道图像的图像处理方法和系统]”的pct/ep/2015/062015中披露了与生成组织区域掩码相关的附加信息和例子。
训练多层神经网络
参考图3a所示,对多层神经网络的训练包括以下步骤:(i)接收样品图像数据(步骤300)(例如,使用图像获取模块202);(ii)从样品图像数据生成基础事实数据(步骤301)(例如,使用基础事实训练模块210;以及(iii)使用样品图像数据和生成的基础事实数据来训练多层神经网络(220)(步骤302)。
给定样品图像,可以在样品图像中识别出不同的组织类型和/或细胞类型。例如,可以将样品图像分割成不同类型的区域,包括肿瘤区域、富含淋巴细胞的区域、基质区域和坏死区域。另外,可以检测样品图像内的细胞并将其分类为肿瘤、淋巴细胞、基质细胞等。鉴于此,生成基础事实数据(步骤301)包括几个步骤,如图3b中概述的,所述步骤包括:(a)识别样品图像数据中的组织区域(步骤311);(b)对样品图像数据中的细胞进行检测和分类(步骤312);(c)使用从医学专业人员(例如,经训练以识别和分类生物样品中的不同细胞类型的病理学家或其他医学专家)接收的输入来调整或校正细胞分类(步骤313);以及(d)在像素级上组合组织区域识别和校正的/调整的细胞分类,使得每个像素包括单个标记(例如,向量)(步骤314和315)。在生成基础事实数据(步骤311至315)之后,将基础事实数据馈送到多层神经网络(220),使得可以训练神经网络(步骤316)。给定病理学家或医学专家的输入,基础事实数据的生成是半监督式的。本文将进一步详细描述这些步骤中的每一个步骤。
区域识别
然后,诸如通过使用区域识别模块203来为样品图像内的各个区域生成基础事实数据。例如,可以从h&e样品图像中生成与各种组织区域(例如正常组织、肿瘤组织、坏死组织、富含淋巴细胞的淋巴细胞区域、基质组织等)有关的基础事实数据。当然,本领域技术人员将认识到,所识别出的区域可以是肿瘤周围区域、免疫边缘区域、血管浸润、神经浸润、粉刺样区域、浆细胞或富含中性粒细胞的区域、活化基质、出血、正常腺泡或导管等等,并且所识别出的区域的类型取决于生物样品的类型。在一些实施方案中,识别出的部分可以是特定生物标记物(例如,特定ihc标记物)的代表性过表达肿瘤区域。
在一些实施方案中,样品图像内的各种组织区域可以由病理学家或医学专业人员手动识别。例如,检查生物样品的病理学家可以通过注释样品图像来注释不同的区域或组织区域。假设组织区域基础事实数据是手动生成的,则可以认为基础事实数据的生成(步骤301)是半自动的。
在一些实施方案中,可以使用在查看器应用(例如,virtuoso/verso(tm)或类似软件)中提供的注释工具来绘制图像注释并且可以以任何放大率或分辨率来绘制注释。在一些实施方案中,病理学家手动注释特定区域(例如肿瘤)的边界,所述边界可通过不同的颜色、纹理、组织学结构和细胞空间排列在视觉上识别。在一些实施方案中,注释边界被转换为实心(例如,封闭的)多边形,每个实心多边形对应于单个结构(例如,单个肿瘤巢)。
可替代地或另外,可以使用图像分析操作来自动地检测肿瘤区域或使用自动图像分析操作(诸如,分割、阈值化、边缘检测等)来检测其他区域,并且基于所检测到的区域来自动地生成fov。在一些实施方案中,根据在2015年1月23日提交的题为“adaptiveclassificationforwholeslidetissuesegmentation[用于整个切片组织分割的自适应分类]”(被公开为wo2015/113895)的pct/ep2015/051302中描述的方法来执行图像分割和组织类型识别。一般而言,wo2015/113895描述了经由与区域的分类相关的操作来从图像中的其他区域分割肿瘤区域、为经分类的网格点指定高置信度得分和低置信度得分中的至少一个、基于被指定有高置信度得分的网格点来修改组织类型的已知特征数据库、并且生成经修改的数据库、并基于经修改的数据库对被指定有低置信度得分的网格点进行重新分类,以分割组织(例如,识别图像中的组织区域),所述区域的分类包括:识别组织图像中的网格点、将网格点分类为多种组织类型之一、并且基于组织类型的已知特征的数据库来生成经分类网格点。
不管是经由手动过程还是经由自动过程来识别组织区域,都可以生成特定组织区域的组织区域掩码,诸如图4的面板(b)和图5的面板(b)所展示的。在美国专利申请公开号2017/0154420中描述了生成组织区域掩码的方法。
细胞检测和分类
在图像获取和/或解混合之后,将输入图像或解混图像通道图像提供给细胞检测模块204以检测细胞,并随后将其提供给细胞分类模块205以对细胞和/或核进行分类(步骤300)。本文所述的过程和算法可以适于基于输入图像内的特征来对各种类型的细胞或细胞核进行识别和分类,包括对肿瘤细胞、非肿瘤细胞、基质细胞和淋巴细胞进行识别和分类。本领域技术人员将认识到,所识别出的细胞类型可能取决于样品图像的类型和染色,例如在免疫细胞的情况下,可能是检测并分类了包括cd3和cd8在内的不同类型的免疫细胞。同样,细胞分类可以是标记物阳性肿瘤细胞或标记物阴性肿瘤细胞。作为另一个例子,在用免疫标记物(诸如cd3或cd8淋巴细胞)染色的ihc图像中,具有膜标记物染色(dab)的细胞是阳性的,而被复染色的淋巴细胞是标记物阴性的。本领域技术人员将认识到,不同的组织由不同的细胞类型组成,例如,甲状腺乳头状癌中的乳突、腺癌中的腺体以及化生性癌中的多核巨细胞。
本领域技术人员将认识到,细胞的核、细胞质和膜具有不同的特性,并且不同地染色的组织样品可能揭示不同的生物学特征。实际上,本领域技术人员将认识到,某些细胞表面受体可以具有定位于膜或者定位于细胞质的染色模式。这样,“膜”染色模式与“细胞质”染色模式在分析上是不同的。同样,“细胞质”染色模式和“细胞核”染色模式在分析上是不同的。这些不同的染色模式中的每一个都可以用作标识细胞和/或核的特征。例如,基质细胞可能由fap强烈染色,而肿瘤上皮细胞可能由epcam强烈染色,而细胞角蛋白可能由panck染色。因此,通过利用不同的染色剂,可以在图像分析期间区别和区分不同的细胞类型以提供分类解决方案。
在美国专利号7,760,927(“‘927专利”)中描述了用于在具有一种或多种染色剂的生物样品图像中对核、细胞膜和细胞质进行识别、分类和/或评分的方法。例如,us7,760,927描述了一种用于同时识别用生物标记物染色的生物组织的输入图像中的多个像素的自动方法,所述方法包括:考虑将输入图像的前景中多个像素的第一颜色平面以用于同时识别细胞质像素和细胞膜像素,其中,已对所述输入图像进行处理以移除所述输入图像的背景部分并移除所述输入图像的复染成分;确定数字图像的前景中的细胞质像素与细胞膜像素之间的阈值水平;以及使用所确定的阈值水平,与从前景选择的像素及其八个相邻像素同时确定所选择的像素是数字图像中的细胞质像素、细胞膜像素还是过渡像素。
在一些实施方案中,通过首先识别出候选核、然后自动区分肿瘤核与非肿瘤核,来自动识别肿瘤核。在组织图像中识别候选核的许多方法是本领域已知的。例如,可以通过应用基于径向对称的方法(诸如对解混后的苏木精图像通道或生物标记物图像通道)来执行自动候选核检测(参见parvin,bahram等人的“iterativevotingforinferenceofstructuralsaliencyandcharacterizationofsubcellularevents[迭代投票以推断亚细胞事件的结构显著性和特性]”,imageprocessing,ieeetransactionson16.3(2007):615-623)。
更具体地,在一些实施方案中,处理作为输入接收的图像以诸如检测核中心(种子)和/或分割核。例如,可以提供指令以使用parvin的技术基于径向对称投票来检测核中心(以上指出的)。在一些实施方案中,使用径向对称来检测核以检测核的中心并且然后基于细胞中心周围的染色强度来对核进行分类。在一些实施方案中,如共同转让的共同未决的专利申请wo/2014/140085a1中所描述的,使用基于径向对称的核检测操作。例如,可以在图像内计算图像大小,并且通过将所选区域内的大小的总和相加来累积在每个像素处的一个或多个投票。可以使用均值平移聚类来找出区域中的局部中心,其中,局部中心表示实际核位置。基于径向对称投票的核检测在彩色图像强度数据上执行并且使显式使用核为具有不同大小和离心率的椭圆形斑点的先验域知识。为了实现这一点,与输入图像中的颜色强度一起,图像梯度信息也用于径向对称投票并且与自适应分割过程组合以精确地检测和定位细胞核。如本文使用的“梯度”是例如通过考虑围绕所述特定像素的一组像素的强度值梯度来针对特定像素计算的像素的强度梯度。每个梯度可以具有相对于由数字图像的两个正交边缘定义x和y轴的坐标系的特定“取向”。例如,核种子检测涉及将种子定义为假设位于细胞核内的点,并且用作定位细胞核的起始点。第一步骤是使用基于径向对称的高度稳健的方法检测与每个细胞核相关联的种子点,以检测椭圆形斑点、类似于细胞核的结构。径向对称方法使用基于内核的投票过程来对梯度图像进行操作。通过处理通过投票内核积累投票的每个像素来创建投票应答矩阵。内核基于在特定像素处计算的梯度方向以及预期的最小和最大核大小以及投票内核角的范围(通常在[π/4,π/8]范围内)。在所得投票空间中,可以求解出具有高于预定阈值的投票值的局部极大值位置作为种子点。稍后可以在后续分割或分类过程期间丢弃外来种子。在美国专利公开号2017/0140246中讨论了其他方法。
可以使用本领域普通技术人员已知的其他技术来识别核。例如,可以根据h&e或ihc图像之一的特定图像通道来计算图像大小,并且在指定大小周围的每个像素可以被指定有基于像素周围区域内的大小总和的多个投票。可替代地,可以执行均值偏移聚类操作以找出投票图像内的表示核的实际位置的局部中心。在其他实施方案中,可以使用核分割来基于现在已知的核中心经由形态操作和局部阈值化来分割整个核。在又其他实施方案中,可以利用基于模型的分割来检测核(即,从训练数据集中学习核的形状模型,并将其作为先验知识来分割测试图像中的核)。
在一些实施方案中,然后随后使用针对每个核单独计算的阈值来分割核。例如,otsu方法可以用于在经识别核周围区域中进行分割,由于认为核区域中的像素强度是变化的。如本领域普通技术人员将理解的,otsu方法用于通过使类内方差最小化来确定最优阈值并且对于本领域技术人员而言是已知的。更具体地,otsu方法用于自动地执行基于聚类的图像阈值或将灰度图像减小为二进制图像。算法假设图像包含遵循双模态直方图的两类像素(前景像素和后景像素)。然后,计算使两个类分离的最佳阈值,使得它们的组合扩展(类内方差)最小或等效(因为成对平方距离的总和是恒定的),因此它们的类间方差是最大的。
在一些实施方案中,所述系统和方法进一步包括自动分析图像中所识别出的核的光谱和/或形状特征,以识别非肿瘤细胞的核。例如,可以在第一步中在第一数字图像中识别斑点。如本文所使用的“斑点”可以是例如数字图像的区域,在所述区域中,一些属性(诸如强度或灰度值)是恒定的或在规定的值范围内变化。在某种意义上,可以将斑点中的所有像素视为彼此相似。例如,可以使用基于数字图像上位置函数的导数的差分方法以及基于局部极值的方法来识别斑点。核斑点是这样的斑点,其像素和/或其轮廓形状表明所述斑点可能是由用第一染色剂染色的核生成的。例如,可以评估斑点的径向对称,以确定所述斑点是应被识别为核斑点还是任何其他结构,例如染色伪影。例如,在斑点具有长的形状并且不是径向对称的情况下,所述斑点不可以被识别为核斑点,而是被识别为染色伪影。取决于实施方案,被识别为“核斑点”的斑点可以表示一组像素,所述一组像素被识别为候选核并且可以被进一步分析以确定所述核斑点是否表示核。在一些实施方案中,任何种类的核斑点都直接用作“识别出的核”。在一些实施方案中,对识别出的核或核斑点进行过滤操作,以识别出不属于生物标记物阳性肿瘤细胞的核,并从已识别出的核的列表中移除所述识别出的非肿瘤核或从一开始就不将所述核添加到识别出的核的列表。例如,可以分析所识别出的核斑点的另外的光谱和/或形状特征,以确定核或核斑点是否是肿瘤细胞的核。例如,淋巴细胞的核大于其他组织细胞(例如肺细胞)的核。在肿瘤细胞源自肺组织的情况下,通过识别出最小大小或直径明显大于正常肺细胞核的平均大小或直径的所有核斑点来识别淋巴细胞的核。与淋巴细胞的核有关的识别出的核斑点可从所述一组已识别出的核中被移除(即“从中滤出”)。通过滤出非肿瘤细胞的核,可以提高所述方法的准确性。取决于生物标记物,非肿瘤细胞也可以在一定程度上表达生物标记物,并且因此可以在第一数字图像中产生不源自肿瘤细胞的强度信号。通过从全部已识别出的核中识别和滤出不属于肿瘤细胞的核,可以提高识别生物标记物阳性肿瘤细胞的准确性。在美国专利公开2017/0103521中描述了这些和其他方法。在一些实施方案中,一旦检测到种子,就可以使用局部自适应阈值方法并且创建检测到的中心周围的斑点。在一些实施方案中,还可以结合其他方法,诸如,还可以使用基于标记物的流域算法来识别检测到的核中心周围的核斑点。在共同未决的申请pct/ep2016/051906(被公开为wo2016/120442)中描述了这些和其他方法。
在检测到核之后,从输入图像内导出特征(或度量)。从核特征导出度量是本领域中所熟知的,并且已知的任何核特征可以用于本公开的上下文中。可以计算度量的非限制性例子包括:
(a)从形态特征导出的度量
如本文中使用的“形态特征”是例如指示核的形状或尺寸的特征。在不希望受任何特定理论的束缚的情况下,认为形态特征提供关于细胞或其核的大小和形状的一些重要信息。例如,可以通过对包含在核斑点或种子中或周围的像素应用各种图像分析算术来计算形态特征。在一些实施方案中,形态特征包括面积、短轴长度和长轴长度、周长、半径、坚固性等。在细胞级上,此类特征用于将核分类为属于健康或患病细胞。在组织级上,组织中这些特征的统计数据可用于是否将组织分类为患病。
(b)从表观特征导出度量
例如,如本文使用的“表观特征”是针对特定核通过对包含在用于识别核的核斑点或种子中或其周围的像素的像素强度值进行比较来计算的特征,由此所比较的像素密度是从不同图像通道中导出的(例如,背景通道、用于对生物标记物进行染色的通道等)。在一些实施方案中,可以根据像素强度的百分数值(例如,第10位、第50位和第95位百分数值)以及根据不同图像通道计算的梯度大小的百分数值来计算从表观特征导出的度量。例如,首先,识别表示兴趣核的核斑点内的多个图像通道ic(例如,三个通道:htx、dab、亮度)中的每一个的像素值的多个(p个)x百分位值(x=10、50、95)。计算表观特征度量可能是有利的,因为导出的度量可以描述核区域的属性以及描述核周围的膜区域。
(c)从背景特征导出的度量
例如,“背景特征”是指示细胞质中的表观和/或染色存在的特征,以及包括从图像中提取背景特征的核的细胞的细胞膜特征。可以例如通过以下各种操作来计算数字图像中描绘的核和相应细胞的背景特征和相应度量:识别表示核的核斑点或种子;分析与所计算的经识别细胞组直接相邻的像素面积(例如,在核斑点边界周围的20个像素(大约9微米)厚的带状结构),因此,在具有这种核的细胞的细胞质和膜以及与该细胞直接相邻的面积中捕获表观和染色存在。这些度量类似于核表观特征,但是在每个核边界周围大约20像素(大约9微米)厚的带状结构中计算,因此捕获具有经识别核的细胞的细胞质和细胞膜以及直接与该细胞相邻的面积中捕获表观和染色存在。在不希望受任何特定理论的束缚的情况下,选择带状结构大小,因为认为其捕获在核周围的可以用于提供用于核区分的有用信息的足够量的背景组织区。这些特征与由“j.kong等人,“acomprehensiveframeworkforclassificationofnucleiindigitalmicroscopyimaging:anapplicationtodiffusegliomas,”isbi,2011,第2128-2131页”披露的特征类似。认为可以使用这些特征来判定周围组织是基质还是上皮细胞(诸如,在h&e染色组织样品中)。在不希望受任何特定理论的束缚的情况下,认为这些背景特征还捕获膜染色模式,所述膜染色模式在使用适当的膜染色剂染色组织样品时是有用的。
(d)从颜色导出的度量。
在一些实施方案中,从颜色导出的度量包括颜色比率(r/(r+g+b))或颜色主要成分。在其他实施方案中,从颜色导出的度量包括对颜色中每种颜色的局部统计(均值/中值/方差/标准偏差)和/或局部图像窗口中的颜色强度相关性。
(e)从强度特征导出的度量
将具有某些特定属性值的所述一组相邻细胞设置在以组织病理学切片图像表示的灰色细胞的黑色阴影与白色阴影之间。颜色特征的相关性定义了大小类的实例,因此这种颜色细胞的强度从其周围的暗细胞聚类中确定受影响的细胞。
(f)从纹理特征导出的度量
pct公开号wo/2016/075095和wo/2016/075096中描述了纹理特征及其衍生方法的例子。
(g)从空间特征导出的度量
在一些实施方案中,空间特征包括:细胞的局部密度;两个相邻检测细胞之间的平均距离;和/或从细胞到分割区域的距离。
(h)从核特征导出的度量
本领域技术人员还将认识到的是,度量也可以从核特征中导出。这种核特征的计算由xing等人的“robustnucleus/celldetectionandsegmentationindigitalpathologyandmicroscopyimages:acomprehensivereview[数字病理学和显微图像中的稳健核/细胞检测和分割:全面综述]”,ieeerevbiomedeng9,234-263,2016年1月进行了描述。
当然,可以考虑本领域普通技术人员已知的其他特征并将其用作特征计算的基础。
作为另一个例子,可以将细胞分类为淋巴细胞,诸如在pct公开号wo/2016/075096中描述的。特别地,pct公开号wo/2016/075096描述了一种计算机实施的方法,所述方法在ihc测定中将染色的组织样品的图像内的细胞针对pd-l1生物标记物的存在进行分类,所述方法包括:从组织样品图像内的核特征计算核特征度量;基于感兴趣核与组织样品的图像来计算上下文信息度量;以及使用核特征度量与上下文信息度量的组合(作为分类器的输入)对组织样品图像内的细胞进行分类,其中,细胞被分类为阳性免疫细胞、阳性肿瘤细胞、阴性免疫细胞、以及阴性肿瘤细胞或其他细胞中的至少一种。在一些实施方案中,所述方法进一步包括创建前景分割掩码以识别细胞内的单个核的步骤。所述公开文件进一步描述了在pd-l1染色的组织的背景下:具有不表达pd-l1生物标记物的淋巴细胞(“阴性淋巴细胞”)的区域由小的蓝色斑点表征;具有确实表达pd-l1生物标记物的淋巴细胞(“阳性淋巴细胞”)的区域由小的蓝色斑点和棕色斑点表征;具有主要表达pd-l1生物标记物的细胞(“阳性肿瘤细胞”)的肿瘤区域由大的蓝色斑点和棕色环表征;以及其中的细胞不表达pd-l1生物标记物(“阴性肿瘤细胞”)的肿瘤区域仅由大的蓝色斑点表征。
在一些实施方案中,图像分析模块207运行不止一次。例如,图像分析模块207第一次运行以提取特征并对第一图像中的细胞和/或核进行分类;并且然后第二次运行以提取特征并对一系列附加图像中的细胞和/或核进行分类,其中,附加图像可以是其他单形图像或解混图像通道图像、或其任意组合。
在导出特征之后,可以将特征单独使用或与训练数据结合使用(例如,在训练期间,示例性细胞与由专家观察者根据本领域普通技术人员已知的过程提供的基础事实标识一起呈现)以对核或细胞进行分类。在一些实施方案中,所述系统可以包括分类器,所述分类器至少部分地基于针对每个生物标记物的一组训练或参考切片而被训练。本领域技术人员将认识到,可以使用不同组的切片来训练每个生物标记物的分类器。因此,对于单个生物标记物,在训练后获得单个分类器。本领域技术人员还将认识到,由于从不同生物标记物获得的图像数据之间存在差异,因此可以针对每个不同生物标记物训练不同的分类器,以确保在看不见的测试数据上具有更好的性能,其中,测试数据的生物标记物类型将是已知的。可以至少部分地基于如何最好地处理训练数据的(例如,在组织类型、染色规程和其他感兴趣特征方面的)差异来选择经训练的分类器,以用于切片解释。
在一些实施方案中,分类模块是支持向量机(“svm”)。一般而言,svm是一种分类技术,其基于非线性输入数据集经由用于非线性情况的内核被转换为高维线性特征空间的统计学习理论。不希望受任何特定理论的束缚,认为支持向量机借助于内核函数k将表示两个不同的类的一组训练数据e投射到高维空间中。在此变换后的数据空间中,对非线性数据进行变换,使得可以生成平线(可区分的超平面)来分离类,从而最大化类分离。然后经由k将测试数据投射到高维空间中,并且在所述测试数据相对于超平面下降位置的基础上对测试数据进行分类。内核函数k定义了将数据投影到高维空间中的方法。
在其他实施方案中,使用adaboost算法执行分类。adaboost是一种结合多个弱分类器来生成强分类器的自适应算法。由病理学家在训练阶段期间识别的图像像素(例如具有特定染色或属于特定组织类型的图像像素)用于为每个单独的纹理特征φj生成概率密度函数,对于j∈{1,...,k},其被认为是弱分类器。贝叶斯定理然后用于为构成弱学习者的每个φj生成可能场景lj=(cj,|j∈{1,....,k})。这些由adaboost算法组合成强分类器πj=∑ti=1αjilji,其中,对于每个像素cj∈cj,πj(cj)是表明像素cj属于类ωt的组合可能性,其中,αji是训练期间为特征φi确定的权重,并且t是迭代次数。
在一些实施方案中,使用分类器来区分肿瘤核与也可以被进一步分类(例如,通过识别淋巴细胞核和基质核)的其他候选核。在一些实施方案中,如本文进一步所述,应用学得的监督分类器来识别肿瘤核。例如,在核特征方面对学得的监督分类器进行训练以识别肿瘤核,并且然后应用所述分类器来将未标记图像中的候选核分类为肿瘤核或非肿瘤核。可选地,可以进一步训练学得的监督分类器以区分不同类的非肿瘤核,诸如淋巴细胞核和基质核。在一些实施方案中,用于识别肿瘤核的学得的监督分类器是随机森林分类器。例如,可以通过以下方式训练随机森林分类器:(i)创建肿瘤核和非肿瘤核的训练集,(ii)提取每个核的特征,以及(iii)基于提取的特征训练随机森林分类器,以区分肿瘤核与非肿瘤核。然后可以将经训练的随机森林分类器应用于将未标记图像中的核分类为肿瘤核和非肿瘤核。可选地,可以进一步训练随机森林分类器以区分不同类的非肿瘤核,诸如淋巴细胞核和基质核。
自动细胞分类的校正
在如上所述使用自动图像分析进行细胞检测和分类之后,病理学家或其他医学专业人员可以调整或以其他方式校正自动分类(步骤314)。以这种方式,细胞分类是半自动或半监督式的。例如,可以将通过自动图像分析算法提供的细胞分类显示为查看器中样品图像上的覆盖图,并且然后医学专业人员可以检查可疑细胞分类,以确认细胞已被正确分类。另一方面,如果对细胞进行了错误分类,则医学专业人员可能有机会调整细胞分类,即手动覆盖自动细胞分类。例如,医学专业人员可以手动选择错误分类的细胞,并用正确的分类对它们进行重新标记。本领域技术人员将认识到,通过检查自动分类结果,可以将更好的基础事实训练数据提供给多层神经网络。步骤314的输出是来自自动图像分析的正确识别的分类与一组调整后的细胞分类的组合,从而提供“最终的”细胞分类。
区域级和细胞级分类数据的组合
在医学专业人员已经对细胞分类结果进行了检查之后,在像素级对组织区域标识和细胞分类进行组合(步骤314)。
所述过程的这一步骤考虑了生物系统的先验知识。本领域技术人员将认识到,在某些类型的组织中可能找不到某些类型的细胞。例如,在富含淋巴细胞的区域中将不可能找到基质细胞。通过丢弃那些生物学上不可行的组合,基础事实数据变得丰富了背景知识,从而有助于改进对多层神经网络的训练。
例如,图6展示了可以被分类的不同区域和不同细胞。虽然20种不同的组合是可能的,但是只有较小的组合子集是在生物学上可行的。在该例子中,生物学上可行的组织区域中的细胞类型组合包括:(i)肿瘤区域中的肿瘤细胞;(ii)肿瘤区域中的基质细胞;(iii)肿瘤区域中的淋巴细胞;(iv)基质区域中的肿瘤细胞;(v)基质区域中的基质细胞;(vi)基质区域中的淋巴细胞;(vii)富含淋巴细胞的区域中的肿瘤细胞;以及(viii)富含淋巴细胞的区域中的淋巴细胞。当然,生物学上可行的组合的程度可能基于样品类型或样品来源而不同,并且本领域技术人员将能够提供指示丢弃某些组合的指令。
在一些实施方案中,所述系统可以识别区域,但是没有细胞类型可以与所识别出的区域内的像素相关联。在这种情况下,可以用区域标记和标记“背景细胞”(例如肿瘤区域,背景细胞)来标记像素,其中,标记“背景细胞”用作“占位符”,其表示所述区域不包含任何相关的细胞类型。这样,标记“背景细胞”指示没有细胞类型。在一些实施方案中,如果检测到的不是感兴趣细胞或可以忽略的伪影,则检测到的细胞也可以被认为是“背景细胞”。
然后为样品图像内的每个像素指定标记(步骤315),所述标记包括所述像素所属的细胞类型和区域类型。例如,像素标记可以是(肿瘤细胞,基质区域)。以这种方式,每个像素都用生物学上可行的标记来标记。
多层神经网络
然后使用样品图像和为样品图像内的每个像素指定的标记来训练多层神经网络(图3a的步骤302;图3b的步骤316)。为此目的,可以实施任何多层神经网络。合适的多层神经网络包括yannlecun提出的lenet;alexkrizhevsky等人提出的alexnet;matthewzeiler等人提出的zfnet;szegedy等人提出的googlenet;karensimonyan等人提出的vggnet;以及kaiminghe等人提出的resnet。在一些实施方案中,多层神经网络为vgg16(simonyan,2014)。在其他实施方案中,多层神经网络为densenet(参见huang等人的“denselyconnectedconvolutionalnetworks[密集连接的卷积网络]”,arxiv:1608.06993)。在一些实施方案中,利用了全卷积神经网络,诸如由long等人的“fullyconvolutionalnetworksforsemanticsegmentation[用于语义分割的全卷积网络]”,computervisionandpatternrecognition(cvpr),2015ieeeconference,20015年6月(inspec登记号:15524435)所描述的。
通常在自然图像的大型且公开可用的图像数据档案库(诸如russakovsky等人于2015年在imagenetlargescalevisualrecognitionchallenge[imagenet大规模视觉识别挑战],ijcv,2015中提出的imagenet)上对神经网络进行训练。鉴于通常用于生物样本(诸如肿瘤样品)的训练图像较少,因此采用迁移学习方法。在一些实施方案中,可以使用“迁移学习”方法,其中,将预训练网络(诸如以上提及的那些)连同其学得的权重一起用于在新的训练图像集上初始化训练过程,并对所述预训练网络和所述权重进行训练以使所述权重适应于目前的特定应用(参见pan、s.j.与yang、q.,2010年。asurveyontransferlearning[迁移学习调查]。ieeetransactionsonknowledgeanddataengineering,22(10),1345-1359.doi:10.1109/tkde.2009.191)。在一些实施方案中,预训练网络在可能或可能不与感兴趣的应用直接相关、并且可能或可能不是生物学性质的现有公共或私有数据集上进行训练。在一些实施方案中,最终层的权重被随机地初始化以避免落入次优局部最小值,而在其他实施方案中,最终层的权重被不变地导入。
在训练了多层神经网络(步骤302)之后,可以向经训练的分类器提供基础事实不可用的生物样品的一个或多个未标记图像(如本文所指出的),以对未标记图像内的区域和细胞进行分类(步骤303和304)。
使用经训练的多层神经网络对区域和细胞进行分类
本公开还提供了使用经训练的多层网络对区域和细胞进行分类的系统和方法(参见图3a)。在一些实施方案中,将未标记图像数据提供给经训练的多层神经网络(步骤303),其中,经训练的多层神经网络对未标记图像中的检测到的细胞进行分类(步骤304)。一旦在未标记图像内对细胞分类,就可以将分类结果用于进一步分析(例如,创建覆盖掩码、评分等)。本文将进一步详细描述这些步骤中的每一个步骤。
参考图3c,在一些实施方案中,系统首先接收要分类的未标记图像(步骤320)。可以使用本文所述的图像获取模块202来获取要分类的未标记图像,或者可以从存储器201(或从存储模块240)中检索所述未标记图像。可以使用本文中标识的任何模块(例如组织区域掩码模块或视野确定模块)对未标记图像进行预处理。
接下来,使用细胞检测模块204在未标记图像内检测细胞核(步骤321)。可以根据本文所述的用于检测细胞以进行基础事实数据生成的任何方法来检测细胞核(例如,使用基于径向对称的细胞检测方法)。在其他实施方案中,可以使用在美国专利申请公开号2017/0098310中描述的方法来确定细胞核。特别地,美国公开号2017/0098310描述了一种用于自动辨别生物组织样品的数字图像中的细胞核边界的方法,所述方法包括:由处理器识别数字图像中的一个或多个初始边缘,并将识别出的初始边缘内的像素用作初始投票像素;由处理器使用张量投票域对每个所述初始投票像素附近的像素进行张量投票操作;由处理器累加每个相邻像素的多个投票,以确定所述相邻像素的张量值,投票量指示所述相邻像素是否可能是与初始投票像素相同核的边缘像素,张量值是由所述相邻像素位于其附近的所有初始投票像素所投下的所有投票的总和;以及由处理器分解每个确定的张量值,以确定分解后的张量值的特征值和特征向量;由处理器评估所确定的特征值和特征向量,以识别出经细化边缘像素,所述经细化边缘像素表示细胞核边界;以及由处理器生成显著边缘强度图像,所述显著边缘强度图像选择性地包括表示细胞核边界的识别出的经细化边缘像素。
在其他实施方案中,根据由veta等人的“automaticnucleisegmentationinh&estainedbreastcancerhistopathologyimages[h&e染色的乳腺癌组织病理学图像中的自动核分割]”,plosone.2013;8(7):e70221所描述的方法进行图像分割。根据该方法,图像分割可以分为四个主要步骤:1)预处理;2)标记物控制的分水岭分割;3)后处理;以及4)来自多个尺度的结果的合并。预处理的目的是在保留核的边界的同时移除不相关的内容。预处理以用于从rgb图像中分离出苏木精染色的颜色解混开始。然后使用一系列形态学操作处理苏木精图像的灰度版本以移除不相关的结构。所述过程的核心部分是标记物控制的分水岭分割。使用以下两种类型的核标记物:使用突显预处理图像的高径向对称区域最小值的结构的图像变换提取的标记物。在后处理步骤中,移除不太可能表示核的区域,并且将剩余区域的轮廓参数化为椭圆。通过在预处理步骤中改变结构元素的大小,可以调整分割过程以寻找不同尺度的核,从而允许多尺度分析。然后通过解析并发区域来合并来自多个尺度和两种标记物类型的分割结果以给出最终分割。
在其他实施方案中,计算前景分割掩码(步骤322),并且使用所生成的前景分割掩码来对未标记图像进行过滤以识别出各个细胞(步骤323)。在一些实施方案中,使用在美国专利申请公开号2017/0337596中描述的方法来生成前景分割掩码。特别地,us2017/0337596描述了通过以下方式来计算前景分割:(1)应用过滤器以增强图像,使得(a)丢弃不太可能具有核的图像区域,并且(b)识别出局部区域内的核;以及(2)进一步应用可选的过滤器,以选择性地移除伪影、移除小的斑点、移除不连续性、填充洞并分裂更大的斑点。在一些实施方案中,所应用的过滤器选自由全局阈值化过滤器、局部自适应阈值化过滤器、形态运算过滤器和分水岭变换过滤器。在一些实施方案中,首先应用全局阈值化过滤器,然后应用局部自适应阈值化过滤器。在一些实施方案中,在应用局部自适应阈值化过滤器之后,应用用于选择性地移除伪影、移除小的斑点、移除不连续性、填充洞并分裂更大的斑点的可选过滤器。在一些实施方案中,对单个核的识别进一步包括对过滤后的输入图像执行连接分量的标记过程。
接下来,将未标记图像提供给经训练的多层神经网络220(步骤324)。本领域技术人员将认识到,多层神经网络220必须已被训练为对未标记图像中存在(或怀疑存在)的细胞类型进行分类。例如,如果将网络训练为对乳腺癌样品中的淋巴细胞和肿瘤细胞进行识别和分类,则未标记图像也必须是乳腺癌样品的图像。在一些实施方案中,多层神经网络可以用源自多种类型的癌症的样品图像(单形的或多重的)进行训练,并对来自该特定类型癌症的样品图像进行分类。在将未标记图像提供给经训练的多层神经网络之后,所述网络会在像素级提供预测分类,即,为未标记图像内的每个像素指定特定的可预测细胞类型标记,例如肿瘤细胞、基质细胞等。
然后使用像素级预测分类,将为每个识别出的细胞指定标记(步骤325)。在一些实施方案中,为每个识别出的单个细胞指定细胞标记包括:(i)量化在所识别出的单个细胞内带有各个预测分类标记的像素的数量;以及(ii)将具有最大数量的预测标记指定为细胞标记。在一些实施方案中,可以将生成的前景掩码覆盖在未标记图像的顶部,并且可以对任何区域(即,所识别出的细胞)内的每个不同的预测像素标记进行量化,然后将具有最大数量的预测标记选择为细胞标记。例如,如果特定的识别出的细胞总共包括100个像素,并且所有这些像素中的55个被预测为肿瘤细胞像素,所有这些像素中的40个被预测为基质细胞,且所有这些像素中的5个被预测为淋巴细胞,则将为所述细胞指定肿瘤细胞的标记,因为大多数像素被预测为肿瘤细胞像素。可替代地,如果没有前景分割,则在标记指定期间可以利用已建立的核中心(即,中心围绕的小的径向圆,例如,具有半径为大约5个像素的圆的像素分组)。在这种情况下,将大多数像素标记用作所述细胞的标记。
在指定细胞标记之后,可以进一步评估图像(步骤326)。例如,在一些实施方案中,可以采用评分模块来生成一个或多个表达分数。在共同受让的且共同未决的于2013年12月19日提交的申请wo/2014/102130a1“imageanalysisforbreastcancerprognosis[针对乳腺癌预后的图像分析]”和2104年3月12日提交的wo/2014/140085a1“tissueobject-basedmachinelearningsystemforautomatedscoringofdigitalwholeslides[用于对整个数字切片进行自动评分的基于组织对象的机器学习系统]”中进一步详细地描述了用于评分的方法。例如,基于检测到的阳性和阴性肿瘤细胞/核,可以使用一种或多种方法来计算各种切片级分数,诸如标记物阳性百分比。
在一些实施方案中,可以为感兴趣的特定区域或视野确定评分。感兴趣区域或fov可以由病理学家或医学专业人员手动选择,或者可以基于未标记图像内的形态来自动选择(例如,参见美国专利申请公开号2017/0154420)。
在其他实施方案中,可以将分类结果可以覆盖在未标记图像上。例如,可以用颜色或其他标志来标记未标记图像内的每个细胞(参见图4的面板(c)和图5的面板(c))。图4和5展示了基础事实图像的例子,其中,对区域进行了手动注释。同样,使用传统方法(例如,如本文所述的径向对称)检测了核中心,并且在每个检测到的中心周围进行了前景分割作为相关联的细胞掩码。使用了(本文中描述的)传统分类器来为细胞指定标记。然后由病理学家使用图像注释平台(在错误分类的地方)手动校正了这些指定的标记。
例子
提供了非限制性例子以说明本文概述的各个步骤。
背景
个性化患者护理需要可预测对于her2靶向治疗和基于蒽环类药物的化疗的反应的生物标记物。基于组织学的肿瘤浸润性淋巴细胞(tils)评估在乳腺癌中显示为预后性的并且可能是治疗预测性的。然而,在这些医学需求的背景下,尚未详尽地且系统地确定til、肿瘤细胞、其他微环境介体之间的相互作用、它们的空间关系、数量以及其他基于图像的特征。为了量化和探索患者人群中的这些方面,我们开发了一种基于深度学习的图像分析算法,用于乳腺癌h&e组织整个切片图像的区域级和细胞级分割和分类。
方法:概述
深度学习(dl)是一种机器学习方法,其中训练了多层(深度)卷积神经网络来自动发现显著特征以区分不同的感兴趣结构。为了自动解释h&e图像,我们打算识别出肿瘤细胞、基质细胞和淋巴细胞以及肿瘤区域和基质区域,并拒绝其他区域(例如坏死、伪影)。对单个深度网络进行了训练,以在像素级进行组合的区域和细胞分类。对区域级基础事实进行了手动注释。然而,手动注释每个细胞很繁琐,而且还容易出错。为了减轻这个问题,使用了一种半自动方法,其中病理学家手动校正由图像分析算法输出的错误分类。
方法:深度学习训练和验证
为了训练网络,将20张放大20倍的整个切片图像用于区域注释和细胞注释(约20,000个区域,500,000个细胞,2×108个像素@0.5um)。
为了进行验证,在八个整个切片中,每个整个切片使用了约1mm×1mm的四个子区域(约10,000个区域,200,000个细胞,108个像素)—参见图__和图__(细胞的颜色编码:肿瘤(绿色)、基质(黄色)、淋巴细胞(红色);区域的颜色编码:肿瘤(青色)、基质(黄色)、淋巴(红色)、伪影(深蓝色))。
结果
在两个实验中,将开发的算法与病理学家的基础事实进行了比较。
在第一实验中,有6个h&e乳腺癌病例,其中具有放大20倍的14个带注释的较小区域。两位病理学家手动将约9000个细胞注释为基础事实。为了进行算法比较,仅保留来自这两位病理学家的一致的那些基础事实注释,而忽略其他的。
在第二实验中,使用了半自动方法(本公开内容)对10,000个区域和200,000个细胞进行基础事实测量。单个病理学家在视觉上检查和校正了通过基于特征的方法输出的细胞标记,而不会丢弃任何模糊的细胞标记。
对于区域级的基础事实验证,重叠的异质区域(诸如基质区和淋巴细胞区)对病理学家进行区域划分提出了挑战;因此,我们评估了组合这些区域的计算准确性。
鉴于以上结果,诸位申请人已经证明了涉及深度卷积神经网络的新颖且稳健的方法对于h&e乳腺癌图像中的组合的区域和细胞分类是可行的。
在图7和图8中描绘了结果。参考图7,在由病理学家手动注释的区域中,使用本文所述的半自动方法来检测、分割细胞并生成细胞分类标记。所示出的细胞标记是细胞级的,即,对于与反映单个轮廓的单个连接的斑点相对应的所有像素,示出了指定的共同细胞标记。区域级标记是像素级的,即,属于一种区域类别的所有像素都以一种特定的颜色示出。对标记进行颜色编码。肿瘤细胞、淋巴细胞和基质细胞分别以绿色、红色和黄色示出。下部图像是使用经训练的多层神经网络对样品图像进行分类的结果。区域标记图像示出在右下方,并且细胞级标记图像示出在左下方。在像素级示出了区域标记图像,并且细胞标记图像在细胞级示出了细胞分类标记。如本文所述,多层神经网络输出经分类的图像,从而为每一个像素指定组合的(区域,细胞)标记。在后续步骤中,针对每个像素将组合的标记分为两个不同的标记—细胞标记和区域标记。在下一步骤中,针对每个细胞将核/细胞中心位置以及相关联的前景斑点分割掩码作为附加输入,针对从像素级的细胞标记中聚合的每个细胞(在细胞级)导出分类标记。
用于实践本公开的实施方案的其他部件
下面描述了可以与本公开的系统和方法结合使用的其他部件(例如,系统或模块)。
解混模块
在一些实施方案中,作为输入接收的图像可以是多重图像,即,接收到的图像是用一种以上染色剂染色的生物样品的图像。在这些实施方案中,并且在进一步处理之前,首先将多重图像解混到其组成通道中,其中每个解混通道对应于特定的染色剂或信号。在一些实施方案中,解混图像(通常被称为“通道图像”或“图像通道图像”)可以被用作本文描述的每个模块的输入。例如,可以利用第一h&e图像、针对多个分化标记物(cd3、cd8等)簇染色的第二多重图像、以及各自针对特定生物标记物(例如er、pr、ki67等)染色的多个单形图像来确定标记物间异质性。在这个例子中,首先将多重图像解混为其组成通道图像,并且可以将这些通道图像与h&e图像和所述多个单形图像一起使用,以确定标记物间异质性。
在一些实施方案中,在包含一种或多种染色剂和苏木精的样品中,可以为一种或多种染色剂和苏木精的每个通道产生单独的图像。不希望受任何特定理论的束缚的情况下,认为这些通道突显组织图像中的不同组织结构,因此,这些组织结构可被称为结构图像通道。在一些实施方案中,解混至少提供了苏木精图像通道图像。在一些实施方案中,获取的图像被解混到表示图像中局部苏木精量和高亮细胞核区域的单独通道中。本领域技术人员将认识到,从这些通道提取的特征可用于描述组织的任何图像中存在的不同生物结构。
成像系统202提供的多光谱图像是与各个生物标记物和噪声成分相关联的基础光谱信号的加权混合物。在任何特定像素,混合权重与组织中特定位置的基础共存生物标记物的生物标记物表达和该位置的背景噪声成比例。因此,混合权重因像素而异。本文公开的光谱解混方法将每个像素处的多通道像素值向量分解成组成生物标记物端元或组分的集合,并估计每个生物标记物的各个组成染色剂的比例。
解混是将混合像素的测量光谱分解成一组组成光谱或端元以及一组相应的分数或丰度的过程,这些分数或丰度表示像素中存在的每个端元的比例。具体而言,解混过程可以提取染色剂特异性通道,以使用标准类型的组织和染色剂组合所熟知的参考光谱来确定单个染色剂的局部浓度。解混可以使用从对照图像检索的或者从观察图像估计的参考光谱。解混每个输入像素的分量信号使得能够检索和分析染色特异性通道,例如h&e图像中的苏木精通道和曙红通道,或者ihc图像中的二氨基联苯胺(dab)通道和复染色(例如苏木精)通道。术语“解混”和“颜色去卷积”(或“去卷积”)等(例如,“去卷积”、“解混”)在本领域中可以互换使用。
在一些实施方案中,使用线性解混,用解混模块205解混多重图像。例如,在'zimmermann的“spectralimagingandlinearunmixinginlightmicroscopy”advbiochemengin/biotechnol(2005)95:第245-265页'和在c.l.lawson和r.j.hanson的"solvingleastsquaresproblems",prenticehall,1974,第23章,第161页"中描述了线性解混。在线性染色剂解混中,在任何像素处测量的光谱(s(λ))被视为染色剂光谱组分的线性混合并且等于在像素处表达的每个单独染色剂的颜色参考(r(λ))的比例或权重(a)之和
s(λ)=a1·r1(λ)+a2·r2(λ)+a3·r3(λ).......ai·ri(λ)
这可以更一般地表达为以下矩阵形式
s(λ)=σai·ri(λ)或s=r·a
如果获取了m个信道图像并且存在n种单独的染色剂,则m×n矩阵r的列是如本文导出的最佳颜色系统、n×1向量a是单独染色剂的未知比例并且m×1向量s是像素处测量的多通道光谱向量。在这些方程中,每个像素中的信号(s)在获取多重图像和参考光谱期间进行测量,即最佳颜色系统如本文所描述的那样导出。各种染色剂的贡献(ai)可以通过计算它们对测量的光谱中的每个点的贡献来确定。在一些实施方案中,使用最小二乘逆拟合法获得解,所述最小二乘逆拟合法通过对以下方程组求解使测量的光谱与计算的光谱之间的平方差最小化:
在这个方程中,j表示检测通道的数量并且i等于染色剂的数量。线性方程解通常包括允许受约束的解混强制权重(a)求和为1。
在其他实施方案中,使用在2014年5月28日提交的题为“imageadaptivephysiologicallyplausiblecolorseparation[像自适应生理上似然颜色分离]”的wo2014/195193中描述的方法来完成解混。一般而言,wo2014/195193描述了一种通过使用迭代优化的参考向量分离输入图像的分量信号来进行解混的方法。在一些实施方案中,来自测定的图像数据与特定于测定特性的预期或理想结果相关,以确定质量度量。在低质量图像或与理想结果相关性差的情况下,调整矩阵r中的一个或多个参考列向量,并且使用调整后的参考向量迭代地重复解混,直到相关性显示出匹配生理和解剖要求的良好质量图像。解剖、生理和测定信息可用于定义应用于测量图像数据的规则,以确定质量度量。这些信息包括组织是如何染色的,组织内的哪些结构是打算染色的或不打算染色的,以及结构、染色剂和特定于正在处理的测定的标记物之间的关系。迭代过程产生特定于染色的向量,所述向量可以生成精确标识感兴趣结构和生物学相关信息的图像,没有任何噪声或不想要的光谱,因此适于分析。参考向量被调整到搜索空间内。搜索空间定义了参考向量可以用来表示染色剂的值的范围。搜索空间可以通过扫描包括已知或常见问题在内的各种代表性训练测定,并确定训练测定的高质量参考向量集来确定。
在其他实施方案中,使用在215年2月23日提交的题为“groupsparsitymodelforimageunmixing[用于图像解混的群稀疏模型]”的wo2015/124772中描述的方法来完成解混。总的来说,wo2015/124772描述了使用组稀疏性框架来解混,其中在“相同的组”内对来自多个共存标记物的染色贡献的分数进行建模,并且在不同的组中对来自多个非共存标记物的染色贡献的分数进行建模,向建模的组稀疏性框架提供多个共存标记物的共同定位信息,使用组套索求解建模的框架以在每个组内产生最小二乘解,其中最小二乘解对应于共存标记物的解混,并且在对应于非共存标记物的解混的组中产生稀疏解。此外,wo2015124772描述了一种通过以下方式进行解混的方法:输入从生物组织样品获得的图像数据;从电子存储器读取参考数据,所述参考数据描述了多种染色剂中的每一种的染色剂颜色;从电子存储器读取共存数据,所述共存数据描述了多组染色剂,每组包括可以在生物组织样品中共存的染色剂,并且每组形成用于组套索标准的组,所述组中的至少一个组具有两个或以上的大小;以及使用参考数据作为参考矩阵来计算用于获得解混图像的组套索标准的解。在一些实施方案中,用于解混图像的方法可以包括生成组稀疏模型,其中来自共定位标记物的一部分染色贡献被指定在单个组内,来自非共定位标记物的一部分染色贡献被指定在单独的组内,并且使用解混算法求解组稀疏模型以在每个组内产生最小二乘解。
其他系统部件
本公开的系统200可以绑定到可以对组织样本执行一个或多个制备过程的样本处理设备。制备过程可以包括但不限于对样本进行脱蜡、对样本进行调节(例如,细胞调节)、对样本进行染色、执行抗原修复、执行免疫组织化学染色(包括标记)或其他反应和/或执行原位杂交(例如,sish、fish等)染色(包括标记)或其他反应、以及用于制备用于显微术、微量分析、质谱法或其他分析方法的样本的其他过程。
处理设备可以将固定剂施加到样本。固定剂可以包括交联剂(诸如醛类(例如甲醛、多聚甲醛和戊二醛)以及非醛类交联剂)、氧化剂(例如,金属离子和复合物,如四氧化锇和铬酸)、蛋白质变性剂(例如,乙酸、甲醇和乙醇)、未知机制的固定剂(例如,氯化汞、丙酮和苦味酸)、组合试剂(例如,卡诺氏固定剂(carnoy'sfixative)、methacarn、波恩氏流体(bouin'sfluid)、b5固定剂、罗斯曼氏流体(rossman'sfluid)、詹德莱氏流体(gendre'sfluid))、微波和混杂固定剂(例如,排出体积固定和蒸气固定)。
如果样本是嵌入石蜡中的样品,则可以使用一种或多种适当的脱蜡流体对样品进行脱蜡。除去石蜡后,可以相继向样本施加任何数量的物质。物质可以用于预处理(例如,用于反转蛋白质交联、暴露核酸等)、变性、杂交、洗涤(例如,严格洗涤)、检测(例如,将视觉或标记物分子与探针链接)、扩增(例如,扩增蛋白质、基因等)、复染、盖玻等。
样本处理设备可以将广泛范围的物质施加到样本。物质包括但不限于染色剂、探针、试剂、冲洗剂和/或调节剂。物质可以是流体(例如,气体、液体或气体/液体混合物)等。流体可以是溶剂(例如,极性溶剂、非极性溶剂等)、溶液(例如,水溶液或其他类型的溶液)等。试剂可以包括但不限于染色剂、润湿剂、抗体(例如,单克隆抗体、多克隆抗体等)、抗原回收流体(例如,基于水性或非水性的抗原修复溶液、抗原回收缓冲液等)等。探针可以是与可检测的标记或报告分子附接的分离的核酸或分离的合成寡核苷酸。标记可以包括放射性同位素、酶底物、辅因子、配体、化学发光或荧光剂、半抗原和酶。
在对样本进行处理之后,用户可以将承载样本的载玻片运送到成像设备。在一些实施方案中,成像设备是明场成像器载玻片扫描仪。一种明场成像器是ventanamedicalsystems公司出售的iscancoreo明场扫描仪。在自动化实施方案中,成像设备是数字病理学装置,如题为imagingsystemandtechniques[成像系统和技术]的国际专利申请号pct/us2010/002772(专利公开号为wo/2011/049608)所公开的或于2011年9月9日提交的题为imagingsystems,cassettes,andmethodsofusingthesame[成像系统、暗盒和其使用方法]的美国专利申请号61/533,114所公开的。
成像系统或设备可以是多光谱成像(msi)系统或荧光显微镜系统。这里使用的成像系统是msi。msi通常通过提供对像素级图像的光谱分布的访问用基于计算机化显微镜的成像系统来配备病理样本的分析。虽然存在各种多光谱成像系统,但是所有这些系统共有的操作方面是形成多光谱图像的能力。多光谱图像是捕获特定波长或跨电磁波谱的特定光谱带宽的图像数据的图像。可以通过光学滤波器或通过使用能够选择预定光谱分量的其他仪器来挑选这些波长,所述预定光谱分量包括在可见光范围之外的波长处的电磁辐射,如例如红外(ir)。
msi系统可以包括光学成像系统,所述光学成像系统的一部分包含光谱选择性系统,所述光谱选择性系统可调谐以定义预定数量的即n个离散光学带。光学系统可以适于对组织样品进行成像、用宽带光源在透射中照射到光学检测器上。在一个实施方案中可以包括放大系统(如例如显微镜)的光学成像系统具有通常在空间上与光学系统的单个光学输出对准的单个光轴。当调整或调谐光谱选择系统(例如用计算机处理器)时,系统形成组织的一系列图像,如以确保在不同的离散光谱带中获取图像。设备可以另外包含显示器,所述显示器中出现来自所获取的图像的序列中的至少一个视觉上可感知的组织图像。光谱选择系统可以包括光学色散元件(如衍射光栅)、光学滤波器(如薄膜干涉滤光器)的集合、或适于响应于用户输入或预编程处理器的命令从光源通过样品朝向检测器透射的光谱中选择特定通带的任何其他系统。
在替代性实施方式中,光谱选择系统定义了对应于n个离散光谱带的若干个光学输出。这种类型的系统从光学系统摄入透射光输出,并且沿着n个空间上不同的光路在空间上重定向这个光输出的至少一部分,其方式为将识别的光谱带中的样品沿着对应于这个识别的光谱带的光路成像到检测器系统上。
本说明书中描述的主题和操作的实施方案可以在数字电子电路系统中或在计算机软件、固件或硬件(包括本说明书中公开的结构及其结构等同物)或其中的一个或多个的组合中实施。可以将本说明书中描述的主题的实施方案实施为一个或多个计算机程序,即在计算机存储介质上编码以用于由数据处理设备来执行或者用于控制数据处理设备的操作的计算机程序指令的一个或多个模块。本文所描述的任何模块可以包括由一个或多个处理器执行的逻辑。如本文所使用的,“逻辑”是指具有可以应用于影响处理器操作的指令信号和/或数据形式的任何信息。软件是逻辑的例子。
计算机存储介质可以是机器可读存储装置、机器可读储存基板、随机或串行存取存储器阵列或装置、或其中的一个或多个的组合。此外,虽然计算机存储介质不是传播信号,但是计算机存储介质可以是以人工生成的传播信号编码的计算机程序指令的来源或目的地。计算机存储介质还可以是或者可以包括在一个或多个单独的物理部件或介质(例如,多个cd、磁盘或其他存储装置)中。可以将本说明书中描述的操作实施为由数据处理设备对存储在一个或多个计算机可读存储装置上或从其他来源接收的数据执行的操作。
术语“编程处理器”包括用于处理数据的所有种类的设备、装置和机器,包括例如可编程微处理器、计算机、芯片上系统或多个芯片上系统、或前述项的组合。设备可以包括专用逻辑电路系统,例如fpga(现场可编程门阵列)或asic(专用集成电路)。除了硬件之外,设备还可以包括为所讨论的计算机程序创造执行环境的代码,例如,构成处理器固件、协议栈、数据库管理系统、操作系统、跨平台运行时环境、虚拟机、或其中的一个或多个的组合的代码。设备和执行环境可以实现各种不同的计算模型基础结构,诸如web服务、分布式计算和网格计算基础结构。
计算机程序(也称为程序、软件、软件应用、脚本或代码)可以以任何形式的编程语言书写,包括编译或解释语言、说明性或者过程性语言,并且计算机程序可以以任何形式部署,包括作为独立程序或者作为模块、部件、子例程、对象或适用于计算环境的其他单元。计算机程序可以但不需要对应于文件系统中的文件。程序可以被存储在保存其他程序或数据的文件的一部分(例如,存储在标记语言文档中的一个或多个脚本)中,被存储在专用于所讨论程序的单个文件中或多个协调的文件(例如,存储一个或多个模块、子程序或部分代码的文件)中。计算机程序可以被部署成在一个计算机上或者在位于一个站点或跨多个站点分布并且通过通信网络互连的多个计算机上执行。
本说明书中描述的过程和逻辑流程可以由一个或多个可编程处理器实行,所述一个或多个可编程处理器执行一个或多个计算机程序以便通过对输入数据进行操作并且生成输出来执行动作。过程和逻辑流程还可以由设备执行,并且设备还可以被实施为专用逻辑电路系统,例如fpga(现场可编程门阵列)或asic(专用集成电路)。
举例来讲,适于执行计算机程序的处理器包括通用和专用两种微处理器以及任何种类的数字计算机的任何一个或多个处理器。通常,处理器将从只读存储器或随机存取存储器或两者接收指令和数据。计算机的必不可少的元件是用于根据指令执行动作的处理器和用于存储指令和数据的一个或多个存储器装置。通常,计算机还将包括用于存储数据的一个或多个大容量存储装置(例如,磁盘、磁光盘或光盘),或者被操作性地耦合以从大容量存储装置接收数据或向大容量存储装置传递数据或两者。然而,计算机不需要有这种装置。此外,计算机可以嵌入另一个装置中,仅举几例,例如移动电话、个人数字助理(pda)、移动音频或视频播放器、游戏控制台、全球定位系统(gps)接收器或便携式存储装置(例如,通用串行总线(usb)闪存驱动器)。适于存储计算机程序指令和数据的装置包括所有形式的非易失性存储器、介质和存储器装置,举例来讲,包括半导体存储器装置(例如,eprom、eeprom和闪存装置)、磁盘(例如,内置硬盘或可移除盘)、磁光盘、以及cd-rom盘和dvd-rom盘。处理器和存储器可以由专用逻辑电路系统补充或并入其中。
为了提供与用户的交互,本说明书中描述的主题的实施方案可以实施在具有用于向用户显示信息的显示装置(例如,lcd(液晶显示器)、led(发光二极管)显示器或oled(有机发光二极管)显示器)以及通过其用户可以向计算机提供输入的键盘和定点装置(例如鼠标或轨迹球)的计算机上。在一些实施方式中,触摸屏可以用于显示信息并接收来自用户的输入。还可以使用其他种类的装置来提供与用户的交互;例如,提供给用户的反馈可以是任何形式的感官反馈,例如,视觉反馈、听觉反馈或触觉反馈;并且可以以任何形式接收来自用户的输入,包括声音、语音或触觉输入。另外,计算机可以通过向用户使用的装置发送文档和从用户使用的装置接收文档(例如,通过响应于从用户的客户端装置上的web浏览器接收的请求将网页发送到web浏览器)来与用户交互。
本说明书中描述的主题的实施方案可以实施在包括以下的计算系统中:后端部件(例如,作为数据服务器)、或中间件部件(例如,应用服务器)、或前端部件(例如,具有图形用户界面或web浏览器的客户端计算机,用户可以通过所述图形用户界面或所述web浏览器与本说明书中描述的主题的实施方式交互)、或者一个或多个这种后端、中间件或前端部件的任何组合。系统的部件可以通过数字数据通信的任何形式或介质(例如,通信网络)进行互连。通信网络的例子包括局域网(“lan”)和广域网(“wan”)、互联网络(例如,互联网)以及对等网络(例如,自组织对等网络)。例如,图1的网络20可以包括一个或多个局域网。
计算系统可以包括任何数量的客户端和服务器。客户端和服务器通常远离彼此并且通常通过通信网络进行交互。客户端与服务器的关系借助于在各自的计算机上运行并且彼此具有客户端-服务器关系的计算机程序产生。在一些实施方案中,服务器将数据(例如,html页面)传输到客户端装置(例如,目的是向与客户端装置交互的用户显示数据和从与客户端装置交互的用户接收用户输入)。可以从服务器处的客户端装置接收在客户端装置处生成的数据(例如,用户交互的结果)。
根据本文公开的实施方案,多层神经网络适用于lenet、alexnet、zfnet、detectnet、googlenet、vggnetresnet、vgg16和densenet。
根据本文公开的实施方案,所述方法进一步包括生成包含每个标记的单个细胞的描述性标志的覆盖图。
尽管已经参考多个说明性实施方案描述了本公开,但是应当理解,本领域技术人员可以设计出落入本公开的原理的精神和范围内的许多其他修改和实施方案。更具体地,在不脱离本公开的精神的情况下,可以在前述公开内容、附图和所附权利要求的范围内对主题组合布置的组成部分和/或布置进行合理的变化和修改。除了组成部分和/或布置的变化和修改之外,替代性用途对于本领域技术人员而言也将是显而易见的。
- 还没有人留言评论。精彩留言会获得点赞!