一种冷冻电镜单颗粒成像数据的分类方法与流程
本发明涉及结构生物学技术领域,更具体地,涉及一种冷冻电镜数据处理中根据构象分类的方法。
背景技术:
将样品通过低温冷冻后使用投射电子显微镜观察的显微技术,叫做冷冻电子显微镜技术,简称冷冻电镜技术。冷冻电镜技术是重要的结构生物学研究方法,它与另外两种技术:x射线晶体学和核磁共振一起构成了高分辨率结构生物学研究的基础。同时,冷冻电镜技术是近年最为热门的结构生物学研究手段。经过多年的发展,冷冻电镜技术近年来取得了突破性进步,已经能够作为研究生物大分子结构与功能的有效手段。冷冻电镜单颗粒成像数据的分类是获取三维结构的重要一步。
因为大分子复合物在功能循环中经常经历构象转变,许多冷冻电镜样品是含有不同构象的混合物。这种类型的结构异质性可以与不完全形成的复合物或未纯化至均匀的样品共存。为了实现高分辨率重建,需要在冷冻电镜单粒子分析过程中处理数据中多种不同结构的存在。许多流行的图像分类方法基于用户定义数量的参考的竞争性细化,这些方法有效地将数据划分为离散数量的类,其中每个类假设在结构上是同质的。在现有分类方法中,无监督分类方法是比较有效的,其不需要了解数据中关于结构生物学的先验知识。通过引入最大似然分类方法,可以实现离散数量的三维状态的无监督分类,这些方法已经在多个图像处理包中实现。
然而,当大分子复合物表现出连续的分子运动时,离散分类方法最终不太适合。这时候就需要引入数据中关于结构生物学的先验知识,来增加分类的准确性。本发明提出的冷冻电镜单颗粒成像数据的分类方法,利用分子动力学模拟的方式,引入了不同成像数据关于结构生物学的先验知识,能够更加准确的区分出数据中分子的运动。
技术实现要素:
为了克服现有冷冻电镜单颗粒成像数据分类方法无法区分大分子复合物表现出的连续分子运动的不足,本发明提供一种基于分子动力学模拟的冷冻电镜单颗粒成像数据的分类方法,可以有效提高分类准确度,更好区分电镜图像中的不同构象变化;并且提前计算了电镜图像的可能方向与位移,降低了分类阶段的计算复杂度,减少了计算资源的开销。
为了解决上述技术问题,本发明采用的技术方案如下:
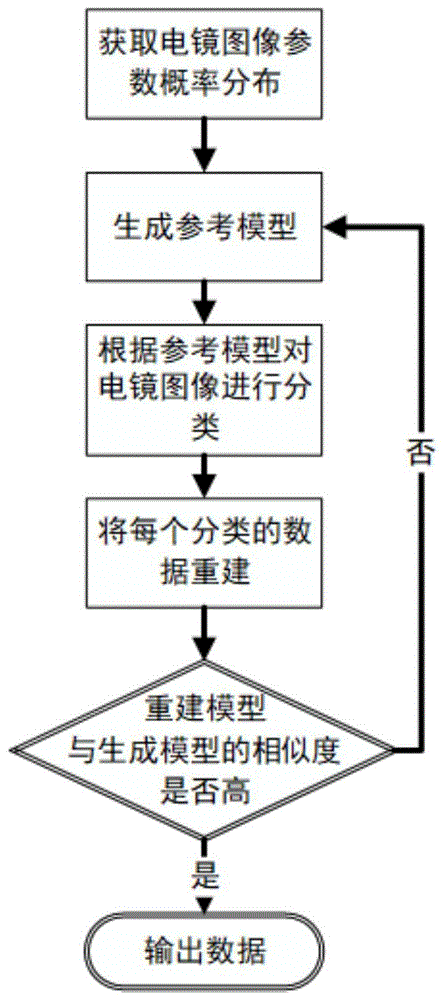
一种冷冻电镜单颗粒成像数据的分类方法,包括以下步骤:
第一步,用最大似然法迭代计算电镜图像的参数概率分布函数,包括以下步骤:
1.1根据已知体系结构生成三维密度图,对三维密度图进行低通滤波,去除以此三维密度图为参考的模型对计算结果的过拟合;
1.2设置采样角度为7.5,在三维空间内对参考模型进行采样,获取参考模型的二维投影图;
1.3将实验数据与采样得到的二维投影图分别计算相关度,获取实验数据在三维空间中不同角度的概率分布函数;
1.4使用上一步得到的概率分布函数,重建得到三维密度图,作为新的参考,同时计算三维密度图的傅里叶壳函数,得到分辨率;
1.5根据得到的分辨率对比判断计算是否收敛:收敛,则停止计算,输出角度、位移等参数信息;否则,使用更小的采样角度,重复第二步;
第二步,根据已知体系结构,模拟构象变化,获取其中相对稳定的若干状态,包括以下步骤:
2.1利用弹性网络以及分子扰动模型,生成结构变化模型;
2.2根据生成模型挑选结构变化合理的若干模型;
2.3将生成的模型转化为三维电子密度图;
第三步,选取生成模型中的两个三维密度图,以0.9度的采样角度,对生成的三维电子密度图进行采样,生成投影图像,并将生成的投影图像与对应角度的电镜图像计算相关度,根据同一图像对不同参考的对应角度投影的相关系数,计算该图像的分类;
第四步,将得到的两个分类数据进行重建;
第五步,比较重建得到的模型与生成模型的差异,如果相似,则分类有效;否则,选取新的两个模型,重复第三步。
本发明提出的冷冻电镜单颗粒成像数据的分类方法,在分类的阶段引入了目标体系的结构生物学知识,生成了可能存在的构象变化,将这些构象变化生成的模型作为参考,得到了电镜图像对于这些模型的分布。
本发明的有益效果表现在:该方法相较于现有的无监督分类方法,可以有效提高分类准确度,更好区分电镜图像中的不同构象变化。并且提前计算了电镜图像的可能方向与位移,降低了分类阶段的计算复杂度,减少了计算资源的开销。
附图说明
图1为根据本发明总体流程图。
具体实施方式
下面结合附图和实施例,对本发明的具体实施方式作进一步详细描述。以下实施例用于说明本发明,但不用来限制本发明的范围。
参照图1,一种冷冻电镜单颗粒成像数据的分类方法,包括以下步骤:
第一步,用最大似然法迭代计算电镜图像的参数概率分布函数,包括以下步骤:
1.1根据已知体系结构生成三维密度图,对三维密度图进行低通滤波,去除以此三维密度图为参考的模型对计算结果的过拟合;
1.2设置采样角度为7.5,在三维空间内对参考模型进行采样,获取参考模型的二维投影图;
1.3将实验数据与采样得到的二维投影图分别计算相关度,获取实验数据在三维空间中不同角度的概率分布函数;
1.4使用上一步得到的概率分布函数,重建得到三维密度图,作为新的参考,同时计算三维密度图的傅里叶壳函数,得到分辨率;
1.5根据得到的分辨率对比判断计算是否收敛:收敛,则停止计算,输出角度、位移等参数信息;否则,使用更小的采样角度,重复第二步;
第二步,根据已知体系结构,模拟构象变化,获取其中相对稳定的若干状态,包括以下步骤:
2.1利用弹性网络以及分子扰动模型,生成结构变化模型;
2.2根据生成模型挑选结构变化合理的若干模型;
2.3将生成的模型转化为三维电子密度图;
第三步,选取生成模型中的两个三维密度图,以0.9度的采样角度,对生成的三维电子密度图进行采样,生成投影图像,并将生成的投影图像与对应角度的电镜图像计算相关度,根据同一图像对不同参考的对应角度投影的相关系数,计算该图像的分类;
第四步,将得到的两个分类数据进行重建;
第五步,比较重建得到的模型与生成模型的差异,如果相似,则分类有效;否则,选取新的两个模型,重复第三步。
本实施例中,基于最大似然法计算电镜图像的参数概率密度分布函数,就是寻找对于一组含有未知噪声的二维图像面内旋转和平移的最大似然表示。对于具有k个不同投影方向的数据集,其二维图像x1,x2,…xn可表示为:
其中
其中αk为第k个投影的可能性,满足αk≥0,且
为了得到最好的估计,需要找到一套参数θ0。在这套参数下,x出现的可能性是最大的,即似然函数p(x|θ)最大。由于xi之间相互独立,可以求其对数函数l(θ)的最大值。由贝叶斯定理有:
在给定的k,φ与θ参数条件下,xi中每一个像素都是一个连续的随机变量,其概率分布由a与高斯噪音σgi决定。对于有j个像素的图片xi而言,其联合分布的形式为:
对于似然函数的最大化,使用最大期望的迭代算法实现,每一轮迭代可以分为两步:e步(expectationstep)与m步(maximizationstep)。
在e步中,根据第n轮估计的参数θ(n),用似然函数的一个下边界q(θ,θ(n))来估计似然函数l(θ),q(θ,θ(n))的表达式为:
由贝叶斯定理有:
p(k,φ|xiθ(n))=p(xi|k,φ,θ(n))p(kφ|θ(n))/q(xi,θ(n))
其中
接下来在m步中最大化q(θ,θ(n)),将得到的参数来作为下一轮估计的参数θ(n+1)。这只需要对q(θ,θ(n))中的每一个变量求偏导并令偏导等于零即可。这样得到了第n+1轮对于ak的估计ak(n+1):
上述方法在算法上是很容易实现的。根据第n轮估计的统计参数,计算每一张单颗粒图片对于每一个类的各个平移旋转操作下的可能性,然后将每一张图平移旋转后的图片按照对应的概率加权起来就是第n+1轮ak的估计。这样每一张图片对各个方向上的投影都有贡献,类似的可以得到其他参数的估计:
弹性网络模式是通过给定cα原子坐标的蛋白质的天然结构,可以通过使用具有单力常数的谐波势来建立蛋白质的弹性网络模型,以解释位于截止距离rc内的所有cα原子之间的成对相互作用,如下面等式:
其中dij表示cα原子运动的模型动态坐标之间的距离,i和j,以及
以上所述是对本发明的优选实施方式进行进一步的说明,并非用于限定本发明的保护范围。凡在本发明的精神和原则之内,所作出的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!