一种结合深度网络特征和机器学习模型的乳腺癌数据分类方法与流程
本发明属于大数据技术领域,尤其涉及一种结合深度网络特征和机器学习模型的乳腺癌数据分类方法。
背景技术:
目前医疗影像技术已经被广泛的应用在肿瘤的临床中,医疗影像技术是以图片数据的方式展示肿瘤,医生在分析肿瘤数据时往往带有很强的经验性和主观性,不能准确的分析出癌症的相关数据。
目前对肿瘤的影像数据的分析均采用小数据集进行分析,在深度学习上会出现过拟合问题。
技术实现要素:
本发明的目的是提供一种结合深度网络特征和机器学习模型的乳腺癌数据分类方法,解决了对肿瘤图片进行数据分类的技术问题。
为实现上述目的,本发明采用如下技术方案:
一种结合深度网络特征和机器学习模型的乳腺癌数据分类方法,包括如下步骤:
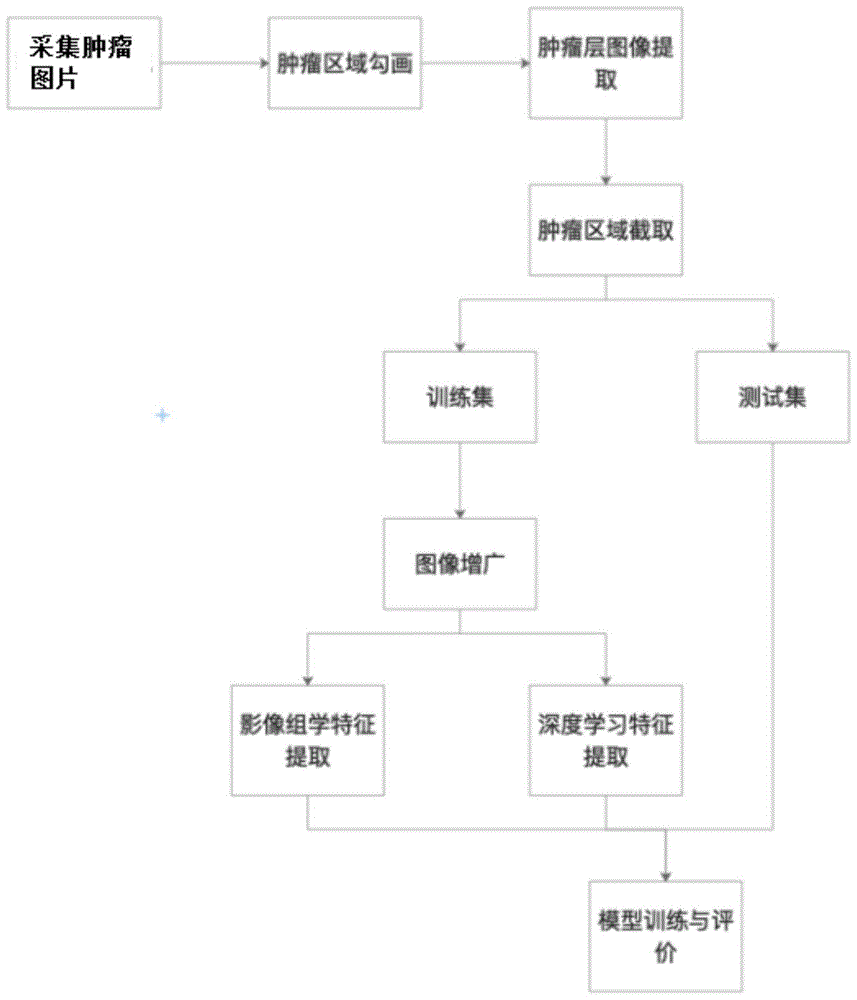
步骤1:建立影像采集服务器、客户端和数据分类服务器,影像图片采集服务器、客户端和数据分类服务器均通过互联网相互通信;
在数据分类服务器中建立图片处理模块、特征提取模块和模型构建模块;
步骤2:影像采集服务器采集肿瘤图片,并将肿瘤图片发送给客户端;
步骤3:医疗人员通过客户端编辑肿瘤图片,在肿瘤图片上勾画出肿瘤区域,生成肿瘤区域图片;
客户端将肿瘤图片和肿瘤区域图片一起发送给数据分类服务器;
步骤4:数据分类服务器接收到肿瘤图片和肿瘤区域图片后,图片处理模块设定一个虚拟矩形,找出肿瘤区域图片的中心点,以该中心点作为虚拟矩形的中心,根据虚拟矩形的边界对肿瘤区域图片进行截取,得到分类图片;
步骤5:按照步骤2到步骤4的方法,对多个肿瘤图片进行处理,得到分类图片的数据集合;
按照训练集70%、测试集30%的规则,利用分层抽样的方法将分类图片的数据集合划分为分类训练集和分类测试集;
对训练组数据进行数据集增广操作,得到扩大版训练集;
步骤6:特征提取模块调取扩大版训练集,从扩大版训练集中的分类图片中提取带有肿瘤的mri图层的图像特征;
步骤7:模型构建模块采用tensorflow和keras提供的网络模型作为基准模型,建立训练模型,其步骤如下:
步骤a1:载入基准模型的初始参数,去掉基准模型最后一层全连接层,加入初始化参数为0的新的全连接层,全连接层使用sigmoid激活函数,输出维度为2,输出值作为预测概率;
步骤a2:将扩大版训练集作为模型训练数据,将其中的分类图片复制为三通道图像,采用5折交叉验证进行模型训练,建立训练模型;
步骤a4:将分类测试集放入训练模型中进行测试,得到模型评价,模型评价的标准为准确率和auc;
步骤8:选择基础模型中的隐藏层最后一层输出的特征图作为深度学习特征,将二维矩阵展平成一维向量,向量维度统一使用64维,大于64维的采用pca算法进行降维,得到39维影像组学特征与64维深度学习特征;
步骤9:将39维影像组学特征与64维深度学习特征组合成103维特征,特征选择使用开源的sklearn包进行,先通过p值检验,筛选出p值<0.05的特征;
步骤10:使用pearson相关性分析对每两个特征之间的相关性进行计算,相关性大于0.85或者小于-0.85的两组特征认为是冗余特征,随机抛弃其中一个特征;
步骤11:使用svm-rfe进行包装法特征筛选,在初始特征集上训练评估器,并且通过feature_importances_属性获得每个特征的重要性,然后从当前的一组特征中丢弃最不重要的特征;
步骤12:在修剪的集合上递归地重复步骤10到步骤11的过程,直到最终到达所需数量的要选择的特征,选择auc作为特征选择的评价标准;
步骤13:采用svm径向基核进行分类模型训练,采用五折交叉验证和网格搜索进行超参数的选择,采用测试数据进行模型评价,评价指标为auc,auc值最高的模型作为最终的分类模型;
步骤14:当影像采集服务器采集到新的肿瘤图片时,首选根据步骤3到步骤4的方法对肿瘤图片进行处理,然后提取带有肿瘤的mri图层的图像特征,最后采用步骤13中得出的最终的分类模型对新的肿瘤图片进行分类;
步骤15:数据分类服务器将步骤14得出的分类结果发送给客户端进行展示。
优选的,在执行步骤6时,所述图像特征包括一阶统计特征19个,2d形状特征10个,纹理特征10个,每层分类图片一共可提取39个影像组学特征。
优选的,在执行步骤a2时,所述三通道图像即为rgb图片。
优选的,在执行步骤a1时,所使用的预训练网络模型包括resnet50、inceptionv4、vgg16、vgg19、densenet、googlenet和alexnet。
本发明所述的一种结合深度网络特征和机器学习模型的乳腺癌数据分类方法,解决了对肿瘤图片进行数据分类的技术问题,本发明结合了迁移学习、深度学习特征提取、影像组学、包装法特征选择、机器学习模型训练等方法,实现了乳腺癌数据的实时分类,提高了分类准确度,本发明利用了深度学习提取的高维度抽象特征,也避免了小数据集在深度学习上的过拟合问题。
附图说明
图1是本发明的步骤2到步骤6的流程图;
图2是本发明的步骤7到步骤8的流程图;
图3是本发明的步骤9到步骤13的流程图;
图4是svm-rfe特征选择图。
具体实施方式
如图1-图4所示的一种结合深度网络特征和机器学习模型的乳腺癌数据分类方法,包括如下步骤:
步骤1:建立影像采集服务器、客户端和数据分类服务器,影像图片采集服务器、客户端和数据分类服务器均通过互联网相互通信;
在数据分类服务器中建立图片处理模块、特征提取模块和模型构建模块;
步骤2:影像采集服务器采集肿瘤图片,并将肿瘤图片发送给客户端;
步骤3:医疗人员通过客户端编辑肿瘤图片,在肿瘤图片上勾画出肿瘤区域,生成肿瘤区域图片;
本实施例中,原始肿瘤图片大小为512×512,虚拟矩形的大小为224×224。
客户端将肿瘤图片和肿瘤区域图片一起发送给数据分类服务器;
步骤4:数据分类服务器接收到肿瘤图片和肿瘤区域图片后,图片处理模块设定一个虚拟矩形,找出肿瘤区域图片的中心点,以该中心点作为虚拟矩形的中心,根据虚拟矩形的边界对肿瘤区域图片进行截取,得到分类图片;
步骤5:按照步骤2到步骤4的方法,对多个肿瘤图片进行处理,得到分类图片的数据集合;
按照训练集70%、测试集30%的规则,利用分层抽样的方法将分类图片的数据集合划分为分类训练集和分类测试集;
对训练组数据进行数据集增广操作,得到扩大版训练集;
增广操作使用keras框架中的指定函数完成,包括0-20像素随机左右平移,0-20像素随机上下平移,图片-30度到30度随机旋转,图像180度旋转,图像镜面翻转,对图片对应的肿瘤勾画文件进行相同的操作,保证肿瘤区域与对应图像肿瘤区域吻合。数据增广后,训练集图片大小变为原来的5倍。
步骤6:特征提取模块调取扩大版训练集,从扩大版训练集中的分类图片中提取带有肿瘤的mri图层的图像特征;
本实施例中,提取工具采用pyradiomics开源包,分别提取一阶统计特征19个,2d形状特征10个,纹理特征10个,每层图片一共可提取39个影像组学特征。
图像特征如表1所示:
表139维特征是采用pyradiomics开源包进行计算得到,也就是表1中的39个特征,本发明把表1中的特征个数当作维。
深度学习每经过卷积和池化操作,就会得到一个特征图,可以把它理解成是一张图像,比如图像大小是28×28,28指的是像素点的个数。
那么将这个图像展平成向量就是784维,可以理解为784个(维)特征,将所有每张图像提取出来的特征放在一起,使用pca方法(成熟的方法)进行降维,降维后的特征个数可以指定,这里本实施例指定为64。
步骤7:模型构建模块采用tensorflow和keras提供的网络模型作为基准模型,建立训练模型,其步骤如下:
tensorflow和keras提供的网络模型都是在imagenet数据集上的预训练模型,医学图像数据集较小,适合采用迁移学习的方法进行模型训练。
步骤a1:载入基准模型的初始参数,去掉基准模型最后一层全连接层,加入初始化参数为0的新的全连接层,全连接层使用sigmoid激活函数,输出维度为2,输出值作为预测概率;
预测概率是深度模型使用的,因为深度学习模型需要预测概率值和真实值进行比较,所以需要计算损失函数从而进行梯度优化和参数优化。
步骤a2:将扩大版训练集作为模型训练数据,将其中的分类图片复制为三通道图像,采用5折交叉验证进行模型训练,建立训练模型;
步骤a4:将分类测试集放入训练模型中进行测试,得到模型评价,模型评价的标准为准确率和auc;
步骤8:选择基础模型中的隐藏层最后一层输出的特征图作为深度学习特征,将二维矩阵展平成一维向量,向量维度统一使用64维,大于64维的采用pca算法进行降维,得到39维影像组学特征与64维深度学习特征;
步骤9:将39维影像组学特征与64维深度学习特征组合成103维特征,特征选择使用开源的sklearn包进行,先通过p值检验,筛选出p值<0.05的特征;
103维特征具有统计学意义。
步骤10:使用pearson相关性分析对每两个特征之间的相关性进行计算,相关性大于0.85或者小于-0.85的两组特征认为是冗余特征,随机抛弃其中一个特征;
步骤11:使用svm-rfe进行包装法特征筛选,在初始特征集上训练评估器,并且通过feature_importances_属性获得每个特征的重要性,然后从当前的一组特征中丢弃最不重要的特征;
步骤12:在修剪的集合上递归地重复步骤10到步骤11的过程,直到最终到达所需数量的要选择的特征,选择auc作为特征选择的评价标准;
本实施例中,修剪的集合指的是修剪的特征集合,是采用svm-rfe进行特征选择。
svm-rfe中的rfe指的是递归特征消除,比如特征初始个数为39+64=103个,采用这种方法进行svm分类器的训练,每训练一轮,就得到一个auc值(auc为评价模型好坏的指标)并给特征重要程度进行排序,丢弃一个最不重要的特征,特征个数可以任意指定,本实施例中指定为1,这时候对剩下的102个特征进行训练,即修剪的特征集合,得到auc并丢弃最不重要的特征,重复进行,直到剩下一个特征,选择auc值最大时的特征组合为最终选择的特征组合。
步骤13:采用svm径向基核进行分类模型训练,采用五折交叉验证和网格搜索进行超参数的选择,采用测试数据进行模型评价,评价指标为auc,auc值最高的模型作为最终的分类模型;
步骤14:当影像采集服务器采集到新的肿瘤图片时,首选根据步骤3到步骤4的方法对肿瘤图片进行处理,然后提取带有肿瘤的mri图层的图像特征,最后采用步骤13中得出的最终的分类模型对新的肿瘤图片进行分类;
步骤15:数据分类服务器将步骤14得出的分类结果发送给客户端进行展示。
优选的,在执行步骤6时,所述图像特征包括一阶统计特征19个,2d形状特征10个,纹理特征10个,每层分类图片一共可提取39个影像组学特征。
优选的,在执行步骤a2时,所述三通道图像即为rgb图片。
优选的,在执行步骤a1时,所使用的预训练网络模型包括resnet50、inceptionv4、vgg16、vgg19、densenet、googlenet和alexnet。
本发明所述的一种结合深度网络特征和机器学习模型的乳腺癌数据分类方法,解决了对肿瘤图片进行数据分类的技术问题,本发明结合了迁移学习、深度学习特征提取、影像组学、包装法特征选择、机器学习模型训练等方法,实现了乳腺癌数据的实时分类,提高了分类准确度,本发明利用了深度学习提取的高维度抽象特征,也避免了小数据集在深度学习上的过拟合问题。
- 还没有人留言评论。精彩留言会获得点赞!