三维扫描中解剖结构的顺序分割的制作方法
[0001]
本发明涉及在容积扫描中分割解剖结构的方法,更具体地涉及在医学3d扫描中分割解剖结构的方法。
背景技术:
[0002]
容积医学扫描中解剖结构的准确分割在当前的临床实践中受到高度关注,因为它在涉及计算机辅助诊断、图像引导介入、放射治疗和放射学的许多任务中扮演重要角色。特别是,定量诊断需要了解解剖器官的准确边界。
[0003]
三维深度学习分割方法在各种器官和模态上示出有希望的结果。如lu等[11]在肝脏分割方面的工作一样,这些方法中的大多数是建立在3d u-net架构[5]上的。dou等[6]提出了一种3d全卷积结构,其通过深度监督层来提高肝脏分割的准确性。yang等[16]使用对抗式训练,以便在ct扫描中获得更好的肝脏3d u-net分割性能。sekuboyina等[14]提出了一种用于在ct扫描中定位和分割脊柱的管道方法。这里,椎骨分割以分块方式执行,以克服存储器限制,并且同时获得细粒度的结果。korez等[9]介绍了一种与多尺度双向cnn相组合的类似的分块方法。
[0004]
上述方法将源自3d图像采集的扫描重采样到容积中,并在3d中应用卷积。这通常涉及缩小规模,以克服存储器限制。因此,以分片方式处理容积数据的方法变得重要。例如,li等[10]首先将u-net [13]架构的分片密集连接变体应用于肝脏分割,并通过使用自动上下文算法的3d模型来细化结果。对于相同的任务,christ等[4]应用分片的u-net来获得粗略的分割,并利用3d中的条件随机场来细化结果。
[0005]
为了适当利用空间信息,仅依靠切片内的数据是不够的。为了解决该问题,除了主要的2d方法之外,上述方法还应用了计算昂贵的3d细化策略。
[0006]
相反,bates等[2]和chen等[3]示出了,将递归神经网络与全卷积方法相组合,允许直接处理切片上的空间信息。所提出的方法使用u-net变量来产生初始分割,所述初始分割随后由卷积长短期记忆网络(lstm)[7]进行细化。
[0007]
本发明的一个方面是提供一种改进的分割方法。
技术实现要素:
[0008]
上述方面通过如权利要求1所述的方法来实现。
[0009]
在从属权利要求中陈述了本发明优选实施例的具体特征。
[0010]
本发明的方法是一种通用的、鲁棒的、端到端的、时间分布卷积和双向卷积lstm块的混合。
[0011]
该方法克服了当前方法的诸如忽略各向异性体素大小、仅考虑切片内信息以及存储器限制之类的缺点。
[0012]
更进一步地,通过在描绘两个不同器官(即肝脏和椎骨)的两个ct数据集上进行评估,示出了视场的不变性,特别是恒定切片计数的不变性。肝脏分割通常是肝病诊断中的必
要步骤,而椎骨分割对于脊柱异常(例如,骨折)的标识或图像引导的脊柱干预是重要的。
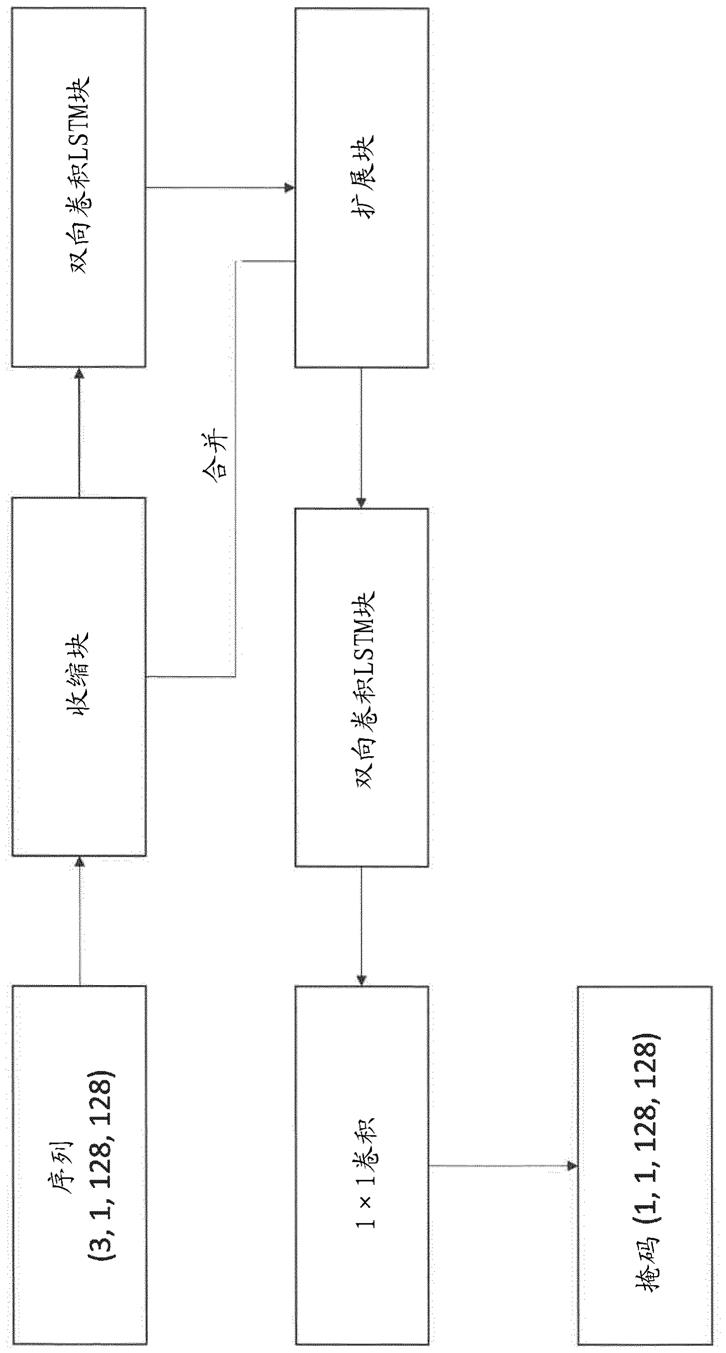
[0013]
从以下描述和附图中,本发明的进一步的优点和实施例将变得显而易见。
附图说明
[0014]
图1示出了本发明方法的基础架构的概述;图2是具有关于输入和输出张量的过滤器和形状的详细信息的表格;图3是对椎骨和肝脏的双重评估结果。
具体实施方式
[0015]
方法一般设置令为一组容积扫描,其中每个由体素和密度组成。
[0016]
更具体地,每个扫描因此是具有像素和密度的一组个切片。
[0017]
针对每个切片,可获得一组真值掩码:其中l对应于语义类别标签以及,大小为的所有二进制矩阵的空间。
[0018]
为了加强输入流形状的再现性,以如下方式构建了新的训练数据集。
[0019]
从每个扫描中,选择大小为的子集,其中可以被奇数整除。
[0020]
随后,每个被划分成长度为的保序子集,并且全部被组合成新的数据集i',该数据集现在由奇数个基数的子集组成。
[0021]
出于训练和评估的目的,数据集i'被划分成不重叠的集合,即和。
[0022]
在训练期间,网络连续地通过小批量,其中是集合的全部分区。
[0023]
针对每个序列,即,对于一些以及一些序列,计算网络的多类别输出:将网络理解为函数将网络理解为函数在单个步骤中针对单个像素导出其具有一定概率的语义分类,其中对应于序列的中间元素。
[0024]
为了估计并且最大化该概率,定义如下损失函数:
该损失函数估计网络结果与期望真值的偏差(误差)。
[0025]
使用由novikov等[12]导出的形式记法,以如下方式定义损失函数。
[0026]
对于距离函数,加权系数和序列,在集合k和全部分区上,损失函数为:。
[0027]
针对训练序列s、特征通道l、真值掩码g
s
、以及sigmoid激活函数的距离函数可以被定义为:其中是特征函数,即,如果在像素x的定位处g
s
为1,则,否则为0。
[0028]
架构为了利用保序切片(的元素)空间-时间相关性并且由于它们的顺序性质,时间分布卷积和双向卷积lstm块已经被组合在端对端的、可训练的、类似u-net的混合架构中。
[0029]
图1示出了所提出的架构的高层次概述。图2利用每层的张量形状补充了图1。
[0030]
所述网络将奇数长度的序列作为输入。
[0031]
然后,该序列被传递给收缩块。通过网络的收缩块独立地处理序列中的每个元素。收缩块由重复的时间分布卷积和最大池化层组成。
[0032]
时间分布卷积是传递给特殊封装器(wrapper)的典型卷积。时间分布封装器允许将任何层独立地应用于输入的每个时间帧(或切片)。在该工作的上下文中,这样的时间帧对应于从容积中提取的训练序列的元素。在本架构中,封装器应用于所有卷积、池化、上采样和级联层中。
[0033]
为了捕捉切片之间的时空相关性,已经将针对输入序列的每个元素提取的特征传递到网络的收缩部分的末端处的卷积lstm (clstm)块[15]。
[0034]
clstm的双向修改与求和运算符一起使用,以便使得网络能够学习两个方向上的切片的时空相关性。
[0035]
该clstm块旨在增加低维高抽象特征的依赖性。
[0036]
该双向clstm块的序列输出然后被传递到扩展路径。序列的每个元素都被独立地处理。
[0037]
扩展部分由时间分布卷积和上采样层组成。在每个上采样层之后,特征与来自收缩部分的相应特征级联。当特征的空间分辨率达到期望的大小时,序列被传递到另一个双向clstm块。对序列进行双向处理,并对输出进行求和。因此,所述块有助于实现两个目标:增加对高维特征的依赖性,并将进入的序列转换为单通道输出。
[0038]
所得到的特征然后被传递到(1,1)卷积层,以便将每个特征向量映射到期望数量的类。所述结果经由独立应用于每个像素的sigmoid激活映射到[0,1]范围中。这导致对输
入序列的中间元素的分割。
[0039]
实验设置训练数据和准备为了证明该架构在不同解剖器官上的可推广性,使用了椎骨和肝脏的公共数据集。
[0040]
对于椎骨分割,使用了csi 2014挑战训练集[17]。它包括覆盖整个腰椎和胸椎的10个ct扫描,以及每个扫描的全椎骨分割掩码。轴向平面内分辨率在0.3125和0.3616 mm2之间变化,切片厚度为1毫米。
[0041]
对于肝脏分割,使用了以下两个相关的数据集:3dircadb-01和3dircadb-02[1]。第一个数据集由10名和10名男性的20个3d计算机断层扫描组成,其中在75%的案例中具有肝脏肿瘤。第二个数据集由具有肝局灶性结节增生的两个匿名扫描组成。轴向平面内分辨率在0.56和0.961 mm2之间变化,并且切片厚度在1.0和4.0 mm之间变化。
[0042]
用于椎骨分割的训练序列中的连续元素是以3mm的距离生成的,并且在椎骨和肝脏区域内以5mm的距离进行肝脏分割。
[0043]
这些数字是基于数据集中的最大切片厚度选取的。在所有的评估中,我们使用了三个切片的序列.对两个器官的评估是以双重(two-fold)方式进行的。所有切片和掩码都被下采样为128
ꢀ×ꢀ
128成像分辨率,以用于及时运行评估。
[0044]
实施细节所有实验均使用python中具有tensorflow后端的keras进行。我们使用固定初始速率为10-5并且标准值为β1 = 0.9,β2 = 0.999的adam [8]优化算法,通过由等式3示出的损失,来训练网络。使用scikit图像库对真值掩码进行下采样。
[0045]
结果图3示出了椎骨和肝脏分割的两个组的dice和jaccard得分。前两列示出的是当考虑到整个容积时,甚至当感兴趣的器官不存在时的情况的分数。第三和第四列仅示出包含感兴趣器官的区域的结果。
[0046]
本发明的重点在于通用的分割方法,因此最终结果不进行后处理,因为通常该过程是特定于任务的,并且需要特定于器官的调整。在该情况下,如预期的那样,对于该上下文,不相关的骨骼结构被部分分割,从而降低了整个容积的得分。
[0047]
为了展示当前的网络比其2d变体有所改进,在相同的训练条件下,在肝脏的组1上构建并评估了另外两个架构。
[0048]
在第一架构中,输入被以如下方式改变:它将采用仅由一个切片组成的序列(我们旨在分割的中间切片)。
[0049]
在第二架构中,输入维度没有改变,但是第一卷积lstm块被移除,并且第二卷积块被聚合层代替,该聚合层将对时间信道上的进入的特征进行求和。
[0050]
当仅考虑器官区域时,这两个架构实现了0.87和0.878的相似dice得分,这显著地低于本发明网络的得分。
[0051]
参考文献1.3d-ircadb数据库,可在https://www.ircad.fr/research/3d-ircadb-01/获得。
[0052]
2.r. bates、b. irving,、b. markelc, j. kaeppler、r. muschel、v. grau、和 j. a. schnabel的“extracting 3d vascular structures from microscopy images using convolutional recurrent networks”, arxiv预印本, arxiv:1705.09597,2017年。
[0053]
3.j. chen、l. yang, y. zhang、m. alber、以及 d. z. chen的“combining fully convolutional and recurrent neural networks for 3d biomedical image segmentation”,nips,3036
–
3044页,2016年。
[0054]
4.p. f. christ、 m. e. a. elshaer、f. ettlinger、s. tatavarty、m. bickel、p. bilic, m. rempfler、m. armbruster、f. hofmann, m. danastasi等的
“ꢀ
automatic liver and lesion segmentation in ct using cascaded fully convolutional neural networks and 3d conditional random fields”,miccai,415
–
423页,springer出版社, 2016年。
[0055]
5.
öꢀ
cicek、a. abdulkadir、s. s. lienkamp、t. brox以及 o. ronneberger的“3d u-net: learning dense volumetric segmentation from sparse annotation”,miccai,424
–
432页, cham, 2016年,springer 国际出版社。
[0056]
6.q. dou, h. chen、y. jin、l. yu、j. qin以及 p.-a. heng的
“ꢀ
3d deeply supervised network for automatic liver segmentation from ct volumes”,miccai,149
–
157页,springer出版社,2016年。
[0057]
7.s. hochreiter 和j. schmidhuber的
“ꢀ
long short-term memory. neural computation”, 9(8):1735
–
1780, 1997年。
[0058]
8.d. kingma和j. ba. adam的“a method for stochastic optimization”,《 iclr》,2015年。
[0059]
9.r. korez、b. likar、f. pernu
ˇ
s以及 t. vrtovec的
“ꢀ
segmentation of pathological spines in ct images using a two-way cnn and a collision-based model”,international workshop and challenge on computational methods and clinical applications in musculoskeletal imaging,95
–
107页,springer出版社, 2017年。
[0060]
10.x. li、h. chen、x. qi、q. dou、c.-w. fu以及 p. a. heng.的“h-denseunet: hybrid densely connected u-net for liver and liver tumor segmentation from ct volumes”,arxiv预印本, arxiv:1709.07330,2017年。
[0061]
11.f. lu、f. wu、p. hu, z. peng以及d. kong的“automatic 3d liver location and seg
-ꢀ
mentation via convolutional neural network and graph cut”,international journal of cars, 12(2):171
–
182,2017年。
[0062]
12.a. a. novikov、d. lenis、d. major、j. hlad
˚
uvka、m. wimmer以及 k. b
ü
hler的
“ꢀ
fully convolutional architectures for multi-class segmentation in chest radiographs”,ieee transactions on medical imaging,pp(99):1
–
1,2018年。
[0063]
13.o. ronneberger、p. fischer以及t. brox的“u-net: convolutional networks for biomedical image segmentation”,miccai,234
–
241页, springer出版社,2015年。
[0064]
14.a. sekuboyina、j. kukacka、j. s. kirschke、b. h. menze以及a. valentinitsch的“attention-driven deep learning for pathological spine segmentation”,international workshop and challenge on computational methods and clinical applications in musculoskeletal imaging, 108
–
119页,springer出版社,2017年。
[0065]
15.x. shi、z. chen、h. wang、d.-y. yeung、w.-k. wong以及 w.-c. woo的“convolutional lstm network: a machine learning approach for precipitation nowcasting”,nips,nips,802
–
810页, mit press,2015年。
[0066]
16.d. yang、d. xu、s. k. zhou、b. georgescu、m. chen、s. grbic、d. metaxas以及d. comaniciu的“automatic liver segmentation using an adversarial image-to-image network”,miccai,507
–
515页,springer出版社, 2017年。
[0067]
17.j. yao、j. e. burns、h. munoz以及r. m. summers的“detection of vertebral body fractures based on cortical shell unwrapping”,miccai,509
–
516页,berlin,heidelberg,2012年,springer berlin heidelberg出版社。
[0068]
已经详细描述了本发明的优选实施例,现在对于本领域技术人员来说应当显而易见的是,在不脱离所附权利要求中限定的本发明的范围的情况下,可以对其进行多种修改。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1