一种基于U-Net网络改进的乳腺MRI分割方法
本发明涉及医疗科技技术领域,特别涉及一种基于u-net网络改进的乳腺mri分割方法。
背景技术:
据世界卫生组织(who)估计,2012年乳腺癌的发病率为167万例(其中78.8万例在较发达国家),52.2万人死亡(198000例在较发达国家),2007-2012年期间全球有620万例(320万例在较发达国家)。世卫组织指出,早期发现是降低癌症死亡率的一个关键因素。尽管世界卫生组织提倡用乳房x射线摄像作为参考筛查工具,但由于mri(非电离辐射)的安全性、高3d分辨率和动态信息,动态对比增强磁共振成像(dce-mri)已越来越多地作为临床成像程序,用于评估新辅助化疗的反应。
dce-mri检查产生4d(3d空间+1d时间)数据,包括静脉注射顺磁造影剂(通常为钆基)前后获得的图像。通常,在每次采样时间里会获得数百张图像。此外,为了进行高质量的数据分析,可能需要多次采样(从10次到100次);每个图像都在数万体素的范围内。分析如此庞大的数据是一项非常耗时且容易出错的任务。许多研究致力于开发计算机辅助检测/诊断(cad)系统,以支持医生进行dce-mri数据筛查。dce-mricad系统的功能包括:乳腺组织分割、运动纠正(减少不自主运动引起的运动伪影)、roi检测、病变恶性程度评估和治疗评估。
技术实现要素:
在本发明中,我们关注cad的第一阶段,这个阶段通常被称为breastmaskextraction,目的是对所有乳腺组织的精细分割。在之前的研究表明,这第一阶段强烈影响后续的分析。运动校正阶段可以依靠乳房边缘更好地模拟软组织的变形。病变检测阶段也可能受益于摘除乳腺面罩(breastmask),避开其他器官,同时包括一些非常敏感的部位,如腋窝或胸肌附近的组织,这些部位可能存在一些病变。此外,通过减少感兴趣的体积和移除不需要的体素来减少所有后续分析的计算工作量。
一种基于u-net网络改进的乳腺mri分割方法,基于动态对比增强磁共振成像(dce-mri),其具体步骤为,
q1.建立一个用于乳腺分割的2d卷积神经网络。
q2.将3d乳腺dce-mri数据进行切片,同时保存2d图像在3d空间中的位置索引信息,
生成2d乳腺数据。
q3.准备数据集,对2d乳腺数据进行预处理,增加对比度,归一化和数据增强(翻转,平移,镜像),数据集包括训练集+测试集。
q4.利用训练集训练神经网络,得到一个全卷积神经网络模型。
q5.使用得到的网络模型分割测试集中乳腺dce-mri数据,得到2d分割结果,将2d分割结果按序拼接得到3d分割结果。
本发明的有益效果。
本发明提供了一种基于u-net网络改进的乳腺mri分割方法,实现对所有乳腺组织的精细分割,利用3d的空间位置信息,提高乳腺面罩(breastmask)的分割精度,为后续乳腺肿瘤的分析计算减少工作量。
附图说明
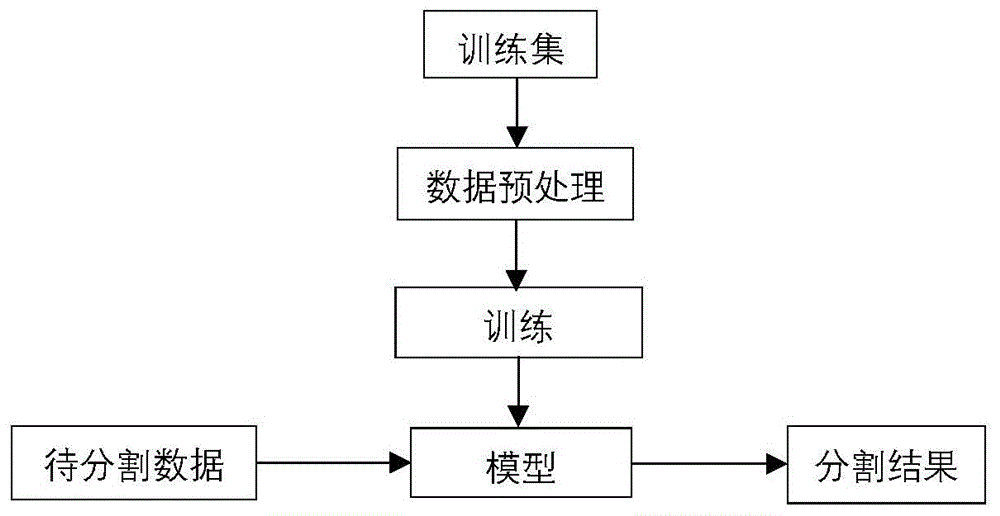
图1为本发明提供的一种基于u-net改进的卷积神经网络用于乳腺dce-mri分割方法的整体步骤流程结构示意图。
图2为本发明中乳腺mri数据三维示意图,及按横断面(transverseplane)切片示意图。
图3为本发明中使用到的注意力模块,为通道空间注意力模块(cbam)。
图4为本发明提供的一种基于u-net网络改进的卷积神经网络结构示意图。
具体实施方式
下面结合符图,对本发明的一个具体实施方式进行详细描述,但应当理解本发明的保护范围并不受具体实施方式的限制。
如图1所示,一种基于u-net网络改进的乳腺mri分割方法,基于动态对比增强磁共振成像(dce-mri),其具体步骤如下:
步骤一:鉴于3d卷积神经网络在模型参数,数据量上占用资源过大,且3d数据集有限。2d卷积神经网络在模型参数、精度、速度和数据量上表现的更好,且数据集充分。本发明采用2d卷积神经网络对3d乳腺dce-mri数据进行分割。首先,建立一个用于乳腺分割的2d卷积神经网络(附图4),卷积神经网络的输入是一个512*512的灰度图和一个长度为108的一维位置索引向量(one-hot编码方式),输出是512*512的二值图像;网络结构由左、右、上三个部分构成,网络的左侧是输入图像的encoder部分,右侧是输入图像的decoder部分,上方是对一维位置索引向量的encoder部分;
网络的卷积层采用denseblock(附图4),每个block由(bn+relu+conv3*3)构成。网络上方索引向量的encoder部分先通过向量和向量转置的乘积生成108*108矩阵,再经过上采样调整成512*512大小的索引图。该索引图经过denseblock和下采样调整通道数和特征图大小,然后与网络左侧每一次下采样前输出的各维特征图进行特征融合(对两个特征图求和)。在网络右侧的上采样过程中,将encoder阶段输出的各维特征图经过通道-空间注意力模块(附图3)后,与decoder阶段的各维输出拼接。
步骤二:制作2d乳腺dce-mri数据,首先,将3d乳腺dce-mri数据从冠状面(transverseplane)进行切片(附图2),得到一系列2d图片,保存的2d文件名以“3d文件名+位置索引+文件扩展名”格式命名,其中“位置索引”即为2d图像在3d空间中的位置索引。(例如对3d“caoyuejuan_002061813”切片得到2d序列,其中“caoyuejuan_002061813_89.png”表示该2d图像在3d数据中的位置索引为89,该索引信息用于解析出索引向量作为网络的另一个输入。)对3d数据进行切片时保存2d平面数据在3d空间中的位置索引信息,从而在步骤四中与2d数据一起作为输入送入神经网络进行训练。至此,完成2d乳腺dce-mri数据制作。
步骤三:准备数据集,对2d乳腺dce-mri数据进行预处理,首先进行数据划分,将数据集按8:2分成训练集和测试集。其次,进行图像增强:1).增加对比度。2).归一化。最后进行数据增强:1).翻转。2).平移。3).镜像。(其中平移操作即保持乳腺区域完整性不变的情况下,将图像进行上下平移。)至此,完成2d乳腺dce-mri数据预处理。
步骤四:利用训练集训练卷积神经网络。首先,读取训练集,对于每一个2d乳腺图像,可以产生两个数据,分别为乳腺图像数据和由位置索引生成的一维向量。其中该一维位置向量采用one-hot编码方式生成,即对应索引位置为1,其它位置为0.然后,将乳腺图像数据和位置向量作为输入,送入卷积神经网络进行训练。最后,得到一个卷积网络模型。
步骤五:使用得到的网络模型分割测试集中乳腺图像数据,得到2d乳腺图像分割结果,将2d分割结果按照位置索引拼接得到3d分割结果。
以上公开的仅为本发明的几个具体实施例,但是,本发明实施例并非局限于此,任何本领域的技术人员能思之的变化都应落入本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!