一种病灶定向合成方法、装置、设备及存储介质与流程
1.本技术实施例涉及图像处理技术领域,尤其涉及一种病灶定向合成方法、装置、设备及存储介质。
背景技术:
2.随着ct分辨率的提高,影像科医生面对大量图像,很容易因工作疲劳而导致漏诊和误诊,难以满足高质量的阅片需求。计算机辅助检测(computer aided detection,cade)系统能够自动检测,并进行定量分析,可以辅助影像科医生阅片,减轻工作负担,提高阅片精度和效率。
3.基于深度学习的肺结节检测算法具有很高的检测精度,但算法的训练需要大量的高质量标注数据,而一个薄层胸部ct序列包含200-500张图像,需要逐层查看进行标注。由于图像和病灶(图像噪声、层厚、病灶类别、尺寸等)分布的不同,例如在一个数据集上训练好的模型,在另一个数据集上测试的效果往往不佳,需要在新的数据集上重新训练或者迁移训练,导致深度学习算法模型的泛化性较弱。
技术实现要素:
4.本技术实施例提供一种病灶定向合成方法、装置、设备及存储介质,以提高病灶图像的多样性。
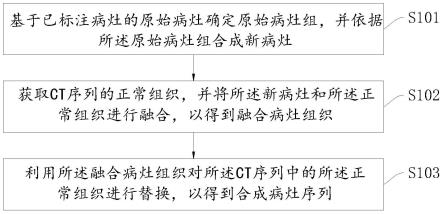
5.在第一方面,本技术实施例提供了一种病灶定向合成方法,包括:
6.基于已标注病灶的原始病灶确定原始病灶组,并依据所述原始病灶组合成新病灶;
7.获取ct序列的正常组织,并将所述新病灶和所述正常组织进行融合,以得到融合病灶组织;
8.利用所述融合病灶组织对所述ct序列中的所述正常组织进行替换,以得到合成病灶序列。
9.在第二方面,本技术实施例提供了一种病灶定向合成装置,包括病灶合成模块、组织融合模块和病灶替换模块,其中:
10.所述病灶合成模块,用于基于已标注病灶的原始病灶确定原始病灶组,并依据所述原始病灶组合成新病灶;
11.所述组织融合模块,用于获取ct序列的正常组织,并将所述新病灶和所述正常组织进行融合,以得到融合病灶组织;
12.所述病灶替换模块,用于利用所述融合病灶组织对所述ct序列中的所述正常组织进行替换,以得到合成病灶序列。
13.在第三方面,本技术实施例提供了一种病灶定向合成设备,包括:存储器以及一个或多个处理器;
14.所述存储器,用于存储一个或多个程序;
15.当所述一个或多个程序被所述一个或多个处理器执行,使得所述一个或多个处理器实现如第一方面所述的病灶定向合成方法。
16.在第四方面,本技术实施例提供了一种包含计算机可执行指令的存储介质,所述计算机可执行指令在由计算机处理器执行时用于执行如第一方面所述的病灶定向合成方法。
17.本技术实施例通过将原始病灶组合成新病灶,并在ct序列中选择正常组织,将正常组织与新病灶进行融合得到融合病灶组织,然后利用融合病灶组织替换掉ct序列中的正常组织,以得到合成病灶序列,合成病灶序列中的融合病灶组织由不同原始病灶合成得到,有效增加病灶图像的多样性,通过不同原始病灶组的定向合成,有效实现在病灶级别上的数据扩增,并且通过将正常组织与新病灶进行融合,得到的融合病灶组织更逼真,将合成病灶序列用于深度学习算法模型的训练,有效提高深度学习算法模型精准度,并提高深度学习算法模型的泛化能力。
附图说明
18.图1是本技术实施例提供的一种病灶定向合成方法的流程图;
19.图2是本技术实施例提供的另一种病灶定向合成方法的流程图;
20.图3是本技术实施例提供的一种新病灶合成效果示意图;
21.图4是本技术实施例提供的一种合成病灶序列效果示意图;
22.图5是本技术实施例提供的一种在不同缩放系数下高斯掩码的中间层面图像示意图;
23.图6是本技术实施例提供的一种融合病灶组织的效果示意图;
24.图7是本技术实施例提供的一种合成病灶序列与原生病灶序列的效果对比示意图;
25.图8是本技术实施例提供的一种融合病灶标注在合成病灶序列中的示意图;
26.图9是本技术实施例提供的一种病灶定向合成装置的结构示意图;
27.图10是本技术实施例提供的一种病灶定向合成设备的结构示意图。
具体实施方式
28.为了使本技术的目的、技术方案和优点更加清楚,下面结合附图对本技术具体实施例作进一步的详细描述。可以理解的是,此处所描述的具体实施例仅仅用于解释本技术,而非对本技术的限定。另外还需要说明的是,为了便于描述,附图中仅示出了与本技术相关的部分而非全部内容。在更加详细地讨论示例性实施例之前应当提到的是,一些示例性实施例被描述成作为流程图描绘的处理或方法。虽然流程图将各项操作(或步骤)描述成顺序的处理,但是其中的许多操作可以被并行地、并发地或者同时实施。此外,各项操作的顺序可以被重新安排。当其操作完成时所述处理可以被终止,但是还可以具有未包括在附图中的附加步骤。所述处理可以对应于方法、函数、规程、子例程、子程序等等。
29.图1给出了本技术实施例提供的一种病灶定向合成方法的流程图,本技术实施例提供的病灶定向合成方法可以由病灶定向合成装置来执行,该病灶定向合成装置可以通过硬件和/或软件的方式实现,并集成在病灶定向合成设备中。
30.下述以病灶定向合成装置执行病灶定向合成方法为例进行描述。参考图1,该病灶定向合成方法包括:
31.s101:基于已标注病灶的原始病灶确定原始病灶组,并依据所述原始病灶组合成新病灶。
32.本实施例提供的原始病灶组包括两个或两个以上已标注病灶的原始病灶,其中对原始病灶的标注类型可以是原始病灶的尺寸、纹理类型、病灶类型、危险程度等。例如,对于一个ct序列,分别对ct序列中的每一层的影像图像进行病灶标注,例如标注出各层影像图像中出现的病灶的尺寸、纹理类型、病灶类型、危险程度等,并由各层对病灶的标注确定该ct序列中的原始病灶以及对应的原始病灶标注。
33.示例性的,在样本数据集中选择两个原始病灶,并将这两个原始病灶共同作为一个原始病灶组。其中,样本数据集保存有多个已标注病灶的原始病灶。可选的,在选择原始病灶构建原始病灶组时,根据原始病灶标注选择相近的两个原始病灶,例如在相同尺寸范围、纹理类型、病灶类型或危险程度内选择两个原始病灶构成一个原始病灶组,并依次在样本数据集中抽选原始病灶构建多个原始病灶组。
34.进一步的,在确定原始病灶组后,将原始病灶组中的原始病灶组进行合成,得到合成后的新病灶。新病灶是原始病灶组中多个原始病灶局部特征的融合,与传统针对单个样例的数据扩增(例如采用旋转、裁剪、缩放等方式实现的数据扩增)相比,具有更强的代表能力,能增加更丰富的病灶样本。
35.s102:获取ct序列的正常组织,并将所述新病灶和所述正常组织进行融合,以得到融合病灶组织。
36.示例性的,选择一个ct序列,并获取该ct序列中的正常组织,将上述得到的新病灶和该正常组织进行融合,得到融合后的融合病灶组织。可选的,将新病灶的边缘与正常组织进行融合,使得新病灶的边缘更接近ct序列中的正常组织。
37.可以理解的是,以肺结节病灶为例,肺结节病灶大多是肺内的类圆形或不规则形的病灶,故在模拟真实病灶时,需要将合成的新病灶融合进肺部组织,与真实病灶越接近越好,若直接将合成的新病灶替换正常的肺部组织,即直接将新病灶替换掉ct序列中的正常组织,新病灶与正常组织的差别较大,会产生较强的突兀感。本实施例利用正常组织与新病灶进行融合,使得新病灶的边缘更接近ct序列中的正常组织,新病灶与正常组织之间的过渡更平滑自然。
38.s103:利用所述融合病灶组织对所述ct序列中的所述正常组织进行替换,以得到合成病灶序列。
39.示例性的,将新病灶与正常组织进行融合得到融合病灶组织后,将融合病灶组织替换掉ct序列中的正常组织,从而将融合病灶组织合成到ct序列中,以得到合成病灶序列。该合成病灶序列可用于深度学习算法模型的训练中,并且本技术通过不同原始病灶组成的原始病灶组,得到不同的新病灶,有效实现病灶级别的数据扩增,并增加病灶图像的多样性。
40.上述,通过将原始病灶组合成新病灶,并在ct序列中选择正常组织,将正常组织与新病灶进行融合得到融合病灶组织,然后利用融合病灶组织替换掉ct序列中的正常组织,以得到合成病灶序列,合成病灶序列中的融合病灶组织由不同原始病灶合成得到,有效增
51.由上表可知,该公开数据集lndb19中结节病灶以微小结节(直径小于5mm的原始病灶)和实性结节为主,分布极不均衡。在指定分布范围中随机选择原始病灶构建原始病灶组,以得到多个原始病灶组。例如在尺寸直径在10-20mm范围中,依次随机抽取两个原始病灶构建一个原始病灶组,直至该分布范围的原始病灶抽取完或剩下的原始病灶不足以构建原始病灶组,此时得到了定向在尺寸直径为10-20mm范围内的原始病灶组;或者是在实性结节的范围中,依次随机抽取两个原始病灶构建一个原始病灶组,直至该分布范围的原始病灶抽取完或剩下的原始病灶不足以构建原始病灶组,此时得到了定向在实性结节范围内的原始病灶组。
52.可以理解的是,本方案中对原始病灶进行分布统计时基于原始病灶的尺寸和纹理类型进行,并按此分布提取所需样本。可以理解的是,分布依据可以替换为其他分布类型,如lung-rads分级等,本技术不做限定。
53.s202:对所述原始病灶组进行尺寸归一化,并依据所述原始病灶组合成新病灶。
54.由上述可知,原始病灶组由不同的原始病灶组成,在合成新病灶之前,需要对原始病灶组进行尺寸归一化,以保证新病灶的合成效果。
55.在一个实施例中,对所述原始病灶组进行尺寸归一化,包括步骤s2021-步骤s2022:
56.s2021:基于所述原始病灶组中的所述原始病灶所对应的原始病灶尺寸,在所述原始病灶尺寸之间随机确定归一化尺寸。
57.具体的,对于每个原始病灶组,确定原始病灶组中各个原始病灶的原始病灶尺寸(可根据原始病灶的标注信息确定),并在这些原始病灶尺寸之间随机确定一个归一化尺寸。
58.以一个原始病灶组包含两个原始病灶为例,假设两个原始病灶的原始病灶尺寸分别为s1和s2,在s1和s2之间随机确定一个尺寸作为归一化尺寸s,设定归一化尺寸s在s1和s2之间服从均匀分布,即s~u(s1,s2),此时归一化尺寸s在s1和s2之间取任意值的概率一样。
59.s2022:按照所述归一化尺寸对所述原始病灶进行尺寸归一化。
60.具体的,在确定原始病灶组对应的归一化尺寸后,按照该归一化尺寸对该原始病灶组中的各个原始病灶进行尺寸归一化,以将各个原始病灶的病灶尺寸转换为相应的归一化尺寸。例如通过拉伸、缩放、裁剪等方式,将原始病灶的病灶尺寸转换为归一化尺寸。此时,原始病灶组中的全部原始病灶的尺寸均转换为归一化尺寸,即同一原始病灶组中的全部原始病灶的尺寸均一致。
61.在对原始病灶组的各个原始病灶进行尺寸归一化后,将原始病灶组合成为新病灶。在一个可能的实施例中,依据所述原始病灶组合成新病灶可以是按照贝塔分布将所述原始病灶组合成新病灶。
62.具体的,按照贝塔分布将所述原始病灶组合成新病灶包括步骤s2023-步骤s2024:
63.s2023:按照贝塔分布确定合成参数,基于所述合成参数确定所述原始病灶组中各个所述原始病灶的合成比例。
64.s2024:基于所述合成比例和所述归一化尺寸,将所述原始病灶组合成新病灶。
65.以两个原始病灶组成一个原始病灶组为例,本实施例提供的新病灶的合成公式
为:
66.patch_new=λ*patch1+(1-λ)*patch2
67.其中,patch_new为新病灶,patch1和patch2为两个原始病灶,λ为合成参数,λ的定义域为[0,1],并且合成参数λ服从贝塔分布,即λ~beta(α,β)。贝塔分布是一个概率的概率分布,其定义域为0~1,即λ∈[0,1],α和β为贝塔分布的两个形状参数,用于控制贝塔分布的分布形状,并且α、β∈(0,∞)。需要进行解释的是,当α《1并且β《1时,贝塔分布的分布形状为u形。本实施例将λ作为原始病灶patch1的合成比例,并将(1-λ)作为原始病灶patch2的合成比例。
[0068]
本示例中选择贝塔分布的形状参数对应设置为贝塔分布所对应的分布形状为u形,例如设置α=β=0.2(可根据实际情况进行设置,本技术不做限定),此时贝塔分布的分布形状为对称u形,贝塔分布的中心为0.5,两侧分布概率较中心高。
[0069]
具体的,基于设定的形状参数,依据贝塔分布确定合成参数λ的值,并将其中一个原始病灶patch1的合成比例为λ,另一个原始病灶patch2的合成比例为(1-λ)。进一步的,基于原始病灶patch1的合成比例λ、原始病灶patch2的合成比例(1-λ)以及归一化尺寸s,将两个原始病灶合称为一个新病灶,并且新病灶的病灶尺寸为归一化尺寸s。
[0070]
图3给出了本技术实施例提供的一种新病灶合成效果示意图,如图3所示,示例性的,图3中patch1和patch2为原始病灶组中的两个原始病灶,patch_new为由patch1和patch2合成的新病灶。其中随机生成的归一化尺寸s为15.1mm,生成的合成参数λ为0.65,那么patch1和patch2的合成比例分别为0.65和0.35,此时patch_new=0.65*patch1+0.35*patch2,并且patch_new的病灶尺寸为15.1mm。合成的新病灶patch_new产生于原始病灶对,新病灶patch_new每一个像素点均由两个原始病灶对应像素点按照合成比例融合得到,是两者局部特征的融合,与传统针对单个样例的数据扩增相比,具有更强的代表能力,能增加更丰富的病灶样本。
[0071]
可以理解的是,本技术提供的合成参数可应用于所有原始病灶组进行原始病灶的融合,还可以是对每组原始病灶组进行原始病灶的融合时,均重新进行合成参数的确定,本技术不做限定。
[0072]
s203:依据所述新病灶的病灶尺寸和三维高斯函数,生成高斯掩码。
[0073]
可以理解的是,在得到新病灶后,需要将新病灶融合到ct序列中,由于对原始病灶组融合得到的新病灶与进行融合的ct序列存在较大差别,若直接将新病灶替换掉ct序列中的正常组织,将产生较大的突兀感。图4为本技术实施例提供的一种合成病灶序列效果示意图,如图4所示,此时直接将新病灶和ct序列进行融合得到的合成病灶序列在融合位置中,可观察到新病灶与周围正常组织存在明显的融合边界。
[0074]
为了实现新病灶与周围正常组织的平滑过渡,本方案引入高斯函数,由于病灶为三维组织结构,本技术引入的高斯函数具体为三维高斯函数。具体的,由上述可知,新病灶的病灶尺寸为归一化尺寸,本实施例依据新病灶的病灶尺寸和三维高斯函数,生成高斯掩码。
[0075]
本方案提供的三维高斯函数的计算公式为:
[0076][0077]
其中,f为三维高斯函数,假设三个维度方向(x、y、z)相互独立,并具有各自不同的方差x、y、z表示到中心点距离,k(k≥1)为缩放系数。
[0078]
在一个可能的实施例中,本方案提供的三维高斯函数三个维度方向的方差各向不同。并且,三维高斯函数三个维度方向的标准差以新病灶的病灶尺寸为基数随机生成。具体的,三维高斯函数各维度方向的标准差的计算公式为:
[0079][0080]
其中,σi为i方向的标准差,di为病灶i方向的直径,t1和t2为所取的最小和最大值。本方案以t1=0.34、t2=0.66为例进行描述,即生成的高斯掩码的半径为标准差的1.51到2.94倍(1/t倍,倒数关系)之间。
[0081]
在具体化缩放系数、三个方向的方差或标准差、三个方向的大小后,可得到对应的高斯掩码,即高斯掩码是三维高斯函数的实例。
[0082]
图5为本技术实施例提供的一种在不同缩放系数下高斯掩码的中间层面图像示意图,图中仅展示高斯掩码的中间层面图像,当k设置为1(图5左)时,高斯掩码各维度方差相同,并且分辨率高,在k设置为2(图5右)时,三维高斯函数各维度方差不同,并且分辨率低。结合图5,从高斯掩码的边缘到中心,三维高斯函数值逐渐增大,当k设置为1时,中心点最大值为1,而边缘最小值取决于各维度方差的大小,当标准差σ小于半径的1/3时,边缘高斯函数值接近0。可以理解的是,新病灶和正常组织融合的目的是让新病灶边缘在ct序列中的过渡更平滑自然,所以只需要进行新病灶边缘和正常组织的融合,而不需要进行整个新病灶的渐进过渡融合,所以本实施例将k设置为2(可根据实际情况进行设置,本技术不做限定),并且当三维高斯函数值大于1时,将三维高斯函数值置为1,此时k可作为对新病灶和正常组织进行融合的控制融合比例系数,其越大则进行融合的新病灶区域就越靠近边缘。
[0083]
s204:随机获取ct序列的正常组织,并利用所述高斯掩码对所述新病灶和所述正常组织进行融合,以得到融合病灶组织。
[0084]
随机选择一个ct序列,并随机获取该ct序列中的正常组织,并且该正常组织的组织尺寸与需要进行融合的新病灶的并在尺寸一致,即正常组织的组织尺寸为归一化尺寸。
[0085]
本方案采用各向异性(各维度方向的方差不同)的三维高斯函数来进行新病灶和正常组织的融合,同时设计了融合比例系数k,可以自由灵活的控制融合比例,使得生成的融合病灶组织更自然、逼真和丰富。
[0086]
进一步的,在确定ct序列中的正常组织后,利用上述得到的高斯掩码对新病灶和正常组织进行融合,以得到融合病灶组织。据此,对新病灶和正常组织进行融合具体包括步骤s2041-步骤s2042:
[0087]
s2041:基于所述高斯掩码确定所述新病灶和所述正常组织所对应的融合比例。
[0088]
具体的,本实施例以两个原始病灶合成一个新病灶为例进行描述,本实施例根据高斯掩码确定新病灶和正常组织对应的融合比例,例如在确定高斯掩码为gmask时,将新病
灶的融合比例确定为gmask,并将正常组织的融合比例确定为(1-gmask)。
[0089]
s2042:按照所述融合比例对所述新病灶和所述正常组织进行融合,以得到融合病灶组织。
[0090]
按照新病灶和正常组织各自对应的融合比例,对新病灶和正常组织进行融合,以得到融合病灶组织。可以理解的是,融合病灶组织的病灶尺寸与归一化尺寸一致,融合病灶组织每一个点的值,与新病灶和正常组织在对应点的值按照融合比例融合后得到的值一致。
[0091]
具体的,本方案提供的融合病灶组织的融合公式为:
[0092]
patch_m=gmask*patch_new+(1-gmask)*patch0
[0093]
其中,patch_m为融合病灶组织,gmask为高斯掩码,patch_new为新病灶,patch_0为正常组织,对应的,新病灶对应的融合比例为gmask,正常组织的融合比例为(1-gmask)。
[0094]
图6为本技术实施例提供的一种融合病灶组织的效果示意图,图6中patch0为正常组织,patch_new为新病灶,patch_m为融合病灶组织。如图6所示,从随机获取的ct序列中提取出正常组织patch0,并根据上述确定的高斯掩码确定新病灶patch_new的融合比例为gmask,正常组织patch0的融合比例为(1-gmask),根据融合公式对新病灶和正常组织进行融合后得到如图6所示的融合病灶组织patch_m。
[0095]
s205:利用所述融合病灶组织对所述ct序列中的所述正常组织进行替换,以得到合成病灶序列。
[0096]
由上述可知,融合病灶组织的病灶尺寸与提取出的正常组织的组织尺寸一致,可直接将融合病灶组织替换掉ct序列中提取的正常组织,以得到合成病灶序列,实现融合病灶组织与ct序列的融合。
[0097]
图7为本技术实施例提供的一种合成病灶序列与原生病灶序列的效果对比示意图,图7中a~d为合成病灶序列的中间层面示意图,e和f为原生病灶序列的中间层面示意图,图中箭头所指向区域为融合病灶组织和原始病灶组织。从图7可看出,融合病灶组织在合成病灶序列中的融合效果和原始病灶组织在原生病灶序列中的显示效果非常接近。
[0098]
s206:基于所述融合病灶组织对应的病灶尺寸和所述融合病灶组织在所述ct序列中的病灶位置,生成所述融合病灶组织对应的融合病灶标注。
[0099]
具体的,在将融合病灶组织融合到ct序列中得到合成病灶序列后,根据融合病灶组织的病灶尺寸(例如与上述确定的归一化尺寸一致),以及融合病灶组织在ct序列中的病灶位置(与被替换掉的正常组织的组织位置一致),生成该融合病灶组织对应的融合病灶标注,并在合成病灶序列中同步标注该融合病灶标注。
[0100]
在一个可能的实施例中,在对融合病灶组织进行标注之前,需要重新确定融合病灶组织的病灶尺寸。据此,在利用所述融合病灶组织对所述ct序列中的所述正常组织进行替换,以得到合成病灶序列之后,还包括步骤s2061-步骤s2062:
[0101]
s2061:以所述高斯掩码的标准差为基准确定所述融合病灶组织对应的病灶尺寸。
[0102]
s2062:基于所述融合病灶组织对应的病灶尺寸和所述融合病灶组织在所述ct序列中的病灶位置,生成所述融合病灶组织对应的融合病灶标注。
[0103]
其中,融合病灶组织在ct序列中的病灶位置与被替换的正常组织的中心位置一致,记为(x0,y0,z0),但是融合病灶组织的病灶尺寸不等于正常组织的组织尺寸。可以理解
的是,在将新病灶和正常组织进行融合时,由于新病灶的边缘部分被融合成背景,融合病灶组织的病灶尺寸要小于提取的正常组织的组织尺寸,所以需要重新估计融合病灶组织的病灶尺寸。
[0104]
由于在对新病灶和正常组织进行融合时采用的是三维高斯函数,本实施例以高斯掩码的标准差σ为基准进行病灶尺寸(半径)的估计,即融合病灶组织在i方向的半径为ri=γσi,其中γ可根据实际情况进行设置,本技术不做限定,例如γ与步骤s203中t1和t2的选择相关。
[0105]
图8是本技术实施例提供的一种融合病灶标注在合成病灶序列中的示意图,其中融合病灶标注按照融合病灶组织的尺寸和位置在合成病灶序列中进行展示。本实施例取γ=1.5为例,生成的融合病灶标注如图8所示,生成的融合病灶标注可以较好的表示生成的病灶,融合病灶标注记为(x0,y0,z0,3σ
x
,3σy,3σz),表示融合病灶标注记在合成病灶序列中的中心点和直径。以高斯掩码的标准差σ为基准进行融合病灶组织半径的重新估计,生成更精准的融合病灶组织的融合病灶标注(位置、大小等),可有效增加标注数据,辅助检测算法提升精度。
[0106]
上述,通过将原始病灶组合成新病灶,并在ct序列中选择正常组织,将正常组织与新病灶进行融合得到融合病灶组织,然后利用融合病灶组织替换掉ct序列中的正常组织,以得到合成病灶序列,合成病灶序列中的融合病灶组织由不同原始病灶合成得到,有效增加病灶图像的多样性,通过不同原始病灶组的定向合成,有效实现在病灶级别上的数据扩增,并且通过将正常组织与新病灶进行融合,得到的融合病灶组织更逼真,将合成病灶序列用于深度学习算法模型的训练,有效提高深度学习算法模型精准度,并提高深度学习算法模型的泛化能力。并且通过对原始病灶组进行尺寸归一化,提高对原始病灶的融合效果,融合得到的新病灶是多个原始病灶的局部特征的融合,与传统针对单个样例的数据扩增相比,具有更强的代表能力,能增加更丰富的病灶样本。通过三维高斯函数实现新病灶的边缘和正常组织的融合,并得到融合病灶组织,使得融合病灶组织在合成病灶序列中的过渡更平滑,并且通过重新估计融合病灶组织的病灶尺寸,并结合融合病灶组织在ct序列中的病灶位置得到融合病灶标注,自动生成融合病灶组织在合成病灶序列中的融合病灶标注,提高融合病灶组织的标注效率。同时,本方案采用原始病灶组和贝塔分布来合成新病灶,相比于单个病灶的增强,具有更强的表示能力,能提供更丰富的病灶样本。另外,本方案可按照对病灶的分布需求,对病灶进行定向合成扩增,有效缓解病灶级别的分布不均衡问题,并有效缓解病灶分布不均衡和标注困难的问题,进而提高病灶检测深度学习算法的泛化能力。
[0107]
图9给出了本技术实施例提供的一种病灶定向合成装置的结构示意图。参考图9,该病灶定向合成装置包括病灶合成模块31、组织融合模块32和病灶替换模块33。
[0108]
其中,所述病灶合成模块31,用于基于已标注病灶的原始病灶确定原始病灶组,并依据所述原始病灶组合成新病灶;所述组织融合模块32,用于获取ct序列的正常组织,并将所述新病灶和所述正常组织进行融合,以得到融合病灶组织;所述病灶替换模块33,用于利用所述融合病灶组织对所述ct序列中的所述正常组织进行替换,以得到合成病灶序列。
[0109]
上述,通过将原始病灶组合成新病灶,并在ct序列中选择正常组织,将正常组织与新病灶进行融合得到融合病灶组织,然后利用融合病灶组织替换掉ct序列中的正常组织,以得到合成病灶序列,合成病灶序列中的融合病灶组织由不同原始病灶合成得到,有效增
加病灶图像的多样性,通过不同原始病灶组的定向合成,有效实现在病灶级别上的数据扩增,并且通过将正常组织与新病灶进行融合,得到的融合病灶组织更逼真,将合成病灶序列用于深度学习算法模型的训练,有效提高深度学习算法模型精准度,并提高深度学习算法模型的泛化能力。
[0110]
在一个可能的实施例中,所述病灶合成模块31具体用于:
[0111]
对已标注病灶的原始病灶进行分布统计,并根据分布统计结果在所述原始病灶中选择原始病灶组;
[0112]
对所述原始病灶组进行尺寸归一化,并依据所述原始病灶组合成新病灶。
[0113]
在一个可能的实施例中,所述病灶合成模块31在对已标注病灶的原始病灶进行分布统计,并根据分布统计结果在所述原始病灶中选择原始病灶组时,具体包括:
[0114]
基于已标注病灶的原始病灶所对应的原始病灶标注,对所述原始病灶进行分布统计;
[0115]
基于分布统计结果,依照设定的标注分布范围在所述原始病灶中随机选择原始病灶组。
[0116]
在一个可能的实施例中,所述病灶合成模块31在对所述原始病灶组进行尺寸归一化时,具体包括:
[0117]
基于所述原始病灶组中的所述原始病灶所对应的原始病灶尺寸,在所述原始病灶尺寸之间随机确定归一化尺寸;
[0118]
按照所述归一化尺寸对所述原始病灶进行尺寸归一化。
[0119]
在一个可能的实施例中,所述病灶合成模块31在依据所述原始病灶组合成新病灶时,具体包括:
[0120]
按照贝塔分布将所述原始病灶组合成新病灶。
[0121]
在一个可能的实施例中,所述病灶合成模块31在按照贝塔分布将所述原始病灶组合成新病灶时,具体包括:
[0122]
按照贝塔分布确定合成参数,基于所述合成参数确定所述原始病灶组中各个所述原始病灶的合成比例;
[0123]
基于所述合成比例和所述归一化尺寸,将所述原始病灶组合成新病灶。
[0124]
在一个可能的实施例中,所述新病灶的合成公式为:
[0125]
patch_new=λ*patch1+(1-λ)*patch2
[0126]
其中,patch_new为新病灶,patch1和patch2为两个原始病灶,λ为合成参数,λ的定义域为[0,1],并且λ服从贝塔分布。
[0127]
在一个可能的实施例中,所述贝塔分布的形状参数对应设置为所述贝塔分布所对应的分布形状为u形。
[0128]
在一个可能的实施例中,所述组织融合模块32具体用于:
[0129]
依据所述新病灶的病灶尺寸和三维高斯函数,生成高斯掩码;
[0130]
随机获取ct序列的正常组织,并利用所述高斯掩码对所述新病灶和所述正常组织进行融合,以得到融合病灶组织。
[0131]
在一个可能的实施例中,所述三维高斯函数的计算公式为:
[0132][0133]
其中,f为三维高斯函数,分别为三维高斯函数三个维度方向的方差,x、y、z表示到中心点距离,k为缩放系数。
[0134]
在一个可能的实施例中,所述三维高斯函数三个维度方向的方差各向不同。
[0135]
在一个可能的实施例中,所述三维高斯函数三个维度方向的标准差以所述新病灶的病灶尺寸为基数随机生成。
[0136]
在一个可能的实施例中,所述三维高斯函数各维度方向的标准差的计算公式为:
[0137][0138]
其中,di为病灶i方向的直径,t1和t2为所取的最小和最大值。
[0139]
在一个可能的实施例中,所述组织融合模块32在利用所述高斯掩码对所述新病灶和所述正常组织进行融合,以得到融合病灶组织时,具体包括:
[0140]
基于所述高斯掩码确定所述新病灶和所述正常组织所对应的融合比例;
[0141]
按照所述融合比例对所述新病灶和所述正常组织进行融合,以得到融合病灶组织。
[0142]
在一个可能的实施例中,所述融合病灶组织的融合公式为:
[0143]
patch_m=gmask*patch_new+(1-gmask)*patch0
[0144]
其中,patch_m为融合病灶组织,gmask为高斯掩码,patch_new为新病灶,patch_0为正常组织。
[0145]
在一个可能的实施例中,所述装置还包括第一标注模块,所述第一标注模块用于:
[0146]
基于所述融合病灶组织对应的病灶尺寸和所述融合病灶组织在所述ct序列中的病灶位置,生成所述融合病灶组织对应的融合病灶标注。
[0147]
在一个可能的实施例中,所述装置还包括第二标注模块,所述第二标注模块用于:
[0148]
以所述高斯掩码的标准差为基准确定所述融合病灶组织对应的病灶尺寸;
[0149]
基于所述融合病灶组织对应的病灶尺寸和所述融合病灶组织在所述ct序列中的病灶位置,生成所述融合病灶组织对应的融合病灶标注。
[0150]
本技术实施例还提供了一种病灶定向合成设备,该病灶定向合成设备可集成本技术实施例提供的病灶定向合成装置。图10是本技术实施例提供的一种病灶定向合成设备的结构示意图。参考图10,该病灶定向合成设备包括:输入装置43、输出装置44、存储器42以及一个或多个处理器41;所述存储器42,用于存储一个或多个程序;当所述一个或多个程序被所述一个或多个处理器41执行,使得所述一个或多个处理器41实现如上述实施例提供的病灶定向合成方法。其中输入装置43、输出装置44、存储器42和处理器41可以通过总线或者其他方式连接,图10中以通过总线连接为例。
[0151]
存储器42作为一种计算设备可读存储介质,可用于存储软件程序、计算机可执行程序以及模块,如本技术任意实施例所述的病灶定向合成方法对应的程序指令/模块(例如,病灶定向合成装置中的病灶合成模块31、组织融合模块32和病灶替换模块33)。存储器
42可主要包括存储程序区和存储数据区,其中,存储程序区可存储操作系统、至少一个功能所需的应用程序;存储数据区可存储根据设备的使用所创建的数据等。此外,存储器42可以包括高速随机存取存储器,还可以包括非易失性存储器,例如至少一个磁盘存储器件、闪存器件、或其他非易失性固态存储器件。在一些实例中,存储器42可进一步包括相对于处理器41远程设置的存储器,这些远程存储器可以通过网络连接至设备。上述网络的实例包括但不限于互联网、企业内部网、局域网、移动通信网及其组合。
[0152]
输入装置43可用于接收输入的数字或字符信息,以及产生与设备的用户设置以及功能控制有关的键信号输入。输出装置44可包括显示屏等显示设备。
[0153]
处理器41通过运行存储在存储器42中的软件程序、指令以及模块,从而执行设备的各种功能应用以及数据处理,即实现上述的病灶定向合成方法。
[0154]
上述提供的病灶定向合成装置、设备和计算机可用于执行上述任意实施例提供的病灶定向合成方法,具备相应的功能和有益效果。
[0155]
本技术实施例还提供一种包含计算机可执行指令的存储介质,所述计算机可执行指令在由计算机处理器执行时用于执行如上述实施例提供的病灶定向合成方法。
[0156]
存储介质——任何的各种类型的存储器设备或存储设备。术语“存储介质”旨在包括:安装介质,例如cd-rom、软盘或磁带装置;计算机系统存储器或随机存取存储器,诸如dram、ddr ram、sram、edo ram,兰巴斯(rambus)ram等;非易失性存储器,诸如闪存、磁介质(例如硬盘或光存储);寄存器或其它相似类型的存储器元件等。存储介质可以还包括其它类型的存储器或其组合。另外,存储介质可以位于程序在其中被执行的第一计算机系统中,或者可以位于不同的第二计算机系统中,第二计算机系统通过网络(诸如因特网)连接到第一计算机系统。第二计算机系统可以提供程序指令给第一计算机用于执行。术语“存储介质”可以包括可以驻留在不同位置中(例如在通过网络连接的不同计算机系统中)的两个或更多存储介质。存储介质可以存储可由一个或多个处理器执行的程序指令(例如具体实现为计算机程序)。
[0157]
当然,本技术实施例所提供的一种包含计算机可执行指令的存储介质,其计算机可执行指令不限于如上所述的病灶定向合成方法,还可以执行本技术任意实施例所提供的病灶定向合成方法中的相关操作。
[0158]
上述实施例中提供的病灶定向合成装置、设备及存储介质可执行本技术任意实施例所提供的病灶定向合成方法,未在上述实施例中详尽描述的技术细节,可参见本技术任意实施例所提供的病灶定向合成方法。
[0159]
上述仅为本技术的较佳实施例及所运用的技术原理。本技术不限于这里所述的特定实施例,对本领域技术人员来说能够进行的各种明显变化、重新调整及替代均不会脱离本技术的保护范围。因此,虽然通过以上实施例对本技术进行了较为详细的说明,但是本技术不仅仅限于以上实施例,在不脱离本技术构思的情况下,还可以包括更多其他等效实施例,而本技术的范围由权利要求的范围决定。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1