一种颜色保真的功能影像体绘制方法
1.本发明涉及一种医学影像数据可视化分析方法,特别涉及一种颜色保真的功能影像体绘制方法,属于医学影像处理领域。
背景技术:
2.随着核医学技术的发展,继ct、mri等解剖结构影像(structural image) 之后,现代医学影像学又为临床诊断提供了spect、pet等功能影像(functionalimage)。医学功能影像是通过分子成像技术得到的,具体的成像过程是:以放射性同位素为示踪剂,将其注射入人体内并使其浓聚在被测器脏上,通过对示踪剂发射出的射线进行图像重建,从而得到影像方式的检测结果。病变组织因其代谢异常会表现出不同于正常组织的示踪剂摄取能力,从而产生不同于正常组织的成像效果。这种成像方法将组织的病理学检查延伸为生化显示,有助于疾病的早期诊断与治疗。在当前常用的功能影像中,spect可反映生命肌体的血液流量和代谢状态,pet可提供高精度的病变组织的生理代谢状态。
3.目前,功能影像数据的采集有二维和三维两种形式。由于散射的原因,三维采集得到影像分辨率通常较低。为了获得分辨率较高的影像数据,一般采用二维采集方式,其结果是影像切片系列。在这种情况下,观察者需要通过查看多张切片来了解被研究个体的信息。尽管切片提供了详细的二维影像,但医学研究的对象实际上是三维个体。观察者面临从二维切片构造或想象三维形态的挑战。
4.为应对这一挑战,可以将二维切片按顺序堆叠成三维体数据,然后采用体绘制技术对其进行可视化处理,直接显示其内部的三维形态结构。此时,如果切片数据是未经伪彩色映射的灰度图像,那么像素值表示的是实际功能上的代谢能力,其体数据可视化结果反应的就是实际的功能代谢情况。但是,在实际应用中,为了更好地观察组织的代谢信息,通常会对切片数据进行伪彩色映射,因此目前常见的pet、spect等功能影像都是彩色的。在这种情况下,对二维切片序列进行三维可视化处理,将面临颜色失真的问题。因为体绘制技术只能处理标量体数据,而伪彩色图像并不是标量数据,如果按照常规的处理策略,将彩色图像其转换为灰度图像,必然会丢失颜色信息。虽然体绘制技术可以通过颜色传输函数对绘制结果重新着色,但是这种着色在没有足够先验知识的情况下,无法与原来二维切片的颜色映射保持一致,而出现颜色失真。颜色失真不仅会增加观察者的分析难度,还可能使观察者产生错觉,甚至误导观察者。
5.为解决对彩色功能影像切片序列进行三维体绘制时面临的颜色失真问题,本发明提出一种颜色保真的功能影像体绘制方法。在给定功能态医学影像彩色切片序列的情况下,该方法首先根据切片像素的颜色和亮度值,对切片像素进行聚类;然后根据像素的类别索引值构造与切片序列对应的三维体数据,同时根据类别索引和聚类中心的关系构造颜色传输函数;最后设置不透明度传输函数,结合所构造的颜色传输函数,对所构造的三维体数据进行体绘制,得到保留原有功能影像颜色映射信息的体绘制结果。该方法不仅通过体绘制直观展示了二维功能影像切片系列所包含的三维形态结构信息,还通过颜色保真提供了
准确的功能代谢信息,有助于提高功能影像的可视化分析效率。
技术实现要素:
6.为解决对彩色功能影像切片序列进行三维体绘制时面临的颜色失真问题,本发明提出一种颜色保真的功能影像体绘制方法。
7.本发明的目的是通过以下技术方案实现的。
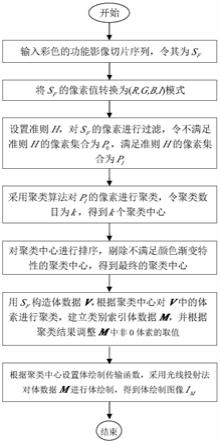
8.一种颜色保真的功能影像体绘制方法,包括以下步骤:
9.步骤1:输入彩色的功能影像切片序列,令其为s
f
。
10.步骤2:将彩色切片序列的像素值转换为(r,g,b,i)模式,其中r、g、b是原像素值的颜色分量,i是原像素值的亮度值。
11.步骤3:设置过滤准则h,对切片序列的像素进行过滤,令不满足准则h 的像素集合为p0,满足准则h的像素集合为p
i
。
12.步骤4:采用聚类算法对p
i
的像素进行聚类,令聚类数目为k,得到的聚类中心为c1(r1,g1,b1,i1),c2(r2,g2,b2,i2),...,c
k
(r
k
,g
k
,b
k
,i
k
)。
13.步骤5:对聚类中心c1(r1,g1,b1,i1),c2(r2,g2,b2,i2),...,c
k
(r
k
,g
k
,b
k
,i
k
)进行排序整理,具体计算过程如下:
14.步骤5.1:从k个聚类中心中选出亮度值最小的作为已排序的聚类中心队列 q
s
的队首,记为q
s1
(r
s1
,g
s1
,b
s1
,i
s1
);
15.步骤5.2:计算所有未排序的聚类中心与q
s
队尾元素的欧式距离,将距离最小的聚类中心作为队尾加入到q
s
;
16.步骤5.3:重复步骤5.2,直到k个聚类中心都加入到q
s
;
17.步骤5.4:按顺序对q
s
表示的颜色进行可视化,剔除不符合颜色渐变特性的聚类中心,得到新的长度为k
′
的聚类中心队列q
s
′
。
18.步骤6:将切片序列按顺序堆叠成三维体数据v,v中每个体素都是一个四元组(r,g,b,i)。
19.步骤7:按照步骤5得到的聚类中心,对v中每个体素进行分类,建立与之对应的类别索引体数据m,具体方法是:如果v的体素值不满足准则h,则令其类别索引值为0,否则其类别索引值为与其距离最近的聚类中心的类别索引值。
20.步骤8:调整体数据m中非0体素的取值,具体方法是:令第i个体素的原有值m
i
=m,按照下面的公式将m
i
修改为
[0021][0022]
其中d
mi
是v中与m
i
对应的体素v
i
到第m个聚类中心的距离,d
mmax
和d
mmin
分别是v中所有属于第m类的体素与第m个聚类中心的最大距离和最小距离。
[0023]
步骤9:根据聚类中心的颜色值和类别索引,构造体绘制颜色传输函数,并根据实际需求设定体绘制不透明度传输函数,然后采用光线投射法对体数据m 进行体绘制,得到体绘制结果图像i
m
。
[0024]
有益效果
[0025]
本发明所述的一种颜色保真的功能影像体绘制方法,与现有技术和方法相比,具有以下几个方面的优势和特点:
[0026]
(1)本发明方法依据像素的颜色分量和亮度信息对像素进行聚类,聚类中心包含了功能影像的颜色和亮度信息。在对类别索引体数据m进行体绘制时,采用根据类别索引和聚类中心构造的颜色传输函数,可以使体绘制结果的颜色信息与原功能影像的颜色信息保持一致。
[0027]
(2)本发明方法对原始功能影像的像素进行了聚类,然后根据类别索引构造了一个新的体数据m,再对m进行体绘制。新的体数据m与类别信息相关,可以在体绘制过程很方便地根据类别信息设置不透明度传输函数,选出重要的或者是感兴趣的代谢信息进行可视化显示。
[0028]
(3)本发明方法以颜色保真的方式将功能影像切片序列的分析从二维空间移入到三维空间,使用户不仅可以直接感知三维空间的形态结构信息,还通过颜色保真,使用户可以像查看二维切片那样,通过颜色编码获取准确的代谢信息,有助于提高功能影像的可视化分析效率。
附图说明
[0029]
图1一种颜色保真的功能影像体绘制方法的处理流程;
[0030]
图2输入切片序列s
f
的部分切片示例;
[0031]
图3与图2所示的输入切片一一对应的重构切片示例;
[0032]
图4(a)原始聚类中心的可视化结果;
[0033]
图4(b)排序后的聚类中心的可视化结果;
[0034]
图4(c)最终采用的聚类中心的可视化结果;
[0035]
图5体绘制颜色传输函数;
[0036]
图6体绘制不透明度传输函数;
[0037]
图7s
f
经本发明方法处理后的结果图像,其中(a)、(b)和(c)是分别从不同角度观察到的视图。
[0038]
图8s
f
经灰度变换后再进行三维体绘制的结果图像;
[0039]
图9pet
‑
fdg彩色切片序列经本发明方法处理后的结果,其中(a)为体绘制颜色传输函数和不透明度传输函数,(c)、(d)和(e)是分别从不同角度观察到的带有裁剪截面的体绘制结果图像,(b)是裁剪截面对应的原始输入切片。
[0040]
具体实施方法
[0041]
下面结合附图和实施例阐述本发明的具体实施方式。
[0042]
图1给出了本发明所述的一种颜色保真的功能影像体绘制方法的处理流程,其主要步骤如下:
[0043]
步骤s1:输入彩色的功能影像切片序列,令其为s
f
。
[0044]
本实施例从harvard medical school公开的the wholebrain atlas数据集 (http://www.med.harvard.edu/aanlib/home.htm)中,选取了一个胶质瘤患者的脑部spect
‑
t1切片序列作为输入,令其为s
f
。从s
f
中提取的三个不同位置的切片作为示例,分别如图2中的(a)、(b)和(c)所示。
[0045]
步骤s2:将彩色切片序列的像素值转换为(r,g,b,i)模式,其中r、g、b是原像素值的颜色分量,i是原像素值的亮度值。
[0046]
该步骤亮度值的计算方法有多种,常用的比较简单的有如下几种:
[0047]
i=max(r,g,b),
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(1)
[0048][0049][0050]
除此之外,还可以将基于rgb颜色模型的像素值转换为hsi颜色模型的像素值,然后取其中的亮度值i。本实施例采用上面的公式(3)计算像素的亮度值。
[0051]
步骤s3:设置过滤准则h,对切片序列的像素进行过滤,令不满足准则h 的像素集合为p0,满足准则h的像素集合为p
i
。
[0052]
设置过滤准则h是原因是:功能影像的背景区域对功能代谢信息的分析没有太大帮助。对背景区域或类似背景的像素进行过滤可以提高后续分析的效率。
[0053]
在本实施例中,过滤准则h是根据像素的亮度值设置的。此处使用的准则 h是i≥0.1
×
i
max
,其中i是像素的亮度值,i
max
是s
f
中所有像素的最大亮度值。
[0054]
步骤s4:采用聚类算法对p
i
的像素进行聚类,令聚类数目为k,得到的聚类中心为c1(r1,g1,b1,i1),c2(r2,g2,b2,i2),...,c
k
(r
k
,g
k
,b
k
,i
k
)。
[0055]
该步骤的任务是对过滤后的像素进行聚类。可以采用常见的聚类算法来完成该任务,例如k
‑
means和k
‑
means++等聚类算法。
[0056]
k
‑
means和k
‑
means++都是经典的基于距离的聚类算法,采用距离作为相似性的评价指标,两个对象的距离越近,其相似性就越大。两者的不同之处在于, k
‑
means聚类算法的初始聚类中心是随机选择的;而k
‑
means++聚类算法除第一个初始聚类中心是随机选择的外,其他初始聚类中心都是按照使初始聚类中心之间的距离尽可能远的原则选取的。
[0057]
鉴于聚类结果与初始聚类中心密切相关,本实施例选择k
‑
means++算法进行聚类,其中采用的距离测度为欧式距离,具体的聚类过程如下:
[0058]
步骤s4
‑
1:从p
i
中随机选择一个像素,以其颜色分量和亮度值构造第一个聚类中心c1(r1,g1,b1,i1);
[0059]
步骤s4
‑
2:采用欧式距离公式计算p
i
中的像素与已有的聚类中心的最短距离,选择距离值最大的像素的颜色分量和亮度值构造下一个聚类中心;
[0060]
步骤s4
‑
3:重复步骤s4
‑
2,直到选出k个聚类中心;
[0061]
步骤s4
‑
4:计算p
i
中的像素与所有聚类中心的欧式距离,寻找距离最小的聚类中心,并将其划分到该聚类中心所属的类别;
[0062]
步骤s4
‑
5:用均值法计算出每个类的新聚类中心;
[0063]
步骤s4
‑
6:计算每对新旧聚类中心之间的欧式距离,如果有某对新旧聚类中心的距离大于阈值t,则重复步骤s4
‑
4到s4
‑
5,直到所有类的新旧聚类中心的距离小于或等于t。
[0064]
在本实施例的实施过程中,聚类数目k=30,阈值t=0.0001,最后得到30 个聚类中心。依次对这30个聚类中心表示的颜色进行可视化,得到原始聚类中心的可视化结果如图4(a)所示。
[0065]
步骤s5:对聚类中心c1(r1,g1,b1,i1),c2(r2,g2,b2,i2),...,c
k
(r
k
,g
k
,b
k
,i
k
)进行排序整理,具体过程如下:
[0066]
步骤s5
‑
1:从k个聚类中心中选出亮度值最小的作为已排序的聚类中心队列q
s
的队首,记为q
s1
(r
s1
,g
s1
,b
s1
,i
s1
);
[0067]
步骤s5
‑
2:计算所有未排序的聚类中心与q
s
队尾元素的欧式距离,将距离最小的聚类中心作为队尾加入到q
s
;
[0068]
步骤s5
‑
3:重复步骤s5
‑
2,直到k个聚类中心都加入到q
s
;
[0069]
步骤s5
‑
4:按顺序对q
s
表示的颜色进行可视化,剔除不符合颜色渐变特性的聚类中心,得到新的长度为k
′
的聚类中心队列q
s
′
。
[0070]
在本实施例中,排序后的聚类中心按颜色可视化显示的结果如图4(b)所示。显然,q
s22
(r
s22
,g
s22
,b
s22
,i
s22
)和q
s30
(r
s30
,g
s30
,b
s30
,i
s30
)不符合颜色渐变的要求,将被剔除。因此,聚类中心队列q
s
′
的长度k
′
=28,其可视化结果如图4(c)所示。
[0071]
从图4(a)可看到,由步骤4得到的聚类中心的颜色是无序的。如果直接用这些聚类中心对体数据v中的体素进行分类,会出现邻近体素颜色属性值比较接近而类别索引值却相差比较大的情况。这将导致后续体绘制结果无法通过颜色正确反映体素取值的变化。因此,本发明方法对聚类中心进行了排序,并剔除了不满足颜色渐变特性的聚类中心。
[0072]
步骤s6:将切片序列按顺序堆叠成三维体数据v,v中每个体素都是一个四元组(r,g,b,i)。
[0073]
步骤s7:按照步骤5得到的聚类中心,对v中每个体素进行分类,建立与之对应的类别索引体数据m,具体方法是:如果v的体素值不满足准则h,则令其类别索引值为0,否则其类别索引值为与其距离最近的聚类中心的类别索引值。
[0074]
在本实施例中,为了检查聚类结果的准确性,根据索引体数据m重构了输入切片序列。重构输入切片的具体过程是:从m提取切片像素的索引值,根据索引值从队列q
s
′
中获取相应聚类中心的颜色值,将其作为切片像素的颜色。图 3给出了与图2所示的输入切片一一对应的重构切片示例。显然,按照这种方法重构的输入切片序列,在视觉感知上与原始输入切片序列没有明显差异。
[0075]
步骤s8:调整体数据m中非0体素的取值,具体方法是:令第i个体素的原有值m
i
=m,按照下面的公式将m
i
修改为
[0076][0077]
其中d
mi
是v中与m
i
对应的体素v
i
到第m个聚类中心的距离,d
mmax
和d
mmin
分别是v中所有属于第m类的体素与第m个聚类中心的最大距离和最小距离。
[0078]
在经过该步骤处理后,体数据m中原取值为m的体素,其值将被调整到区间[m
‑
1,m]内。该调整可以使最终体绘制结果的颜色变化是连续的,从而呈现更好的视觉效果。实际上,当k值较大时,由于分类精度已经比较高,省略该步骤也是可以的。
[0079]
步骤s9:根据聚类中心的颜色值和类别索引,构造体绘制颜色传输函数,并根据实际需求设定体绘制不透明度传输函数,然后采用光线投射法对体数据 m进行体绘制,得到体绘制结果图像i
m
。
[0080]
在本实施例中,根据聚类中心的颜色值和类别索引构造的颜色传输函数如图5所示,根据观察需要设置的不透明度传输函数如图6所示,最终得到的体绘制结果如图7所示,其中(a)、(b)和(c)分别是从不同角度观察到的结果视图。为了展示本发明方法保留功能影像切片颜色的能力,在图7的体绘制结果中增加了一个裁剪截面,以便在截面上直接观察到与图2(b)对应的切片视图。对比图 7(a)和图2(b),可以看到两者的颜色映射是一致的。用户可以像观察二维切片那样,根据颜色映射解码三维空间结构上的代谢信息。
[0081]
为了说明本发明方法的优势和特征,将本发明方法与常规的功能影像体绘制方法进行了对比。常规方法一般将s
f
中的彩色切片转换为灰度图,然后按顺序堆叠成体数据,最后用体绘制技术对体数据进行可视化。对于本实施例的输入数据,采用常规方法进行处理后,得到的体绘制结果如图8所示。图8的观察角度与图7(a)是一致的。对比两者可以看到:图7(a)中与原切片一致的大块红色肿瘤区域,在图8中表现为蓝色,类似于周边正常的组织区域。这种颜色失真将给后续数据分析工作带来困扰甚至是误导。相比之下,本发明方法很好地规避了这个问题,体绘制结果的颜色信息跟原始输入切片的颜色信息是一致的。
[0082]
为了说明本发明方法的可行性和通用性,采用pet
‑
fdg彩色切片序列对其做了进一步的测试。其中采用的像素过滤准则h为i≥0.2
×
i
max
,聚类算法为 k
‑
means++,聚类数目k=40,阈值t=0.0001,得到的聚类中心都满足颜色渐变要求。在对新构造的体数据进行体绘制时,采用的颜色传输函数和不透明度传输函数如图9(a)所示。图9中的(c)、(d)和(e)是分别从不同角度观察到的体绘制结果图像。这些不同角度的视图,提供了重要的空间结构信息。为了验证聚类结果的准确性,图9(c)、(d)和(e)展示的都是带有裁剪截面的体绘制结果,裁剪截面对应的原始输入切片如图9(b)所示。显然,图9(c)中截面显示的功能代谢信息与图9(b)是一致的。因此,本发明方法对于pet
‑
fdg彩色切片序列的处理是有效的。
[0083]
上述步骤及实例说明了本发明所述的一种颜色保真的功能影像体绘制方法的全部过程。
[0084]
应该理解的是,本实施方式只是本发明实施的具体实例,不应该是本发明保护范围的限制。在不脱离本发明的精神与范围的情况下,对上述内容进行等效的修改或者变更均应包含在本发明所要求保护的范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1