基于不确定度校准和区域分解的端到端染色体分割方法
本发明涉及一种基于不确定度校准和区域分解的端到端染色体分割方法,属于图像识别。
背景技术:
1、由于染色体的数目异常或结构畸变均可能引发如智力障碍、先天性畸形等遗传类疾病,因此目前需要通过对染色体进行观测,从而对胎儿健康进行预测,目前主要通过孕期产前筛查和产前诊断等加以预防。羊水穿刺染色体核型分析作为目前产前诊断的金标准,是将样本羊水细胞分裂中期的染色体显带成像后进一步分割、排序以识别异常。然而,细胞中染色体游离柔性,多重叠交叉且形态多变,导致目前核型分析仍主要依赖专业人士,代价高昂,效率低下且极度依赖经验,亟需高效精确的智能辅助手段。
2、现有技术中存在的问题:染色体分割作为核型分析的首要任务,是后续分类和识别的基础,重点需要解决重叠交叉染色体的分割。和自然图像分割不同,染色体图像的分割存在如下几大大挑战:1)染色体非刚性。染色体是一种非刚性的物质,其多以弯曲状态游离存在于人体细胞核中,造成染色体的多姿态复杂性,给染色体分割任务带来困难。2)多簇性。由于染色体是以游离的状态分布于细胞核中,这就导致染色体容易出现两条或者多条的簇集,造成染色体接触、重叠。3)标记数不足。染色体图像由于其私密性,缺少标注的数据集。
3、现有技术中,申请号:202210548618.6公开一种重叠染色体分割方法,对重叠染色体图像进行边缘提取,并获得所有重叠染色体的边缘轮廓,将所述的边缘轮廓点全部加入到待选切割点集合;对重叠染色体图像提取其骨架、骨架端点和骨架交叉点;根据获取的重叠染色体边缘轮廓和染色体骨架及骨架端点、交叉点确定重叠染色体的切割点;根据所述切割点对所述重叠染色体进行自动分割。其利用重叠染色体的重叠特征通过结合染色体的轮廓和骨架的关系,将所有轮廓点看作切割点并作为候选集合,通过欧氏距离结合染色体骨架精准定位最佳切割点。但是其算力需求大,而且对图像的精细程度要求高,并不能满足快速高校的分割需求。
技术实现思路
1、针对上述存在的技术不足,提供一种解决重叠交叉染色体的分割,能够更好的应对交叉、重叠染色体的漏检和定位不准的问题,能够更好地重叠交叉染色体引起的语义歧义问题的基于不确定度校准和区域分解的端到端染色体分割方法。
2、为实现上述技术目的,本发明提供一种基于不确定度校准和区域分解的端到端染色体分割方法,其步骤如下:
3、步骤1、首先对需要进行端到端染色体分割的染色体图片进行预处理:
4、步骤2、通过迁移学习方法在染色体分类数据集上预训练骨干网络resnet101提取染色体基本特征;
5、步骤3、引入不确定度分支输出不确定度学习检测框的可靠性,利用不确定度修正染色体检测框的位置来实现对染色体更加精确的定位,
6、步骤4、最后利用分解因子将区域生成网络rpn产生的所有建议框roi分解、自适应融合,减轻重叠染色体分割时产生的语义歧义,最终得到分割更完整的染色体。
7、进一步,步骤1中预处理的具体步骤为:
8、1a、将染色体图像放缩到800×1088×3像素;
9、1b、利用labelme软件根据g带染色体数据集对染色体图像中的染色体g带信息进行标注,包括染色体的定位、类别、分割标签向量信息。
10、进一步,步骤2具体步骤如下:
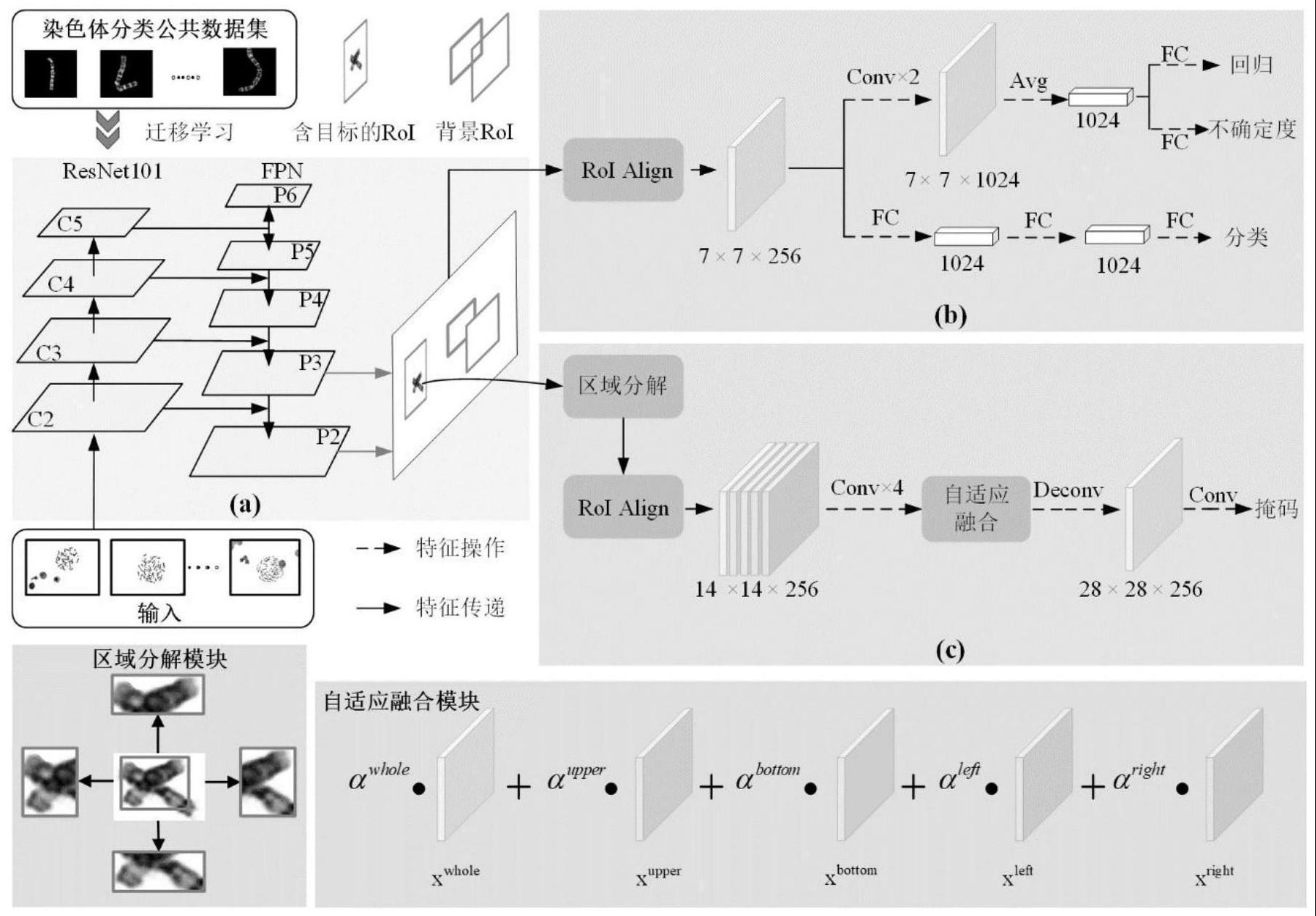
11、2a、将原始染色体图片作为输入送入主干特征提取网络resnet101,特征提取后利用长宽压缩了两次、三次、四次、五次的特征图c2,c3,c4,c5进行后续特征金字塔的构造,其中resnet101网络为通过迁移学习在公共染色体分类公共数据集上预训练微调过的骨干网络,通过迁移学习得到的骨干网络相比在imagenet自然图像上训练骨干网络可以提取染色体的基本特征,有利于后续针对染色体设计的分割网络;
12、2b将四张特征图c2,c3,c4,c5送入特征金字塔fpn以多尺度提取特征,得到不同尺度的四张特征图p2,p3,p4,p5;计算labelme软件标注了染色体g带信息中所有单条染色体的面积得出染色体为小目标的结论,仅需要特征金字塔fpn的多尺度作用在小尺度上,因此只选择p2,p3层作为特征层输出即可;
13、2c将特征金字塔fpn的输出的特征图p2,p3,p4,p5送入区域生成网络rpn进行第一阶段的回归和分类任务,得到的染色体定位框roi,由于染色体是小目标,只需要输出特征图p2,p3,利用染色体定位roi到特征图p2,p3上进行roialign截取出每个roi对应的特征图。
14、进一步,其特征在于步骤3具体步骤如下:
15、3a截取出每个roi对应的特征图分别接回归分支和分类分支,其中在回归分支设置两个卷积层conv,在分类分支设置两个全连接层fc;
16、3b在回归分支额外设计不确定度分支,利用不确定度分支输出的不确定度建立高斯分布模型,染色体目标对应所有候选框的回归参数平均值作为高斯分布的均值,同时将对应候选框不确定度分支的预测值经过softmax操作后得到的处于[0,1]之间的值作为高斯分布的标准差,建立每一条染色体对应的所有候选框的高斯分布;在训练过程中,使经过回归后被认定可以覆盖染色体的候选框的分布接近真实框的分布计算损失函数。
17、进一步,步骤4具体步骤如下:
18、4a将roi区域多次分割以避免由两条及两条以上染色体重叠引起的语义歧义,具体做法为:在roialign之前将roi根据人工预设的分解因子s分解为上下左右四个区域,分解因子s定义为分解区域长/宽与原图长/宽的比值;
19、4b分解后各子区域分别通过roialign提取14×14特征图,记为分别代表经过区域分解后得到的上下左右四部分特征图,ij为该特征图的点(i,j)处;同时提取roi的完整特征得到特征图
20、4c利用权值对各个区域和完整区域的特征图进行自适应融合,得到最终的特征图,权值分别为各子区域权值,由特征图经1×1卷积、再通过softmax映射为权值,自适应融合的公式如下:
21、
22、式中表示1×1卷积后得到的特征图,e为指数操作函数。
23、一种基于不确定度校准和区域分解的端到端染色体分割方法使用的ucrd-net网络,ucrd-net包括染色体特征提取模块fe、不确定度的染色体检测框校准模块uc和基于区域分解的染色体分割模块rd;其中resnet101和fpn均属于染色体特征提取模块fe;
24、染色体特征提取模块fe,利用公共g带染色体分类数据集完成resnet101骨干网络预训练以提取基本特征,再结合端到端检测时单条染色体的小目标特点,选择底层特征图及相应大小的锚框增强特征;
25、不确定度的染色体检测框校准模块uc,根据分类回归在特征学习中关注特征不同的特点设计双分支头分别用于分类回归任务,再增加不确定度分支输出不确定度以修正定位误差较大的检测框,减少由于检测框的不精确而导致的分割掩码缺失;
26、基于区域分解的染色体分割模块rd,将roi分解为四个方向的位置区域而后经过roi align提取四个位置方向的特征,再将四个方向特征和整个roi的特征自适应融合,通过多方向的推理以强化roi内待分割染色体的语义响应,避免roi内其他染色体造成的语义歧义。
27、进一步,对ucrdnet网络训练,设定初始学习率le-5,网络优化采用随机梯度下降sgd法。
28、一种使用ucrd-net网络的染色体分割预测方法,其步骤如下:
29、4a首先输入染色体图片经过ucrd-net的主干网络resnet101和特征金字塔fpn得到特征图后经过区域生成网络得到roi;
30、4b通过roialign操作后首先进入回归分支和分类分支进行定位和分类;
31、此时,经过不确定度分支预测出不确定度,在非极大值抑制阶段修正候选框的排序分数,原始排序分数为分类置信度,通过下式进行修正排序分数:
32、score=λ×(cls_score)+(1-λ)×(1-η)(2)
33、式中,λ为权重参数,本文通过网格搜索设置λ=0.3,score为校准后的分数,cls-score表示不正确的置信度/分类置信度;η为候选框的输出不确定度。
34、4c从4b中得到的经非极大值抑制并修正后的候选框送入分割头进行分割,此时会对该候选框进行相应的分解和自适应融合以输出最后的分割结果。
35、有益效果:
36、1.本发明通过利用公共g带染色体数据集进行迁移学习,预训练调整后的resnet101网络,实现了在数据量较少的情况下有效提取染色体特征;
37、2.本发明定位校准策略能够更好的应对交叉、重叠染色体的漏检和定位不准的问题;
38、3.本发明提出的区域分解模块能够更好地重叠交叉染色体引起的语义歧义问题。
- 还没有人留言评论。精彩留言会获得点赞!