一种微生物培养过程中多种物质实时检测的方法
本发明涉及光谱检测分析,尤其涉及一种微生物培养过程中多种物质实时检测的方法。
背景技术:
1、生物制药领域,过程分析技术(pat)利用先进的分析手段实时监测和控制生物制药生产过程中的多个参数和指标。以确保产品的质量、一致性和生产效率,减少产品失败率并降低生产成本。拉曼检测技术作为一种典型的过程分析技术,以非侵入性、高灵敏度等特点在药物生产过程和质量分析具有广泛的应用。
2、2010年,众多学者利用拉曼技术结合化学计量学方法来实现大肠杆菌微生物的快速检测、分离和识别,同时也尝试采用更先进的拉曼成像技术来对微生物内的化学成分进行可视化分析,推动了该技术在微生物研究领域的应用。2011年nr abu-absi,bm kenty等首次实现了对生物反应器中多项参数葡萄糖、乳酸、铵、谷氨酰胺和存活细胞密度的同时监测。到目前为止,培养过程中如滴定度、16种蛋白氨基酸浓度等更多关键指标被证明可以通过拉曼技术结合化学计量学的方式实时监测。
3、2018年shuai he,yi mon ei kyaw研究团队提出了一种新的无标记定量测量细胞外蛋白激酶a(pka)活性的方法。通过对sers谱进行主成分分析(pca),研究团队成功识别到了725和1395cm-1处的两个拉曼峰,它们的比例强度变化反映了pka对kem的磷酸化程度,从而实现了对pka活性的定量测量。这证明了拉曼光谱技术在蛋白质检测方面的可行性,为实时定量研究奠定了基础。
4、偏最小二乘回归(plsr)是目前使用最为普遍的一种化学计量学算法,常用与多元统计分析,处理多变量数据之间的相关性和预测。plsr能够降低数据维度,在保留原始数据的信息的同时,把多个自变量x和因变量y之间的线性关系提取出来,并用少量的“潜在变量”来映射回归关系。plsr在满足朗伯-比尔定律的线性回归模型中效果表现优异,但最近研究表明在处理非线性问题以及多批次数据适用情况下导致的模型泛化能力表现不佳。由于训练的模型过于依赖于当前输入数据,使得模型鲁棒性没有取得令人的满意结果。
5、随着机器学习的发展,将其他回归算法如支持向量回归(svr)、随机森林回归(rfr)与神经网络回归(annr)等非线性算法结合拉曼光谱能够较好的克服上述线性模型的缺点。
6、生物多批次培养具有明显的特异性,提高模型的鲁棒性需要使用到不同批次的过程数据进行模型的训练。一定程度上数据集的丰富性扩大,模型的鲁棒性也会随之增长,但过多的数据会使得模型的准确性趋于低饱和。且由于间断采样导致数据灾难,数据集存在分布不均的偏差。部分的样本数量过多,使得模型拟合不准确而出现错误的预测结果。
7、深度网络中神经元之间的复杂联系,以及多层深度能够容纳海量的数据,使之成为更为理想的高参数回归模型。2012年卷积神经网络(cnn)结构alexnet,在imagenet图像分类大赛上获得第一名。cnn的局部连接和权值共享特性,减小了网络的参数数量和计算复杂度,同时增加模型的稳定性。cnn在分类和回归任务上都取得良好结果。
8、细菌培养过程中某一时刻的所有信息都分布在同一条拉曼谱图不同波数的峰位上。由于生物大分子之间的耦合效应以及生物荧光干扰,很难通过常规的谱图识别定位特征峰。
技术实现思路
1、针对现有技术中所存在的不足,本发明提供了一种微生物培养过程中多种物质实时检测的方法。其解决了现有技术中存在的微生物培养中物质实时检测不准确的问题。
2、本发明第一方面,提供一种微生物培养过程中多种物质实时检测的方法,包括以下步骤:
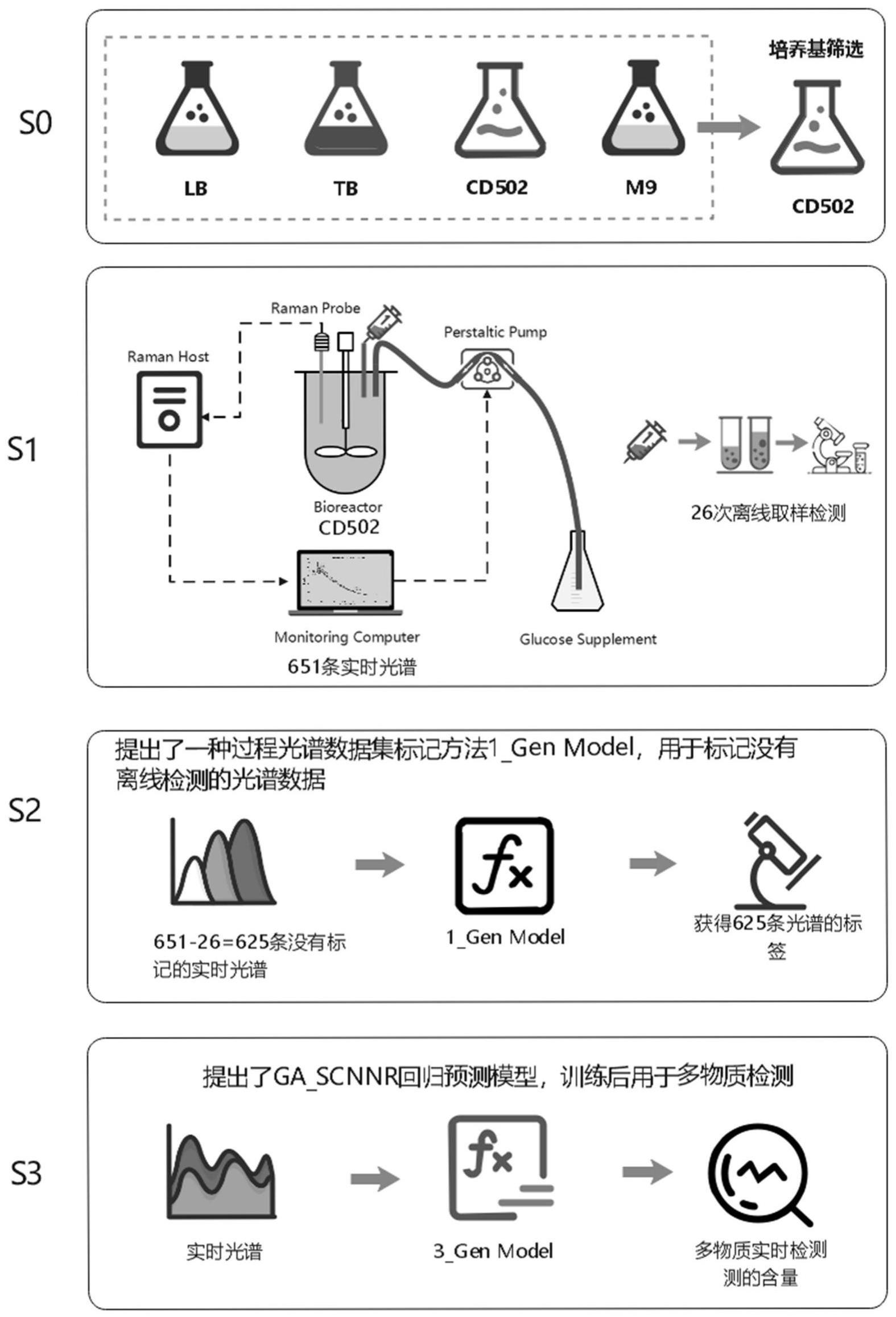
3、s0将微生物进行发酵培养,得到发酵样品;
4、s1建立数据集:在培养过程中连续采集发酵样品的拉曼光谱数据得到过程数据集;同时间断取样并检测发酵样品中物质浓度得到离线数据集;
5、s2标记数据集:根据离线数据集,将过程数据集划分为有标签的过程数据集和无标签的过程数据集,并对无标签的过程数据集进行标记,得到无标签的过程标记数据集;
6、s3建立回归模型:有标签的过程数据集与无标签的过程标记数据集共同组成半监督数据集,用来训练遗传算法-半监督卷积神经网络模型,建立拉曼光谱与物质浓度之间的回归模型。
7、本发明一实施例中,步骤s1中,连续采集发酵样品的拉曼光谱数据的方法包括:将拉曼光谱仪与发酵样品连接,实时检测发酵样品的拉曼光谱数据;
8、间断取样并检测发酵样品中物质浓度的方法包括:每间隔一定时间,采集发酵样品并检测吸光值和物质浓度;
9、优选间隔时间为1h;
10、优选物质包括葡萄糖、乳酸、铵离子、od600和目标蛋白。
11、本发明一实施例中,步骤s2中,根据离线数据集,将过程数据集划分为有标签的过程数据集和无标签的过程数据集的方法包括:将过程数据集中与取样时间对应的拉曼光谱数据划分为有标签的过程数据集,其余拉曼光谱数据划分为无标签的过程数据集;
12、优选地,对无标签的过程数据集进行标记的方法包括模型计算法和插值法;
13、更优选地,所述模型计算法为利用有标签的过程数据集训练完成的模型对无标签的过程数据集进行标记;
14、更优选地,所述插值法为利用三样条插值法对无标签的过程数据集进行标记。
15、本发明一具体实施例中,利用有标签的过程数据集训练完成模型的方法包括:对有标签的过程数据集进行扩充和预处理,用于训练plsr和svr模型;通过模型集成方式计算plsr和svr模型的回归参数,通过boosting模型的迭代方法得到1_gen模型;
16、优选地,所述扩充的方法包括:添加基线斜率、引入高斯噪声和比例数法;
17、优选地,所述预处理的方法包括利用基线校正法、savitzky-golay滤波法、标准正态变量变换法、归一化对拉曼光谱数据进行处理;
18、优选地,所述三样条插值法包括利用插值函数对过程数据进行标记,所述插值函数如式(1)所示:
19、
20、其中,si表示第i个点的插值函数值,xi表示第i个样本点,yi表示第i个样本点的标签值,s0表示插值函数左区间段点函数值,sn表示插值函数右区间段点函数值。
21、本发明一实施例中,步骤s3中,训练遗传算法-半监督卷积神经网络模型的方法包括:通过遗传算法对半监督数据集进行物质的特征波段提取,并对特征波段进行强化;构成训练样本集,用于训练半监督卷积神经网络模型。
22、本发明一具体实施例中,所述特征波段提取的方法包括:以目标函数的均方根误差(rmse)取反的方式作为适应度函数,利用遗传算法对有监督过程数据集的特征波段进行提取;
23、所述对特征波段进行强化的方法包括:采用横向三次样条插值的方式对特征波段进行强化,使之还原为原始光谱长度。
24、本发明一实施例中,还包括s4迁移学习:根据深度卷积神经网络,在训练完成的遗传算法-半监督卷积神经网络模型基础上,引入少量目标物质的拉曼光谱数据和离线数据,即得目标物质的半监督卷积神经网络模型;
25、优选地,所述目标物质为目标蛋白;
26、优选地,在训练完成的遗传算法-半监督卷积神经网络模型基础上,引入少量目标物质的拉曼光谱数据和离线数据的方法包括:在吸光值的半监督数据集预训练scnnr模型为基础,通过添加全连接层,引入少量的目标蛋白拉曼光谱数据集和离线数据集,即可得到目标蛋白的scnnr;
27、优选地,利用目的蛋白的数据集对预训练scnnr模型参数进行调整。
28、本发明第二方面,提供一种微生物培养过程中多种物质实时检测装置,包括:数据集标记模块、回归模型建立模块和迁移学习模块;
29、所述数据集标记模块用于标记过程数据集;
30、所述回归模型建立模块用于建立拉曼光谱与物质浓度之间的回归模型;
31、所述迁移学习模块用于建立少量目标物质的半监督卷积神经网络回归模型。
32、本发明第三方面,提供一种微生物培养过程中多种物质实时检测装置,其特征在于,包括处理器、存储器以及存储在所述存储器中且被配置为由所述处理器执行的计算机程序,所述处理器执行所述计算机程序时实现上述的微生物培养过程中多种物质实时检测的方法。
33、本发明第四方面,提供一种计算机可读存储介质,其特征在于,所述计算机可读存储介质包括存储的计算机程序,其中,在所述计算机程序运行时控制所述计算机可读存储介质所在设备执行上述的微生物培养过程中多种物质实时检测的方法。
34、本发明的技术原理为:
35、在发明以大肠杆菌培养过程中多种物质检测为目标,在深度学习模型cnn的基础上提出了更加具有适用性的遗传、半监督卷积神经网络回归模型(ga_scnnr),用于重组大肠杆菌培养的拉曼模型建立。
36、本发明使用遗传算法关联回归模型,以均方根误差(rmse)取反的方式作为适应度函数,实现了在同一光谱对不同检测物质进行特征位置划分,从2048个输入光谱变量(x1,x2,……,x2048)提取不同的最佳适应特征波段。
37、cnn的网络参数训练通常需要大量的数据,为克服采样数据集的不足,本发明分别使用了有标签的离线数据集和无标签的过程数据,两者共同构成半监督数据集来训练模型。由于离线检测数据的稀有,使用了添加基线斜率、引入高斯噪声和插值计算三种一维数据增强方式扩充标签数据集,强化了拉曼光谱信息与被测物质含量的关联性。因生物反应器的过程拉曼光谱采集是具有时间序列的,可通过时间序列模型回归、插值拟合等方式标记无标签数据集,从而更加细致地捕捉被检测物质的连续变化过程,契合实际情况中物质含量是模拟量的变化特点。
38、同时,ga_scnnr可通过迁移学习的方式增强模型的实际适应力,在保留第n-1批次训练完成的模型前提下,添加额外的全连接层,便能构成第n批次的新模型,以适应新的应用场景,形成(训练-应用-训练)的动态模型结构。具体来说,本发明在已完成训练的scnnr(od600)模型的基础上,添加了两个空白全连接层,分别包含128和64个神经元,构建了迁移学习模型。收集了20条目的蛋白参考数据,并通过数据增强技术将数据集扩大到320条,扩充了16倍。接着,使用目的蛋白数据集对预训练模型进行微调,进一步在目标任务上进行训练,调整模型的参数以适应该任务。通过以上方法,成功构建了适用于目的蛋白表达任务的迁移学习模型。该模型利用了scnnr(od600)模型的先验知识,并通过微调操作,在目标任务上取得了更好的性能和适应性。这种方法为解决目的蛋白表达任务中难以获取离线参考值的问题提供了一种有效的解决方案。
39、相比于现有技术,本发明具有如下有益效果:
40、(1)本发明提出了遗传半监督卷积神经网络(ga_scnnr)框架,以实时拉曼光谱为基础,在重组大肠杆菌培养过程中建立了多参数回归模型,可实现大量的无标记的过程光谱数据用于模型的训练,训练集样本从52个扩增到1302个,极大程度上提升了过程检测的实时性和鲁棒性。
41、(2)本发明提出来一种光谱标记方法,以过程谱标记方法以扩充模型的训练数据集,利用了大量无标签的过程光谱数据,使得模型有更大的数据集用于训练,在少量离线样本的前提下也能训练出高精度的模型。
42、(3)本发明针对不同的检测物质遗传算法的引入,可准确的在同一条拉曼光谱数据中解析出最适的特征位置,提升了多参数目标检测的准确性。
43、(4)本发明使用了深度卷积神经网络利用迁移学习,能够在少量数据的情况下依然能训练出高精度的模型,在及其少量目标蛋白数据集的情况下,成功实现了目标蛋白表达的检测。
44、(5)本发明模型预测的实用性得到了验证,可用于指导培养过程的产量与趋势检测。
45、因此,与传统的化学计量学模型相比,ga_scnnr框架还拥有以下优势:cnn网络独特的卷积结构,可代替光谱数据预处理过程并且具有更高的精确度。实际的生物培养过程,检测参数和培养体系时常变化,ga_scnnr框架通过迁移学习拥有可扩展的能力,在保留之前训练的参数的前提下用少量样本下便可重新适应新的检测任务,为生物检测通用型模型的开发提供了新的思路。
- 还没有人留言评论。精彩留言会获得点赞!