一种基于多种群联合搜索的蛋白质构象空间优化方法与流程
本发明涉及生物信息学、计算机应用领域,尤其涉及的是一种基于多种群联合搜索的蛋白质构象空间优化方法。
背景技术:
二十世纪末以来,生命科学领域飞速发展,作为生物体内分布最广、功能最复杂的一类大分子,蛋白质受到尤为广泛的关注和研究。蛋白质的三维结构决定了其特定的生物功能,因此,获得蛋白质的三维结构对于了解其生物功能、疾病诊断和药物设计是至关重要的。
目前,获得蛋白质三维结构的途径有两种,第一种途径是实验方法直接测定蛋白质的三维结构,此类方法获得的蛋白质三维结构精度高,但是极其消耗时间和资源、对实验环境要求苛刻,并且不能对所有类型的蛋白质进行测定;第二种途径是利用计算机技术结合生物学信息根据蛋白质氨基酸序列预测蛋白质三维结构。由于通过实验测定蛋白质三维结构的方法存在局限,并且氨基酸序列测定的速度与蛋白质三维结构测定的速度之间的鸿沟不断扩大,利用计算机技术预测蛋白质三维结构已经成为获取蛋白质三维结构的重要手段。根据氨基酸序列预测蛋白质三维结构的方法主要分为同源建模法和从头预测法。其中从头预测法不依赖目标蛋白的同源信息,能够探索新型蛋白质的三维结构。目前比较成功的从头蛋白质结构预测方法有baker团队开发的rosetta和张阳团队开发的quark等。
根据氨基酸序列预测蛋白质三维结构实质上是在能量模型引导下的构象空间优化问题。蛋白质构象空间非常庞大,因此如何高效的探索有效的构象空间就显得尤为重要。目前的蛋白质构象空间优化方法存在对构象空间采样能力不足以及量函数不精确导致的引导方向偏差等问题,导致整体的预测精度不高。
因此,目前的构象空间优化方法存在搜索效率低和预测精度较低等问题,需要改进。
技术实现要素:
为了克服现有构象空间优化方法存在搜索效率低和预测精度较低的不足,为了提高构象空间的采样能力并缓解能量函数不精确所带来的不利影响,本发明提供一种基于多种群联合搜索的蛋白质构象空间优化方法。该方法利用种群间个体的信息交互结合片段组装技术增强对蛋白质构象空间的探测能力,利用多种群联合进化、汇合选择能量居中的个体组成新的种群,缓解能量函数不精确导致的引导方向偏差问题,从而提高整体的预测精度。
本发明解决其技术问题所采用的技术方案是:
一种基于差分进化局部扰动的蛋白质构象空间优化方法,所述方法包括以下步骤:
1)输入预测蛋白质的序列信息;
2)设置参数:种群规模np,辅助种群数量m,迭代次数g;
3)迭代rosetta协议第一、二阶段,生成初始种群p={p1,p2,...,pnp},其中pn表示种群p中的第n个个体,n∈{1,2,...,np};
4)设m=1,其中m∈{1,2,...,m};
5)迭代rosetta协议第一、二阶段,生成一个新种群
6)种群p独立进化,过程如下:
6.1)设g=1,其中g∈{1,2,...,g};
6.2)设n=1,其中n∈{1,2,...,np};
6.3)种群中个体信息交互,过程如下:
6.3.1)从种群p中随机选择三个与pn不同并且互不相同的个体pbase、pselect1、pselect2;
6.3.2)从个体pn、pselect1和pselect2中随机选择3个不同的9片段,分别替换个体pbase中相应的片段,生成新的个体pcombi;
6.4)从个体pcombi中随机选择一个位置执行一次9片段的片段组装,生成片段组装后的个体ptrial;
6.5)用rosettascore3能量函数计算ptrial和pn的能量etrial和etarget,按如下方式决定是否用ptrial替换种群中的个体pn:
6.5.1)若etrial≤etarget,用ptrial替换pn,转至步骤6.6);否则,令
6.5.3)生成随机均匀小数prand,prand∈[0,1];
6.5.4)若prand≤p,用ptrial替换pn;否则,保持pn不变;
6.6)令n=n+1;若n≤np,转至步骤6.3);
6.7)令g=g+1;若g≤g,转至步骤6.2);
7)种群pnew采用与种群p同样的进化策略,参见步骤6);
8)按能量由低至高的顺序分别对种群p和种群pnew中的个体进行排序;
9)分别选择种群p和种群pnew中能量排名第
10)令种群p=p′;
11)令m=m+1;若m≤m,转至步骤5);
12)对种群p执行进化操作,该进化操作过程与步骤6)的处理过程相同;
13)利用spicker聚类算法对所有成功替换种群中个体的构象进行聚类,以最大类的类心作为预测结果。
本发明的有益效果为:利用种群间信息交互和片段组装技术增强算法的探测能力,利用多种群联合搜索缓解能量函数不精确造成的不利影响,从而提高总体的预测精度。
附图说明
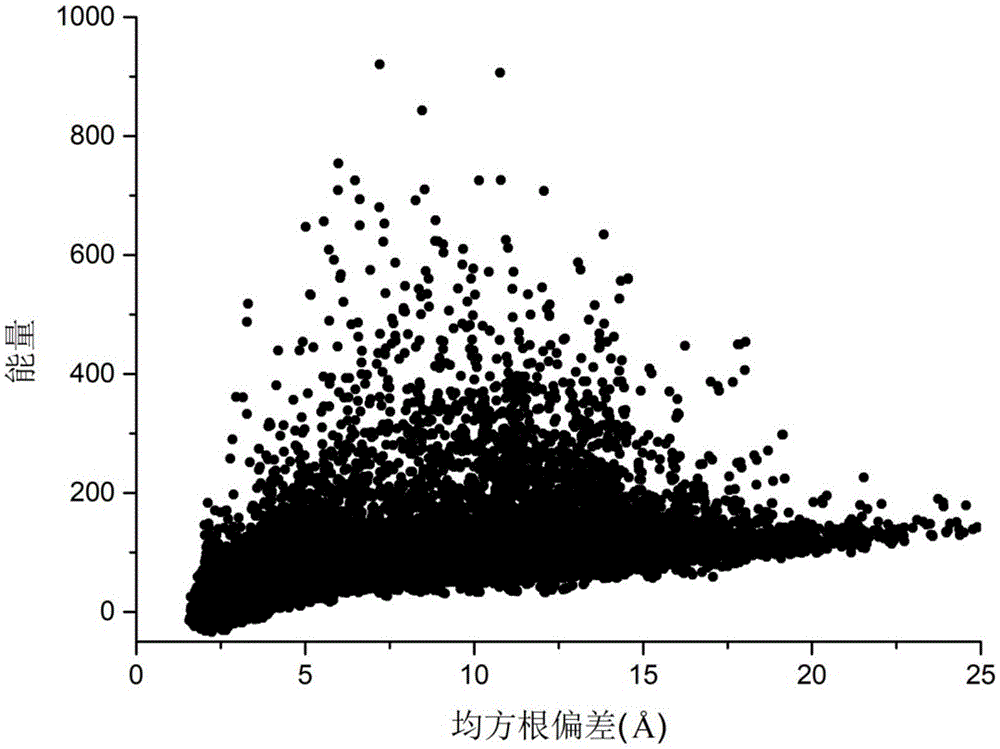
图1是一种基于多种群联合搜索的蛋白质构象空间优化方法对蛋白质1dtj进行结构预测时的构象更新示意图。
图2是一种基于多种群联合搜索的蛋白质构象空间优化方法对蛋白质1dtj进行结构预测得到的三维结构图。
具体实施方式
下面结合附图对本发明作进一步描述。
参照图1和图2,一种基于多种群联合搜索的蛋白质构象空间优化方法,包括以下步骤:
1)输入预测蛋白质的序列信息;
2)设置参数:种群规模np,辅助种群数量m,迭代次数g;
3)迭代rosetta协议第一、二阶段,生成初始种群p={p1,p2,...,pnp},其中pn表示种群p中的第n个个体,n∈{1,2,...,np};
4)设m=1,其中m∈{1,2,...,m};
5)迭代rosetta协议第一、二阶段,生成一个新种群
6)种群p独立进化,过程如下:
6.1)设g=1,其中g∈{1,2,...,g};
6.2)设n=1,其中n∈{1,2,...,np};
6.3)种群中个体信息交互,过程如下:
6.3.1)从种群p中随机选择三个与pn不同并且互不相同的个体pbase、pselect1、pselect2;
6.3.2)从个体pn、pselect1和pselect2中随机选择3个不同的9片段,分别替换个体pbase中相应的片段,生成新的个体pcombi;
6.4)从个体pcombi中随机选择一个位置执行一次9片段的片段组装,生成片段组装后的个体ptrial;
6.5)用rosettascore3能量函数计算ptrial和pn的能量etrial和etarget,按如下方式决定是否用ptrial替换种群中的个体pn:
6.5.1)若etrial≤etarget,用ptrial替换pn,转至步骤6.6);否则,令
6.5.3)生成随机均匀小数prand,prand∈[0,1];
6.5.4)若prand≤p,用ptrial替换pn;否则,保持pn不变;
6.6)令n=n+1;若n≤np,转至步骤6.3);
6.7)令g=g+1;若g≤g,转至步骤6.2);
7)种群pnew采用与种群p同样的进化策略,参见步骤6);
8)按能量由低至高的顺序分别对种群p和种群pnew中的个体进行排序;
9)分别选择种群p和种群pnew中能量排名第
10)令种群p=p′;
11)令m=m+1;若m≤m,转至步骤5);
12)对种群p执行进化操作,该进化操作过程与步骤6)的处理过程相同;
13)利用spicker聚类算法对所有成功替换种群中个体的构象进行聚类,以最大类的类心作为预测结果。
本实施例以序列长度为74的蛋白质1dtj为实施例,一种基于多种群联合搜索的蛋白质构象空间优化方法,包括以下步骤:
1)输入预测蛋白质1dtja的序列信息;
2)设置参数:种群规模np=100,辅助种群数量m=5,迭代次数g=100;
3)迭代rosetta协议第一、二阶段,生成初始种群p={p1,p2,...,pnp},其中pn表示种群p中的第n个个体,n∈{1,2,...,np};
4)设m=1,其中m∈{1,2,...,m};
5)迭代rosetta协议第一、二阶段,生成一个新种群
6)种群p独立进化,过程如下:
6.1)设g=1,其中g∈{1,2,...,g};
6.2)设n=1,其中n∈{1,2,...,np};
6.3)种群中个体信息交互,过程如下:
6.3.1)从种群p中随机选择三个与pn不同并且互不相同的个体pbase、pselect1、pselect2;
6.3.2)从个体pn、pselect1和pselect2中随机选择3个不同的9片段,分别替换个体pbase中相应的片段,生成新的个体pcombi;
6.4)从个体pcombi中随机选择一个位置执行一次9片段的片段组装,生成片段组装后的个体ptrial;
6.5)用rosettascore3能量函数计算ptrial和pn的能量etrial和etarget,按如下方式决定是否用ptrial替换种群中的个体pn:
6.5.1)若etrial≤etarget,用ptrial替换pn,转至步骤6.6);否则,令
6.5.3)生成随机均匀小数prand,prand∈[0,1];
6.5.4)若prand≤p,用ptrial替换pn;否则,保持pn不变;
6.6)令n=n+1;若n≤np,转至步骤6.3);
6.7)令g=g+1;若g≤g,转至步骤6.2);
7)种群pnew采用与种群p同样的进化策略,参见步骤6);
8)按能量由低至高的顺序分别对种群p和种群pnew中的个体进行排序;
9)分别选择种群p和种群pnew中能量排名第
10)令种群p=p′;
11)令m=m+1;若m≤m,转至步骤5);
12)对种群p执行进化操作,该进化操作过程与步骤6)的处理过程相同;
13)利用spicker聚类算法对所有成功替换种群中个体的构象进行聚类,以最
大类的类心作为预测结果。
以氨基酸序列长度为74的蛋白质1dtj为实施例,运用以上方法得到了该蛋白质的近天然态构象,其构象更新示意图如图1所示,预测的蛋白质的均方根偏差为
以上阐述是本发明给出的一个实施的预测效果,本发明不仅适合上述实施例,在不偏离本发明基本思想及不超出本发明实质内容的前提下可对其做种种改进加以实施。
- 还没有人留言评论。精彩留言会获得点赞!