一种新型构建HCV病毒感染可视化报告系统的细胞修饰方法与流程
本发明属于生物技术领域,具体地说,本发明涉及一种新型构建hcv病毒感染可视化报告系统的细胞修饰方法。
背景技术:
1989年人类首次发现并克隆了丙型肝炎病毒(hepatitiscvirus,hcv)(chooetal,science,1989),经过20多年的研究,人们逐渐在其分子生物学特性、致病机理及与宿主的关系上取得较大进展。同时hcv带来的危害也逐渐被人们所认知,根据世界卫生组织统计,目前全球hcv感染率为3%,并且多数患者会转为持续感染,继而诱发肝硬化、肝纤维化和肝癌,已成为威胁人类健康的主要病原之一。
hcv属于黄病毒科肝炎病毒属。hcv经血液传播,主要通过输血和血制品传播,也可以通过破损的皮肤与粘膜传播。目前hcv最常用的治疗手段为干扰素(peg-ifn)和利巴韦林(ribavirin),但是该方法不仅治疗费用昂贵,而且副作用强,易导致肌痛、关节痛、头痛等,同时50%的患者无法根除病毒。虽然有高效的直接抗病毒药物上市,但是极高的价格无法惠及众多患者。而且hcv的基因组差异较大,已发现7种基因型及多个亚型,同时由于hcv复制酶(ns5b)保真性差而导致其基因组变异率高,因此给hcv的治疗带来了极大的挑战。hcv对直接抗病毒药物的逃逸也逐渐被发现和报道(xiaof,etal.plospathog,2014)。
相比原型株,本发明人对hcv患者体内的准种(quasispecies)更感兴趣,它不仅代表了hcv基因的多态性,而且能真实地反映出这种差异在临床上的表现。但是目前还没有相关方法来有效筛选患者体内病毒粒子。本发明人希望通过建立一种新型的hcv检测方法,一方面能有效地从hcv患者血清中分离出hcv病毒粒子,分析其基因组的差异性与临床表现,并最终获得多株hcv感染性克隆用于进一步的基础研究。另一方面针对hcv患者治病费用高昂的情况,本发明人希望能利用一种hcv报告系统帮助患者做药物治疗前检测,测试治疗药物对该病人的有效性;同时为筛选广谱特异性的抗hcv药物提供技术平台。
技术实现要素:
本发明的目的在于提供一种新型的病毒感染的报告系统,来检测病毒的感染。
本发明的另一目的是提供一种新型构建hcv病毒感染可视化报告系统的细胞修饰方法。
在本发明的第一方面,提供了一种重组蛋白,所述重组蛋白包括相互独立的第一融合蛋白和第二融合蛋白,所述第一融合蛋白的结构如下式i所示:
nls-pn-in,(i);
所述第二融合蛋白的结构如下式ii所示:
ic-pc-nls-ips,(ii);
其中,
nls为核定位信号;
ips为线粒体定位信号;
in为内含肽n端片段,ic为内含肽c端片段;
pn为报告蛋白n端片段,pc为报告蛋白c端片段;
并且,当第一融合蛋白和第二融合蛋白空间接触时,所述in和ic结合时形成具有活性的内含肽,所述内含肽剪接pn和pc,从而使pn和pc结合形成具有报告蛋白特性的多肽。“-”表示肽键或肽段(如肽接头)。
在另一优选例中,所述第二融合蛋白中包括切割位点,所述切割位点位于ips内部和/或所述nls和所述ips之间。
在另一优选例中,所述切割位点为病毒蛋白的切割位点。
在另一优选例中,所述切割位点为hcv的ns3/4a蛋白的切割位点、hav的3abc蛋白切割位点、ev71的2a蛋白切割位点、或其组合。
在另一优选例中,所述切割位点为hcv的ns3/4a蛋白的切割位点。
在另一优选例中,所述nls具有如下多肽序列:
dpkkkrkv,seqidno.:1。
在另一优选例中,所述ips具有如下多肽序列:
segtfgihvaenpsiqllegnpgppadpdggprpqadrkfqerevpchrpspgalwlqvavtgvlvvtllvvlyrrrlh,seqidno.:2。
在另一优选例中,所述in和ic多肽序列分别如下:
in:claadtevltveygpiaigklveenircqvyccnpdgyiysqpigqwhqrgeqevieyelsdgriiratadhrfmteegemlsldeiferslelkqiptpllaiaqpsplata,seqidno.:3
ic:vkivrrrslgvqpvydlgvatvhnfvlanglvasncfn,seqidno.:4。
在另一优选例中,所述报告蛋白选自下组:
绿色荧光蛋白(gfp)、和红色荧光蛋白(mcherry)。
在另一优选例中,所述pn和pc选自下组:
gfp-pn:vskgeelftgvvpilveldgdvnghkfsvsgegegdatygkltlkficttgklpvpwptlvttltygvqcfsrypdhmkqhdffksampegyvqertiffkddgnyktraevkfegdtlvnrielkgidfkedgnilghkleynynshnvyimadkq,seqidno.:5
gfp-pc:kngikvnfkirhniedgsvqladhyqqntpigdgpvllpdnhylstqsalskdpnekrdhmvllefvtaagitlgmdelyk,seqidno.:6;和
mcherry-pn:vskgeednmaiikefmrfkvhmegsvnghefeiegegegrpyegtqtaklkvtkggplpfawdilspqfmygskayvkhpadipdylklsfpegfkwervmnfedggvvtvtqdsslqdgefiykvklrgtnfpsdgpvmqkktmgweassermyped,seqidno.:7
mcherry-pc:galkgeikqrlklkdgghydaevkttykakkpvqlpgaynvniklditshnedytiveqyeraegrhstggmdelyk,seqidno.:8。
本发明的第二方面,提供了一种多核苷酸,所述多核苷酸包括编码所述第一融合蛋白的多核苷酸和/或编码所述第二融合蛋白的多核苷酸。
本发明的第三方面,提供了一种表达载体,所述表达载体含有本发明第二方面所述的多核苷酸。
本发明的第四方面,提供了一种宿主细胞,所述的宿主细胞含有本发明第三方面所述的表达载体,或者在基因组中整合有本发明第二方面所述的多核苷酸。
在另一优选例中,所述宿主细胞包含编码所述第一融合蛋白的第一载体和/或编码所述第二融合蛋白的第二载体。
在另一优选例中,所述宿主细胞基因组中整合有编码所述第一融合蛋白的第一多核苷酸,和/或编码所述第二融合蛋白的第二多核苷酸。
在另一优选例中,所述宿主细胞的细胞核定位有所述第一融合蛋白,所述宿主细胞的线粒体定位有所述第二融合蛋白。
本发明的第五方面,提供了一种试剂盒,所述试剂盒包括:
(1)本发明第一方面所述的重组蛋白;
(2)编码所述第一融合蛋白的第一多核苷酸和/或编码所述第二融合蛋白的第二多核苷酸;
(3)包含编码所述第一融合蛋白的多核苷酸的第一载体和/或包含编码所述第二融合蛋白的多核苷酸的第二载体;和/或
(4)本发明第四方面所述的宿主细胞。
本发明的第六方面,提供了一种构建基因工程细胞的方法,所述方法包括步骤:
(1)提供一宿主细胞,将外源的表达第一融合蛋白的多核苷酸导入所述宿主细胞,从而使所述宿主细胞表达所述第一融合蛋白;
(2)将外源的表达第二融合蛋白的多核苷酸导入所述宿主细胞,从而使所述宿主细胞表达所述第二融合蛋白;
其中,所述第一融合蛋白和所述第二融合蛋白分别如上所述。
在另一优选例中,所述第一融合蛋白定位于所述基因工程细胞的细胞核内。
在另一优选例中,所述第二融合蛋白定位于所述基因工程细胞的线粒体。
在另一优选例中,所述宿主细胞为对hcv易感的肝癌细胞系。
本发明的第七方面,提供了一种用于本发明第六方面所述的方法的融合蛋白,所述融合蛋白的结构如下式i所示:
nls-pn-in,(i);或者
所述融合蛋白的结构如下式ii所示:
ic-pc-nls-ips,(ii);
其中,
nls为核定位信号;
ips为线粒体定位信号;
in为内含肽n端片段,ic为内含肽c端片段;
pn为报告蛋白n端片段,pc为报告蛋白c端片段。
本发明的第八方面,提供了一种构建hcv病毒感染报告系统的方法,所述方法包括步骤:
(1)提供一宿主细胞,将外源的表达第一融合蛋白的多核苷酸导入所述宿主细胞,从而使所述宿主细胞表达所述第一融合蛋白;
(2)将外源的表达第二融合蛋白的多核苷酸导入所述宿主细胞,从而使所述宿主细胞表达所述第二融合蛋白;
其中,所述第一融合蛋白和所述第二融合蛋白分别如上所述;
当所述宿主细胞被hcv感染时,hcv非结构蛋白ns3/4a蛋白切割所述第二融合蛋白中的切割位点,从而使所述第二融合蛋白片段游离并在所述nls的驱动下移至核内,与所述第一融合蛋白接触,在内含肽的作用下剪接形成完整的报告蛋白,从而在所述宿主细胞上显示报告蛋白特性。
本发明的第九方面,提供了本发明第一方面所述的重组蛋白,本发明第二方面所述的多核苷酸、本发明第三方面所述的表达载体、本发明第四方面所述的宿主细胞、本发明第五方面所述的试剂盒的用途,用于筛选抗病毒的药物。
本发明的第十方面,提供了一种筛选抗病毒药物的方法,所述方法包括:
(1)根据本发明第六方面所述的方法构建基因工程细胞;
(2)向步骤(1)中构建的基因工程细胞培养体系中加入病毒和待筛选物质作为实验组;
(3)检测实验组中宿主细胞中的报告蛋白特性;
如果实验组中报告蛋白的特性降低,则说明该物质能够抑制该病毒复制。
应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
图1显示了visi-gfp细胞系验证试验的结果。
图2显示了visi-gfp与huh7.5.1在hcv病毒复制和病毒产量上的比较。
图3显示了hcv病毒感染实验。
图4显示了药物抑制实验。
图5显示了visi-mcherry的验证实验。
具体实施方式
本发明人通过广泛而深入的研究,构建了病毒感染可视化报告系统,在宿主细胞中将报告蛋白分开表达,使报告蛋白的两个片段分别定位在细胞核和线粒体上,当细胞感染病毒后,病毒蛋白将定位在线粒体上的报告蛋白片段切割下来,并游离到细胞核内,与定位在细胞核内的蛋白接触,在内含肽的作用下发生剪接形成完整的报告蛋白,从而在宿主细胞上观察到报告蛋白特性。实验结果表明,所述报告系统能够方便、灵敏、高效的显示宿主细胞是否感染病毒。本发明还提供了所述报告系统在药物筛选中的用途。在此基础上,完成了本发明。
在描述本发明之前,应当理解本发明不限于所述的具体方法和实验条件,因为这类方法和条件可以变动。还应当理解本文所用的术语其目的仅在于描述具体实施方案,并且不意图是限制性的,本发明的范围将仅由所附的权利要求书限制。
除非另外定义,否则本文中所用的全部技术与科学术语均具有如本发明所属领域的普通技术人员通常理解的相同含义。如本文所用,在提到具体列举的数值中使用时,术语“约”意指该值可以从列举的值变动不多于1%。例如,如本文所用,表述“约100”包括99和101和之间的全部值(例如,99.1、99.2、99.3、99.4等)。
虽然在本发明的实施或测试中可以使用与本发明中所述相似或等价的任何方法和材料,本文在此处例举优选的方法和材料。
具体地,本发明提供了一种新型的hcv病毒感染可视化报告系统的细胞修饰方法,随后在肝细胞系huh7.5.1细胞中运用该修饰方法建立起一种简单灵敏的hcv感染报告系统,清晰准确的显示被感染细胞,本方法可应用于hcv感染活细胞分选及抗hcv药物的高通量筛选及抗毒活性高效评估。
病毒
病毒(virus)是由一个核酸分子(dna或rna)与蛋白质构成的非细胞形态的靠寄生生活的生命体。病毒无细胞结构,需要依靠宿主细胞完成自身的繁殖。病毒感染细胞后,依靠宿主表达出病毒蛋白,用于组装新的病毒。其中部分病毒蛋白具有切割功能。本发明中所述“切割功能”是指某些病毒蛋白具有酶活性,能特异切割宿主蛋白以干扰宿主的抗病毒过程。实例性的病毒包括:hcv(丙型肝炎病毒)、hav(甲型肝炎病毒)、ev71(肠道病毒71型)等。这些病毒中具有切割动能的蛋白如:hcv中的ns3/4a蛋白、hav中的3abc蛋白、ev71中的2a蛋白。在(lixd,etal.pnas,2005;yangy,etal.pnas,2007;wangb,etal.plospathog,2013)文献中有关于这些蛋白的详细报道。以下以hcv为例,详细介绍。
hcv属于黄病毒科肝炎病毒属,为单股正连rna病毒,基因组全长9.6kb,编码一个多聚蛋白,经胞内激酶和自身非结构蛋白的切割形成3个结构蛋白(core、e1、e2)和7个非结构蛋白(p7、ns2、ns3、ns4a、ns4b、ns5a、ns5b)。核心蛋白core是其核衣壳的主要组成部分,用来包裹hcv基因组;e1、e2为囊膜蛋白,可形成二聚体包裹于核衣壳外;p7为疏水性的离子通道蛋白;ns2既能切割多聚蛋白同时也能参与病毒的组装;ns3n端180个氨基酸具有丝氨酸蛋白酶活性,能与辅酶ns4b一起切割多聚蛋白多个节点,而其c端具有atp水解酶和解旋酶活性,是hcv基因组复制所必需的;ns5a为磷酸化蛋白,可调节hcvrna的复制与组装,而且能与宿主多个因子结合;ns5b是hcv的复制酶。复制而得hcvrna与结构蛋白在细胞内脂类的共同作用下形成新的病毒粒子并释放到胞外,再通过识别细胞表面受体后再进入细胞并完成下一次循环。
利用本发明的技术方案,可以构建一种新型的hcv感染的报告系统,来检测hcv的感染。也可以利用本发明的技术方案,构建其它病毒(优选为能够表达切割蛋白的病毒)感染的报告系统,用来检测病毒感染。
nls(核定位信号)
nls(nuclearlocalizationsignal)是蛋白质的一个结构域,通常为一短的氨基酸序列,它能与入核载体相互作用,使蛋白能被运进细胞核。
在优选地实施方式中,本发明中的nls序列为:
dpkkkrkv(seqidno.:1)。
ips(线粒体定位信号)
ips(ifn-betapromoterstimulator)是ifn-β启动子刺激因子。
在优选地实施方式中,本发明中的ips序列如下:
segtfgihvaenpsiqllegnpgppadpdggprpqadrkfqerevpchrpspgalwlqvavtgvlvvtllvvlyrrrlh(seqidno.:2)。
内含肽
内含肽(intein),是位于宿主蛋白质中的一段插入序列。与内含肽相对应的另一专用术语是外显肽。内含肽基因不是一个独立的,必须插人于外显肽基因才能复制转录,可从前体蛋白中切除并将两侧外显肽连接起来成为成熟蛋白质。其对应的核苷酸序列嵌合在宿主蛋白对应的核酸序列之中,与宿主蛋白基因存在于同一开放阅读框架(openreadingframe,orf)内,并与宿主蛋白质基因进行同步转录和翻译,当翻译形成蛋白质前体后,内含肽从宿主蛋白质中切除,从而形成成熟的具有活性的蛋白。
在优选地实施方式中,本发明中的内含肽为分离性内含肽(splictintein)。分离性内含肽的两个剪接区域分裂成两份,存在于不同的多肽片段上,所以称为分离内含肽。分离内含肽的两个多肽片段可以分开表达,当两个多肽片段接触时,结合成为具有剪接功能的完整内含肽。
优选地本发明中的内含肽为聚球藻属synechococcuselongates(sel)的分离性内含肽,genbank:cp000100.1,其分开的两段分别为:
in:claadtevltveygpiaigklveenircqvyccnpdgyiysqpigqwhqrgeqevieyelsdgriiratadhrfmteegemlsldeiferslelkqiptpllaiaqpsplata(seqidno.:3)
ic:vkivrrrslgvqpvydlgvatvhnfvlanglvasncfn(seqidno.:4)
报告蛋白
如本文所用,报告蛋白(reporterprotein)是一种可被检测的蛋白质或酶,也就是说,是一个非常容易被鉴定的蛋白。常用的报告蛋白包括:抗生素抗性蛋白、β半乳糖苷酶、荧光素酶、荧光蛋白。
在本发明优选的实施方式中,使用荧光蛋白,如绿色荧光蛋白(gfp)和红色荧光蛋白(mcherry)。
绿色荧光蛋白(gfp,238aa,参考genbank:u55762.1)等报告基因的蛋白序列在特定的区域可分裂成两个片段:gfpn(1-157aa)和gfpc(158-237aa),二者均不产生荧光,但借助内含肽(intein)的剪接功能将分开的片段重新拼接成完整的gfp后又会发出荧光。
gfpn(1-157aa)的氨基酸序列如下:
gfp-pn:vskgeelftgvvpilveldgdvnghkfsvsgegegdatygkltlkficttgklpvpwptlvttltygvqcfsrypdhmkqhdffksampegyvqertiffkddgnyktraevkfegdtlvnrielkgidfkedgnilghkleynynshnvyimadkq(seqidno.:5)
gfpc(158-237aa)的氨基酸序列如下:
gfp-pc:kngikvnfkirhniedgsvqladhyqqntpigdgpvllpdnhylstqsalskdpnekrdhmvllefvtaagitlgmdelyk(seqidno.:6)。
红色荧光蛋白mcherry-pn(1-158aa)的氨基酸序列如下:
mcherry-pn:vskgeednmaiikefmrfkvhmegsvnghefeiegegegrpyegtqtaklkvtkggplpfawdilspqfmygskayvkhpadipdylklsfpegfkwervmnfedggvvtvtqdsslqdgefiykvklrgtnfpsdgpvmqkktmgweassermyped(seqidno.:7)
mcherry-pc(159-235aa)的氨基酸序列如下:
mcherry-pc:galkgeikqrlklkdgghydaevkttykakkpvqlpgaynvniklditshnedytiveqyeraegrhstggmdelyk(seqidno.:8)。
hcv感染可视化报告系统
本发明提供的hcv感染可视化报告系统中包括一种重组蛋白,所述重组蛋白包括相互独立的第一融合蛋白和第二融合蛋白,所述第一融合蛋白的结构如下式i所示:
nls-pn-in,(i);
所述第二融合蛋白的结构如下式ii所示:
ic-pc-nls-ips,(ii);
其中,
nls为核定位信号;
ips为线粒体定位信号;
in为内含肽n端片段,ic为内含肽c端片段;
pn为报告蛋白n端片段,pc为报告蛋白c端片段;
并且,当第一融合蛋白和第二融合蛋白空间接触时,所述in和ic结合时形成具有活性的内含肽,所述内含肽剪接pn和pc,从而使pn和pc结合形成具有报告蛋白特性的多肽。
在一个优选地实施方式中,所述第二融合蛋白中包括切割位点,所述切割位点位于ips内部和/或所述nls和所述ips之间;优选地,所述切割位点为hcv非结构蛋白ns3/4a蛋白的切割位点。
在本发明的一个优选地实施方式中,本发明提供的一种hcv感染可视化报告系统(viralinfectionactivatedsplit-inteinbasedreportersystem,visi)中,选用了synechococcuselongates(sel),一种来自蓝藻的分离性内含肽(splictintein,genbank:cp000100.1),蛋白序列可分为dnaen(ncbireferencesequence:wp_011378362.1,758-870aa)和dnaec(ncbireferencesequence:wp_011243264.1,1-38aa)。之后将gfp分裂片段(gfpn/gfpc)与内含肽分裂片段(dnaen/dnaec)相互组合形成融合蛋白(dnaen+gfpn/dnaec+gfpc),利用核定位信号(nls,dpkkkrkv)将内含肽一端(nls-gfpn-dnaen)稳定表达并定位于huh7.5.1细胞核内,而另一端(dnaec-gfpc-nls-ips)则利用线粒体定位信号(ips,462-540aa,genbank:ab232371.1,)稳定表达并定位于huh7.5.1细胞线粒体上,该细胞系本发明人称之为visi-gfp,定位不同造成二者空间位置不同,内含肽无法发挥剪接功能,从而使得visi-gfp细胞无荧光信号出现。
上述线粒体定位信号(ips)自身蛋白序列上存在hcv病毒复制必需蛋白ns3/4a的切割位点,所以当hcv感染后,翻译出的复制所需蛋白ns3/4a将切割ips,造成定位于线粒体一端融合蛋白(dnaec-gfpc-nls-ips)发生游离,在该序列中核定位信号(nls)的驱动下移至核内,因此在空间上接近了定位于核内的一端融合蛋白(nls-gfpn-dnaen),二者在内含肽的作用下,发生剪接形成完整的gfp蛋白,从而使得visi-gfp细胞出现可见的核内绿色荧光。
在本发明中,“重组蛋白”指具有式i或ii所示结构的融合蛋白,也指一种重组蛋白的组合,在该重组蛋白的组合中包括式i和ii所示结构的融合蛋白。此外,应理解,所述术语还包括融合蛋白的活性片段和衍生物。
本发明的多核苷酸可以是dna形式或rna形式。dna形式包括cdna、基因组dna或人工合成的dna。dna可以是单链的或是双链的。dna可以是编码链或非编码链。
本发明还涉及上述多核苷酸的变异体,其编码与本发明有相同的氨基酸序列的蛋白质片段、类似物和衍生物。此多核苷酸的变异体可以是天然发生的等位变异体或非天然发生的变异体。这些核苷酸变异体包括取代变异体、缺失变异体和插入变异体。如本领域所知的,等位变异体是一个多核苷酸的替换形式,它可能是一个或多个核苷酸的取代、缺失或插入,但不会从实质上改变其编码多肽的功能。
如本文所用,术语“引物”指的是在与模板配对,在dna聚合酶的作用下能以其为起点进行合成与模板互补的dna链的寡居核苷酸的总称。引物可以是天然的rna、dna,也可以是任何形式的天然核苷酸。引物甚至可以是非天然的核苷酸如lna或zna等。引物“大致上”(或“基本上”)与模板上一条链上的一个特殊的序列互补。引物必须与模板上的一条链充分互补才能开始延伸,但引物的序列不必与模板的序列完全互补。比如,在一个3'端与模板互补的引物的5'端加上一段与模板不互补的序列,这样的引物仍大致上与模板互补。只要有足够长的引物能与模板充分的结合,非完全互补的引物也可以与模板形成引物-模板复合物,从而进行扩增。
本发明融合蛋白的核苷酸全长序列或其片段通常可以用pcr扩增法、重组法或人工合成的方法获得。对于pcr扩增法,可根据已公开的有关核苷酸序列,尤其是开放阅读框序列来设计引物,并用市售的cdna库或按本领域技术人员已知的常规方法所制备的cdna库作为模板,扩增而得有关序列。当序列较长时,常常需要进行两次或多次pcr扩增,然后再将各次扩增出的片段按正确次序拼接在一起。
一旦获得了有关的序列,就可以用重组法来大批量地获得有关序列。这通常是将其克隆入载体,再转入细胞,然后通过常规方法从增殖后的宿主细胞中分离得到有关序列。
此外,还可用人工合成的方法来合成有关序列,尤其是片段长度较短时。通常,通过先合成多个小片段,然后再进行连接可获得序列很长的片段。
应用pcr技术扩增dna/rna的方法被优选用于获得本发明的基因。用于pcr的引物可根据本文所公开的本发明的序列信息适当地选择,并可用常规方法合成。可用常规方法如通过凝胶电泳分离和纯化扩增的dna/rna片段。
本发明也涉及包含本发明的多核苷酸的载体,以及用本发明的载体或融合蛋白编码序列经基因工程产生的宿主细胞,以及经重组技术产生本发明所述蛋白质的方法。
通过常规的重组dna技术,可利用本发明的多核苷酸序列可用来表达或生产重组蛋白。一般来说有以下步骤:
(1)用本发明的编码本发明蛋白的多核苷酸(或变异体),或用含有该多核苷酸的重组表达载体转化或转导合适的宿主细胞;
(2)在合适的培养基中培养的宿主细胞;
(3)从培养基或细胞中分离、纯化蛋白质。
本领域的技术人员熟知的方法能用于构建含本发明蛋白的编码dna序列和合适的转录/翻译控制信号的表达载体。这些方法包括体外重组dna技术、dna合成技术、体内重组技术等。所述的dna序列可有效连接到表达载体中的适当启动子上,以指导mrna合成。表达载体还包括翻译起始用的核糖体结合位点和转录终止子。
此外,表达载体优选地包含一个或多个选择性标记基因,以提供用于选择转化的宿主细胞的表型性状,如真核细胞培养用的二氢叶酸还原酶、新霉素抗性等,或用于大肠杆菌的四环素或氨苄青霉素抗性。
包含上述的适当dna序列以及适当启动子或者控制序列的载体,可以用于转化适当的宿主细胞,以使其能够表达蛋白质。
宿主细胞可以是原核细胞,如细菌细胞;或是低等真核细胞,如酵母细胞;或是高等真核细胞,如哺乳动物细胞。代表性例子有:大肠杆菌,链霉菌属的细菌细胞;真菌细胞如酵母;植物细胞;果蝇s2或sf9的昆虫细胞;cho、ns0、cos7、或293细胞的动物细胞等。
用重组dna转化宿主细胞可用本领域技术人员熟知的常规技术进行。当宿主为原核生物如大肠杆菌时,能吸收dna的感受态细胞可在指数生长期后收获,用cacl2法处理,所用的步骤在本领域众所周知。另一种方法是使用mgcl2。如果需要,转化也可用电穿孔的方法进行。当宿主是真核生物,可选用如下的dna转染方法:磷酸钙共沉淀法,常规机械方法如显微注射、电穿孔、脂质体包装等。
获得的转化子可以用常规方法培养,表达本发明的基因所编码的多肽。根据所用的宿主细胞,培养中所用的培养基可选自各种常规培养基。在适于宿主细胞生长的条件下进行培养。当宿主细胞生长到适当的细胞密度后,用合适的方法(如温度转换或化学诱导)诱导选择的启动子,将细胞再培养一段时间。
在上面的方法中的蛋白质可在细胞内表达。如果需要,可利用其物理的、化学的和其它特性通过各种分离方法分离和纯化蛋白。这些方法是本领域技术人员所熟知的。这些方法的例子包括但并不限于:常规的复性处理、用蛋白沉淀剂处理(盐析方法)、离心、渗透破菌、超处理、超离心、分子筛层析(凝胶过滤)、吸附层析、离子交换层析、高效液相层析(hplc)和其它各种液相层析技术及这些方法的结合。
如本文所用,术语“融合蛋白”还包括第一融合蛋白或第二融合蛋白的变异形式。这些变异形式包括(但并不限于):1-3个(通常为1-2个,更佳地1个)氨基酸的缺失、插入和/或取代,以及在c末端和/或n末端添加或缺失一个或数个(通常为3个以内,较佳地为2个以内,更佳地为1个以内)氨基酸。例如,在本领域中,用性能相近或相似的氨基酸进行取代时,通常不会改变蛋白质的功能。又比如,在c末端和/或n末端添加或缺失一个或数个氨基酸通常也不会改变蛋白质的结构和功能。此外,所述术语还包括单体和多聚体形式的本发明多肽。该术语还包括线性以及非线性的多肽(如环肽)。
本发明还包括上述融合蛋白的活性片段、衍生物和类似物。如本文所用,术语“片段”、“衍生物”和“类似物”是指基本上保持本发明融合蛋白的功能或活性的多肽。本发明的多肽片段、衍生物或类似物可以是(i)有一个或几个保守或非保守性氨基酸残基(优选保守性氨基酸残基)被取代的多肽,或(ii)在一个或多个氨基酸残基中具有取代基团的多肽,或(iii)抗原肽与另一个化合物(比如延长多肽半衰期的化合物,例如聚乙二醇)融合所形成的多肽,或(iv)附加的氨基酸序列融合于此多肽序列而形成的多肽(与前导序列、分泌序列或6his等标签序列融合而形成的融合蛋白)。根据本文的教导,这些片段、衍生物和类似物属于本领域熟练技术人员公知的范围。
一类优选的活性衍生物指与式ia或式ib的氨基酸序列相比,有至多3个,较佳地至多2个,更佳地至多1个氨基酸被性质相似或相近的氨基酸所替换而形成多肽。这些保守性变异多肽最好根据表a进行氨基酸替换而产生。
表a
本发明还提供本发明融合蛋白的类似物。这些类似物与第一融合蛋白或第二融合蛋白的差别可以是氨基酸序列上的差异,也可以是不影响序列的修饰形式上的差异,或者兼而有之。类似物还包括具有不同于天然l-氨基酸的残基(如d-氨基酸)的类似物,以及具有非天然存在的或合成的氨基酸(如β、γ-氨基酸)的类似物。应理解,本发明的多肽并不限于上述例举的代表性的多肽。
修饰(通常不改变一级结构)形式包括:体内或体外的多肽的化学衍生形式如乙酰化或羧基化。修饰还包括糖基化,如那些在多肽的合成和加工中或进一步加工步骤中进行糖基化修饰而产生的多肽。这种修饰可以通过将多肽暴露于进行糖基化的酶(如哺乳动物的糖基化酶或去糖基化酶)而完成。修饰形式还包括具有磷酸化氨基酸残基(如磷酸酪氨酸,磷酸丝氨酸,磷酸苏氨酸)的序列。还包括被修饰从而提高了其抗蛋白水解性能或优化了溶解性能的多肽。
本发明的主要优点在于:
(1):目前的hcv感染可视化都是建立在对病毒基因组的遗传改造上的;比如在病毒基因组中插入荧光蛋白基因。但是这种遗传改造通常会信号弱并影响病毒的生物学特性。本发明通过对hcv宿主细胞系进行改造,避免了对野生型病毒的影响。野生型hcv感染后通过病毒蛋白酶ns3/4a切割,核转运,内含肽的高效剪接,在细胞核内重新拼接并恢复荧光蛋白的结构和荧光。
(2)病毒感染该hcv感染可视化宿主细胞系统前,细胞中的荧光蛋白基因被分段表达并分别定位于线粒体和细胞核内,所以该系统具有极低的荧光背景和hcv感染后优秀的信噪比。该系统构建的细胞模型与现有技术中的细胞模型相比具有更高的灵敏度,更好的特异性等优点。
(3)依照本方法构建的细胞培养报告系统是一种实时监测hcv感染的细胞系统,感染细胞的荧光信号强弱可以指示hcv感染复制的水平,存在“开关”效应。
下面结合具体实施例,进一步详陈本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明详细条件的实验方法,通常按照常规条件如美国sambrook.j等著《分子克隆实验室指南》(黄培堂等译,北京:科学出版社,2002年)中所述的条件,或按照制造厂商所建议的条件。除非另外说明,否则百分比和份数按重量计算。以下实施例中所用的实验材料和试剂如无特别说明均可从市售渠道获得。
材料
1、质粒:含绿色荧光蛋白(greenfluorescenceprotein,gfp,plasmid#60360))、红色荧光蛋白(mcherry,plasmid#36084)及ips((plasmid#52135)基因序列的质粒购自addgene,内含肽(intein)基因序列由生工生物工程(上海)股份有限公司合成。
2、细胞:huh7.5.1(参考文献zhongj,等.pnas,2005;us8506969;us8143022;可以获自中国科学院上海巴斯德研究所)。
3、病毒:hcvjc1(参考文献lindenbachb.d,等.science,2005;us7439227;可以获自中国科学院上海巴斯德研究所),jcr2a(poenischm,等.plospathog,2015;可以获自中国科学院上海巴斯德研究所)通过人工改造,删除其结构蛋白e1e2(2051bp-3647bp)序列而成jcr2a-deltae1e2毒株(可以获自中国科学院上海巴斯德研究所)。
通用方法
分子克隆、免疫荧光、病毒生长曲线测定等实验方法参考文献(dacosta,etal.jvirol,2012),核质分离实验参考文献(dum,etal.jimmunol,2015)。
分子克隆:通过合成引物再用pcr方法扩增所需基因,随后利用酶切位点将该片段连接到质粒载体上。
免疫荧光:将贴壁细胞用多聚甲醛固定在玻璃片上,再用0.5%的聚乙二醇辛基苯基醚(tritonx-100)在细胞上打孔以方便抗体的进入,随后用5%的血清封闭,而后孵育一抗,1小时后洗涤并孵育荧光二抗,经几次洗涤后将玻璃片置于封片剂中以防止荧光淬灭,随后用激光共聚焦显微镜拍照。
hcvrna的制备与电转化:含有hcv基因组的质粒通过体外转录获得hcv全长的rna,随后用电转仪(975μf,270v,+∞ω,4mm)将hcvrna转移到细胞内并继续培养该细胞.
病毒生长曲线测定:将病毒基因组rna电转到细胞中,随后继续培养该细胞,在不同的时间点(0h、12h、24h、48h、72h、96h)收取细胞上清,病毒上清收齐后再通过半数组织培养感染剂量(tcid50)实验检测其在各个点的感染性。
核质分离:收集一定数量的细胞后,离心获得沉淀,加入hypotonicbuffer(10mmhepes,ph7.6;1.5mmmgcl2;10mmkcl;1mmedta;proteinaseinhibitors),冰上孵育10分钟后离心后转移上清即为细胞质悬液,洗涤数次后再加入highsaltbuffer(20mmhepes,ph7.6;0.5mnacl;1.5mmmgcl2;1mmedta;proteinaseinhibitors),冰上孵育20分钟,离心后获得上清即为细胞核悬液。
本发明人通过上述方法,成功构建了visi-gfp细胞系和visi-mcherry细胞系。
实施例1visi-gfp验证实验
visi-gfp细胞系构建完成后,随即验证了该策略的可行性。本发明人在visi-gfp细胞系内过表达hcvns3/4a蛋白,表达ns3/4a后可见核内出现明显荧光,而阴性对照组(mock)未出现任何荧光,而且背景信号低(图1a)。核质分离实验同样显示存在ns3/4a蛋白时,核内出现大量完整gfp蛋白(39kd)(图1b)。验证实验结果证明该策略可行,相比charlesmrice等(jonesct,etal.natbiotechnol,2010)建立的实时监测hcv感染的细胞模型,visi细胞具有极低的背景,更加清晰的视野,能够快速准确地发现感染细胞。
实施例2visi-gfp细胞系与huh7.5.1的病毒学比较
huh7.5.1是培养hcv常用的细胞系之一,本发明人以huh7.5.1为靶细胞构建visi-gfp细胞系并对huh7.5.1和visi-gfp细胞系在病毒复制及病毒产量上进行了比较。首先在两种细胞系中分别电转包含renillaluciferase报告基因的hcvjcr2a-deltae1e2rna,在不同时间点(0h-96h)收集样品测renillaluciferase值从而检测hcv在该细胞的复制情况,结果发现visi-gfp与huh7.5.1相比,二者在病毒复制水平上无明显差异(图2)。其次在两种细胞系中分别转染hcv基因组jc1后于不同时间点收集病毒上清,并检测其感染能力,图2显示二者在病毒产量上并无差别,综上所述:visi-gfp细胞系的建立并未影响细胞本身在病毒复制和病毒产生方面的能力。
实施例3hcv病毒感染实验
为检测hcv病毒感染能否引起验证实验中同样的核荧光,本发明人用huh7.5.1细胞培养收获的hcvjc1病毒感染visi-gfp细胞系,结果发现病毒感染细胞后翻译产生的ns3/4a同样会产生核内可见荧光(图3)。同时用hcvjc1病毒感染huh7.5.1及visi-gfp后,分别收集不同时间点的细胞,图3显示随着病毒蛋白的合成,visi-gfp细胞内gfp蛋白也随之产生,48h即可出现大量荧光蛋白。与liangjt等(huz,etal.antimicrobagentschemother,2013)建立的hcv检测方法相比.本系统无需对hcv病毒本身进行修改,不改变病毒本身特性,病毒复制即可产生可见荧光。综上所述:visi-gfp细胞系可应用于灵敏特异地可视化检测野生型hcv感染。
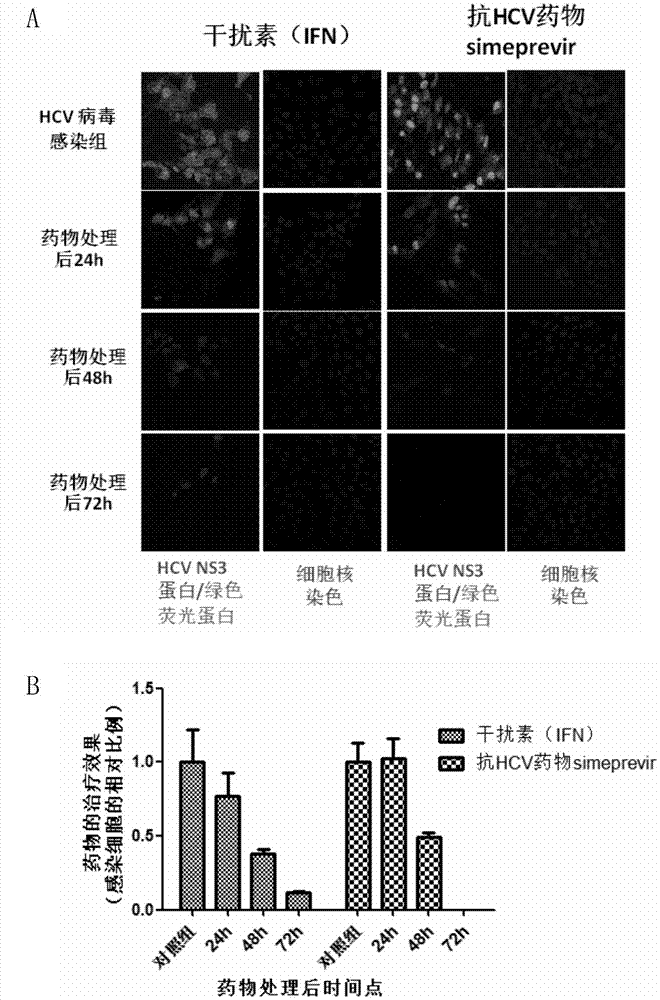
实施例4药物抑制实验
通常hcv在体内会建立起持续感染,能长时间存在于肝脏内而无法清除。dornerm等(marcusdorner,etal.methods,2013)将hcv-cre/loxp系统运用到其建立的hcv感染的小鼠模型中,将hcv-crerna注射到小鼠体内可见明显的报告基因信号,但是该系统存在一定的局限性:一方面该系统对病毒自身序列进行改造(cre插入到hcv基因中),会直接影响病毒自身特性;另一方面该系统一旦信号开启就无法关闭,限制其在抗病毒药物筛选上的应用。前面实验证明visi系统不需要改造病毒自身序列,所以如果要将visi系统运用到小鼠模型内,本发明人必须在模拟其体内持续感染机制的同时验证其是否存在“开关”效应。本发明人首先用hcvjc1病毒感染visi-gfp细胞系,大部分细胞出现可见的核荧光后再加入抗病毒药物(ifn购自上海索莱宝生物科技有限公司、simeprevir购自medchemexpress公司),分别于不同时间点收集细胞并进行免疫染色,观察荧光,结果发现不管是ifn治疗组还是simeprevir治疗组,均出现可见核荧光会随时间的增加而降低,存在明显的“开关”效应,直接反映出药物的抗病毒能力(图4),同理该系统也能用于中和抗体的评估。
实施例5visi荧光蛋白的替换
上述实验中本发明人选用绿色荧光蛋白(gfp)构建visi检测系统,为满足不同的实验需求,后期又将红色荧光蛋白(mcherry,235aa,参考genbank:kp238582.1)蛋白序列在第158位分离成的n端(1-158aa)、c端(159-235aa)两部分,分别替换visi-gfp系统中gfp的n端和c端,从而构建了红色荧光蛋白(mcherry)的visi系统(visi-mcherry)。感染实验证明:在hcvns3/4a蛋白存在时,细胞呈现核红色(如图5),显示出visi-gfp相同的结果;说明此种hcv感染报告系统修饰方法可以实现多色可视化。
总结
体外实验证明hcv病毒感染即可在细胞培养报告系统中产生可见荧光,间接免疫荧光(ifa)实验同样证实出现荧光的细胞存在hcv复制所需的ns3/4a蛋白。hcv药物处理后可见明显的荧光减弱,证明该系统应用于抗病毒药物筛选的适用性。
本发明人建立了一种新型构建hcv感染多色可视化报告系统的细胞修饰方法,利用该方法构建的hcv感染活细胞报告系统,能灵敏高效地显示hcv感染的阳性细胞;其次,药物抑制实验证明该方法可用于抗病毒药物的活性评估和筛选。
参考文献
1:chooql,kuog,weineraj,overbylr,bradleydw,&houghtonm(1989).isolationofacdnaclonederivedfromablood-bornenon-a,non-bviralhepatitisgenome.science,244(4902),359-362.
2:dum,liuj,chenx,xiey,yuanc,xiangy,sunb,lank,chenm,jamessj,zhangy,zhongj,xiaoh(2015).caseinkinaseiicontrolstbk1/irf3activationinifnresponseagainstviralinfection.jimmunol,194(9):4477-88
3:huz,lankh,hes,swaroopm,hux,southalln,zhengw,liangtj(2013).novelcell-basedhepatitiscvirusinfectionassayforquantitativehigh-throughputscreeningofanti-hepatitiscviruscompounds.antimicrobagentschemother,58(2):995-1004
4:jonesct,catanesemt,lawlm,khetanisr,syderaj,plossa,ricecm(2010).real-timeimagingofhepatitiscvirusinfectionusingafluorescentcell-basedreportersystem.natbiotechnol,28(2),167-171.doi:10.1038/nbt.1604
5:marcusdorner,joshuaa.horwitz,justinb.robbins,waltert.barry,qianfeng1,kathymu,christophert.jones,johnw.schoggins,mariateresacatanese,dennisr.burton,mansunlaw,charlesm.rice&alexanderploss(2013).ageneticallyhumanizedmousemodelforhepatitiscvirusinfection.nature,474(7350):208-11.doi:10.1038/nature10168.
6:xiaof,fofanai,heydmannl,barthh,souliere,habersetzerf,doffoelm,bukhj,patelah,zeiselmb,baumerttf(2014).hepatitiscviruscell-celltransmissionandresistancetodirect-actingantiviralagents.plospathog,10(5):e1004128
7:li,xd.sun,l.seth,rb.pineda,g.chen,zj(2005).hepatitiscvirusproteasens3/4acleavesmitochondrialantiviralsignalingproteinoffthemitochondriatoevadeinnateimmunity.procnatlacadsciusa,102(49):17717-22
8:yang,y.liang,y.qu,l.chen,z.yi,m.li,k.lemon,sm(2007).disruptionofinnateimmunityduetomitochondrialtargetingofapicornaviralproteaseprecursor.procnatlacadsciusa,104(17):7253-8
9:wang,b.xi,x.lei,x.zhang,x.cui,s.wang,j.jin,q.zhao,z(2013).enterovirus71protease2aprotargetsmavstoinhibitanti-viraltypeiinterferonresponses.plospathog,9(3):e1003231
10:zhong,j.gastaminza,p.cheng,g.kapadia,s.kato,t.burton,d.r.wieland,s.f.uprichard,s.l.wakita,t.chisari,f.v(2005).robusthepatitiscvirusinfectioninvitro.procnatlacadsciusa,102(26):9294-9
11:lindenbach,b.d.evans,m.j.syder,a.j.wolk,b.tellinghuisen,t.l.liu,c.c.maruyama,t.hynes,r.o.burton,d.r.mckeating,j.a.rice,c.m(2005).completereplicationofhepatitiscvirusincellculture.science,309(5734):623-6
12:poenisch,m.metz,p.blankenburg,h.ruggieri,a.lee,j.y.rupp,d.rebhan,i.diederich,k.kaderali,l.domingues,f.s.albrecht,m.lohmann,v.erfle,h.bartenschlager,r(2015).identificationofhnrnpkasregulatorofhepatitiscvirusparticleproduction.plospathog,11(1):e1004573
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
- 还没有人留言评论。精彩留言会获得点赞!