与芥菜型油菜多室性状相关的两室基因BjMc1和三室基因Bjmc1及其应用的制作方法
本发明属于油菜育种技术领域。具体涉及与芥菜型油菜多室性状相关的两室基因BjMc1和三室基因Bjmc1及其应用。本发明涉及的隐性基因Bjmc1是显性基因BjMc1的功能缺陷突变体。芥菜型油菜两室基因BjMc1的克隆、验证和应用可以加快三室性状在油菜等作物的新品种的选育中的应用,从而加快油菜高产量新品种的选育。
技术背景:
油菜是世界四大主要油料作物之一,在我国种植已有两千多年的历史,在食用油中占有重要的地位,自20世纪90年代以来,我国已成为世界油菜种植面积最大的国家。随着人们生活水平的提高,对优质菜籽油的需要也越来越大,但是我国当前油菜的生产量远不能满足大众的需求。因此提高油菜的产量是当前油菜遗传改良和育种中的主要目标。对于油菜高产品种的选育,角果是其中最为关键的一环。作为影响油菜产量的直接器官,对于角果性状的研究显得极其重要。多室油菜与两室油菜相比,多室油菜的单株产量要远高于两室油菜,这是由于多室油菜与两室油菜的单株角果数和千粒重相差不大,但是多室油菜的每角果粒数要显著高于两室油菜(吕泽文,2011)。在当前油菜的遗传改良中,衡量油菜单位面积产量构成的主要因素有:单位面积有效角果数、每角果粒数和千粒重。而增加每角果粒数和千粒重是新品种培育的重点(祝丽霞,2010)。且当前油菜新品种的发展趋势是向着直播、密植和机械化的方向发展(宋稀等,2010)。所以提高油菜单株产量是培育油菜新品种,实现油菜高产稳产的一个必经途径之一。
目前研究发现,在白菜型油菜、芥菜型油菜和甘蓝型油菜中均发现了角果的多室性状(何余堂等,2003;朱彦涛,2005;赵文路,2007;蒙祖庆和宋丰萍,2012;Paritosh et al.2013;Xiao et al.2013;Xu et al.2014;Fan et al.2014)。一般认为该白菜型多室角果性状受一对隐性核基因控制(何余堂等,2003;Fan et al.2014),芥菜型油菜的多室性状是受两对独立遗传的隐性核基因的控制(Choudhary和Solanki,2007;Xiao et al.2013;Xu et al.2014)。目前,仅见控制白菜型油菜的多室基因BrCLV3被克隆(Fan et al.2014),芥菜型油菜和甘蓝型油菜未见相关多室基因克隆报道。在三大类型油菜中,甘蓝型油菜较其它品种明显具有抗病、耐寒、耐湿、耐肥,产量高而稳定的特点,因此被广泛种植。尽管甘蓝型油菜的多室性状已经被发现,但是甘蓝型油菜的多室性状并不稳定,存在严重的嵌合现象(即两室角果和多室角果并存于同一个植株上),而且有关甘蓝型油菜多室性状的分子标记和基因克隆也未有公布,所以将甘蓝型油菜的多室性状应用到育种中培育高产新品种还有很长的路要走。因此,可将白菜型油菜和芥菜型油菜的多室基因导入到甘蓝型油菜以获得新品种。但是,三大类型油菜存在远缘杂交不亲和的现象,难以通过传统的杂交育种手段来实现高产品种的培育。
目标基因的克隆及通过现代科技手段将目的基因转移到甘蓝型油菜中,成为培育新的高产新品种的新方法。白菜型油菜和甘蓝型油菜的多室性状均存在嵌合现象,不利于新的高产品种的选育。在本发明中,申请人发现,在芥菜型油菜中,Bjmc1基因控制的三室性状稳定,不存在嵌合现象。因此BjMc1基因和Bjmc1基因的克隆及功能研究对甘蓝型油菜高产新品种的培育具有重大的理论意义和实用价值。
技术实现要素:
本发明的一个目的在于提供了一种与芥菜型油菜多室性状相关的两室基因BjMc1,其序列为SEQ ID NO.1所示,编码的蛋白质为SEQ ID NO.4所示,利用两室基因BjMc1可以将芥菜型油菜三室品种J163-4的三室角果恢复为两室角果。
本发明的另一个目的在于提供了一个芥菜型油菜三室基因Bjmc1,其序列为SEQ ID NO.3所示,编码的蛋白质为SEQ ID NO.4所示,利用三室基因Bjmc1可增加拟南芥的多室品种的心皮数目。
本发明的还有一个目的是提供了一种与芥菜型油菜多室性状相关的分子标记M1-1引物,所述的引物为:
M1-1L:GCCTTTGATTGGGTCAGAACTC
M1-1F:AACTAACCGTTTGATCGCGAC
M1-1R:CTAACGCGTCCAGGACAAACC。
本发明的最后一个目的在于提供了一种与芥菜型油菜多室性状相关的分子标记M1-1引物在油菜多室品种的选育中的应用。
为了达到上述目的,本发明采取以下技术措施:
申请人通过图位克隆技术,从芥菜型油菜中分离得到一种能够将芥菜型油菜三室品种J163-4的三室角果恢复为两室角果的两室基因BjMc1(来源于芥菜型油菜两室品种J248),其核苷酸序列为SEQ ID NO:1所示,编码的氨基酸序列如序列表SEQ ID NO:2所示;同时分离得到一种芥菜型油菜三室基因Bjmc1(来源于芥菜型油菜三室品种J163-4),其核苷酸序列如序列表SEQ ID NO:3所示。该基因编码的氨基酸序列如序列表SEQ ID NO:4所示。
基因序列分析发现,芥菜型油菜三室基因Bjmc1的产生是由于在三室油菜中原来的两室基因BjMc1被一个长度为4961bp的copia-LTR型逆转录酶转座子的插入,从而导致了该基因的转录中断造成的,该copia-LTR型逆转录酶转座子如序列表SEQ ID NO:5所示。
申请人通过两室基因BjMc1、三室基因Bjmc1以及copia-LTR型逆转录酶转座子的序列开发分子标记M1-1,所述的分子标记的引物包括:
M1-1L:GCCTTTGATTGGGTCAGAACTC
M1-1F:AACTAACCGTTTGATCGCGAC
M1-1R:CTAACGCGTCCAGGACAAACC。
在本发明的实施例部分,申请人阐述了显性基因BjMc1和隐性基因Bjmc1的克隆、功能验证和分子标记的开发及应用过程以及这两个基因的特点。
与现有技术相比,本发明具有以下优点:
本发明所提供的芥菜型油菜两室基因BjMc1和三室基因Bjmc1均是首次从芥菜型油菜中克隆所得,可利用本发明的技术对油菜两室基因进行基因工程改造来获得产量较高的三室油菜。本发明所提供的分子标记M1-1可以分别特异性扩增两室基因BjMc1和三室基因Bjmc1,运用分子标记进行具备三室性状的芥菜型油菜的辅助选择可以克服传统育种中依靠表型进行选择的特点,减少育种工作量,缩短育种年限,加快了芥菜型多室油菜育种的进程。
附图说明
图1为本发明鉴定、分离、克隆并验证芥菜型油菜两室基因BjMc1和它的基因功能的流程图。
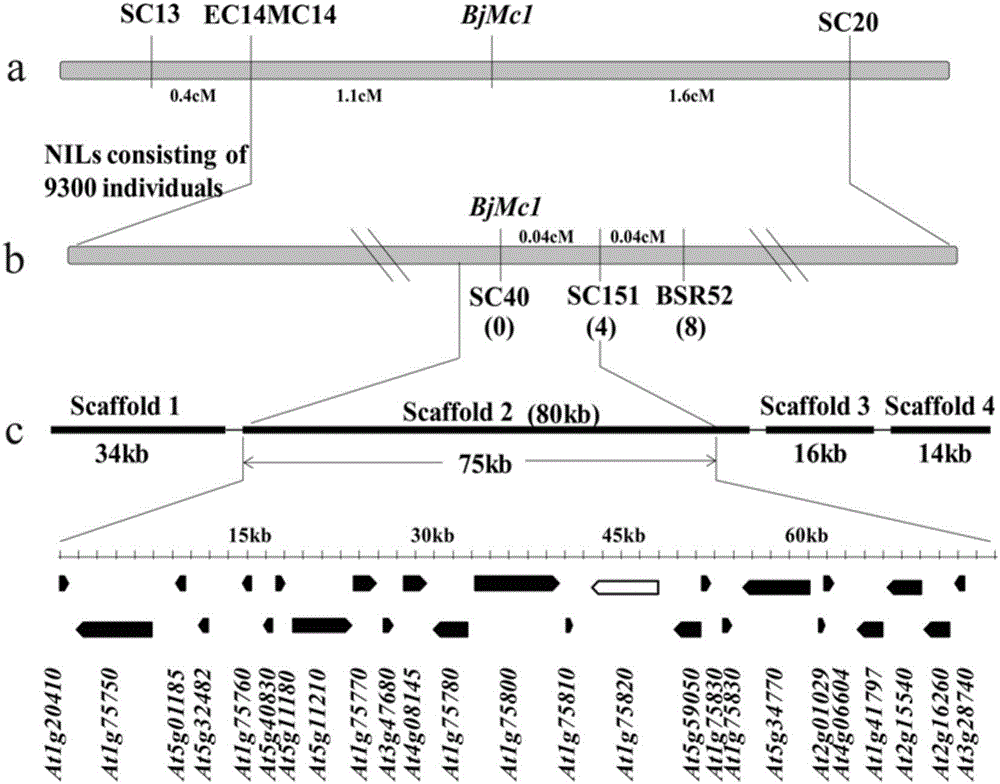
图2为覆盖芥菜型油菜两室基因BjMc1区段的物理图谱。
图中:a表示BjMc1基因初定位的遗传图谱;b表示BjMc1基因精细定位的遗传图谱;分子标记下面的数字表示在近等基因系群体中检测到的BjMc1位点与分子标记之间发生重组交换的单株数;c表示细菌人工染色体(bacterial artificial chromosome,BAC)克隆。刻度线表示菌人工染色体(bacterial artificial chromosome,BAC)克隆序列中,候选基因所在的scaffold2的部分序列。刻度线下面的五边形对应的是相应位置上拟南芥的同源基因,其中,空心五边形表示候选基因及其对应的拟南芥同源基因。
图3为芥菜型油菜功能互补测验过表达载体p35S::BjMc1的构建图。
图4为具有J163-4遗传背景的T0代遗传转化芥菜型油菜植株角果表型与J163-4和J248表型的比较。
结果显示:J163-4遗传转化T0代植株携带显性基因BjMc1的候选基因,角果表现为两室性状。
图5为三室基因Bjmc1过表达载体p35S::Bjmc1构建图。
图6为拟南芥突变体clv1-1的角果表型和以clv1-1为受体的p35S::Bjmc1载体的转基因T0代植株AT-TG100角果表型。
图7为拟南芥突变体clv1-1的角果表型和以clv1-1为受体的p35S::BjMc1载体的转基因T0代植株AT-TG200角果表型。
图8为利用分子标记M1-1的引物在芥菜型油菜两室株系和三室株系及F1的基因组DNA的扩增结果。图中,P1代表两室芥菜型油菜J248,P2代表三室芥菜型油菜J163-4,BC5F1代表J248为母本J163-4为父本的回交5代单株,M代表DNA marker(片段大小依次为:5000bp、3000bp、2000bp、1000bp、750bp)。
具体实施方式
以下结合具体实施例,进一步定义本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的试验方法,通常按照常规条件如《分子克隆:实验室指南》(New York:Cold Spring Harbor Laboratory,1989)中所述的条件,或者按照生产厂商提供的操作手册中建议的条件。
实施例1:
芥菜型油菜两室基因BjMc1和三室基因Bjmc1序列的获得:
1.实验材料
本实验所用的材料为芥菜型油菜两室品种J248和芥菜型油菜三室品种J163-4作为亲本构建了BC4F1群体进行了芥菜型油菜两室基因BjMc1的初定位(Xu et al.2014)。在此基础上,继续以近等基因系中的两室植株为供体亲本,三室植株为轮回亲本,通过兄妹交后得到近等基因系定位群体。
该近等基因系定位群体是由BC5F1近等基因系(3700个单株)和BC6F1近等基因系(5600个单株)构成。近等基因系的单株DNA提取方法参见:Stewart and Via,1993,14(5):748-749。
2.筛选包含BjMc1基因区段的BAC克隆
根据初步定位的结果该基因位于AFLP(Amplified fragment lengthpolymorphism)标记EC14MC14和SCAR(Sequence characterized amplified region)标记SC20之间(Xu et al.2014)。即BjMc1位点位于EC14MC14和SC20之间。利用芥菜型油菜品种紫叶芥的细菌人工染色体(bacterial artificial chromosome,BAC)文库,本发明筛选到了一个包含BjMc1基因位点的BAC克隆。用于筛选BAC克隆的引物序列如下:
C1-1正向引物GGAGGAGCGATCACGGATTCAC
C1-1反向引物AGCAAGCCCAAAATCAGCAACA
3.包含目标基因BAC克隆的测序和序列拼接
筛选到的目标克隆83D02利用TruSeq DNA Sample Prep v2Kit(Illumina,San Diego,USA)构建Illumina标准DNA文库,利用Illumina Hiseq 2000进行测序。测序后的序列过滤低质量、接头和重复序列后,剩余接近100倍覆盖率的高质量序列利用SOAPdenovo软件进行拼接。最终测序结果获得4个Scaffolds序列,分别为Scaffold1(34kb)、Scaffold2(80kb)、Scaffold3(16kb)和Scaffold4(14kb)(图2)。用于精细定位的引物多核苷酸序列见表1所示。
表1精细定位的引物多核苷酸序列
注:引物名称中最后一个英文字母含义分别为:L为正向引物;R为反向引物。
4.精细定位两室基因BjMc1
为了进一步缩小目标基因区域的范围,基于BAC克隆获得的scaffolds的序列开发分子标记,发现有1个SSR分子标记(BSR52)和两个SCAR(SC40和SC151)分子标记在两室和三室材料之间表现出多态性。分析发现,分子标记SC40与目标基因共分离,BSR52和SC151位于目标基因的同一侧(图2)。
预测SC151所位于的Scaffold2的75kb序列(且该序列包含共分离标记SC40所在位点)区段的开放阅读框(open reading frame,ORF),并用拟南芥的同源基因进行注释分析。进一步分析发现,共分离分子标记SC40的位点正好位于拟南芥的心皮发育相关的基因CLV1(At1g75750)所注释的ORF上,而且只有这一个基因与心皮发育相关,所以该ORF被确定为两室基因BjMc1的候选基因,命名为BjCLV1。
5.候选基因BjCLV1确定为芥菜型油菜的两室基因BjMc1
本实施例采用改造后含有2×35S启动子的pCAMBIA2300载体(即将2×35S启动子逆时针方向插入到植物表达载体pCAMBIA2300的HindⅢ和Pst Ⅰ酶切位点)作为芥菜型油菜转基因载体。通过分析携带候选基因的BAC克隆序列的酶切位点和候选基因的关系,以BC5F1群体中的两室单株的基因组DNA为模板,利用高保真PCR方法扩增获得一个3.9kb的片段,该片段不包含Pst Ⅰ和Kpn Ⅰ酶切位点。用于扩增该片段的引物序列如下:
E48-2正向引物:CGATTCACGCTTCCTCAACACATACCTT
E48-2反向引物:ATGGAGATGAGACTTTTGAAAACACACCT。
片段起始端包括基因区间3154bp的序列和下游非翻译区767bp的序列。将扩增片段克隆到pMD18-T载体(购自宝生物工程(大连)有限公司)上,测序结果表明序列完全准确,没有错配碱基存在。提取正确克隆的质粒,用Pst Ⅰ和Kpn Ⅰ双酶切后,回收3.9kb的片段,连接到Pst Ⅰ和Kpn Ⅰ双酶切的改造后的表达载体pCAMBIA2300上,连接产物转化大肠杆菌菌株Trans5α(购自全式金公司),在含有50ug/ml卡那霉素的LB培养基上筛选转化子,挑选单菌落提取质粒,并且通过测序检测确定核苷酸序列完全正确,成功构建了转化植株的载体p35S::BjMc1(见图3)。正确的重组质粒通过冻融法(HOLSTERS M等.1978,Transfection and transformation of Agrobacterium tumefaciens.Mol Gen Cent,183:181-187)导入农杆菌菌株GV1301.油菜遗传转化在常规农杆菌转化法(陈苇等,2006)的基础上加以改进,转化芥菜型油菜三室品种J163-4。种子灭菌用75%酒精浸泡3min后,放入无菌水中冲洗1min。然后放入0.1%升汞中浸泡10min后,无菌水冲洗6-7次。将灭菌后的种子播种到种子萌发培养基上(所述的种子萌发培养基为:MS基本培养基+7g/L琼脂粉,pH值为5.9)。于25℃按培养7天。在超净工作台内将获得的试管幼苗下胚轴切成1cm左右的小段外植体,接种至含有农杆菌(悬浮过夜至对数生长期)的MS液体培养基(pH值为5.9)侵染10-15min后,吸干液体,于25℃黑暗条件下在共培养基上(所述的共培养基包括:MS培养基+30g/L蔗糖+18g/L甘露醇+1mg/L 2,4-D+0.3mg/L Kinetin+100um/L乙酰丁香酮+6g/L琼脂粉,pH值为5.9)共培养36h。将外植体转移到愈伤诱导培养基上(所述的愈伤诱导培养基包括:MS培养基+30g/L蔗糖+18g/L甘露醇+1mg/L 2,4-D+0.3mg/L Kinetin+300mg/L特美汀+50mg/L卡那霉素+0.03uM/L[Ag(NO3)2]3-+6g/L琼脂粉,pH值为5.9),25℃光照培养12-14d。将外植体转入不定芽诱导培养基上(所述的不定芽诱导培养基包括:MS培养基+30g/L蔗糖+1.5g/L TDZ+0.1g/L NAA+300mg/L特美汀+50mg/L卡那霉素+5.0mg/L硝酸银+7g/L琼脂粉,pH值为5.9),25℃光照培养12-14d。获得的带有不定芽点的愈伤组织转移到不定芽生长培养基中,进行生长培养。还未生长出不定芽点的外植体,则继续放到不定芽诱导培养基中进行诱导培养,待长出不定芽点后,转移到不定芽生长培养基中(所述的不定芽生长培养基包括:MS培养基+10g/L葡萄糖+0.25g/L木糖+0.6g/L MES+2.0mg/L反式-Zeatin+0.1mg/L IAA+300mg/L特美汀+25mg/L卡那霉素+6g/L琼脂粉,pH值为5.9)。每15-20d继代一次,直至生长成幼苗。当幼苗长至2-3cm高时,将幼苗转入生根培养基中培养(所述生根培养基包括:MS培养基+10g/L蔗糖+10g/L琼脂粉,pH值为5.9)。对已生根的小苗进行炼苗,然后按常规技术移栽到温室里,进行土培生长。
从获得阳性植株上提取叶片总DNA(参见:Doyle JJ,Doyle JL.Isolation of plant DNA from fresh tissue.Focus,1990,12:13-15),用引物M1-1L和M1-1R通过PCR进一步鉴定转化植株。鉴定引物序列如下:
M1-1L:GCCTTTGATTGGGTCAGAACTC
M1-1R:CTAACGCGTCCAGGACAAACC。
在转基因T0代观察角果表型,验证导入的候选基因的功能。结果显示在27株转基因T0代植株中,有16株表现为形成两室角果(见图4)。将T0代植株自交后收货T1代种子,将T1代播于温室中,结角果后调查角果的表型,并提取叶片总DNA,用引物M1-1L和M1-1R通过PCR鉴定,结果显示在T1代植株中,所有的阳性苗均表现为两室角果性状。PCR扩增的反应体系是:30ng模板DNA,各35ng正向和反向特异引物,1×PCR缓冲液,1UTaq酶,0.15mmol/L dNTPs和2.0mmol/L Mg2+,加ddH2O到总体积为15ul。PCR循环参数为94℃(30s)—55℃(30s)—72℃(90s),共35个循环。1.0%琼脂凝胶检测扩增产物。
最终可确定候选基因BjCLV1即为芥菜型油菜的两室基因BjMc1,其核苷酸序列为SEQ ID NO:1所示,编码的氨基酸序列如序列表SEQ ID NO:2所示。
另外,利用针对两室基因BjMc1的序列设计引物M2,使用BC5F1群体中的三室植株的基因组DNA为模板,利用高保真PCR方法扩增,得到三室基因Bjmc1,其核苷酸序列为SEQ ID NO:3所示,编码的氨基酸序列如序列表SEQ ID NO:4所示。引物序列如下:
M2L:GCCTTTGATTGGGTCAGAACTC
M2R:ATGGAGATGAGACTTTTGAAAACAC。
扩增的片段克隆到pMD18-T载体(购自宝生物工程(大连)有限公司)上进行测序。测序结果表明:三室基因Bjmc1与两室基因BjMc1的基因结构相比较,Bjmc1在第一个外显子区域有一个4961bp(其核苷酸序列为SEQ ID NO:5所示)的转座子序列插入。
实施例2:
三室基因Bjmc1和两室基因BjMc1在拟南芥中的遗传转化:
1.三室基因Bjmc1在拟南芥中的遗传转化
三室基因Bjmc1过表达载体p35S::Bjmc1的骨架选用改造后的pMDC83载体(所述的改造后的pMDC83载体是经Sca I酶切后又连接的pMDC83载体),该载体在植物体内抗性为潮霉素,在细菌体内的抗性为卡那霉素。从BC5F1群体的三室植株基因组DNA中分离目标基因Bjmc1的片段3036bp,其中包括终止密码子下游687bp的片段。扩增引物为:
HY-4L:ATGGAGATGAGACTTTTGAAAACAC
HY-4R:CTGCAAGACGGACTGAGTTAACA。
克隆片段从逆时针方向插入经改造后的pMDC83载体的Xba I-Sac I位点(图5)。基因片段克隆操作与实施例1载体构建一致,得到p35S::Bjmc1载体。
拟南芥的遗传转化使用农杆菌菌液蘸花,通过花粉管通道侵染的方式(Clough SJ and Bent AF 1998)。使用p35S::Bjmc1载体侵染拟南芥突变体clv1-1,该突变体从The Arabidopsis Information Resource(http://www.arabidopsis.org/)引进。拟南芥clv1-1突变体的角果为4个果瓣。侵染后收获拟南芥种子,并在含有潮霉素抗性的MS培养基上播种,筛选有抗性的阳性苗。大约14天后,将阳性苗转移到温室进行土培,同时观察转基因T0代观察角果表型,验证导入的候选基因的功能。结果显示,在62株转基因阳性苗中,25株的植株上面表现出5个果瓣的角果,即角果在发育的过程中形成了5个心皮(图6)。将T0代植株自交后播于温室中,结角果后调查角果的表型,并提取叶片总DNA,用引物M2-1L和M2-1R通过PCR进一步鉴定转化植株。鉴定引物序列如下:
M2-1L:CCACTATCCTTCGCAAGACC
M2-1R:CCGTTGTGACCAGTCATTGAGG。
PCR扩增的反应体系是:30ng模板DNA,各35ng正向和反向特异引物,1×PCR缓冲液,1U Taq酶,0.15mmol/L dNTPs和2.0mmol/L Mg2+,加ddH2O到总体积为15ul。PCR循环参数为94℃(30s)—55℃(30s)—72℃(45s),共35个循环。1.0%琼脂凝胶检测扩增产物。能够扩增出条带,则为阳性植株。明确在拟南芥clv1-1突变体中过表达Bjmc1基因后获得的五果瓣角果可以在后代中稳定遗传。
2.两室基因BjMc1在拟南芥中的遗传转化
使用含有p35S::BjMc1载体(即将两室基因BjMc1从逆时针方向插入经改造后的pMDC83载体的Xba I-Sac I位点)的农杆菌侵染拟南芥突变体clv1-1,侵染及阳性苗的筛选过程与p35S::Bjmc1载体的筛选过程相同。结果显示,共获得32株阳性苗,其中17株的阳性面表现出两室角果的表型(图7)。将T0代植株自交后播于温室中,结角果后调查角果的表型,并提取叶片总DNA,用引物M1-1L和M1-1R通过PCR进一步鉴定转化植株,确定在拟南芥clv1-1突变体中过表达BjMc1基因后获得的两室表型可以在后代中稳定遗传。
实施例3:
一种与芥菜型油菜多室性状相关的分子标记M1-1的获得:
本发明根据已获得的BjMc1基因、Bjmc1基因、copia-LTR逆转录酶转座子序列及其在Bjmc1基因中的插入位点,开发共分离分子标记M1-1。预期M1-1(包含M1-1L、M1-1F和M1-1R三条引物)在PCR的条件下扩增芥菜型两室油菜J248和芥菜型三室油菜J163-4的基因组DNA,在J248中扩增出大约1200bp的片段,而在J163-4中扩增出大约250bp的片段。
本发明利用上述步骤设计的引物进行PCR扩增。PCR的反应体系如下:1×PCR buffer,1.35mM MgCl2,0.08mM dNTPs,1.0U DNA聚合酶(均购自MBI Fermentas,Lithuanin公司),100ng DNA,两条正向引物M1-1L和M1-1F各加0.5uM,反向引物加1uM,ddH2O补充至终体积20ul。热循环参数为:94℃5min;94℃30sec,55℃30sec,72℃90sec,35个循环;72℃10min,1个循环;4℃保存,反应是在PTC-ALD1244PCR仪上完成。扩增产物在水平电泳槽上1.0%琼脂糖凝胶电泳检测。引物对M1-1在芥菜型两室油菜J248的基因组DNA中扩增出1200bp左右的片段,在芥菜型三室油菜J163-4的基因组DNA中扩增出250bp左右的片段,而在F1植株的基因组DNA中两个片段均能扩增出来(图8)。因此该引物M1-1可以作为芥菜型油菜三室性状的共显性分子标记。
实施例4.
一种与芥菜型油菜多室性状相关的分子标记M1-1在有才多室育种中的应用:
BC5F1群体中随机挑选200个植株,利用共显性标记M1-1进行分析。结果证明,其中104个单株与亲本J248中扩增片段一样(即利用引物M1-1L、M1-1F和M1-1R扩增有两个条带,记为“A带型”),另外96个单株与亲本J163-4中扩增片段一样(即利用引物M1-1L、M1-1F和M1-1R扩增有一个条带,记为“B带型”)(表2)。通过分析表中数据可知,凡是M1-1标记进行检测,检测结果为“A带型”的,其角果表型均为两室性状,而检测结果为“B带型”的其角果表型为三室性状。这说明在回交群体中该标记是与芥菜型油菜三室性状共分离的。因此,利用引物对M1-1可在早期有效鉴定出含有两室性状的芥菜型油菜单株,并提高了选择的有效性和准确性。
表2本发明中芥菜型油菜BC5F1群体中角果性状考察及M1-1标记检测结果
SEQUENCE LISTING
<110> 华中农业大学
<120> 与芥菜型油菜多室性状相关的两室基因BjMc1和三室基因Bjmc1及其应用
<130> 与芥菜型油菜多室性状相关的两室基因BjMc1和三室基因Bjmc1及其应用
<160> 8
<170> PatentIn version 3.1
<210> 1
<211> 3154
<212> DNA
<213> 人工序列
<400> 1
atggagatga gacttttgaa aacacacctt ctgtttctgc atcttcacta cgttatctcg 60
attttgcttc tatgtttctc accatgcttc gcttccactg acatggacca tctcctcaac 120
ctcaaatcct caatgactgg tcacaacgga cacggtctcc acgactgggt tcactccact 180
tctcccacgg ctcactgttc tttctccggc gtctcatgcg acgccgacgc tcgtgttgtc 240
tcactcaacg tctctttcac tcctctgttc ggaaccatct caccggagat tgggatgctg 300
aaccgtttgg tgaatctgac gttagccgcg aataacttct ccggtaggtt gccgctggag 360
atgaagagtc tcacttcact aaaggttctc aacatctcca acaacgtaaa cctcaacgga 420
accttccccg gagagattct cacttccatg gtcgacctcg aagtcctcga cgcctataac 480
aacaacttca ctgggccgtt accgccggag attcccgggc tgaacaagct caaacacctc 540
tctctcggag gaaacttctt caccggagag attcccgaga gttacggaga tatccaaagc 600
ttggagtatc tcggcctcaa cggagccgga ctctccggcg aatctccggc gttcttgtca 660
cgtctcaaga atctcagaga aatgtacgtc ggctacttca acagctacac cggcggcgtt 720
ccgccggagt tcggcgaatt aacgaagcta gaaatcctcg acatggcgag ctgtactctc 780
accggagaga ttccgacgac actgagtaat ctgaaacact tgcacacgct gttcctccac 840
atcaacaact taaccggaaa catcccacca gaactctccg gtttaatcag cttaaaatct 900
ctcgatctat caattaacca gctaaccgga gagattcctc agagcttcat ctctctggga 960
aacatcactc tcatcaacct cttcagaaac aacctccacg ggccgatacc ggagttcatc 1020
ggagaaatgc cgaacctcca agtcttccag gtgtgggaga acaacttcac gttagagtta 1080
ccggcgaata tcggccggaa cgggaatctg aaaaagctcg acgtctctga aaaccatctc 1140
accggactca tccccgtgga tttatgcaga ggtgggaagc tggaggttct ggtactctcc 1200
aacaacttct tcttcggctc catcccggag aagctaggtc aatgcaaatc tctaaacaag 1260
atcagaatcg tcaagaatct cctcaacggg accgttccgg cgggactatt caacttacca 1320
ctcgttacaa tcatcgagct cgcggataac ttcttctccg gggaacttcc tacggagatg 1380
tccggcgacg ttctcgatca tatctaccta tctaacaact ggtttaccgg tttaatccct 1440
ccggctatcg gtaatttcaa aaatctacag gatctgttct tagaccggaa ccggtttagc 1500
gggaatattc cgagagaagt tttcgagctg aagcatctca cgaagatcaa cacgagcgct 1560
aacaacctaa ccggcgatat ccctgactca ttctcgcgat gcacttcctt aatctccgtc 1620
gatctcagcc gtaacagaat cggcggagat atcccgaaag acatccacga cgtgattaac 1680
ttaggaactc tcaatctctc cgggaatcag ctcaccggct cgatcccgat cggaatcggg 1740
aagatgacga gcttaaccac tctcgatctc tccttcaacg acctctccgg gagagtacca 1800
ctcggcggtc agttcctagt cttcaacgac acttccttcg ccggaaaccc ttacctctgc 1860
ctccctcacc acgcctcctg cctaacgcgt ccaggacaaa cctctgatcg catccacgcg 1920
gcgctgttct ctccgtcgag gatcgtcatc acgatcatcg cggcgatcac ggcgttgatc 1980
ctcatcagcg tcgcgatccg tcagatgaac aagaagaaac acgagagatc cctctcgtgg 2040
aagctaaccg ccttccagcg actcgatttc aaggcggaag acgtcctcga gtgccttcaa 2100
gaagagaaca taatcggcaa aggcggagcg gggatcgtct accgcggatc catgccgaac 2160
aacgtagacg tcgcgatcaa acggttagtt ggacgcggaa cggggaggag cgatcacgga 2220
ttcacggcgg agattcagac gttaggaaga atccgccacc gtcatatagt gagactactc 2280
ggatacgtgg cgaacaagga cacgaacctg cttctatacg agtacatgcc taacgggagc 2340
ctcggcgagc ttttgcacgg gtctaaaggc ggtcatcttc agtgggagac gaggcacaga 2400
gtagccgttg aagcggcgaa aggactgtgt tatcttcacc atgactgttc gccgttgatc 2460
ttgcacagag acgttaagtc caataacatt ctcctggact ctgatttcga agcccatgtt 2520
gctgattttg ggcttgctaa gttcttagtg gacggtgctg cttctgagtg tatgtcctca 2580
atagctggct cctacggtta catcgctcca ggttagttta aacgtgtttt atataacaaa 2640
ctatgccatg ttctttaaat gttactgaga ctaatcatat gttagagttt gttgtccgtt 2700
ttattgtcgt atagctgatt actatgattt gtgacacaca aatgaatgtg actttaacaa 2760
ctaaaatatg tactaatcat tgtttgtttt gggtttgata gagtatgcat acactctcaa 2820
agtagacgag aagagtgatg tgtatagctt tggagtggtc ttgttggagc tgatagctgg 2880
gaagaaaccg gttggtgagt ttggggaagg agtggatata gtgaggtggg tgaggaacac 2940
ggagggtgag atacctcagc cttcggatgc agctactgtt gttgcgatcg ttgacccgag 3000
gttgactggt tacccgttga ccagtgtgat tcatgtgttc aagatagcga tgatgtgtgt 3060
ggaggatgag gccgcgtcaa ggcctacgat gagggaagtt gtgcacatgc taactaactc 3120
tcctaagtcc gtgactaact tgatcgcctt ctga 3154
<210> 2
<211> 987
<212> PRT
<213> 人工序列
<400> 2
Met Glu Met Arg Leu Leu Lys Thr His Leu Leu Phe Leu His Leu His
1 5 10 15
Tyr Val Ile Ser Ile Leu Leu Leu Cys Phe Ser Pro Cys Phe Ala Ser
20 25 30
Thr Asp Met Asp His Leu Leu Asn Leu Lys Ser Ser Met Thr Gly His
35 40 45
Asn Gly His Gly Leu His Asp Trp Val His Ser Thr Ser Pro Thr Ala
50 55 60
His Cys Ser Phe Ser Gly Val Ser Cys Asp Ala Asp Ala Arg Val Val
65 70 75 80
Ser Leu Asn Val Ser Phe Thr Pro Leu Phe Gly Thr Ile Ser Pro Glu
85 90 95
Ile Gly Met Leu Asn Arg Leu Val Asn Leu Thr Leu Ala Ala Asn Asn
100 105 110
Phe Ser Gly Arg Leu Pro Leu Glu Met Lys Ser Leu Thr Ser Leu Lys
115 120 125
Val Leu Asn Ile Ser Asn Asn Val Asn Leu Asn Gly Thr Phe Pro Gly
130 135 140
Glu Ile Leu Thr Ser Met Val Asp Leu Glu Val Leu Asp Ala Tyr Asn
145 150 155 160
Asn Asn Phe Thr Gly Pro Leu Pro Pro Glu Ile Pro Gly Leu Asn Lys
165 170 175
Leu Lys His Leu Ser Leu Gly Gly Asn Phe Phe Thr Gly Glu Ile Pro
180 185 190
Glu Ser Tyr Gly Asp Ile Gln Ser Leu Glu Tyr Leu Gly Leu Asn Gly
195 200 205
Ala Gly Leu Ser Gly Glu Ser Pro Ala Phe Leu Ser Arg Leu Lys Asn
210 215 220
Leu Arg Glu Met Tyr Val Gly Tyr Phe Asn Ser Tyr Thr Gly Gly Val
225 230 235 240
Pro Pro Glu Phe Gly Glu Leu Thr Lys Leu Glu Ile Leu Asp Met Ala
245 250 255
Ser Cys Thr Leu Thr Gly Glu Ile Pro Thr Thr Leu Ser Asn Leu Lys
260 265 270
His Leu His Thr Leu Phe Leu His Ile Asn Asn Leu Thr Gly Asn Ile
275 280 285
Pro Pro Glu Leu Ser Gly Leu Ile Ser Leu Lys Ser Leu Asp Leu Ser
290 295 300
Ile Asn Gln Leu Thr Gly Glu Ile Pro Gln Ser Phe Ile Ser Leu Gly
305 310 315 320
Asn Ile Thr Leu Ile Asn Leu Phe Arg Asn Asn Leu His Gly Pro Ile
325 330 335
Pro Glu Phe Ile Gly Glu Met Pro Asn Leu Gln Val Phe Gln Val Trp
340 345 350
Glu Asn Asn Phe Thr Leu Glu Leu Pro Ala Asn Ile Gly Arg Asn Gly
355 360 365
Asn Leu Lys Lys Leu Asp Val Ser Glu Asn His Leu Thr Gly Leu Ile
370 375 380
Pro Val Asp Leu Cys Arg Gly Gly Lys Leu Glu Val Leu Val Leu Ser
385 390 395 400
Asn Asn Phe Phe Phe Gly Ser Ile Pro Glu Lys Leu Gly Gln Cys Lys
405 410 415
Ser Leu Asn Lys Ile Arg Ile Val Lys Asn Leu Leu Asn Gly Thr Val
420 425 430
Pro Ala Gly Leu Phe Asn Leu Pro Leu Val Thr Ile Ile Glu Leu Ala
435 440 445
Asp Asn Phe Phe Ser Gly Glu Leu Pro Thr Glu Met Ser Gly Asp Val
450 455 460
Leu Asp His Ile Tyr Leu Ser Asn Asn Trp Phe Thr Gly Leu Ile Pro
465 470 475 480
Pro Ala Ile Gly Asn Phe Lys Asn Leu Gln Asp Leu Phe Leu Asp Arg
485 490 495
Asn Arg Phe Ser Gly Asn Ile Pro Arg Glu Val Phe Glu Leu Lys His
500 505 510
Leu Thr Lys Ile Asn Thr Ser Ala Asn Asn Leu Thr Gly Asp Ile Pro
515 520 525
Asp Ser Phe Ser Arg Cys Thr Ser Leu Ile Ser Val Asp Leu Ser Arg
530 535 540
Asn Arg Ile Gly Gly Asp Ile Pro Lys Asp Ile His Asp Val Ile Asn
545 550 555 560
Leu Gly Thr Leu Asn Leu Ser Gly Asn Gln Leu Thr Gly Ser Ile Pro
565 570 575
Ile Gly Ile Gly Lys Met Thr Ser Leu Thr Thr Leu Asp Leu Ser Phe
580 585 590
Asn Asp Leu Ser Gly Arg Val Pro Leu Gly Gly Gln Phe Leu Val Phe
595 600 605
Asn Asp Thr Ser Phe Ala Gly Asn Pro Tyr Leu Cys Leu Pro His His
610 615 620
Ala Ser Cys Leu Thr Arg Pro Gly Gln Thr Ser Asp Arg Ile His Ala
625 630 635 640
Ala Leu Phe Ser Pro Ser Arg Ile Val Ile Thr Ile Ile Ala Ala Ile
645 650 655
Thr Ala Leu Ile Leu Ile Ser Val Ala Ile Arg Gln Met Asn Lys Lys
660 665 670
Lys His Glu Arg Ser Leu Ser Trp Lys Leu Thr Ala Phe Gln Arg Leu
675 680 685
Asp Phe Lys Ala Glu Asp Val Leu Glu Cys Leu Gln Glu Glu Asn Ile
690 695 700
Ile Gly Lys Gly Gly Ala Gly Ile Val Tyr Arg Gly Ser Met Pro Asn
705 710 715 720
Asn Val Asp Val Ala Ile Lys Arg Leu Val Gly Arg Gly Thr Gly Arg
725 730 735
Ser Asp His Gly Phe Thr Ala Glu Ile Gln Thr Leu Gly Arg Ile Arg
740 745 750
His Arg His Ile Val Arg Leu Leu Gly Tyr Val Ala Asn Lys Asp Thr
755 760 765
Asn Leu Leu Leu Tyr Glu Tyr Met Pro Asn Gly Ser Leu Gly Glu Leu
770 775 780
Leu His Gly Ser Lys Gly Gly His Leu Gln Trp Glu Thr Arg His Arg
785 790 795 800
Val Ala Val Glu Ala Ala Lys Gly Leu Cys Tyr Leu His His Asp Cys
805 810 815
Ser Pro Leu Ile Leu His Arg Asp Val Lys Ser Asn Asn Ile Leu Leu
820 825 830
Asp Ser Asp Phe Glu Ala His Val Ala Asp Phe Gly Leu Ala Lys Phe
835 840 845
Leu Val Asp Gly Ala Ala Ser Glu Cys Met Ser Ser Ile Ala Gly Ser
850 855 860
Tyr Gly Tyr Ile Ala Pro Glu Tyr Ala Tyr Thr Leu Lys Val Asp Glu
865 870 875 880
Lys Ser Asp Val Tyr Ser Phe Gly Val Val Leu Leu Glu Leu Ile Ala
885 890 895
Gly Lys Lys Pro Val Gly Glu Phe Gly Glu Gly Val Asp Ile Val Arg
900 905 910
Trp Val Arg Asn Thr Glu Gly Glu Ile Pro Gln Pro Ser Asp Ala Ala
915 920 925
Thr Val Val Ala Ile Val Asp Pro Arg Leu Thr Gly Tyr Pro Leu Thr
930 935 940
Ser Val Ile His Val Phe Lys Ile Ala Met Met Cys Val Glu Asp Glu
945 950 955 960
Ala Ala Ser Arg Pro Thr Met Arg Glu Val Val His Met Leu Thr Asn
965 970 975
Ser Pro Lys Ser Val Thr Asn Leu Ile Ala Phe
980 985
<210> 3
<211> 2349
<212> DNA
<213> 人工序列
<400> 3
atggagatga gacttttgaa aacacacctt ctgtttctgc atcttcacta cgttatctcg 60
attttgcttc tatgtttctc accatgcttc gcttccactg acatggacca tctcctcaac 120
ctcaaatcct caatgactgg tcacaacgga cacggtctcc acgactgggt tcactccact 180
tctcccacgg ctcactgttc tttctccggc gtctcatgcg acgccgacgc tcgtgttgtc 240
tcactcaacg tctctttcac tcctctgttc ggaaccatct caccggagat tgggatgctg 300
aaccgtttgg tgaatctgac gttagccgcg aataacttct ccggtaggtt gccgctggag 360
atgaagagtc tcacttcact aaaggttctc aacatctcca acaacgtaaa cctcaacgga 420
accttccccg gagagattct cacttccatg gtcgacctcg aagtcctcga cgcctataac 480
aacaacttca ctgggccgtt accgccggag attcccgggc tgaacaagct caaacacctc 540
tctctcggag gaaacttctt caccggagag attcccgaga gttacggaga tatccaaagc 600
ttggagtatc tcggcctcaa cggagccgga ctctccggcg aatctccggc gttcttgtca 660
cgtctcaaga atctcagaga aatgtacgtc ggctacttca acagctacac cggcggcgtt 720
ccgccggagt tcggcgaatt aacgaagcta gaaatcctcg acatggcgag ctgtactctc 780
accggagaga ttccgacgac actgagtaat ctgaaacact tgcacacgct gttcctccac 840
atcaacaact taaccggaaa catcccacca gaactctccg gtttaatcag cttaaaatct 900
ctcgatctat caattaacca gctaaccgga gagattcctc agagcttcat ctctctggga 960
aacatcactc tcatcaacct cttcagaaac aacctccacg ggccgatacc ggagttcatc 1020
ggagaaatgc cgaacctcca agtcttccag gtgtgggaga acaacttcac gttagagtta 1080
ccggcgaata tcggccggaa cgggaatctg aaaaagctcg acgtctctga aaaccatctc 1140
accggactca tccccgtgga tttatgcaga ggtgggaagc tggaggttct ggtactctcc 1200
aacaacttct tcttcggctc catcccggag aagctaggtc aatgcaaatc tctaaacaag 1260
atcagaatcg tcaagaatct cctcaacggg accgttccgg cgggactatt caacttacca 1320
ctcgttacaa tcatcgagct cgcggataac ttcttctccg gggaacttcc tacggagatg 1380
tccggcgacg ttctcgatca tatctaccta tctaacaact ggtttaccgg tttaatccct 1440
ccggctatcg gtaatttcaa aaatctacag gatctgttct tagaccggaa ccggtttagc 1500
gggaatattc cgagagaagt tttcgagctg aagcatctca cgaagatcaa cacgagcgct 1560
aacaacctaa ccggcgatat ccctgactca ttctcgcgat gcacttcctt aatctccgtc 1620
gatctcagcc gtaacagaat cggcggagat atcccgaaag acatccacga cgtgattaac 1680
ttaggaactc tcaatctctc cgggaatcag ctcaccggct cgatcccgat cggaatcggg 1740
aagatgacga gcttaaccac tctcgatctc tccttcaacg acctctccgg gagagtacca 1800
ctcggcggtc agttcctagt cttcaacgac acttccttcg ccggaaaccc ttacctctgc 1860
ctccctcacc acgcctcctg cctaacgcgt ccaggacaaa cctctgatcg catccacgcg 1920
gcgctgttct ctccgtcgag gatcgtcatc acgatcatcg cggcgatcac ggcgttgatc 1980
ctcatcagcg tcgcgatccg tcagatgaac aagaagaaac acgagagatc cctctcgtgg 2040
aagctaaccg ccttccagcg actcgatttc aaggcggaag acgtcctcga gtgccttcaa 2100
gaagagaaca taatcggcaa aggcggagcg gggatcgtct accgcggatc catgccgaac 2160
aacgtagacg tcgcgatcaa acggttagtt ggacgcggaa cggggaggag cgatcacgga 2220
ttcacggcgg agattcagac gttaggaaga atccgccacc gtcatatagt gagactactc 2280
ggatacgtgg cgaacaagga cacgaacctg cttctatacg agtacatgcc taacgggagc 2340
ctcggctga 2349
<210> 4
<211> 791
<212> PRT
<213> 人工序列
<400> 4
Met Glu Met Arg Leu Leu Lys Thr His Leu Leu Phe Leu His Leu His
1 5 10 15
Tyr Val Ile Ser Ile Leu Leu Leu Cys Phe Ser Pro Cys Phe Ala Ser
20 25 30
Thr Asp Met Asp His Leu Leu Asn Leu Lys Ser Ser Met Thr Gly His
35 40 45
Asn Gly His Gly Leu His Asp Trp Val His Ser Thr Ser Pro Thr Ala
50 55 60
His Cys Ser Phe Ser Gly Val Ser Cys Asp Ala Asp Ala Arg Val Val
65 70 75 80
Ser Leu Asn Val Ser Phe Thr Pro Leu Phe Gly Thr Ile Ser Pro Glu
85 90 95
Ile Gly Met Leu Asn Arg Leu Val Asn Leu Thr Leu Ala Ala Asn Asn
100 105 110
Phe Ser Gly Arg Leu Pro Leu Glu Met Lys Ser Leu Thr Ser Leu Lys
115 120 125
Val Leu Asn Ile Ser Asn Asn Val Asn Leu Asn Gly Thr Phe Pro Gly
130 135 140
Glu Ile Leu Thr Ser Met Val Asp Leu Glu Val Leu Asp Ala Tyr Asn
145 150 155 160
Asn Asn Phe Thr Gly Pro Leu Pro Pro Glu Ile Pro Gly Leu Asn Lys
165 170 175
Leu Lys His Leu Ser Leu Gly Gly Asn Phe Phe Thr Gly Glu Ile Pro
180 185 190
Glu Ser Tyr Gly Asp Ile Gln Ser Leu Glu Tyr Leu Gly Leu Asn Gly
195 200 205
Ala Gly Leu Ser Gly Glu Ser Pro Ala Phe Leu Ser Arg Leu Lys Asn
210 215 220
Leu Arg Glu Met Tyr Val Gly Tyr Phe Asn Ser Tyr Thr Gly Gly Val
225 230 235 240
Pro Pro Glu Phe Gly Glu Leu Thr Lys Leu Glu Ile Leu Asp Met Ala
245 250 255
Ser Cys Thr Leu Thr Gly Glu Ile Pro Thr Thr Leu Ser Asn Leu Lys
260 265 270
His Leu His Thr Leu Phe Leu His Ile Asn Asn Leu Thr Gly Asn Ile
275 280 285
Pro Pro Glu Leu Ser Gly Leu Ile Ser Leu Lys Ser Leu Asp Leu Ser
290 295 300
Ile Asn Gln Leu Thr Gly Glu Ile Pro Gln Ser Phe Ile Ser Leu Gly
305 310 315 320
Asn Ile Thr Leu Ile Asn Leu Phe Arg Asn Asn Leu His Gly Pro Ile
325 330 335
Pro Glu Phe Ile Gly Glu Met Pro Asn Leu Gln Val Phe Gln Val Trp
340 345 350
Glu Asn Asn Phe Thr Leu Glu Leu Pro Ala Asn Ile Gly Arg Asn Gly
355 360 365
Asn Leu Lys Lys Leu Asp Val Ser Glu Asn His Leu Thr Gly Leu Ile
370 375 380
Pro Val Asp Leu Cys Arg Gly Gly Lys Leu Glu Val Leu Val Leu Ser
385 390 395 400
Asn Asn Phe Phe Phe Gly Ser Ile Pro Glu Lys Leu Gly Gln Cys Lys
405 410 415
Ser Leu Asn Lys Ile Arg Ile Val Lys Asn Leu Leu Asn Gly Thr Val
420 425 430
Pro Ala Gly Leu Phe Asn Leu Pro Leu Val Thr Ile Ile Glu Leu Ala
435 440 445
Asp Asn Phe Phe Ser Gly Glu Leu Pro Thr Glu Met Ser Gly Asp Val
450 455 460
Leu Asp His Ile Tyr Leu Ser Asn Asn Trp Phe Thr Gly Leu Ile Pro
465 470 475 480
Pro Ala Ile Gly Asn Phe Lys Asn Leu Gln Asp Leu Phe Leu Asp Arg
485 490 495
Asn Arg Phe Ser Gly Asn Ile Pro Arg Glu Val Phe Glu Leu Lys His
500 505 510
Leu Thr Lys Ile Asn Thr Ser Ala Asn Asn Leu Thr Gly Asp Ile Pro
515 520 525
Asp Ser Phe Ser Arg Cys Thr Ser Leu Ile Ser Val Asp Leu Ser Arg
530 535 540
Asn Arg Ile Gly Gly Asp Ile Pro Lys Asp Ile His Asp Val Ile Asn
545 550 555 560
Leu Gly Thr Leu Asn Leu Ser Gly Asn Gln Leu Thr Gly Ser Ile Pro
565 570 575
Ile Gly Ile Gly Lys Met Thr Ser Leu Thr Thr Leu Asp Leu Ser Phe
580 585 590
Asn Asp Leu Ser Gly Arg Val Pro Leu Gly Gly Gln Phe Leu Val Phe
595 600 605
Asn Asp Thr Ser Phe Ala Gly Asn Pro Tyr Leu Cys Leu Pro His His
610 615 620
Ala Ser Cys Leu Thr Arg Pro Gly Gln Thr Ser Asp Arg Ile His Ala
625 630 635 640
Ala Leu Phe Ser Pro Ser Arg Ile Val Ile Thr Ile Ile Ala Ala Ile
645 650 655
Thr Ala Leu Ile Leu Ile Ser Val Ala Ile Arg Gln Met Asn Lys Lys
660 665 670
Lys His Glu Arg Ser Leu Ser Trp Lys Leu Thr Ala Phe Gln Arg Leu
675 680 685
Asp Phe Lys Ala Glu Asp Val Leu Glu Cys Leu Gln Glu Glu Asn Ile
690 695 700
Ile Gly Lys Gly Gly Ala Gly Ile Val Tyr Arg Gly Ser Met Pro Asn
705 710 715 720
Asn Val Asp Val Ala Ile Lys Arg Leu Val Gly Arg Gly Thr Gly Arg
725 730 735
Ser Asp His Gly Phe Thr Ala Glu Ile Gln Thr Leu Gly Arg Ile Arg
740 745 750
His Arg His Ile Val Arg Leu Leu Gly Tyr Val Ala Asn Lys Asp Thr
755 760 765
Asn Leu Leu Leu Tyr Glu Tyr Met Pro Asn Gly Ser Leu Gly Glu Asn
770 775 780
Lys Arg Arg Gln Arg Glu Phe
785 790
<210> 5
<211> 4961
<212> DNA
<213> 人工序列
<400> 5
tgagaacaag aggagacaga gagaatttta ataaaacttc cttttgtatt cactttctca 60
aaacgtttct ttacaagata atcgagtata tatacaacct caagataaac cctagttccc 120
ttaactttgg accactcaac acgtgtcttc gtcacacaca ctctacacct tgcaaggcat 180
cgtaacggct agacaaagct attccacagt ttactccagc tggtgcattt gtcttcttgt 240
tctgtacttg tacgaactct tcattactct gtttcagagt ttgtctttgg gccagcatac 300
ttccttcagt taatggacca gtcgctgata taccaaggcc catcatggct tgtttactgt 360
catccccccg caaactgatg atgggtggag caccaacact cagtttggct ctgagactct 420
gaaaggaatg gcgtgggaga gatttagtga agatatctgc aagttgcagt gcagaaggaa 480
tgtgctgaac ctccaagaca ccaagagcca cccgctccct aacataatgg taatccgttt 540
caatatgttt ggaccgaccg tgaagaaccg ggtttgctgt aagataaact gcagagaggt 600
tatcacagtg aagaacagcc ggctttggtt gtttgatctt catatcacgc atcacaaacg 660
aaatccatgt taactcagtc cgtcttgcag agctcgatat tcagcctcag tggacgaccg 720
cgagacagtc ggttggcgct ttgccgacca ggagacaatg ttagaaccaa tcatgacaca 780
aaaaccacca gtcgaccgtc ttgtctccct acaccctgcc cagtcactat cactatacgc 840
gacaacatcc aaaccagagt tcttacggaa gtaaatcccc atggatgaag taccacgaag 900
gtagcgaaga acccgcttca acaaatgaaa gtcagcttca gtgggagaat gcattctctg 960
acaaatgaaa ttcacagcaa actgaatgtc gggtcgagtg atagtgagat actgtaactt 1020
cccagcaaga ctccgaaagt agctaggctc agggaacggc cgagtatctt cataaacttg 1080
atcgagtcgc aaagggagag gtgtatgaac aggattacac gcagacatat tggcatggtg 1140
aagaatctct tcagtgtact tggattgaga gaggaaaaga ccagaatctg taatctgagc 1200
ctgaacgccc aagaagtagt gcagattacc caaatctttc atggagaatc ggctactgag 1260
ctcagtgata agggacttga acaaggatgg atttgagcca gtgagaagta tatcatccac 1320
ataaaggagg agaactaatg tgtcgccatt gtgatgataa acaaagagag agggatcagc 1380
tttactgcag acaaagccat actcaagaag gaagttacta aatttatcaa accaagcccg 1440
tggagcttgt tttaaaccgt acagagcctt cttgagagag cacacatgat gaggtttagt 1500
gcaatcttca aagccgggtg gttgtagcat atagactgct tcttgaagat ctccatgaag 1560
gaaagcattt ttaacatcaa gctgagtgat gttccagttc tttgaagcag ctacagcaag 1620
aactatccga atggtagaag tcctaaccac tggactgtat gtctctgtga aatctactcc 1680
ttcttcttga tcaaacccct tagcaactaa ccgcgcttta agtttattga ctgtcccatc 1740
tgcgttaagc ttcactgtaa aaacccatct gcaaccaaga acattcatag tttctgtagc 1800
ttccgttaaa acccaagtgt ggttctgata aatcgagtct agttcatcca acattgcttg 1860
acgccatcca gggtgcgcca aagcttcaga tatagtctgt ggtaaggagg gaatcgtctt 1920
gcttgtgacc agagcgtaac gtggattagg tttgctaata ccagctcgtg atctcgtcat 1980
cataggatga ctaggcgctg ctacgtctgg tgaggcacga ggaggtgatt gagcaacctc 2040
aggaagactt gtggtggata cgtcgccttc agacagctcg aagtcttcct gcccatgatc 2100
agaacgagat ggggttgcaa taggcgtatc ttgacccgtg gtgataggag tatcctgttg 2160
aggatactct gtaacttgag gacactctgt aacttgagaa ggtactgtag gcagatagac 2220
cggaggagct tcaaatgcag taccatcttg ccaagcttta agcaaaccag agttgtgttt 2280
cacaagcaga tgtttatact gtccagtgaa tgggaaggtc tgctcatcga agataacgtg 2340
gcgagagatg taaactcgac ctgttggtgg atgcaaacac ctgtaacctt tgtactgagg 2400
atgataaccg aggaagacac actgtaagga tcggggctcg aacttgtgtt gagcataggg 2460
acgcaagcac ggatagcacg cagagccaaa gattctgaga aatgagtagt ctggtgcttt 2520
actagagaga acctctgaag ggcttttgtt gttcagaaca gaagaaggga tcatgttgct 2580
gatgaagtta gcagagtaga aggcctcaac ccaatattta agaggagtgt gactttgaaa 2640
taacatagaa agacccaatt cagtaagatg tctatgtttt ctctcagcaa taccattttg 2700
ttgaggagtt gaaggacatg aaacaagatg aacaatccct tgatctctga gatgattgcg 2760
aaaccttgtg ctcataaact caccaccccc atcgctctga aagactttca gtttcttgtt 2820
aaactgattt tcgatttgtt tttgaaagac aatgaacaca tcacaaaaat cagacttggc 2880
tttaagaggt attatccatg agtagcgact aaagttgtca acaaagactg cataatactt 2940
gaacccttga actgatacaa ccggagaagg accccacaga tcacaatgta tccgatctat 3000
gggttccttg acactagact ctgaaataaa aaaaggaagc tgactgctct ttcccatctg 3060
gcaaggtcca caaacgggag aaatgctgct cttattgact atgattgcct tgctttgctt 3120
gaggagctga agaatctgga agttcgcgtg cccaagtcgc tgatgccaca ccatatcact 3180
agctgcacac tgacgattgg agaagtaagc cgcaaactcc ttcttctcca gcacatacaa 3240
gcctctattt cgagttccct ttgtcaccac cttctgagca tcgaggtcga tgacatagac 3300
agcattagag tcaaagaaca ccccacaggg gtaatcatca cacagtttgg atacagataa 3360
tagtgatttt tgcattgaag gacagacaag aacatcgttt agaggaaggc tacctgtctc 3420
agtggtgagc gttgtggaac caacgtgggt aatcggaagg aaattgccat ctgccaccat 3480
aacatgctca gaaccattgt atggagtagt gttctgcatc tgagttgtat taggaaccat 3540
gtgagcagtg gcacctgagt cagctatcca ttccctgcca ctaggatcag agactggaaa 3600
agcggacaga gcatgaggaa catcgtcact ctgataagag ttatcaaagc ggttccagca 3660
acgaagagcg gagtggccgg tgcggccaca gatctgacac acagggcgag agttgttgtt 3720
gttattgcca ccaccagagt tgctgacttg ttgagtgaaa cctcttccac gagttgagaa 3780
gccaccacga ccagaatagc caccgcgacc acgagaataa ttattgcttc gtcctctgtt 3840
cgagttgttg tagtaccctc ggtgagtctg gaaagccatg ttgggagtaa catctgatgt 3900
tgcttcatac gactgaagac gagcgtcaaa accagagaca tcagaaatca catcgttcaa 3960
agtcggagga gggattcgag tcatggagct ctgaacgaca gtgataattg gatcatattc 4020
tcttccaagg ccattgataa atgagaagat cttcatggat tcctctatag gcttcccaat 4080
ggagctcaac ttgtcacaaa tggctcgaaa ctcacgacag taggtagtaa agtctttacc 4140
tttcttagtc atcaactgga gagaccgtct gagctcaaac tcacgagcaa tagaactctt 4200
gttgtagttg tcagctaacg ataaccagac atcacgagat gtagatagac catgtactgt 4260
accaaggact tcctcagtaa gtgttccaaa gatccaagaa atgacgagtt gatccgtaca 4320
agtccaagct tcaaagcgag gatttggggc ttgaacgttg acgttgtcga ccacggttgt 4380
gactagctcc ggcggaggtg gaatctgacc agtagcaaag cccaacagct tctgactgcg 4440
aagaagagac tccatttgag tcttccatag gagatagttg ctgtcactca gtttaatggt 4500
aacagaactt gtaatgtgaa cactggtggg aaaagggtat ggatcttgct cagccatgac 4560
agcacctgtt agggttaaat gagctctgat accatgagaa caagaggaga cagagagaat 4620
tttaataaaa cttccttttg tattcacttt ctcaaaacgt ttctttacaa gataatcgag 4680
tatatataca acctcaagat aaaccctagt tcccttaact ttggaccact caacacgtgt 4740
cttcgtcaca cacactctac accttgcaag gcatcgtaac ggctagacaa agctattcca 4800
cagtttactc cagctggtgc atttgtcttc ttgttctgta cttgtacgaa ctcttcatta 4860
ctctgtttca gagtttgtct ttgggccagc atacttcctt cagttaatgg accagtcgct 4920
gatataccaa ggcccatcat ggcttgttta ctgtcatcgg c 4961
<210> 6
<211> 22
<212> DNA
<213> 人工序列
<400> 6
gcctttgatt gggtcagaac tc 22
<210> 7
<211> 21
<212> DNA
<213> 人工序列
<400> 7
aactaaccgt ttgatcgcga c 21
<210> 8
<211> 21
<212> DNA
<213> 人工序列
<400> 8
ctaacgcgtc caggacaaac c 21
- 还没有人留言评论。精彩留言会获得点赞!