家蚕核型多角体病毒诱导型39K启动子及其重组载体和应用的制作方法
本发明属于生物技术领域,涉及家蚕核型多角体病毒诱导型39k启动子,还涉及含有该启动子的重组载体和应用。
背景技术
家蚕是具有重要经济价值的鳞翅目模式昆虫。蚕丝业为世界经济、文化和社会发展做出了重要贡献。家蚕和家蚕核型多角体病毒作为高效表达外源蛋白的生物反应器,在生物工程领域具有重要的应用价值。天然启动子的低表达活性和非特异性在生物工程的应用中存在一定的局限性。在前期研究工作中,发明人所在课题组发明了bmnpv39k诱导型启动子(zl201010231957.9)以及改造后的增强型en39k(zl201310641709.5)。该启动子具有bmnpv诱导启动活性,能使细胞在受到bmnpv感染时启动外源基因的表达,但其启动子长度太长,核心调控区域未知,已不能满足现实需要。迫切需要在家蚕(鳞翅目)中构建高效的人工合成诱导型启动子满足不同生物工程需求。
诱导型启动子是诱导型调控序列或诱导型增强子,是一组能在特定物理、化学或病原体信号刺激下增强外源基因表达的启动子。一般而言,诱导型启动子与转录激活子一样,以非活性形式存在,并可通过相应的信号刺激直接或间接激活。目前,诱导型启动子(cre/loxp、tet-on/tet-off、蜕皮激素和病原体诱导系统)的几种技术方法被广泛应用于动植物基因工程领域,包括基因功能鉴定或品种改良。昆虫是地球上最大的生物类群。很多经济昆虫(如蚕和蜜蜂)具有非常重要的经济价值。然而,在昆虫基因工程研究中还没有一个较好的诱导系统,因此在抗病育种和基因治疗中构建致病诱导型启动子具有重要意义。
人工合成的启动子是通过结合不同启动子元件的不同组合来构建更强的表达水平,并用启动子的不同组合替换或重新设计序列。植物中的合成启动子的研究相对较多,大多集中在合成诱导型合成启动子上。合成启动子主要是利用顺式调控元件来融合核心启动子。构建不同病原诱导型人工启动子可有效提高转基因抗病基因在植物抗病育种中的广谱性。或者,构建与组织特异性启动子(根、茎、叶等)和诱导型启动子结合的人工诱导启动子有助于特定组织诱导表达,以提高作物品质和提高作物抗性和抗病性。人工合成的启动子也被报道在动物,其构建方法主要是通过不同表达调控序列的同向组装,应用于靶向治疗疾病,以及外源基因的特异性组织表达。人工合成启动子在昆虫研究中才刚刚起步,特别是在昆虫疾病育种领域。
在以前的研究中,筛选了bmnpv诱导启动子(vp1054、p33、bm21、bm122、39k、p143和p6.9)活性,发现39k启动子具有最高的bmnpv诱导的转录活性(zl201010231957.9)。并使用hr3、h5、polh和pu的增强子进一步增强bmnpv39k启动子的病毒诱导活性。可诱导的39k启动子控制的外源hycu-ep32基因的过度表达在转基因品系中具有高的抗病毒能力。此外,我们构建了一个家蚕核型多角体病毒诱导的rna干扰系统,它可以抑制bmnpv复制,受到病毒感染的严格控制,并且对宿主细胞没有毒性。并且高效的利用病毒诱导的39k启动子crispr/cas9基因编辑系统,降低了潜在的靶效和高的编辑效率,增强了家蚕细胞的抗病毒能力。因此,为了使病毒诱导的39k启动子在基因功能研究、家蚕抗性育种和害虫防治中更有效,人工合成诱导型启动子在昆虫中是非常迫切的。
技术实现要素:
有鉴于此,本发明的目的之一在于提供家蚕核型多角体病毒诱导型39k启动子;本发明的目的之二在于提供家蚕核型多角体病毒诱导型39k启动子原件;本发明的目的之三在于提供人工合成家蚕核型多角体病毒诱导型启动子;本发明的目的之四在于提供所述人工合成家蚕核型多角体病毒诱导型启动子的重组表达载体;本发明的目的之五在于提供家蚕核型多角体病毒诱导型39k启动子或所述家蚕核型多角体病毒诱导型39k启动子原件在家蚕基因工程育种或家蚕核型多角体病毒表达系统中的应用;本发明的目的之六在于提供所述重组表达载体在家蚕基因工程育种或家蚕核型多角体病毒表达系统中的应用。
为实现上述发明目的,本发明提供如下技术方案:
1、家蚕核型多角体病毒诱导型39k启动子,所述启动子的核苷酸序列如seqidno.2、seqidno.4或seqidno.5所示。优选的,所述启动子的核苷酸序列如seqidno.2所示。
2、家蚕核型多角体病毒诱导型39k启动子原件,所述启动子原件核苷序列如seqidno.3所示。
3、人工合成家蚕核型多角体病毒诱导型启动子,其特征在于:含有如seqidno.3所示的家蚕核型多角体病毒诱导型39k启动子原件和其它家蚕核型多角体病毒启动子序列。优选的,所述家蚕核型多角体病毒启动子为p33启动子,其核苷酸序列如seqidno.6所示。
4、含有所述家蚕核型多角体病毒诱导型39k启动子或所述人工合成家蚕核型多角体病毒诱导型启动子的重组表达载体。
可以在p39k-1启动子或启动子调控原件39k(-310~-355)的下游连接目的基因,包括结构基因、调节基因、结构基因的反义基因、调节基因的反义基因、能够干扰内源基因表达的小rna等构建重组表达载体,在bmnpv感染或相关因子的诱导下驱动结构基因、调节基因、结构基因的反义基因、调节基因的反义基因、天然小rna或人工合成小rna的表达。
优选方案一,所述重组表达载体为报告基因重组表达载体,所述报告基因由人工合成病毒诱导型启动子p39k-1或启动子调控原件39k(-310~-355)控制表达。报告基因包括但不限于:荧光素酶(fluc)基因、红色荧光蛋白(dered)基因、绿色荧光蛋白(gfp)基因、增强型绿色荧光蛋白(egfp)基因等。该重组表达载体家蚕核型多角体病毒诱导型启动子的相关研究。
优选方案二,目的基因为家蚕抗性基因。
优选方案三,目的基因为rnai和基因编辑序列。
所述重组表达载体能够在bmnpv感染或ie-1蛋白表达情况下激活表达。
所述人工合成病毒诱导型启动子原件为39k(-310~-355),所述39k(-310~-355)原件bmnpvie-1蛋白结合原件,由bmnpv感染或ie-1蛋白表达控制表达。该启动子原件39k(-310~-355)可用于制备防治家蚕感染bmnpv的药物、家蚕抗bmnpv品系的基因工程育种以及诱导型家蚕核型多角体病毒表达系统。
5、所述家蚕核型多角体病毒诱导型39k启动子或所述人工合成家蚕核型多角体病毒诱导型启动子在家蚕基因工程育种或家蚕核型多角体病毒表达系统中的应用
6、所述重组表达载体在家蚕基因工程育种或家蚕核型多角体病毒表达系统中的应用。
本发明的有益效果在于:本发明从bmnpv39k启动子母体启动子中获得,启动子长度降低为原来的39%,但启动子活性仍为原来的87%以上,同时我们获得了bmnpvie-1蛋白结合启动子原件为39k(-310~-355),可用于合成不同需要的人工合成启动子,提高基本启动子诱导活性;p39k-1或启动子调控原件39k(-310~-355)可以驱动外源基因在昆虫细胞或昆虫个体中经bmnpv感染或相关因子的诱导下高效表达,不但适用于基因功能分析等分子生物学理论研究,同时适用于利用基因工程技术进行的家蚕品种改良,特别适用于家蚕高效抗bmnpv品系的培育以及通过表达外源致死基因、标记基因、rnai以及基因编辑系统以达到消除病害扩散的家蚕品种培育,具有很好的应用前景。
附图说明
为了使本发明的目的、技术方案和有益效果更加清楚,本发明提供如下附图进行说明:
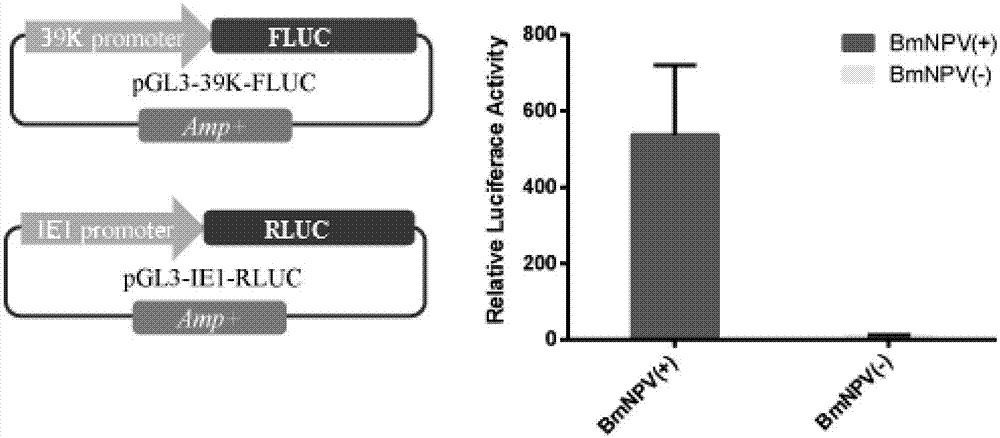
图1为双荧光素酶报告系统示意图。
图2为39k启动子5’末端截短相对荧光素酶活性检测。
图3为39k启动子中间缺失相对荧光素酶活性检测。
图4为39k启动子3’末端截短相对荧光素酶活性检测
图5为39k启动子核苷酸序列及调控原价分析。
图6为人工合成病毒诱导性39k启动子构建及荧光素酶活性检测。
图7为分别用重组载体piz-ie0,piz-ie1,piz-ie2,piz-pe38、piz-me53和pgl3-39k-dsred共转染细胞72小时后的红色荧光发光情况。
图8为分别用重组载体piz-ie0,piz-ie1,piz-ie2,piz-pe38、piz-me53和pgl3-39k-dsred共转染72小时后,dsred基因表达变化分析。
图9为分别用重组载体piz-ie0,piz-ie1,piz-ie2,piz-pe38、piz-me53和pgl3-39k-dsred共转染72小时后荧光素酶酶活检测。
图10为emsa分析ie-1蛋白转录激活39k启动子的结合序列。
图11为emsa分析ie-1蛋白与39k(-310~-355)调控序列结合特征。
图12为39k(-310~-355)启动子调控原件构建人工合成病毒诱导型启动子示意图及酶活检测。
图13为人工合成病毒诱导型启动子系统激活原理。
具体实施方式
下面将结合附图,对本发明的优选实施例进行详细的描述。
实施例1、39k启动子的结构与功能分析
为了产生更优化的病毒诱导特异性启动子,使用截短和突变策略逐渐去除了39k启动子核心区,并分析了39k启动子活性的变化。将启动子控制的萤火虫荧光素酶(fluc)和对照质粒ie1(seqidno.7)启动子控制的海参荧光素酶(rluc)共转染到bmn-swu1细胞中,再用bmnpv感染细胞,于感染后72h收集细胞,裂解,离心取上清,用双荧光素酶报告基因检测试剂盒测定裂解上清的ppl和prl,计算相对荧光素酶活性,考察转染bmn-swu1细胞在感染bmnpv后39k启动子活性。通过检测相对荧光素酶fluc/rluc的变化来鉴定启动子活性(图1)。
为了鉴定39k启动子表达所需的核心区域,进行逐步缺失,以~773~+134为原启动子的启动子序列(seqidno.1),与原序列相比,每个启动子逐步截短降低了50bp(图2)。在bmn-swu1细胞中共转染了15个5’截短的荧光素酶检测质粒和pgl3-ie1-rluc质粒。转染48小时后,加入bmnpv或培养基48小时后,测定荧光素酶活性。结果表明,随着启动子长度的减少,启动子活性逐渐下降。p573启动子的长度相对于39k启动子有200bp的下降,但启动子活性仅下降14.5%(图2)。表明,片段773~573对39k启动子活性影响不大。然而,相对于39k启动子,p323启动子的启动子活性降低了97.21%。暗示-323~-573片段始终具有39k诱导型启动子的重要调控元件。p273、p323和p373具有较强的组成启动子活性,p273启动子比p23高12.27倍,表明-223~-273片段与39k启动子的组成启动子活性有关(图2)。
为了进一步分析39k启动子调控基序,对39k启动子进行了中间片段缺失。结果表明,p-1~273、p223~273、p337~473对39k启动子活性无显著影响(图3)。另外,p-1~223、p1~-373和p-1~473启动子的缺失形式使启动子活性迅速下降(图3)。因此,结合5’端缺失结果和选择最佳启动子的原理,确定缺失为-1~273片段。39k启动子的+1~+134片段为核心区,3’端逐渐截断,并分析启动子活性。结果表明,与39k相比,p+116和p+62的启动子活性分别提高了35.4%和97%。这些结果表明,+134~+116和+76~+62的缺失增加39k启动子的活性,这两个片段对启动子活性有抑制作用(图4)。为了获得最佳启动子,可以根据该结果在3’端删除+136~+62片段。
实施例2、人工诱导型39k启动子的构建
基于对39k启动子的缺失分析,粗略地确定了对39k启动子活性有影响的区域。同时,通过启动子预测程序对启动子核心区的关键调控元件进行了分析。在线分析结果表明,39k启动子包含核心成分,如两个增强子组分cgtgcgc、六个caat位点、两个转录抑制剂tgac、两个顺式调控原ccat和两个tata盒(图5)。结合39k启动子核心元件和关键调控区的位置,首先构建了3个人工诱导启动子,包括p39k-1(-573~-273和+1~+62片段)(seqidno.2)、p39k-5(-573~-273和+1~+134)(seqidno.4)、p39k-9(-773~-273和+1~+134)(seqidno.5)。p39k-1、p39k-5和p39k-9启动子的活性分别达到39k启动子活性的87.24%、75.94%和112.34%(图6)。p39k-1、p39k-5和p39k-9启动子的启动子长度分别为362bp、436bp和636bp。构建人工启动子的目的是在不影响启动子活性的前提下最小化启动子的长度。因此,p39k-1启动子的长度仅为39k启动子序列的39.91%,但启动子活性仍能达到原来的87.24%,这是较好的人工诱导启动子。以前的研究表明,caat位点的突变对cggt能显著增加启动子活性。因此,在p39k-1、p39k-5和p39k-9启动子中分别构建了-326个位点、-399个位点和两个位点(-326和-399)同时突变的9个人工诱导启动子(-326和-399位点caat变为cggt)。双荧光素酶报告分析表明,这九个点突变的启动子没有显著增加它们相对于p39k-1、p39k-5和p39k-9启动子的启动子活性(图6)。p39k-1人工诱导启动子仍含有增强子样成分cgtgcgc、caat位点、转录抑制剂tgac。在前两个tata盒被删除后,一个新的tata盒出现在人工诱导启动子p39k-1)的转录起始位点的-70bp位置(结合上述结果,p39k-1仍具有启动子调控原基,是最理想的人工诱导启动子。
实施例3、鉴定诱导型启动子39k的转录调控蛋白
家蚕核型多角体病毒基因的表达由级联调节,并且每个后续的基因表达都依赖于前一个阶段。家蚕核型多角体病毒39k基因是一个延迟的早期表达基因。为了确定39k启动子转录结合蛋白,首先从美国国立生物技术信息中心(ncbi)下载bmnpv全基因组序列(nc001962.1)设计并合成如下表达5个即刻早期基因(ie-0,ie-1,ie-2,pe38和me53)蛋白的引物并在引物两段加上相应的限制性酶切位点(下列引物序列中下划线序列):
ie0(ecori)-f:5’-cggaattcatgataagaaccagcagtc-3’(seqidno.11);
ie0(noti)-r:5’-ataagaatgcggccgctttatacgatgtcctgca-3’(seqidno.12);
ie1(ecori)-f:5’-cggaattcatgacgcaaattaatt-3’(seqidno.13);
ie-1(noti)-r:5’-ataagaatgcggccgcattaaattcaatttttttatat-3’(seqidno.14);
ie2(bamhi)-f:5’-cgcggatccatgagtcgccaaatcaacgc-3’(seqidno.15);
ie2(noti)-r:5’-ataagaatgcggccgcttaaggtttagacatctcaatagtg-3’(seqidno.16);
pe38(ecori)-f:5’-cggaattcatggacaaacgtgcc-3’(seqidno.17);
pe38(noti)-r:5’-ataagaatgcggccgccaatttatcacatttacg-3’(seqidno.18);
me53(ecori)-f:5’-cggaattcatgaaccgttttttt-3’(seqidno.19);
me53(noti)-r:5’-ataagaatgcggccgcgacattgttgtttac-3’(seqidno.20);
pcr条件为:94℃预变性5分钟;94℃变性50秒,52℃退火50秒,72℃延伸2分钟,30个循环;最后72℃延伸10分钟。以bmnpv全基因组为模板,采用pu-f/pu-r引物对pcr扩增pu序列。pcr条件为:94℃预变性5分钟;94℃变性50秒,56℃退火50秒,72℃延伸3分钟,30个循环;最后72℃延伸10分钟。pcr产物纯化后,分别通过t克隆与piz-v5/his质粒连接,连接产物转化大肠杆菌dh5α感受态细胞,用含有zeocin的lb平板筛选阳性克隆,挑取阳性克隆单菌落,用含有zeocin的lb培养基过夜培养后提取质粒,测序,将测序正确的阳性克隆质粒命名为:piz-ie0、piz-ie1、piz-ie2、piz-pe38和piz-me53。
利用pgl3-39k-dsred共转染了piz-ie0、piz-ie1、piz-ie2、piz-pe38和piz-me53,然后通过荧光显微镜家蚕dsred蛋白表达。结果表明,仅在病毒感染和piz-ie1转染的bmn-swu1细胞中观察到dsred蛋白的表达,但在piz-ie0、piz-ie2、piz-pe38、piz-me53和未感染的细胞中未检测到dsred蛋白的表达(图7)。表明,bmnpv感染和ie-1蛋白表达可迅速激活dsred蛋白表达。此外,为了检测诱导的敏感性,研究了ie-1蛋白和bmnpv诱导的dsred基因的转录。结果表明,病毒和ie-1蛋白诱导了dsred基因的大规模转录(图8)。在piz-ie0、piz-ie2、piz-pe38、piz-me53转染和未感染细胞中均未检测到dsred基因转录水平的变化。荧光素酶分析同样表明只有ie-1蛋白能有效诱导39k启动子活性。其他早期基因对39k启动子没有转录调控(图9)。
实施例4、emas分析ie-1蛋白转录结合39k启动子区域
为了进一步确定ie-1蛋白是39k启动子的直接转录结合靶位点的结论,通过凝胶移位竞争试验(emas)分析ie-1蛋白潜在的结合位点序列,探针序列使用一个生物素标记的寡核苷酸进行。通过在线程序预测,设计了总共4个含有多个潜在结合位点的探针。这些探针分别命名为探针1(-486~-532)(seqidno.8)、探针2(-386~-431)(seqidno.9)、探针3(-310~-355)(seqidno.3)和探针4(+2~+47)(seqidno.10)。ie-1蛋白的原核表达和纯化用于探针孵育。生物素标记探针3(-310~-355)与ie-1蛋白的孵育,使emsa发生明显的条带移位,加入未标记探针dna的竞争探针条带消失(图10)。相反,在emsa检测中,探针1(-486~-532)、探针2(-386~-431)和探针4(+2~+47)孵育后,没有发现明显的带位移现象。
为了进一步研究探针3与ie-1蛋白的结合活性,分析了生物素标记探针和未标记dna浓度对条带移位的影响。结果表明,探针3与ie-1蛋白的孵育导致条带的迁移,随着生物素标记的探针3浓度的增加而增加,并且随着竞争探针未标记dna浓度的增加而减少(图11)。在没有ie-1蛋白孵育的探针中没有检测到显著的带移位。表明在bmnpvie-1蛋白诱导的39k转录激活中,ie-1特异性结合于39k启动子探针3(-310~-355)。
实施例5、人工合成病毒诱导型启动子的应用
为了扩大人工诱导的39k启动子在昆虫基因工程中的应用,通过结合不同的家蚕核型多角体病毒启动子如p33(seqidno.6)和39k(-310~-355)结合序列,合成了新的启动子p33+39k(-310~-355)。结果表明,人工合成诱导型启动子p33+39k(-310~-355)启动子结合39k启动子调控原件39k(-310~-355)后,启动子的诱导活性显著高于原序列。bmnpv感染后,p33+39k(-310~-355)的启动子活性增加了4.46倍,比原始序列高出1.48倍(图12)。以上结果表明,39k启动子的片段可用于人工诱导启动子的构建,以提高不同启动子和基因工程的诱导活性。人工合成病毒诱导型启动子启动原理为在bmnpv未感染的条件现靶标基因不会转录,翻译为蛋白,处于未激活状态不会影响宿主;在bmnpv感染或者ie-1蛋白表达的调剂下,进而结合转录调控序列39k(-310~-355),激活人工合成启动子的诱导表达,实现调控的目的(图13)。
最后说明的是,以上优选实施例仅用以说明本发明的技术方案而非限制,尽管通过上述优选实施例已经对本发明进行了详细的描述,但本领域技术人员应当理解,可以在形式上和细节上对其作出各种各样的改变,而不偏离本发明权利要求书所限定的范围。
序列表
<110>西南大学
<120>家蚕核型多角体病毒诱导型39k启动子及其重组载体和应用
<160>20
<170>siposequencelisting1.0
<210>1
<211>910
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>1
aaggctgtcttgctgtgtgcccgtcgcgcgtaccggagcgcgaacgcgcccgccgccgac60
ataaacgacacttttctagaaaaaatttccataccacgaggtcatcgcgattgttgcgac120
gcaaaagtttacgaaactgccgtgcgcgagtttgtggaagaaactggccggttttttgac180
agcgcgttcatctacaagtttccatttacgttacaatggaaagacgacggtgtcacttac240
aagtatttgatatacgtaggcgtcgtgcgcggcaacttgattgacgtgaacgccaaaccc300
aacacgtacaccgtgaagttgttgccgggcacgtttggcaacgactatcgtataatgtta360
aaaccgcgacgcttcaattgcgaaataacgcgcagcctggccatcgtgccgctcaacaaa420
tattttaattatatgaacgacaaacaattgatcacgtacgattacagcaattacattgaa480
ttttttagttttgtgcgcagcatcaagaagcgtttcgataataggcaattgcaagacttt540
ttctacgccactctaaaaaagatagacaacaacgatgccccccaaaaattgcacgcactt600
aggcgggtgtaattcggactgcttgacccgaagcgaaatacaagcgctgttcagggaagc660
catcaacacgctcaagcacacaatgaatacagaagacgtctgcgcgcacatgttggacat720
cgtgtcgtttgagcgtataaaagaatatataagagctaatttaggccatttcacagtaat780
taccgacaaatgttctaagcgtaaggtgtgtcttcatcacaaacgaattgccaggctgtt840
gggcattaaaaaaatatatcatcaagaatacaagcgggtcgtttcaaaggtttacaaaaa900
tcaaacatgg910
<210>2
<211>362
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>2
tccatttacgttacaatggaaagacgacggtgtcacttacaagtatttgatatacgtagg60
cgtcgtgcgcggcaacttgattgacgtgaacgccaaacccaacacgtacaccgtgaagtt120
gttgccgggcacgtttggcaacgactatcgtataatgttaaaaccgcgacgcttcaattg180
cgaaataacgcgcagcctggccatcgtgccgctcaacaaatattttaattatatgaacga240
caaacaattgatcacgtacgattacagcaattacattgaattttttagttttgtgcgcag300
agtaattaccgacaaatgttctaagcgtaaggtgtgtcttcatcacaaacgaattgccag360
gc362
<210>3
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>3
aatattttaattatatgaacgacaaacaattgatcacgtacgatta46
<210>4
<211>436
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>4
tccatttacgttacaatggaaagacgacggtgtcacttacaagtatttgatatacgtagg60
cgtcgtgcgcggcaacttgattgacgtgaacgccaaacccaacacgtacaccgtgaagtt120
gttgccgggcacgtttggcaacgactatcgtataatgttaaaaccgcgacgcttcaattg180
cgaaataacgcgcagcctggccatcgtgccgctcaacaaatattttaattatatgaacga240
caaacaattgatcacgtacgattacagcaattacattgaattttttagttttgtgcgcag300
agtaattaccgacaaatgttctaagcgtaaggtgtgtcttcatcacaaacgaattgccag360
gctgttgggcattaaaaaaatatatcatcaagaatacaagcgggtcgtttcaaaggttta420
caaaaatcaaacatgg436
<210>5
<211>636
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>5
aaggctgtcttgctgtgtgcccgtcgcgcgtaccggagcgcgaacgcgcccgccgccgac60
ataaacgacacttttctagaaaaaatttccataccacgaggtcatcgcgattgttgcgac120
gcaaaagtttacgaaactgccgtgcgcgagtttgtggaagaaactggccggttttttgac180
agcgcgttcatctacaagtttccatttacgttacaatggaaagacgacggtgtcacttac240
aagtatttgatatacgtaggcgtcgtgcgcggcaacttgattgacgtgaacgccaaaccc300
aacacgtacaccgtgaagttgttgccgggcacgtttggcaacgactatcgtataatgtta360
aaaccgcgacgcttcaattgcgaaataacgcgcagcctggccatcgtgccgctcaacaaa420
tattttaattatatgaacgacaaacaattgatcacgtacgattacagcaattacattgaa480
ttttttagttttgtgcgcagagtaattaccgacaaatgttctaagcgtaaggtgtgtctt540
catcacaaacgaattgccaggctgttgggcattaaaaaaatatatcatcaagaatacaag600
cgggtcgtttcaaaggtttacaaaaatcaaacatgg636
<210>6
<211>916
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>6
tgaatacaataaacatggatttaaattccttgatatcaaagtttgcggtgccgcgcgtca60
tggtcacattactagatgtgctggccgcgtttggcgatttaactccatagtcgtatacgg120
tgcccagaaaaatgctgttagtgcccactttgttggagccctctttgactatgtcgtcta180
tagatagcggtttttcggcaatatacatttggctcaatttattatcgccgtgtgcgatgc240
gcaaattggccacccgggcgttattaagctttacgtttaatcgtttgttctcgtctgatt300
ccaccgattcgctgctctgccctaaactgggcgacgcctcggttaaggaatttaaattta360
atgcattcgtccaatagagataaatcagtacgagcaaaacgatcaataacacgattttcc420
acatgatttgttttaatttacaatttcaattccaatgagatttaggttgtgcaaatgttt480
caaaagtacacttaaattatcgttttcccaattgacgggcacggccacaacgattttact540
gctgagcgtaaaaatttcgaaaatggccgtgataaactgttcgcaatagttaataacgtt600
gctgccgaacgcagactcgtcgaccaaattcacatatgtcttatacataatcattgcatc660
gaacaatacgtggtgtttgttaaaaacgccctcgtttttaagcaccaagtgtttgtatac720
ttttttcataaataaggcttgctcctgtttgagttcagcaaaataagtaaacttgtcgca780
ttggtcgtccgtaagacattctgtaatattttcgaaataaataataggctcgtcggcgtc840
cgtgtcggcgctaacgtacaacgacgctgtggccggcgcctgacacaaatacagcacgat900
cgttttgctagtcgcc916
<210>7
<211>638
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>7
ttgcagttcgggacataaatgtttaaatatatcaatgtctttgtgatgcgcgcgacattt60
ttgtaagttattaataaaatgcaccgacacgttgcccgacattatcattaaatccttggc120
gtagaatttgtcgggtccgttgtccgtgtgcgctagcatgcccgtaacggaccttgagct180
tttggcttcaaaggttttgcgcacagacaaaatgtgccacacttgcagctctgcttgtgt240
acgcgttaccacaaatcccaacggcgcagtgtacttgttatatgtaaataaatctcgata300
aaggcgcggcgcgcgaatgcagctgatcacgtacgctcctcgtgttccgttcaaggacgg360
tgttatcgacctcagattaatatttatcggccgactgttttcgtatccgctcaccaaacg420
ggtttttgcattaacattgtatgtcggcggatgttctgtatctaatttgaataaataaat480
gataaccgcattggttttagagggcataataaaaaaaatattattatcgtgttcgccatt540
ggggcagtataaattgacgttcatgttgaatattgtttcagttgcaagttgacattggcg600
gcgacacgatcgtgaacaaccaaacgactagggatcta638
<210>8
<211>47
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>8
agtatttgatatacgtaggcgtcgtgcgcggcaacttgattgacgtg47
<210>9
<211>46
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>9
gactatcgtataatattaaaaccgcgacgcttcaattgcgaaataa46
<210>10
<211>46
<212>dna
<213>家蚕核型多角体病毒(bombyxmorinucloepolyhedrivirus)
<400>10
agtaattaccgacaaatgttctaagcgtaaggtgtgtcttcatcac46
<210>11
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>11
cggaattcatgataagaaccagcagtc27
<210>12
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>12
ataagaatgcggccgctttatacgatgtcctgca34
<210>13
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>13
cggaattcatgacgcaaattaatt24
<210>14
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>14
ataagaatgcggccgcattaaattcaatttttttatat38
<210>15
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>15
cgcggatccatgagtcgccaaatcaacgc29
<210>16
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>16
ataagaatgcggccgcttaaggtttagacatctcaatagtg41
<210>17
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>17
cggaattcatggacaaacgtgcc23
<210>18
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>18
ataagaatgcggccgccaatttatcacatttacg34
<210>19
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>19
cggaattcatgaaccgttttttt23
<210>20
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>20
ataagaatgcggccgcgacattgttgtttac31
- 还没有人留言评论。精彩留言会获得点赞!