用于检测CRISPR/Cas9基因编辑系统切割基因产生移码突变的基因元件及应用的制作方法
本发明涉及基因工程中的克隆技术和分子检测技术领域,具体而言,涉及一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的基因元件及应用。
背景技术:
规律成簇的间隔短回文重复序列(clusteredregularlyinterspacedshortpalindromicrepeats,crispr)是大多数细菌及古细菌中的一种获得性免疫方式。2012年,jinek等使用crispr/cas9基因编辑系统对基因组dna进行操作,并证实了这一系统可以在活体细胞实现rna介导的基因编辑。至今,crispr/cas9基因编辑系统广泛应用于目的基因编辑。特别是在诱导性多功能干细胞(inducedpluripotentstemcells,ipscells)或胚胎干细胞(embryonicstemcell,escs)中的应用,使用crispr/cas9基因编辑系统修改基因组所获的细胞系可以模仿基因病患者,用于目的新药、特效药的研发;或失活特定突变的基因,达到基因编辑治疗疾病的目的。另一方面,由于crispr/cas9基因编辑系统是rna介导的,所以grna是否具有专一的切割位点是最核心的,制约crispr/cas9基因编辑系统使用的因素,是决定前述目的实现的关键要素之一。因此,grna设计好坏决定目标基因组被切割的效率;专一性的grna可以减少脱靶几率,提高基因编辑的正确率,避免额外错误的编辑。所以,需要一种检测方法来快速鉴定所设计的grna对基因组的切割效率和编辑产生移码突变后的基因组碱基序列是怎样分布的。
技术实现要素:
本发明旨在提供一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的基因元件及应用,以提供一种快速的、可在活细胞胞内鉴定grna对基因组的切割效率的方法。
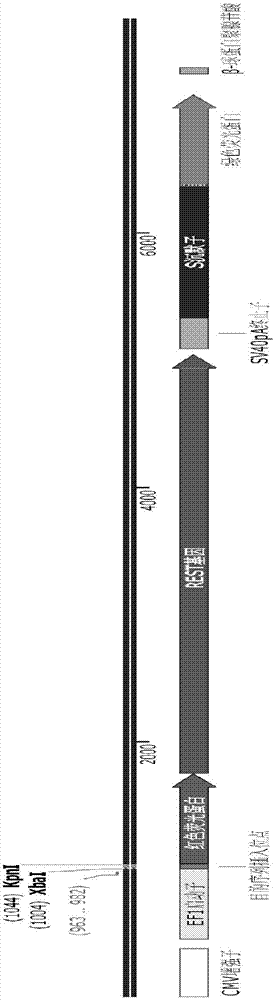
为了实现上述目的,根据本发明的一个方面,提供了一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的基因元件(crispr/cas9indelassaysystem,cias)。cias基因元件包括有效连接的启动子、第一报告基因、rest基因、sv40pa基因、s沉默子、第二报告基因和终止子,其中,启动子与第一报告基因基因之间可插入被切割目标dna序列,第一报告基因不同于第二报告基因,rest基因的表达能够抑制沉默子后面第二报告基因的表达。
进一步地,基因元件还包括有效连接的增强子。
进一步地,增强子为cmv增强子。
进一步地,第一报告基因为红色荧光蛋白基因,第二报告基因为绿色荧光蛋白基因。
进一步地,启动子为ef1启动子,终止子为β-球蛋白聚腺苷酸。
进一步地,基因元件含有双酶切位点,双酶切位点位于启动子和第一报告之间,被切割目标dna序列连接至双酶切位点之间。
进一步地,双酶切位点为xbai和kpni酶切位点。
进一步地,rest基因和s沉默子扩增于人源h9细胞系。
进一步地,基因元件具有如seqidno:1所示的核苷酸序列。
根据本发明的另一方面,提供了一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的载体。该载体包括上述任一种基因元件。
进一步地,载体为puc系列载体、peasy、pbluescriptii或pbr322。
根据本发明的另一方面,提供了一种宿主细胞。该宿主细胞包括上述任一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的载体。
进一步地,宿主细胞为大肠杆菌。
根据本发明的再一方面,提供了一种检测crispr/cas9基因编辑系统切割基因产生移码突变的方法。该方法为采用上述任一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的载体进行检测。
进一步地,包括以下步骤:s1,将上述任一种载体插入被切割目标dna序列,然后转染至细胞系中,同时转入装载有grna的载体(包含表达cas9蛋白的dna序列);s2,观察第一报告基因和第二报告基因的表达情况,根据第一报告基因和第二报告基因的表达情况判断grna对目标dna序列的编辑是否产生了移码突变。
进一步地,还包括:当第二报告基因正常表达时,判断为crispr/cas9基因编辑系统切割基因产生了移码突变,并扩增目标dna序列进行测序。
进一步地,细胞系为hek293细胞系。
用于检测crispr/cas9基因编辑系统切割基因产生移码突变的基因元件(简称cias)可作为核心元件连接到载体中,可以用来在活细胞胞内鉴定grna对基因组的切割效率,该检测具有产生移码突变时是即时荧光表现型、高稳定性的荧光表型和易操作性,并且该载体构建具有简单、快速、省时、省力等遗传操作的显著优点。
附图说明
构成本申请的一部分的说明书附图用来提供对本发明的进一步理解,本发明的示意性实施例及其说明用于解释本发明,并不构成对本发明的不当限定。在附图中:
图1示出了基因元件cias组成示意图。;
图2a和2b分别为本发明的基因元件cias装载的cias-puc19和pcias的质粒图谱。
图3a和3b分为装载目的dna序列的target-pcias和target-1-pcias。
图4a、4b、4c和4d分别为装载grna序列的质粒图谱。
图5a、5b、5c和5d分别为目的dna被编辑和没有被编辑的菌株的光镜和荧光照片。
具体实施方式
需要说明的是,在不冲突的情况下,本申请中的实施例及实施例中的特征可以相互组合。下面将参考附图并结合实施例来详细说明本发明。
针对现有技术中存在的一些技术问题,本发明提供一种检测crispr/cas9基因编辑系统切割基因产生移码突变的基因元件、载体、宿主细胞及方法。
根据本发明一种典型的实施方式,提供一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的基因元件。该基因元件包括有效连接的启动子、第一报告基因、rest基因、sv40pa基因、s沉默子、第二报告基因和终止子,其中,启动子与第一报告基因基因之间可插入被切割目标dna序列,第一报告基因不同于第二报告基因,rest基因的表达能够抑制沉默子后面第二报告基因的表达。
本发明中的“有效连接”即生物技术领域的常规含义,即连接后的基因元件能够起到其预定的作用。
目标dna序列被连接至上述基因元件中,grna/cas9复合体切割目标dna序列产生移码突变时,第一报告基因和rest基因也会产生移码突变,是不能表达正确的第一报告基因-rest融合蛋白,即为无法产生第一报告基因的蛋白,也不能抑制s沉默子后面第二报告基因的表达。这时,第二报告基因的蛋白是能够正常表达的,可使宿主细胞系产生第二报告基因的信号。
目标dna序列被连接至基因元件cias中,grna/cas9复合体切割目标dna序列不产生移码突变时,第一报告基因和rest基因表达正确的第一报告基因-rest融合蛋白,即为产生第一报告基因的蛋白,同时s沉默子抑制后面第二报告基因的表达。这时,第一报告基因是能够正常表达的,而第二报告基因是不表达的,所以第一报告基因的信号。
当第一报告基因为红色荧光蛋白基因,第二报告基因为绿色荧光蛋白基因时,目标dna序列被连接至基因元件cias中,grna/cas9复合体切割目标dna序列产生移码突变时,基因元件红色荧光蛋白和rest基因也会产生移码突变,是不能表达正确的红色荧光-rest融合蛋白,即为无法产生红色荧光蛋白和不抑制沉默子后面基因绿色荧光蛋白的表达。这时,绿色荧光蛋白是能够正常表达的,可使宿主产生绿色荧光。目标dna序列被连接至基因元件cias中,grna/cas9复合体切割目标dna序列不产生移码突变时,基因元件红色荧光蛋白和rest基因表达正确的红色荧光-rest融合蛋白,即为产生红色荧光蛋白和抑制沉默子后面基因绿色荧光蛋白的表达。这时,红色荧光蛋白是能够正常表达的,而绿色荧光是不表达的,所以宿主产生红色荧光。
上述基因元件cias所包含的序列可以通过基因合成或扩增自含有这一基因元件的目标核酸序列获得,并通过融合pcr或同源重组的方法连接在一起。组成序列不同来源,可以应用于不同细胞系。其中,根据本发明一种典型的实施方式,rest基因和s沉默子扩增于人源ips或h9细胞系,更适合人源细胞系的基因移码突变检测。
优选的,基因元件还包括有效连接的增强子,更优选的增强子为cmv增强子。
根据本发明一种典型的实施方式,启动子为ef1启动子,终止子为β-球蛋白聚腺苷酸。
便于目标dna序列,基因元件cias含有双酶切位点,双酶切位点位于启动子和第一报告之间,被切割目标dna序列连接至双酶切位点之间。优选的,双酶切位点为xbai和kpni酶切位点。
优选的,基因元件cias具有如seqidno:1所示的核苷酸序列。
seqidno:1
tgtaaaacgacggccagtgaattcgacattgattattgactagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatggaatagcaacagacatacaaactaaagaattacaaaaacaaattacaaaaattcaaaattttatcgatactagtaaggatctgcgatcgctccggtgcccgtcagtgggcagagcgcacatcgcccacagtccccgagaagttggggggaggggtcggcaattgaacgggtgcctagagaaggtggcgcggggtaaactgggaaagtgatgtcgtgtactggctccgcctttttcccgagggtgggggagaaccgtatataagtgcagtagtcgccgtgaacgttctttttcgcaacgggtttgccgccagaacacagctgaagcttcgaggggctcgcatctctccttcacgcgcccgccgccctacctgaggccgccatccacgccggttgagtcgcgttctgccgcctcccgcctgtggtgcctcctgaactgcgtccgccgtctaggtaagtttaaagctcaggtcgagaccgggcctttgtccggcgctcccttggagcctacctagactcagccggctctccacgctttgcctgaccctgcttgctcaactctacgtctttgtttcgttttctgttctgcgccgttacagatccaagctgtgaccggcgcctacatgtctagannnnnnnnnnnnnnnnnnnnnnnnnnnnnnggtaccgtgtctaagggcgaagagctgattaaggagaacatgcacatgaagctgtacatggagggcaccgtgaacaaccaccacttcaagtgcacatccgagggcgaaggcaagccctacgagggcacccagaccatgagaatcaaggtggtcgagggcggccctctccccttcgccttcgacatcctggctaccagcttcatgtacggcagcaaaaccttcatcaaccacacccagggcatccccgacttctttaagcagtccttccctgagggcttcacatgggagagagtcaccacatacgaagacgggggcgtgctgaccgctacccaggacaccagcctccaggacggctgcctcatctacaacgtcaagatcagaggggtgaacttcccatccaacggccctgtgatgcagaagaaaacactcggctgggaggcctccaccgagatgctgtaccccgctgacggcggcctggaaggcagaagcgacatggccctgaagctcgtgggcgggggccacctgatctgcaacttgaagaccacatacagatccaagaaacccgctaagaacctcaagatgcccggcgtctactatgtggacagaagactggaaagaatcaaggaggccgacaaagaaacctacgtcgagcagcacgaggtggctgtggccagatactgcgacctccctagcaaactggggcacaaacttaattaagccacccaggtaatggggcagtcttctggaggaggagggctgtttaccagcagtggcaacattggaatggccctgcctaacgacatgtatgacttgcatgacctttccaaagctgaactggccgcacctcagcttattatgctggcaaatgtggccttaactggggaagtaaatggcagctgctgtgattacctggtcggtgaagaaagacagatggcagaactgatgccggttggggataacaacttttcagatagtgaagaaggagaaggacttgaagagtctgctgatataaaaggtgaacctcatggactggaaaacatggaactgagaagtttggaactcagcgtcgtagaacctcagcctgtatttgaggcatcaggtgctccagatatttacagttcaaataaagatcttccccctgaaacacctggagcggaggacaaaggcaagagctcgaagaccaaaccctttcgctgtaagccatgccaatatgaagcagaatctgaagaacagtttgtgcatcacatcagagttcacagtgctaagaaattttttgtggaagagagtgcagagaagcaggcaaaagccagggaatctggctcttccactgcagaagagggagatttctccaagggccccattcgctgtgaccgctgcggctacaatactaatcgatatgatcactatacagcacacctgaaacaccacaccagagctggggataatgagcgagtctacaagtgtatcatttgcacatacacaacagtgagcgagtatcactggaggaaacatttaagaaaccattttccaaggaaagtatacacatgtggaaaatgcaactatttttcagacagaaaaaacaattatgttcagcatgttagaactcatacaggagaacgcccatataaatgtgaactttgtccttactcaagttctcagaagactcatctaactagacatatgcgtactcattcaggtgagaagccatttaaatgtgatcagtgcagttatgtggcctctaatcaacatgaagtaacccgccatgcaagacaggttcacaatgggcctaaacctcttaattgcccacactgtgattacaaaacagcagatagaagcaacttcaaaaaacatgtagagctacatgtgaacccacggcagttcaattgccctgtatgtgactatgcagcttccaagaagtgtaatctacagtatcacttcaaatctaagcatcctacttgtcctaataaaacaatggatgtctcaaaagtgaaactaaagaaaaccaaaaaacgagaggctgacttgcctgataatattaccaatgaaaaaacagaaatagaacaaacaaaaataaaaggggatgtggctggaaagaaaaatgaaaagtccgtcaaagcagagaaaagagatgtctcaaaagagaaaaagccttctaataatgtgtcagtgatccaggtgactaccagaactcgaaaatcagtaacagaggtgaaagagatggatgtgcatacaggaagcaattcagaaaaattcagtaaaactaagaaaagcaaaaggaagctggaagttgacagccattctttacatggtcctgtgaatgatgaggaatcttcaacaaaaaagaaaaagaaggtagaaagcaaatccaaaaataatagtcaggaagtgccaaagggtgacagcaaagtggaggagaataaaaagcaaaatacttgcatgaaaaaaagtacaaagaagaaaactctgaaaaataaatcaagtaagaaaagcagtaagcctcctcagaaggaacctgttgagaagggatctgctcagatggaccctcctcagatggggcctgctcccacagaggcggttcagaaggggcccgttcaggtggagccgccacctcccatggagcatgctcagatggagggtgcccagatacggcctgctcctgacgagcctgttcagatggaggtggttcaggaggggcctgctcagaaggagctgctgcctcccgtggagcctgctcagatggtgggtgcccaaattgtacttgctcacatggagctgcctcctcccatggagactgctcagacggaggttgcccaaatggggcctgctcccatggaacctgctcagatggaggttgcccaggtagaatctgctcccatgcaggtggtccagaaggagcctgttcagatggagctgtctcctcccatggaggtggtccagaaggagcctgttcagatagagctgtctcctcccatggaggtggtccagaaggaacctgttaagatagagctgtctcctcccatagaggtggtccagaaggagcctgttcagatggagttgtctcctcccatgggggtggttcagaaggagcctgctcagagggagccacctcctcccagagagcctccccttcacatggagccaatttccaaaaagcctcctctccgaaaagataaaaaggaaaagtctaacatgcagagtgaaagggcacggaaggagcaagtccttattgaagttggcttagtgcctgttaaagatagctggcttctaaaggaaagtgtaagcacagaggatctctcaccaccatcaccaccactgccaaaggaaaatttaagagaagaggcatcaggagaccaaaaattactcaacacaggtgaaggaaataaagaagcccctcttcagaaagtaggagcagaagaggcagatgagagcctacctggtcttgctgctaatatcaacgaatctacccatatttcatcctctggacaaaacttgaatacgccagagggtgaaactttaaatggtaaacatcagactgacagtatagtttgtgaaatgaaaatggacactgatcagaacacaagagagaatctcactggtataaattcaacagttgaagaaccagtttcaccaatgcttcccccttcagcagtagaagaacgtgaagcagtgtccaaaactgcactggcatcacctcctgctacaatggcagcaaatgagtctcaggaaattgatgaagatgaaggcatccacagccatgaaggaagtgacctaagtgacaacatgtcagagggtagtgatgattctggattgcatggggctcggccagttccacaagaatctagcagaaaaaatgcaaaggaagccttggcagtcaaagcggctaagggagattttgtttgtatcttctgtgatcgttctttcagaaagggaaaagattacagcaaacacctcaatcgccatttggttaatgtgtactatcttgaagaagcagctcaagggcaggagtaaagctgaatctaagtcgacttaagaaccgctcgaggccggcaaggccggatccagacatgataagatacattgatgagtttggacaaaccacaactagaatgcagtgaaaaaaatgctttatttgtgaaatttgtgatgctattgctttatttgtaaccattataagctgcaataaacaagttaacaacaacaattgcattcattttatgtttcaggttcagggggaggtgtgggaggttttttaaagcaagtaaaacctctacaaatgtggtatggctgattatgatctcattcatccttttctcctcgtccctccttcattcattcatagccccccgccctgcccgcttcagcatttcattcattcattcattcattcatttcccggagctccgctagcgcacaccccttcagccgaaggccccagcgcgcaggcgcaggccgggagaggcaggcaccctccaatcgtcgggcgtccttcctcctccgggcggccgcccgcttccccatgaatgaacattgacgtcaatggggcggggcgcgcccacgtgaccccgcgcgctcccctttataaggcggtggaggcgcgggcgctgtccagcgtgctgaagccggagcgagctagccgcccggagccgcgccgacccagctgagcccagcccacgggacgccagacctcgaccgtcgctcctaccccggccaccgctcggagccgaggcggacgcgtcccgatcttcccctgtccccaccctgccccgaccctcctctccacctctcgcgtcgtgacaccagctggtaaatactccgctgttcgtccctcaaaccctcggcagccagccgtgggcgtgagggagggttctctctcctctcgatgggggtgttgcaaacacagcggggagccccctggtaagggtccccggtaaacgggggagtcgcagctttttctcttgctgctgaagtcgcccacgcaccatccggggagtcctacggggagggagcagagatttttttttcccccatattgctgctgcttagtacgtgggcgatggcagtgagatggctcagggaaggggccgaggaggccctgggtaagcgagggcttcgggggttattttcccatttacacggctccagagatcggcacaacatcttcctcctttgctcctaaacgttcctcttctgggtaaggtttgggggatcagggaagccccgggtttcctgctgaaaggtgggggaagggaacgtagacctagagaggggaattcttacagaaatcctctttttttggtcccttctatttttcagtctccggcagcctcttggtcatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctaccccgaccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcacccagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaagtaaacgcgtgaattcactcctcaggtgcaggctgcctatcagaaggtggtggctggtgtggccaatgccctggctcacaaataccactgagatctttttccctctgccaaaaattatggggacatcatgaagccccttgagcatctgacttctggctaataaaggaaatttattttcattgcaatagtgtgttggaattttttgtgtctctcactcggaaggacatatgggagggcaaatcatttaaaacatcagaatgagtatttggtttagagtttggcaacatatgcccatatgctggctgccatgaacaaaggttggctataaagaggtcatcagtatatgaaacagccccctgctgtccattccttattccatagaaaagccttgacttgaggttagattttttttatattttgttttgtgttatttttttctttaacatccctaaaattttccttacatgttttactagccagatttttcctcctctcctgactactcccagtcatagctgtccctcttctcttatggagatccctcgacctgcagcccaagcttggcgtaatcatggtcatagctgtttcctg。
根据本发明一种典型的实施方式,提供一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的载体。该载体包括上述任一种基因元件。基因元件cias可以连接至puc系列或其他克隆质粒或其他功能性质粒或宿主基因组中实现cias核酸序列的稳定遗传,可以以不同的载体形式运载基因元件cias转入不同的宿主中行使功能。
根据本发明一种典型的实施方式,提供一种宿主细胞。该宿主细胞包括上述任一种用于检测crispr/cas9基因编辑系统切割基因产生移码突变的载体,例如宿主细胞为大肠杆菌。将用于检测crispr/cas9基因编辑系统切割基因产生移码突变的载体转化到大肠杆菌中,从阳性克隆中提取质粒。基因元件cias可以进行不同物种的密码子优化,即可在不同宿主细胞行使功能。
根据本发明一种典型的实施方式,提供一种检测crispr/cas9基因编辑系统切割基因产生移码突变的方法。该方法包括采用上述用于检测crispr/cas9基因编辑系统切割基因产生移码突变的载体进行检测。
优选的,包括以下步骤:s1,将上述载体插入被切割目标dna序列,然后转染至细胞系中,同时转入装载有grna的载体;s2,观察第一报告基因和第二报告基因的表达情况,根据第一报告基因和第二报告基因的表达情况判断grna对目标dna序列的编辑是否产生了移码突变。优选的,当第二报告基因正常表达时,判断为crispr/cas9基因编辑系统切割基因产生了移码突变,并扩增目标dna序列进行测序。其中,优选的,细胞系为hek293细胞系。hek293细胞系为易转染细胞系,便于快速检测。
构建本发明的基因元件cias有助于鉴定细胞内grna/cas9复合体切割目的dna序列和其他可能的配对dna序列后产生的移码突变情况,为进一步对设计的grna提供胞内编辑的信息和为基因编辑应用提供背景信息。本发明构建的pcias质粒(即包括上述基因元件的载体,例如:seqidno:1)可以直接连接目的dna序列,转染至hek293细胞系中,同时转入设计的装载grna的载体。48小时后,在荧光显微镜下观察细胞表达出来的荧光蛋白来确定grna对目的dna序列的编辑是否产生了移码突变。针对表现出来的绿色荧光株抽提dna后,pcr扩增目的dna序列,使用t载体装载之后测定核酸序列,用来鉴定被编辑的dna序列。
根据本发明一种典型的实施方式,基因元件cias如图1所示,是通过dna序列合成的方式获得。合成的cias序列需要通过sanger测序验证,将cias装载至puc19质粒。
根据本发明一种典型的实施方式,功能性载体的使用方法,包括以下步骤:a.使用pcr扩增将cias线性化;b.将目的片段cias连接至线性化的ppuro质粒中而获得目的质粒cias-ppuro(或称为pcias);c.将步骤b获得的质粒转化至大肠杆菌中,筛选正确的克隆子,并通过sanger测序验证;d.使用xbai和kpni酶切割pcias质粒后,连接目的dna序列至pcias质粒。在一个实施例中,分别转入不同目的dna序列至pcias质粒,并转入grna载体,来验证切割活性和造成移码突变后的碱基序列。
该pcias质粒可以作为检测目的dna序列的初始载体,并且该载体具有简单、快速、省时、省力等遗传操作的显著优点,可用于检测grna对目的dna切割活性验证和编辑后的dna序列分析确认。
本发明做构建的检测crispr/cas9基因编辑系统切割基因产生移码突变的载体可以快速检测设计的grna所产生的基因组切割后的突变是否是移码突变和产生移码突变后所产生的碱基敲除类型及类型几率。本发明所述的方法能够快速、高效、即时和稳定的检测设计的grna/cas9复合体切割基因组后,所修复的基因型是否符合实验预期,在基因功能研究方面有着重要的意义。
在本发明一种典型的实施方式中,基因元件cias的作用机理的技术优势是,将目的dna序列装载至pcias(pcias是cias连接至载体上的名称)质粒上后,同时转grna载体至hek293细胞系中,能够及时快速检测grna是否能够切割目的dna序列。由于pcias可以携带有puro抗性筛选基因,所以添加puro后,能去除没有转入目的质粒的细胞,降低了高背景的缺点。
为了验证基因元件cias快速检测grna的切割活性和被编辑后的碱基序列,本发明在一个实施例中,装载目的dna序列至pcias上,同时转grna载体质粒一起转至hek293细胞系中。经过puro要筛选后,使用荧光显微镜观察报告基因的表达情况。如果细胞表达绿色荧光,表示grna是能够切割目的dna序列的;如果细胞表达红色荧光,表示grna是不能切割目的dna序列的。另外,通过提取细胞系的dna来鉴定目标dna序列被编辑的碱基情况,来分析编辑结果而确定适合的grna。
下面将结合实施例进一步说明本发明的有益效果。本发明中为详细描述的步骤或试剂可以采用本领域的常规技术手段实现或采用本领域的常规试剂。
实施例1
在下述实施例中,所用的酶购自neb,hek293细胞系来自北京赛贝生物科技有限公司。基因元件cmv增强子、ef1启动子、红色荧光蛋白基因、rest基因、sv40pa终止子、s沉默子、绿色荧光蛋白基因和β-球蛋白聚腺苷酸的序列信息来自ncbi数据库,合成由金唯智公司完成,并转载到puc19质粒上(见图2a)。参考图1为基因元件cias示意图谱。
本发明通过遗传操作过程获得pcias质粒(图2b),示例性过程如下:
将300ng的质粒ppuro的dna与0.5μl限制性内切酶xhai混合,进行酶切,37℃水浴1h,回收后获得线性化载体片段ppuro。以cias-puc19质粒为模板,以cia-p1(seqidno:2)和cia-p2(seqidno:3)为引物扩增cias序列。使用slic克隆方法连接上述2片段。将100ng的ppuro和插入片段cias按照摩尔比1:2,利用0.5u的t4dna聚合酶于室温下处理2.5分钟,随后冰浴并转化至大肠杆菌中,涂布在含有amp平板上筛选正确的克隆子,获得质粒pcias,并且进行测序验证。
seqidno:2
gtaaaacgacggccagtgaattcgacattgattattgactagttattaatag
seqidno:3
gccaagcttgggctgcaggtcgagggatctccataagagaag
实施例2
以h9细胞系基因组总dna为模板,使用引物target-p-1(seqidno:4)和target-p-2(seqidnno:5)进行pcr反应。反应程序:98℃预变性5分钟;98℃变形10s,60℃退火15s,72℃延伸1min,35个循环;72℃延伸10min。回收后,获得目的dna序列(即为target序列,seqidno:6)。
将300ng的质粒pcias的dna与限制性内切酶xbai和kpni酶切(分别为0.5μl和0.5μl)混合,进行酶切,37℃水浴1h,回收后获得线性化载体片段pcias。使用slic克隆方法连接上述2片段。将100ng的pcias和插入片段target序列按照摩尔比1:2,利用0.5u的t4dna聚合酶于室温下处理2.5分钟,随后冰浴并转化至大肠杆菌中,涂布在含有amp平板上筛选正确的克隆子,获得质粒target-pcias(图3a),并且进行测序验证。
为了验证是否是由于dna序列所致的结果,本发明中使用了hla-e基因内的一段dna序列作为dna序列对比实验。根据以上所述的方法,使用引物target-1-p-1(seqidno:7)和target-1-p-2(seqidnno:8),扩增得到target-1序列(seqidnno:9),连接至载体上获得质粒target-1-pcias(图3b),并且进行测序验证。
seqidno:4
gtctagactccctgatcgcctatagatc
seqidno:5
cggtacccatgaagaaagcaggtgtggg
seqidno:6
catgaagaaagcaggtgtgggtcctggaccaatagccctcctgaggtctgtcctcagggaccttcccctgtgacttgtgactgctgggatcaggtcccatcaccgccgtaatcaaggtgataaatctgtccttcattttaacaggtgctttacaaaagagtaagtgctggcacacagggcccaggctgggtcggcccatgattgtggaaggtgcttcccagtaatgagacagggcacatttctagctggggcttggaaccctcagtgagacaagaaatctcagaccccacccttcaccccttctccacctgagctcttcctcctccacatcacggcagcgaccacagctccagtgatcacagctccaaggagaaccaggccagcaatgatgcccacgatggggatggtgggctgggaagacagctctgggaaaagaggggaaggtgaggggccctgaccctgctaaaggtctccagagaggctcctgctttccctaagagacatgacacccccatctccctccttaccccatctcagggtgaggggcttgggcagaccctcatgctgcacatggcaggtgtatctctgctcctctccagaaggcaccaccacagccgcccacttctggaaggttccatcccctgcaggcctggtctccacgagctccgtgtcctgggtctggtcctccccatcccgctgccaggtcagtgtgatctccgcagggtagaagcccagggcccagcacctcagggtggcctcatggtcagagatggggtggtgggtcatatgtgtcttgggggggtctgacgggaagagtcagaaaattcaggcattttgcatctgtcatgggacactccaccagcacgcatgtggccatcttgagaatggacaggacacccgggatggggaagagagcacagaacccagacaccagcctggacacaggcacctgggataatcttctattccctgagaagggaacagcgacttctggtcctgacctgagtggaggctgagagactcagaagtgctggactcagacccccacacacattgagtgtgaagcagagaacaaggcctgagaggaaaagtcacgggcccaaggctgctgccggtgtcaaagggaaccactcatcagtattcgagggatcgtcttcccgtcactccttcagagattttatcccttaattgtgtcagagagcagggcggaacctcagagtcactctctggtacaggatctggaaacccaggaggattcctctccctcaggaccagagggagggtgatattctagtgttggtcccaattgtctcccctccttgtgggaggccagcccgggagatctataggcgatcagggag
seqidno:7
cggtaccgcagactcagttctcattcc
seqidno:8
gtctagagcctgtgtggatgctgagtg
seqidno:9
gcagactcagttctcattcccaatgggtgtcgggtttctagagaagccaatcagcgtcgccacgactcccgactataaagtccccatccggactcaagaagttctcaggactcagaggctgggatcatggtagatggaaccctccttttactcctctcggaggccctggcccttacccagacctgggcgggtgagtgcggggtcgggatggaaacggcctctaccgggagtagagaggggccggcccggcgggggcgaaggactcggggagccgcgccgggaggagggtcgggccgatctcagcccctcctcgcccccaggctcccactccttgaagtatttccacacttccgtgtcccggcccggccgcggggagccccgcttcatctctgtgggctacgtggacgacacccagttcgtgcgcttcgacaacgacgccgcgagtccgaggatggtgccgcgggcgccgtggatggagcaggaggggtcagagtattgggaccgggagacacggagcgccagggacaccgcacagattttccgagtgaatctgcggacgctgcgcggctactacaatcagagcgaggccggtgagtgaccccggccaggggagcaggtcacgacccctccccatcccccacggacggcgcgggtcccctcgaatcttcgggtcccagattcaccccaaggctgcggaacccgcccagaccctagaccggggagagtctcaggcgcctttacccggttctttttcagtttaggccaaaatgcccacagggtggtggcgacgggggcggggcttggtgggcgggactgactaaggggcggggccagggtctcacaccctgcagtggatgcatggctgcgagctggggcccgacgggcgcttcctccgcgggtatgaacagttcgcctacgacggcaaggattatctcaccctgaatgaggacctgcgctcctggaccgcggtggacacggcggctcagatctccgagcaaaagtcaaatgatgcctctgaggcggagcaccagagagcctacctggaagacacatgcgtggagtggctccacaaatacctggagaaggggaaggagacgctgcttcacctgggtaagagggtccacagggctactctcccatctccttcttgggctaggactgtgcccacagctgacagacctcaaacagtagaagaaacagggatggaggccagaataccactcctcccttggatcaggagagggagctgtcacctgaggtacaggagatcctataccacagagtgactctcttaaagggccagacctctctcaggggcaattaaggaatctagtctcgctggagattccatccttcagatgaactgatgagcagttctctttgactcccagtattaggaatcacgggggagtttctctcgtgcctgattctcagccccacaccaagagtttttggaggtctgactccagcttttctcagtcactcagcatccacacaggc
实施例3
为了检验目标基因的grna的切割活性。首选需要将目标grna装载至载体上,所需要的操作如下:
本发明中针对目标hla-a基因进行grna设计后,使用引物grna12b-p-01(seqidno:10)和grna12b-p-02(seqidno:11)混合后在pcr仪中执行退火程序,获得grna12b片段。
将300ng的质粒phs-cas9的dna与限制性内切酶kpni酶切(为0.5μl)混合,进行酶切,37℃水浴1h,回收后获得线性化载体phs-cas9。
将上述产物使用t4连接酶连接,并转化至大肠杆菌中,涂布在含有amp平板上筛选正确的克隆子,获得质粒grna12b-phs-cas9(图4a),并且进行测序验证。
如果需要检测多grna时,则需转入多个装载grna的载体即可实现。
另外,对目标基因hla-a进行设计grnaa,使用的引物是grnaa-p-01(seqidno:12)和grnaa-p-02(seqidno:13),所切割的位点不在target序列中,是为了检测当目的序列没有cas9/grnaa的切割位点时,是会引导红色荧光蛋白表达,所使用的构建方法为上述构建方法,本发明构建了质粒grnaa-phs-cas9(图4b)
同时,按着以上的设计原则,本发明中对另一段dna序列(seqidno:9)基因编辑进行分析。所设计的grna分别为sg01rna-phs-cas9(图4c)和sg04rna-phs-cas9(图4d)。
seqidno:10
gtggaaaggacgaaacaccgcgtggagaccaggcctgcag
seqidno:11
gctatttctagctctaaaacctgcaggcctggtctccacg
seqidno:12
gtggaaaggacgaaacaccgttacaagtaagacctactcc
seqidno:13
gctatttctagctctaaaacggagtaggtcttacttgtaa
实施例4
按照lipofectamine3000的实验方案,将质粒target-pcias、grna12b-phs-cas9、target-1-pcias和sg01rna-phs-cas9分别同时转入hek293细胞系中。37度孵育细胞2-4天后,在荧光显微镜下观察细胞,发现细胞呈现绿色荧光(图5a和图5c),说明目标dna序列是产生了移码突变。
抽提这一细胞系的dna,使用引物jd-p-1(seqidno:14)和jd-p-2(seqidno:15)扩增片段。利用t载体连接上述片段,转化至大肠杆菌中,涂布在含有amp平板上。将20个克隆子随机挑取扩培后,送测序公司进行序列测定验证。
seqidno:14
ggtgccctccatgtacagc
seqidno:15
ctgcgccgttacagatccaa
实施例5
按照实施例3和实施例4所述的方法,构建质粒grnaa-phs-cas9(图4b)和grnab-phs-cas9(图4d)并且分别同时和target-pcias、target-1-pcias转至hek293细胞系中。37度孵育细胞2-4天后,在荧光显微镜下观察细胞,发现细胞呈现红色荧光(图5b和图5d),说明grna序列没有切割目标dna序列。
抽提这一细胞系的dna,使用引物jd-p-1(seqidno:14)和jd-p-2(seqidno:15)扩增片段。利用t载体连接上述片段,转化至大肠杆菌中,涂布在含有amp平板上。将20个克隆子随机挑取扩培后,送测序公司进行序列测定验证。
从以上的描述中,可以看出,本发明上述的实施例实现了如下技术效果:
1)相比于在直接检测目的宿主的基因组编辑情况,本发明所构建的质粒pcias,当目的dna序列装载至pcias后转至hek293细胞系中,可快速检测grna的切割活性和分析被切割后的碱基序列;
2)使用本发明所构建的质粒和检测方法所具有的优势是能够快速、简单检测目的dna序列被不同的grna编辑情况,可以极大节约试验时间和操作环节,降低实验成本,使科研效率的到提高;
3)对于多grna检测要求,本发明方法也是可以提供检测grna切割活性和编辑后的基因组编辑情况,并且可以进行分析off-target序列是否存在被编辑的情况。。
以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
序列表
<110>北京赛贝生物技术有限公司
<120>用于检测crispr/cas9基因编辑系统切割基因产生移码突变的基因元件及应用
<130>pn90870sbsw
<160>15
<170>siposequencelisting1.0
<210>1
<211>7703
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(7703)
<223>基因元件cias
<400>1
tgtaaaacgacggccagtgaattcgacattgattattgactagttattaatagtaatcaa60
ttacggggtcattagttcatagcccatatatggagttccgcgttacataacttacggtaa120
atggcccgcctggctgaccgcccaacgacccccgcccattgacgtcaataatgacgtatg180
ttcccatagtaacgccaatagggactttccattgacgtcaatgggtggagtatttacggt240
aaactgcccacttggcagtacatcaagtgtatcatatgccaagtacgccccctattgacg300
tcaatgacggtaaatggcccgcctggcattatgcccagtacatgaccttatgggactttc360
ctacttggcagtacatctacgtattagtcatcgctattaccatggaatagcaacagacat420
acaaactaaagaattacaaaaacaaattacaaaaattcaaaattttatcgatactagtaa480
ggatctgcgatcgctccggtgcccgtcagtgggcagagcgcacatcgcccacagtccccg540
agaagttggggggaggggtcggcaattgaacgggtgcctagagaaggtggcgcggggtaa600
actgggaaagtgatgtcgtgtactggctccgcctttttcccgagggtgggggagaaccgt660
atataagtgcagtagtcgccgtgaacgttctttttcgcaacgggtttgccgccagaacac720
agctgaagcttcgaggggctcgcatctctccttcacgcgcccgccgccctacctgaggcc780
gccatccacgccggttgagtcgcgttctgccgcctcccgcctgtggtgcctcctgaactg840
cgtccgccgtctaggtaagtttaaagctcaggtcgagaccgggcctttgtccggcgctcc900
cttggagcctacctagactcagccggctctccacgctttgcctgaccctgcttgctcaac960
tctacgtctttgtttcgttttctgttctgcgccgttacagatccaagctgtgaccggcgc1020
ctacatgtctagannnnnnnnnnnnnnnnnnnnnnnnnnnnnnggtaccgtgtctaaggg1080
cgaagagctgattaaggagaacatgcacatgaagctgtacatggagggcaccgtgaacaa1140
ccaccacttcaagtgcacatccgagggcgaaggcaagccctacgagggcacccagaccat1200
gagaatcaaggtggtcgagggcggccctctccccttcgccttcgacatcctggctaccag1260
cttcatgtacggcagcaaaaccttcatcaaccacacccagggcatccccgacttctttaa1320
gcagtccttccctgagggcttcacatgggagagagtcaccacatacgaagacgggggcgt1380
gctgaccgctacccaggacaccagcctccaggacggctgcctcatctacaacgtcaagat1440
cagaggggtgaacttcccatccaacggccctgtgatgcagaagaaaacactcggctggga1500
ggcctccaccgagatgctgtaccccgctgacggcggcctggaaggcagaagcgacatggc1560
cctgaagctcgtgggcgggggccacctgatctgcaacttgaagaccacatacagatccaa1620
gaaacccgctaagaacctcaagatgcccggcgtctactatgtggacagaagactggaaag1680
aatcaaggaggccgacaaagaaacctacgtcgagcagcacgaggtggctgtggccagata1740
ctgcgacctccctagcaaactggggcacaaacttaattaagccacccaggtaatggggca1800
gtcttctggaggaggagggctgtttaccagcagtggcaacattggaatggccctgcctaa1860
cgacatgtatgacttgcatgacctttccaaagctgaactggccgcacctcagcttattat1920
gctggcaaatgtggccttaactggggaagtaaatggcagctgctgtgattacctggtcgg1980
tgaagaaagacagatggcagaactgatgccggttggggataacaacttttcagatagtga2040
agaaggagaaggacttgaagagtctgctgatataaaaggtgaacctcatggactggaaaa2100
catggaactgagaagtttggaactcagcgtcgtagaacctcagcctgtatttgaggcatc2160
aggtgctccagatatttacagttcaaataaagatcttccccctgaaacacctggagcgga2220
ggacaaaggcaagagctcgaagaccaaaccctttcgctgtaagccatgccaatatgaagc2280
agaatctgaagaacagtttgtgcatcacatcagagttcacagtgctaagaaattttttgt2340
ggaagagagtgcagagaagcaggcaaaagccagggaatctggctcttccactgcagaaga2400
gggagatttctccaagggccccattcgctgtgaccgctgcggctacaatactaatcgata2460
tgatcactatacagcacacctgaaacaccacaccagagctggggataatgagcgagtcta2520
caagtgtatcatttgcacatacacaacagtgagcgagtatcactggaggaaacatttaag2580
aaaccattttccaaggaaagtatacacatgtggaaaatgcaactatttttcagacagaaa2640
aaacaattatgttcagcatgttagaactcatacaggagaacgcccatataaatgtgaact2700
ttgtccttactcaagttctcagaagactcatctaactagacatatgcgtactcattcagg2760
tgagaagccatttaaatgtgatcagtgcagttatgtggcctctaatcaacatgaagtaac2820
ccgccatgcaagacaggttcacaatgggcctaaacctcttaattgcccacactgtgatta2880
caaaacagcagatagaagcaacttcaaaaaacatgtagagctacatgtgaacccacggca2940
gttcaattgccctgtatgtgactatgcagcttccaagaagtgtaatctacagtatcactt3000
caaatctaagcatcctacttgtcctaataaaacaatggatgtctcaaaagtgaaactaaa3060
gaaaaccaaaaaacgagaggctgacttgcctgataatattaccaatgaaaaaacagaaat3120
agaacaaacaaaaataaaaggggatgtggctggaaagaaaaatgaaaagtccgtcaaagc3180
agagaaaagagatgtctcaaaagagaaaaagccttctaataatgtgtcagtgatccaggt3240
gactaccagaactcgaaaatcagtaacagaggtgaaagagatggatgtgcatacaggaag3300
caattcagaaaaattcagtaaaactaagaaaagcaaaaggaagctggaagttgacagcca3360
ttctttacatggtcctgtgaatgatgaggaatcttcaacaaaaaagaaaaagaaggtaga3420
aagcaaatccaaaaataatagtcaggaagtgccaaagggtgacagcaaagtggaggagaa3480
taaaaagcaaaatacttgcatgaaaaaaagtacaaagaagaaaactctgaaaaataaatc3540
aagtaagaaaagcagtaagcctcctcagaaggaacctgttgagaagggatctgctcagat3600
ggaccctcctcagatggggcctgctcccacagaggcggttcagaaggggcccgttcaggt3660
ggagccgccacctcccatggagcatgctcagatggagggtgcccagatacggcctgctcc3720
tgacgagcctgttcagatggaggtggttcaggaggggcctgctcagaaggagctgctgcc3780
tcccgtggagcctgctcagatggtgggtgcccaaattgtacttgctcacatggagctgcc3840
tcctcccatggagactgctcagacggaggttgcccaaatggggcctgctcccatggaacc3900
tgctcagatggaggttgcccaggtagaatctgctcccatgcaggtggtccagaaggagcc3960
tgttcagatggagctgtctcctcccatggaggtggtccagaaggagcctgttcagataga4020
gctgtctcctcccatggaggtggtccagaaggaacctgttaagatagagctgtctcctcc4080
catagaggtggtccagaaggagcctgttcagatggagttgtctcctcccatgggggtggt4140
tcagaaggagcctgctcagagggagccacctcctcccagagagcctccccttcacatgga4200
gccaatttccaaaaagcctcctctccgaaaagataaaaaggaaaagtctaacatgcagag4260
tgaaagggcacggaaggagcaagtccttattgaagttggcttagtgcctgttaaagatag4320
ctggcttctaaaggaaagtgtaagcacagaggatctctcaccaccatcaccaccactgcc4380
aaaggaaaatttaagagaagaggcatcaggagaccaaaaattactcaacacaggtgaagg4440
aaataaagaagcccctcttcagaaagtaggagcagaagaggcagatgagagcctacctgg4500
tcttgctgctaatatcaacgaatctacccatatttcatcctctggacaaaacttgaatac4560
gccagagggtgaaactttaaatggtaaacatcagactgacagtatagtttgtgaaatgaa4620
aatggacactgatcagaacacaagagagaatctcactggtataaattcaacagttgaaga4680
accagtttcaccaatgcttcccccttcagcagtagaagaacgtgaagcagtgtccaaaac4740
tgcactggcatcacctcctgctacaatggcagcaaatgagtctcaggaaattgatgaaga4800
tgaaggcatccacagccatgaaggaagtgacctaagtgacaacatgtcagagggtagtga4860
tgattctggattgcatggggctcggccagttccacaagaatctagcagaaaaaatgcaaa4920
ggaagccttggcagtcaaagcggctaagggagattttgtttgtatcttctgtgatcgttc4980
tttcagaaagggaaaagattacagcaaacacctcaatcgccatttggttaatgtgtacta5040
tcttgaagaagcagctcaagggcaggagtaaagctgaatctaagtcgacttaagaaccgc5100
tcgaggccggcaaggccggatccagacatgataagatacattgatgagtttggacaaacc5160
acaactagaatgcagtgaaaaaaatgctttatttgtgaaatttgtgatgctattgcttta5220
tttgtaaccattataagctgcaataaacaagttaacaacaacaattgcattcattttatg5280
tttcaggttcagggggaggtgtgggaggttttttaaagcaagtaaaacctctacaaatgt5340
ggtatggctgattatgatctcattcatccttttctcctcgtccctccttcattcattcat5400
agccccccgccctgcccgcttcagcatttcattcattcattcattcattcatttcccgga5460
gctccgctagcgcacaccccttcagccgaaggccccagcgcgcaggcgcaggccgggaga5520
ggcaggcaccctccaatcgtcgggcgtccttcctcctccgggcggccgcccgcttcccca5580
tgaatgaacattgacgtcaatggggcggggcgcgcccacgtgaccccgcgcgctcccctt5640
tataaggcggtggaggcgcgggcgctgtccagcgtgctgaagccggagcgagctagccgc5700
ccggagccgcgccgacccagctgagcccagcccacgggacgccagacctcgaccgtcgct5760
cctaccccggccaccgctcggagccgaggcggacgcgtcccgatcttcccctgtccccac5820
cctgccccgaccctcctctccacctctcgcgtcgtgacaccagctggtaaatactccgct5880
gttcgtccctcaaaccctcggcagccagccgtgggcgtgagggagggttctctctcctct5940
cgatgggggtgttgcaaacacagcggggagccccctggtaagggtccccggtaaacgggg6000
gagtcgcagctttttctcttgctgctgaagtcgcccacgcaccatccggggagtcctacg6060
gggagggagcagagatttttttttcccccatattgctgctgcttagtacgtgggcgatgg6120
cagtgagatggctcagggaaggggccgaggaggccctgggtaagcgagggcttcgggggt6180
tattttcccatttacacggctccagagatcggcacaacatcttcctcctttgctcctaaa6240
cgttcctcttctgggtaaggtttgggggatcagggaagccccgggtttcctgctgaaagg6300
tgggggaagggaacgtagacctagagaggggaattcttacagaaatcctctttttttggt6360
cccttctatttttcagtctccggcagcctcttggtcatggtgagcaagggcgaggagctg6420
ttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagttc6480
agcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagttcatc6540
tgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgacctacggc6600
gtgcagtgcttcagccgctaccccgaccacatgaagcagcacgacttcttcaagtccgcc6660
atgcccgaaggctacgtccaggagcgcaccatcttcttcaaggacgacggcaactacaag6720
acccgcgccgaggtgaagttcgagggcgacaccctggtgaaccgcatcgagctgaagggc6780
atcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaactacaacagc6840
cacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatc6900
cgccacaacatcgaggacggcagcgtgcagctcgccgaccactaccagcagaacaccccc6960
atcggcgacggccccgtgctgctgcccgacaaccactacctgagcacccagtccgccctg7020
agcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgcc7080
gggatcactctcggcatggacgagctgtacaagtaaacgcgtgaattcactcctcaggtg7140
caggctgcctatcagaaggtggtggctggtgtggccaatgccctggctcacaaataccac7200
tgagatctttttccctctgccaaaaattatggggacatcatgaagccccttgagcatctg7260
acttctggctaataaaggaaatttattttcattgcaatagtgtgttggaattttttgtgt7320
ctctcactcggaaggacatatgggagggcaaatcatttaaaacatcagaatgagtatttg7380
gtttagagtttggcaacatatgcccatatgctggctgccatgaacaaaggttggctataa7440
agaggtcatcagtatatgaaacagccccctgctgtccattccttattccatagaaaagcc7500
ttgacttgaggttagattttttttatattttgttttgtgttatttttttctttaacatcc7560
ctaaaattttccttacatgttttactagccagatttttcctcctctcctgactactccca7620
gtcatagctgtccctcttctcttatggagatccctcgacctgcagcccaagcttggcgta7680
atcatggtcatagctgtttcctg7703
<210>2
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(52)
<223>引物cia-p1
<400>2
gtaaaacgacggccagtgaattcgacattgattattgactagttattaatag52
<210>3
<211>42
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(42)
<223>引物cia-p2
<400>3
gccaagcttgggctgcaggtcgagggatctccataagagaag42
<210>4
<211>28
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(28)
<223>引物target-p-1
<400>4
gtctagactccctgatcgcctatagatc28
<210>5
<211>28
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(28)
<223>引物target-p-2
<400>5
cggtacccatgaagaaagcaggtgtggg28
<210>6
<211>1361
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(1361)
<223>目的dna序列
<400>6
catgaagaaagcaggtgtgggtcctggaccaatagccctcctgaggtctgtcctcaggga60
ccttcccctgtgacttgtgactgctgggatcaggtcccatcaccgccgtaatcaaggtga120
taaatctgtccttcattttaacaggtgctttacaaaagagtaagtgctggcacacagggc180
ccaggctgggtcggcccatgattgtggaaggtgcttcccagtaatgagacagggcacatt240
tctagctggggcttggaaccctcagtgagacaagaaatctcagaccccacccttcacccc300
ttctccacctgagctcttcctcctccacatcacggcagcgaccacagctccagtgatcac360
agctccaaggagaaccaggccagcaatgatgcccacgatggggatggtgggctgggaaga420
cagctctgggaaaagaggggaaggtgaggggccctgaccctgctaaaggtctccagagag480
gctcctgctttccctaagagacatgacacccccatctccctccttaccccatctcagggt540
gaggggcttgggcagaccctcatgctgcacatggcaggtgtatctctgctcctctccaga600
aggcaccaccacagccgcccacttctggaaggttccatcccctgcaggcctggtctccac660
gagctccgtgtcctgggtctggtcctccccatcccgctgccaggtcagtgtgatctccgc720
agggtagaagcccagggcccagcacctcagggtggcctcatggtcagagatggggtggtg780
ggtcatatgtgtcttgggggggtctgacgggaagagtcagaaaattcaggcattttgcat840
ctgtcatgggacactccaccagcacgcatgtggccatcttgagaatggacaggacacccg900
ggatggggaagagagcacagaacccagacaccagcctggacacaggcacctgggataatc960
ttctattccctgagaagggaacagcgacttctggtcctgacctgagtggaggctgagaga1020
ctcagaagtgctggactcagacccccacacacattgagtgtgaagcagagaacaaggcct1080
gagaggaaaagtcacgggcccaaggctgctgccggtgtcaaagggaaccactcatcagta1140
ttcgagggatcgtcttcccgtcactccttcagagattttatcccttaattgtgtcagaga1200
gcagggcggaacctcagagtcactctctggtacaggatctggaaacccaggaggattcct1260
ctccctcaggaccagagggagggtgatattctagtgttggtcccaattgtctcccctcct1320
tgtgggaggccagcccgggagatctataggcgatcagggag1361
<210>7
<211>27
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(27)
<223>引物target-1-p-1
<400>7
cggtaccgcagactcagttctcattcc27
<210>8
<211>27
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(27)
<223>引物target-1-p-2
<400>8
gtctagagcctgtgtggatgctgagtg27
<210>9
<211>1500
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(1500)
<223>target-1序列
<400>9
gcagactcagttctcattcccaatgggtgtcgggtttctagagaagccaatcagcgtcgc60
cacgactcccgactataaagtccccatccggactcaagaagttctcaggactcagaggct120
gggatcatggtagatggaaccctccttttactcctctcggaggccctggcccttacccag180
acctgggcgggtgagtgcggggtcgggatggaaacggcctctaccgggagtagagagggg240
ccggcccggcgggggcgaaggactcggggagccgcgccgggaggagggtcgggccgatct300
cagcccctcctcgcccccaggctcccactccttgaagtatttccacacttccgtgtcccg360
gcccggccgcggggagccccgcttcatctctgtgggctacgtggacgacacccagttcgt420
gcgcttcgacaacgacgccgcgagtccgaggatggtgccgcgggcgccgtggatggagca480
ggaggggtcagagtattgggaccgggagacacggagcgccagggacaccgcacagatttt540
ccgagtgaatctgcggacgctgcgcggctactacaatcagagcgaggccggtgagtgacc600
ccggccaggggagcaggtcacgacccctccccatcccccacggacggcgcgggtcccctc660
gaatcttcgggtcccagattcaccccaaggctgcggaacccgcccagaccctagaccggg720
gagagtctcaggcgcctttacccggttctttttcagtttaggccaaaatgcccacagggt780
ggtggcgacgggggcggggcttggtgggcgggactgactaaggggcggggccagggtctc840
acaccctgcagtggatgcatggctgcgagctggggcccgacgggcgcttcctccgcgggt900
atgaacagttcgcctacgacggcaaggattatctcaccctgaatgaggacctgcgctcct960
ggaccgcggtggacacggcggctcagatctccgagcaaaagtcaaatgatgcctctgagg1020
cggagcaccagagagcctacctggaagacacatgcgtggagtggctccacaaatacctgg1080
agaaggggaaggagacgctgcttcacctgggtaagagggtccacagggctactctcccat1140
ctccttcttgggctaggactgtgcccacagctgacagacctcaaacagtagaagaaacag1200
ggatggaggccagaataccactcctcccttggatcaggagagggagctgtcacctgaggt1260
acaggagatcctataccacagagtgactctcttaaagggccagacctctctcaggggcaa1320
ttaaggaatctagtctcgctggagattccatccttcagatgaactgatgagcagttctct1380
ttgactcccagtattaggaatcacgggggagtttctctcgtgcctgattctcagccccac1440
accaagagtttttggaggtctgactccagcttttctcagtcactcagcatccacacaggc1500
<210>10
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(40)
<223>引物grna12b-p-01
<400>10
gtggaaaggacgaaacaccgcgtggagaccaggcctgcag40
<210>11
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(40)
<223>引物grna12b-p-02
<400>11
gctatttctagctctaaaacctgcaggcctggtctccacg40
<210>12
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(40)
<223>引物grnaa-p-01
<400>12
gtggaaaggacgaaacaccgttacaagtaagacctactcc40
<210>13
<211>40
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(40)
<223>引物grnaa-p-02
<400>13
gctatttctagctctaaaacggagtaggtcttacttgtaa40
<210>14
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(19)
<223>引物jd-p-1
<400>14
ggtgccctccatgtacagc19
<210>15
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>primer_bind
<222>(1)..(20)
<223>引物jd-p-2
<400>15
ctgcgccgttacagatccaa20
- 还没有人留言评论。精彩留言会获得点赞!