通过在高甘油含量的培养基上发酵来改善1,3-丙二醇产生的微生物和方法与流程
本发明涉及一种用于从具有高甘油含量的培养基产生1,3-丙二醇的新方法和微生物,优选地其中所述甘油为工业甘油。更特别地,该微生物过表达hcpr和/或frdx基因。
背景技术:
1,3-丙二醇(1,3-propanediol)(pdo),也称为丙二醇(trimethyleneglycol)或丙二醇(propyleneglycol),是最古老的已知发酵产物之一。它最早于1881年由augustfreund在含有巴氏梭菌(clostridiumpasteurianum)的甘油发酵培养物中鉴定。pdo是甘油发酵的一种典型产物,尽管它已在其他有机底物的厌氧转化中被发现。很少生物体(全部都是细菌)能够形成pdo。这些包括克雷伯氏菌(klebsiella)属(肺炎克雷伯菌(k.bneumoniae))、肠杆菌属(enterobacter)(聚团肠杆菌(e.agglomerans))、柠檬酸杆菌属(citrobacter)(弗氏柠檬酸杆菌(c.freundii))、乳杆菌属(lactobacilli)(短乳杆菌(l.brevis)和布氏乳杆菌(l.buchneri))和梭菌属(clostridia)(丁酸梭菌(c.butyricum)、巴氏梭菌(c.pasteurianum))的肠细菌。其中,就产量和滴度而言,丁酸梭菌(c.butyricum)被认为是pdo的最佳“天然产生者”。
pdo,作为一种双官能有机化合物,可以潜在地用于许多不同的合成反应中,包括作为缩聚反应的单体,以产生聚酯、聚醚和聚氨酯,尤其是聚对苯二甲酸亚丙基酯(ptt)。考虑到pdo的结构和反应性,它还可以用作溶剂、粘合剂、去污剂、化妆品、纺织品(例如服装纤维或地板)和塑料(例如汽车工业、包装或作为涂料)中的成分。
尽管可以使用各种化学方法产生pdo,但它们会产生包含极度污染物质的废物流,从而使以化学方法产生的pdo与石化二醇(例如1,2-乙二醇、1,2-丙二醇和1,4-丁二醇)在成本上没有竞争力。尽管已描述了使用大肠杆菌(escherichiacoli)将d-葡萄糖生物转化为pdo的一种更加环境友好的方法,但该方法具有若干主要缺点。值得注意的是,由于产生菌株的不稳定性,培养是不连续的,并且还需要添加昂贵的辅因子维生素b12。确实,尽管pdo可以通过维生素b12依赖性途径在大肠杆菌中产生,但大肠杆菌本身不产生这种辅因子。
由于由生物柴油工业产生的包含甘油的大量工业甘油的可用性,使用工业甘油作为底物的连续、无维生素b12且碳收率较高的方法将是有利的。
尽管纯甘油具有广泛的应用(例如,作为食品、药物或化妆品添加剂),但生物柴油合成过程中产生的工业甘油通常包含与盐和水混合的80-85%的甘油,因此在其可以用作添加剂之前需要额外的纯化步骤。因此,与其他碳源(诸如葡萄糖或纯甘油)相比,工业甘油被视为废品,而不是有价值的商品,因此代表了pdo的丰富而廉价的发酵底物。
梭状芽胞杆菌代表用于产生pdo的非常有希望的菌株。确实,丁酸梭菌能够使用纯甘油,但也可以使用工业甘油,作为在分批和两阶段连续发酵中通过b12非依赖途径产生pdo的唯一碳源(papanikolaou等,2000)。但是,在最高甘油浓度下,以0.02h-1的稀释率获得的最大pdo滴度为48.1g/l,对应于0.96g/l/h的产生率。在进料培养基中最大甘油浓度为90g/l的情况下在酵母提取物(一种昂贵的含有机氮的化合物,已知会增加细菌生物量)的存在下进行培养。
wo2006/128381公开了工业甘油在使用pdo的“天然产生者”,诸如肺炎克雷伯菌、丁酸梭菌或巴氏梭菌的分批和补料分批培养物中产生pdo的用途。然而,wo2006/128381中使用的培养基也包含酵母提取物。达到的最大产生率也与papanikolaou等人,2000中发现的产生率相似,在0.8至1.1g/l/h之间。
gonzalez-pajuelo等人,2005中描述了被称为丙酮丁酸梭菌dg1pspd5的重组丙酮丁酸梭菌菌株的性能,该菌株含有来自丁酸梭菌的维生素b12非依赖性甘油脱水酶和pdo脱氢酶。此菌株最初在含有高达120g/l的纯甘油的进料培养基中生长并产生pdo。此外,使用含有60g/l的纯甘油或工业甘油的补料培养基进行的分析未显示任何差异。这些结果也是在酵母提取物的存在下获得的。然而,未测试包含浓度高于60g/l的甘油的工业甘油。
最近,wo2010/128070已经公开了丙酮丁醇梭菌dg1pspd5菌株,其已进一步适应于在高浓度的工业甘油上和不存在酵母提取物的情况下生长。丙酮丁醇梭菌dg1pspd5适应菌株的所得群体能够在含有相对高质量工业甘油的培养基中产生pdo,该工业甘油来源于novance(compiègne,法国),其中甘油浓度高达120g/l,pdo滴度高达53.5g/l,产量高达0.53g/g,以及产生率高达2.86g/l/h。
在专利申请wo2012/062832中,发明人描述了从通过与wo2010/128070中描述的相同的方法获得的丙酮丁醇梭菌dg1pspd5适应菌株群体中分离克隆“c08”。此克隆能够在包含源自novance(compiègne,法国)的相对高质量的工业甘油的培养基中产生pdo,该工业甘油甘油的甘油浓度约为105g/l。对于初始群体观察到pdo滴定度最高达50.45g/l,产量最高达0.53g/g,以及产生率最高达3.18g/l/h,而分离出的克隆c08在相同条件下显示出增加的pdo产量,pdo滴度最高达51.30g/l,产量高达0.50g/g,产生率最高达3.05g/l/h。
尽管有这些改进,但仍需要来自甘油特别是来自工业甘油的增加的pdo产量(例如产量、滴度和/或产生率)。还需要从具有较高杂质(其可抑制pdo产生)含量的工业甘油和/或从不同来源获得的工业甘油产生pdo的方法和微生物。确实,工业甘油的组成可因制造商不同而有所变化,甚至在批次之间也有所变化。此外,工业甘油越来越多地被污染,其具有增加水平的会抑制生长和/或pdo产生的被称为有机非甘油物质(mong)的杂质,包括脂肪酸(例如油酸、亚油酸)、醇、盐和金属。最后,需要具有降低水平的残留甘油的方法和微生物。确实,降低的残留甘油水平有助于下游pdo纯化。
本发明提供了一种用于改进pdo的产生,特别是从工业甘油底物的pdo的产生的方法和微生物。确实,发明人惊奇地发现hcpr和/或frdx基因的过表达进一步改善了pdo产生,因为与其中未过表达hcpr和/或frdx基因的未修饰的菌株的性能相比时,观察到更高的pdo滴度和更高的产量。另外,本发明人惊奇地发现在连续培养过程中存在较少的残留甘油。发明人还发现,过表达hcpr和/或frdx基因的菌株能够在高浓度的在工业甘油中含有的甘油(例如最高达约105g/l)的存在下从越来越不纯的工业甘油以及从来自不同来源的工业甘油以这种增加的水平产生pdo。
技术实现要素:
本发明涉及一种发酵产生pdo的方法,该方法包括在包含甘油的培养基上培养将甘油转化为pdo并且过表达一氧化氮响应性转录调节因子和/或铁氧还蛋白-3样蛋白的重组微生物。优选地,编码所述蛋白质的基因分别是hcpr和frdx基因。优选地,hcpr和/或frdx基因被过表达。
在本发明方法的一个优选实施方案中,hcpr和/或frdx基因通过遗传修饰而过表达,所述遗传修饰诸如不限于通过使调节hcpr和/或frdx基因表达的启动子突变,通过使hcpr和frdx基因之间的基因间区域突变,通过基因复制,或通过从质粒过表达hcpr和/或frdx基因。
在本发明的方法的一个优选实施方案中,通过使两个基因之间的基因间区域突变,优选通过插入,更优选通过包含至少一个核苷酸的插入,在重组微生物中过表达hcpr和/或frdx基因,其中所述至少一个核苷酸优选是“a”核苷酸。在一个特别优选的实施方案中,所述基因间区域被单个碱基(优选为“a”核苷酸)插入突变。根据一个优选的实施方案,所述插入发生在包含重复的“a”核苷酸(优选包含至少7个“a”核苷酸)的区域中。优选地,至少一个“a”核苷酸插入被并入丙酮丁醇梭菌atcc824基因组(ncbi参考序列:nc_003030.1)的位置1014234和1014240之间。
在本发明的方法的一个具体实施方案中,重组微生物适应于在高浓度甘油特别是工业甘油的存在下生长。优选地,工业甘油中的甘油浓度为90至120g/l,优选约105g/l。优选地,工业甘油包含至少5%脂肪酸。工业甘油优选是生物柴油产生的副产物。最终,pdo优选是纯化的。
在本发明的方法的另一个实施方案中,重组微生物是细菌,其优选选自梭状芽胞杆菌属或克雷伯菌属的种,更优选选自丙酮丁醇梭菌、丁酸梭菌、巴氏梭菌和肺炎克雷伯菌。
根据该方法的一个具体实施方案,将重组微生物与至少一种其他微生物在微生物聚生体中共培养,优选与梭状芽胞杆菌属的至少一种其他微生物共培养,更优选与生孢梭菌或楔形梭菌共培养,甚至更优选与生孢梭菌和楔形梭菌共培养。
本发明还涉及用于从甘油产生pdo的重组微生物,其中所述微生物将甘油转化为pdo并过表达一氧化氮响应性转录调节因子和/或铁氧还蛋白-3样蛋白。优选地,在所述重组微生物中,编码所述蛋白质的基因分别是hcpr和frdx基因。优选地,hcpr和/或frdx基因被过表达。优选地,hcpr和/或frdx基因通过遗传修饰而过表达,所述遗传修饰诸如不限于,通过使调节hcpr和/或frdx基因表达的启动子突变,通过使hcpr和/或frdx之间的基因间区域突变,通过基因复制或通过从质粒过表达hcpr和/或frdx基因。
在一个优选的实施方案中,通过使两个基因之间的基因间区域突变,优选通过插入,更优选通过包含至少一个核苷酸的插入,在重组微生物中过表达hcpr和/或frdx基因,其中所述至少一个核苷酸优选是“a”核苷酸。在一个特别优选的实施方案中,基因间区域被单个碱基插入(优选地为“a”核苷酸)突变。根据一个优选的实施方案,所述插入发生在包含重复的“a”核苷酸,优选包含至少7个“a”核苷酸的区域中。优选地,所述至少一个“a”核苷酸插入被并入丙酮丁醇梭菌atcc824基因组(ncbi参考序列:nc_003030.1)的位置1014234和1014240之间。
特别地,本发明涉及适应于在具有高浓度工业甘油的培养基上生长的重组微生物,优选其中工业甘油中的甘油浓度为90至120g/l。工业甘油可包含杂质。在一个实施方案中,工业甘油包含至少5%脂肪酸。
在另一个实施方案中,本发明的重组微生物是细菌,其优选选自梭状芽胞杆菌属或克雷伯菌属的种,更优选选自丙酮丁醇梭菌、丁酸梭菌、巴氏梭菌和肺炎克雷伯菌。在一个具体的实施方案中,将本发明的微生物与至少一种其他微生物在微生物聚生体中共培养,优选与梭状芽胞杆菌属的至少一种其他微生物共培养,更优选与生孢梭菌或楔形梭菌共培养,甚至更优选与生孢梭菌和楔形梭菌共培养。
本发明进一步涉及根据本文所述方法的重组微生物。
发明详述
在详细描述本发明之前,应当理解,本发明不限于具体示例的方法,并且当然可以改变。还应理解,本文所使用的术语仅出于描述本发明的特定实施方案的目的,而不意欲进行限制,其仅由所附权利要求来限制。
本文引用的所有出版物、专利和专利申请,无论是上文还是下文,均通过引用以其整体并入本文。然而,引用本文提及的出版物是为了描述和公开在所述出版物中报道的并且可以与本发明结合使用的方案、试剂和载体。本文中的任何内容均不应解释为承认本发明无权凭借在先发明而早于这种公开。
此外,除非另有说明,否则本发明的实施采用本领域技术范围内的常规微生物学和分子生物学技术。这样的技术对于技术人员是公知的,并且在文献中有充分的解释。参见,例如,prescott等(1999)和sambrook等(1989)(2001)。
必须注意的是,如本文和所附权利要求中所使用的,单数形式的“一个”,“一种”和“该”包括复数形式,除非上下文另外明确指出。因此,例如,对“微生物”的提及包括多种这样的微生物,而对“内源基因”的提及是对一种或多种内源基因的提及,依此类推。除非另有定义,否则本文使用的所有技术和科学术语具有与本发明所属领域的普通技术人员通常所理解的相同含义。尽管与本文所述的材料和方法相似或等同的任何材料和方法均可用于实施或测试本发明,但现在描述优选的材料和方法。如本文所使用的,以下术语可以用于解释权利要求和说明书。
在所附的权利要求和本发明的先前描述中,除非上下文由于表达语言或必要的暗示而另外需要,否则词语“包括(comprise)”或诸如“包含(comprises)”或“包含(comprising)”的变体以包括性含义使用,即,在本发明的各种实施方案中,指所陈述的特征的存在但不排除其他特征的存在或增加。
本发明涉及用于发酵产生pdo的新方法和微生物。
如本文所用,术语“微生物”是指通常可在自然界中发现的所有类型的单细胞生物,包括诸如细菌的原核生物以及诸如酵母和真菌的真核生物。在本发明的上下文中,微生物优选是细菌,更优选选自肠杆菌科(enterobacteriaceae)、芽孢杆菌科(bacillaceae)、梭状芽胞杆菌科(clostridiaceae)、链霉菌科(streptomycetaceae)和棒状杆菌科(corynebacteriaceae)。术语“埃希氏杆菌属(escherichia)”、“克雷伯菌属(klebsiella)”、“芽孢杆菌属(bacillus)”、“梭状芽胞杆菌(clostridium)”、“梭状芽胞杆菌(clostridia)”和“棒状杆菌属(corynebacterium)”是指属于这些科或属的所有细菌种。作为非限制性实例,细菌种可以选自埃希氏杆菌属种(escherichiasp.)(优选大肠杆菌)、克雷伯菌属种(优选肺炎克雷伯菌)、芽孢杆菌属种(优选枯草芽孢杆菌)、梭状芽胞杆菌属种(优选丙酸丁醇梭菌、丁酸梭菌和巴氏梭菌)以及棒状杆菌属种(优选谷氨酸棒状杆菌)。
如本文所用,术语“重组微生物”或“经遗传修饰的微生物”是指已经例如通过适应而被遗传修饰或遗传工程改造的微生物或微生物菌株。根据这些术语的通常含义,这意味着本发明的微生物在自然界中不存在,并且与该微生物衍生自其的“亲代”微生物相比被遗传修饰。“亲代”微生物可以天然存在(即野生型微生物)或可以已经先前被修饰但是不表达或过表达本发明的一种或多种蛋白质(即hcdr和/或frdx)。因此,本发明的重组微生物已被修饰以表达或过表达至少在亲代微生物中未表达或未过表达的hcdr和/或frdx蛋白。
优选地,亲代微生物选自本文列出的微生物。在一个具体的实施方案中,亲代微生物选自梭状芽胞杆菌属种丙酮丁醇梭菌、丁酸梭菌、巴氏梭菌和相关分离株,或选自克雷伯氏菌属种,诸如肺炎克雷伯菌和相关分离株。更优选地,亲代微生物选自描述于gonzalez-pajuelo等人2005或pct专利申请号wo2010/128070或wo2012/062832中的丙酮丁醇梭菌菌株。甚至更优选地,亲代微生物选自丙酮丁醇梭菌dg1pspd5菌株,诸如dg1pspd5pd0001ve05菌株。
可以对本发明的重组微生物进行多种遗传修饰。作为非限制性实例,内源基因可以在重组微生物中被减毒、缺失或过表达,而外源基因可以被导入,由质粒携带或整合到菌株的基因组中,以在细胞内表达。这样的修饰可以例如通过基因工程改造、通过适应来进行,其中在以下条件下培养微生物:对微生物施加特定压力并诱导诱变,或者在特定选择压力下通过结合定向诱变和进化通过强迫新的代谢途径的发展和进化。
在本发明的上下文中,用于发酵产生pdo的方法包括在包含甘油的培养基上培养将甘油转化为pdo并过表达一氧化氮响应性转录调节因子和/或铁氧还蛋白-3样蛋白的微生物。
本文描述的一氧化氮响应性转录调节因子,也称为hcpr,在数据库http://regprecise.lbl.gov/regprecise/中被描述为crp家族的转录调节因子。它包含camp结合域和camp依赖性蛋白激酶的调节亚基。它优选由丙酮丁醇梭菌的基因ca_c0884编码。示例性的基因和氨基酸序列分别显示在seqidno:1和2中。
本文所述的铁氧还蛋白3-样蛋白,也称为frdx,显著地包含4fe-4s铁氧还蛋白铁-硫结合域。其优选由丙酮丁醇梭菌的基因ca_c0885编码。示例性的基因和氨基酸序列分别显示在seqidno:3和4中。
上述基因的核苷酸序列或由所述基因编码的氨基酸序列,根据其在数据库中的登录号和版本和/或根据其序列标识被描述于下表1中。
表1:本发明的hcpr和frdx基因和蛋白质
本文所用的术语“过表达(overexpress)”、“过表达(overexpression)”等应广泛地理解为包括与在相同条件下未修饰的或“亲代”微生物中的表达水平蛋白相比,相应蛋白表达的任何增加。不应认为以任何特定水平表达该蛋白。如本文所用,术语“表达水平”是指在微生物中表达的感兴趣的蛋白质(或编码所述蛋白质的基因或mrna)的量(例如相对量、浓度),其可以通过本领域公知的方法测量,诸如通过qrt-pcr、蛋白质印记-免疫印记、酶联免疫吸附测定(例如elisa)、定量蛋白质组学方法等。
本领域技术人员可以容易地诱导感兴趣的蛋白质在微生物中的过表达。
作为非限制性实例,一种或多种内源基因,诸如hcpr和/或frdx基因,可以通过以下方式过表达:通过除了内源调节元件之外还引入有利于上调的异源序列、或者通过用这类异源序列替代那些内源调节元件、或通过在微生物内染色体内(即引入染色体中)或染色体外(例如引入质粒或载体内)引入一个或多个补充拷贝的内源基因。在此方面,可以通过本领域公知的方法,例如通过基因重组,将几个拷贝的基因引入染色体上。特别地,可以在丙酮丁醇梭菌中使用染色体修饰的标准技术,例如,根据专利申请wo2008/040387中描述的用于梭状芽胞杆菌属的同源重组的方法。在本发明的一个优选实施方案中,将基因的第二个拷贝引入染色体中(即基因重复)。替代性地或另外地,可通过质粒或载体将基因引入微生物中,并在染色体外表达。作为非限制性实例,它可以由不同类型的质粒携带,这些质粒的复制起点可能不同,这取决于它们可以在其中复制的微生物以及它们在细胞中的拷贝数。例如,取决于所选质粒的复制起点的性质,由质粒转化的微生物可包含1至5个拷贝的质粒,约20个拷贝,或甚至多达500个拷贝。在复制起点和细胞中拷贝数方面不同的多种质粒是本领域公知的,并且本领域技术人员可以容易地为此目的进行选择。可以在丙酮丁醇梭菌中复制的质粒的实例包括但不限于psos质粒(tummala等,1999)、psyl系列的质粒(lee,1992)和pmtl系列的质粒(chambers等,1988)等。
过表达内源基因的另一种方式是将其启动子(即野生型启动子)与更强的启动子交换。适用于此类目的的启动子可以是同源的(源自相同种)或异源的(源自不同种),并且在本领域中是公知的。确实,本领域技术人员可以容易地选择合适的启动子来诱导内源基因,诸如hcpr和/或frdx基因的表达。最方便用于增加基因表达水平的启动子是本领域技术人员公知的:这些启动子包括,尤其是,启动子ptrc、ptac、plac和λ启动子pr和pl。这些启动子可通过特定化合物或通过特定外部条件(诸如温度或光照)“诱导”,和/或可以是同源或异源的。导致基因高度过表达的丙酮丁醇梭菌启动子的具体实例包括thl、adc、ptb启动子(tummala等,1999)。
也可以通过将突变引入基因的编码序列或引入非编码序列来提高内源基因表达水平。当相应氨基酸中未发生修饰时,这些突变可以是同义的,或者当相应氨基酸被改变时,这些突变可以是不同义的。如果突变序列位于调节因子的结合位点中,则同义突变对翻译的蛋白质的功能没有任何影响,但可能对相应基因甚至其他基因的调节有影响。根据突变序列的性质,非同义突变可能会影响翻译的蛋白质的功能以及调节。
特别地,非编码序列中的突变可以位于编码序列的上游(即,在启动子区域中,在增强子、沉默子或绝缘子区域中,在特定转录因子结合位点中)或在编码序列的下游。在启动子区域中引入的突变可以在核心启动子、近端启动子或远端启动子中。可通过使用例如聚合酶链反应(pcr)的定点诱变,通过随机诱变技术(例如通过诱变剂(紫外线或化学试剂,如亚硝基胍(ntg)或甲磺酸乙酯(ems))),或dna改组或易错pcr或使用对微生物施加特定压力并诱导诱变的培养条件来引入突变。在位于基因上游的区域中插入一个或多个补充核苷酸可以显著调节基因表达。作为非限制性实例,可将一种或多种突变引入位于hcpr和/或frdx基因之间的基因间区域(包含hcpr和frdx基因的全序列以及在seqidno:15的序列中示出的亲代基因间区域,而seqidno:16的序列中仅示出了基因间区域的亲代序列)中。作为非限制性实例,“a”核苷酸的插入可以被引入基因间区域。seqidno:17的序列中显示了这种插入的一个实例。
在本发明的上下文中,重组微生物优选过表达hcpr和/或frdx基因。优选地,hcpr和/或frdx基因被过量表达至少1.5倍,更优选至少约2倍,甚至更优选至少3倍或约4倍。
根据第一个优选的实施方案,通过使两个基因之间的基因间区域突变,优选地通过插入,使hcpr和/或frdx基因过表达。优选地,所述基因间突变修饰调控hcpr和/或frdx基因表达的启动子区域,从而诱导过表达。优选地,所述基因间突变诱导hcpr和frdx基因二者的过表达。确实,hcpr和frdx基因在丙酮丁醇梭菌中作为双向基因对排布。但是,根据基因间突变的类型,hcpr和frdx基因中只有一个可以被过表达。优选地,所述基因间突变在丙酮丁醇梭菌atcc824基因组的位置1014117和1014239之间。
优选地,通过插入选自“a”、“c”、“t”和“g”核苷酸中的至少一个核苷酸来使基因间区域突变。优选地,通过插入至少一个“a”核苷酸,更优选为一个“a”核苷酸,来使基因间区域突变。优选地,将至少一个核苷酸插入区域中,其中相同核苷酸被重复至少两次、三次、四次、五次、六次、七次或更多次(例如,将“a”核苷酸插入包含“aaaaaa”的核苷酸延伸中)。当所述插入物是“a”核苷酸时,所述插入物可以进一步位于丙酮丁醇梭菌atcc824基因组的位置1014240处。优选地,所述至少一个“a”被掺入丙酮丁醇梭菌atcc824基因组的位置1014234和1014240之间。
根据第二个优选的实施方案,通过将所述基因引入微生物中外源载体或质粒上,更优选在诱导型启动子的控制下,过表达hcpr和/或frdx基因。
根据第三个优选的实施方案,通过在染色体中引入所述基因的至少一个另外的拷贝(即基因重复)来过表达hcpr和/或frdx基因。
鉴于本文提供的基因和氨基酸序列,并使用数据库(诸如uniprot(用于蛋白质)、genbank(用于基因)或ncbi(用于蛋白质或基因))中可用的信息,技术人员可以容易地确定微生物的特定蛋白质和/或基因的序列,并鉴定其他微生物中的等同蛋白质或基因或其同源物。例如,可以通过将微生物的特定基因(或蛋白质)序列与可在上述数据库中发现的其他微生物的基因(或蛋白质)序列或基因组(或蛋白质组)进行比对来执行此常规工作。使用altschul等人(1990)开发的blast算法可以有利地进行这种序列比对。一旦在序列之间建立了序列同源性,就可以导出共有序列并用于设计简并探针,以克隆相关微生物的相应同源基因(并因此克隆同源蛋白)。这些常规的分子生物学方法是本领域技术人员公知的。
应当进一步理解,在本发明的上下文中,如果编码感兴趣的蛋白质的外源基因在特定微生物中表达,则此基因的合成形式优选用所述微生物的编码相同氨基酸的优选密码子替换非优选密码子或次优选密码子来构建。在本领域中确实公知的是,密码子的使用在微生物种之间变化,这可能影响感兴趣的蛋白质的重组表达水平。为了克服此问题,已经开发了密码子优化方法,并且graf等人(2000)、deml等人(2001)和davis&olsen(2011)对此进行了广泛的描述。值得注意的是,已经开发出了几种用于密码子优化确定的软件程序,例如
在本发明的方法和微生物的上下文中,重组微生物可包含关于亲代菌株的额外修饰。作为非限制性实例,重组微生物可能先前已经适应于从包含甘油的培养基中增加pdo产生。将甘油代谢导向为pdo产生的方法是已知的(参见例如wo2006/128381、gonzález-pajuelo等,2006)。
在一个优选的实施方案中,如果重组微生物是丙酮丁醇梭菌,则其先前优选地已经被适应,更优选地通过厌氧连续过程,用于从具有高甘油含量的培养基中生长和产生pdo,呈现出增加的pdo产生的通量。优选通过厌氧连续过程进行对丙酮丁醇梭菌菌株的适应,这是本领域技术人员公知的技术。在本领域技术人员已知的此方法的细节中,例如可以提到的是,将补料培养基连续添加到发酵罐中,并且同时从系统中除去等量的具有微生物的转化营养液。营养物交换的速率表示为稀释率。因此,将稀释率应用于培养基,稀释率考虑微生物的最大生长速率并影响培养基的摄取和排出速率。
丙酮丁醇梭菌菌株可以通过引入来自丁酸梭菌的额外拷贝的pdo操纵子来进行适应,所述pdo操纵子编码参与维生素b12非依赖性pdo途径的酶。特别地,来自丁酸梭菌的pdo操纵子可以通过质粒过表达或被整合到待适应的丙酮丁醇梭菌菌株的染色体中。例如,pspd5质粒可用于丙酮丁醇梭菌中pdo操纵子的过表达(gonzalez-pajuelo等,2006)。
可替代地或另外地,所述重组微生物可以通过wo2010/128070中公开的选择压力培养方法(具体参见第7页第10行至第8页第23行和实施例2、3和4)进行适应以在具有高甘油含量,特别是高浓度的源自工业甘油的甘油的培养基中生长。
为了从作为唯一碳源的甘油产生pdo而进行了遗传修饰的丙酮丁醇梭菌菌株是本领域已知的,并且特别是在申请wo2001/04324和wo2010/128070中公开。最后,丙酮丁醇梭菌菌株可以通过wo2012/062832中描述的方法预先进行适应。
作为非限制性实例,可以使用含有90至120g/l甘油,优选约105g/l粗甘油的进料培养基,在连续培养中培养待适应的丙酮丁醇梭菌。通过在包含高工业甘油的培养基上以低稀释率培养微生物,并选择能够在具有高浓度的源于工业甘油的甘油的培养基上生长的经适应的微生物,来实现对产生性微生物的“适应”。0.005和0.02h-1之间的稀释率对应于“低稀释率”,而0.02和0.1h-1之间的稀释率对应于普通稀释率。作为非限制性实例,可以在24小时至10天,优选超过2天,更优选约8天的时间内以低稀释率培养待适应的丙酮丁醇梭菌菌株。稀释率通常在0.005至0.1h-1之间,优选在0.005至0.02h-1之间。稀释率可以在适应方法期间改变,最终第一步在0.005至0.02h-1之间进行,第二步将稀释率提高到高达0.1h-1,更优选高达0.06h-1,甚至更优选高达0.07h-1。
在本发明的一个具体实施方案中,使用含有90至120g/l甘油,优选约105g/l粗甘油的进料培养基,以介于0.005和0.02h-1,优选0.02h-1的低稀释率,在连续培养中培养待适应的丙酮丁醇梭菌菌株。在长达10天的时间内,优选在5到8天之间的时间内,丙酮丁醇梭菌菌株适应于进料培养基中存在的高甘油浓度,并且稀释率可以提高至0.1h-1,优选多达0.07h-1。
“培养基”是指适于重组微生物的生长和二醇产生的培养基,其中所述培养基包含甘油,甘油可以是或可以不是培养基中存在的唯一碳源。培养基可以包含或可以不包含有机氮源。氮是天然存在的元素,对于植物和动物的生长和繁殖至关重要。它存在于氨基酸以及许多其他有机和无机化合物中。根据本发明,“有机氮”是指获自活生物体的含氮有机化合物。用于细菌培养的有机氮的常用来源包括酵母提取物。
甘油可以是培养基中存在的唯一碳源。可替代地,培养基中可以存在一种或多种额外碳源(例如葡萄糖)。培养基中存在的甘油可以是纯甘油或工业甘油。
优选地,培养基不包含酵母提取物。甚至更优选地,培养基不包含任何有机氮源。优选地,甘油是存在于培养基中的唯一碳源。
术语“甘油(glycerol)”在本文中可以与术语“甘油(glycerine)”互换使用,并且可以包括纯和/或工业甘油。工业甘油可以作为来自生物柴油产生的生物产物获得(例如,通过甘油酯与短链醇的酯交换反应)。可替代地,可例如通过皂化,酯交换和/或水解反应从植物或动物油和脂肪中获得工业甘油。
“纯甘油”是指纯度大于按重量计95%的甘油的甘油产物。在一些情况下,纯甘油的纯度可大于97%,甚至大于99%或99.2%。甘油产物的其余部分优选由水(例如按重量计约3至5%)组成。纯甘油几乎没有残留杂质。特别地,纯甘油可以通过使用一种或多种方法诸如蒸馏来纯化而获得。
“工业甘油”是指未经实质性纯化而从工业过程中获得的甘油产物。工业甘油(industrialglycerine)也可以称为“粗甘油(rawglycerine)”、“粗甘油(rawglycerol)”、“粗制甘油(crudeglycerol)”或“工业甘油(industrialglycerol)”。工业甘油通常包含按重量计5-95%的甘油,特别是按重量计40-90%的甘油,更特别地为按重量计60-90%的甘油,甚至更特别地为约按重量计65-85%的甘油,或约按重量计70-85%的甘油。工业甘油的其余级分可包含也称为水分的水、矿物质盐和有机非甘油物质(mong),其包含其他非甘油有机化合物和甘油酯。特别地,工业甘油还包含杂质,例如醇(例如甲醇)、无机盐、未反应的单、二和三酰基甘油、甲酯和一种或多种脂肪酸(例如棕榈酸、硬脂酸、油酸、亚麻酸、亚油酸和花生酸)。
根据一个优选的实施方案,工业甘油优选包含按重量计大于约60%、70%或80%的甘油和按重量计小于约15%的水和杂质。根据一个优选的实施方案,矿物盐的浓度小于10%,优选小于5%。根据一个优选的实施方案,mong中包含的脂肪酸的浓度在分析证明中小于20%,优选小于10%,甚至更优选小于5%。根据另一优选的实施方案,脂肪酸或mong的浓度优选为至少5%,更优选为至少10%,甚至更优选为5至10%。
从中获得作为副产物的工业甘油的工业过程尤其是将脂肪和油(特别是植物或动物来源的脂肪和油)加工成工业产品(例如洗涤剂或润滑剂)的制造方法。在此类制造方法中,工业甘油被认为是副产物。工业甘油的确切组成将取决于初始甘油来源(例如动物脂肪或植物油,诸如葵花籽油、菜籽油、大豆油,芥菜籽等),以及甘油提取和下游处理中使用的方法和条件,例如在生物柴油产生中。
在一个特定的实施方案中,工业甘油是来自生物柴油的副产物。优选地,工业甘油包含从生物柴油产生获得的甘油的已知杂质,其包含约80-85%的甘油与盐、水和一些其他有机化合物(即,mong),其包含诸如脂肪酸的化合物,诸如以上列出的那些。从生物柴油产生中获得的工业甘油尚未经过进一步的纯化步骤。
如本文所用,术语“高甘油含量”或“高甘油浓度”是指培养基中的甘油浓度等于或高于90g/l的甘油。在优选的实施方案中,培养基包含浓度为90至120g/l,优选为105至110g/l,更优选为约105g/l或109g/l的甘油。
在一些情况下,可以通过在所谓的微生物聚生体中在至少一种其他微生物的存在下共培养重组微生物来进一步增加pdo的产生和/或进一步降低残留甘油的水平。术语“微生物聚生体”或“共培养”可互换使用,以表示在发酵程中使用两种或多种微生物种。优选地,用于共培养的一种或多种另外的菌株不发酵甘油或不产生pdo。
作为非限制性实例,微生物聚生体可包含至少两种梭状芽孢杆菌菌株,例如一种丙酮丁醇梭菌菌株和选自梭状芽孢杆菌属的菌株(诸如生孢梭菌菌株和楔形梭菌菌株)的至少一种菌株。作为进一步的实例,微生物聚生体可包含至少三种梭状芽孢杆菌菌株,诸如至少一种丙酮丁醇梭菌菌株,至少一种生孢梭菌和至少一种楔形梭菌菌株。在上述情况的任一者下,大多数微生物聚生体可属于丙酮丁醇梭菌种。例如,考虑到培养物中含有的细胞的总数对应于100%,微生物聚生体可包含超过85%的丙酮丁醇梭菌、0.001%至0.2%的生孢梭菌和/或0.1%至15%的楔形梭菌。特别地,微生物聚生体包含85%-99.8%的丙酮丁醇梭菌、0.001%-0.15%的生孢梭菌和/或0.2%-15%的楔形梭菌,或,90%-99.8%的丙酮丁醇梭菌、0.002%-0.13%的生孢梭菌和/或0.2%-10%的楔形梭菌。
因此,在一个优选的实施方案中,本发明的微生物在微生物聚生体中共培养。所述微生物聚生体优选包含与至少一种,优选两种其他微生物共培养的本文公开的重组微生物,或如本文公开的本发明的方法中所述的重组微生物。根据一个特别优选的实施方案,将重组微生物与至少一种楔形梭菌菌株,更优选与至少一种楔形梭菌菌株和至少一种生孢梭菌共培养。优选地,本发明的重组微生物是丙酮丁醇梭菌菌株,甚至更优选地经适应以从具有高甘油含量且特别是具有工业甘油中含有的高浓度甘油的培养基生长和产生pdo。
在一个优选的实施方案中,考虑到培养物中含有的细胞的总数对应于100%,本发明的微生物聚生体包含超过85%的丙酮丁醇梭菌、0.001%至0.2%的生孢梭菌和/或0.1%至15%的楔形梭菌。在一个更优选的实施方案中,微生物聚生体包含85%至99.8%的丙酮丁醇梭菌,0.001%至0.15%的生孢梭菌和/或0.2%至15%的楔形梭菌。在一个甚至更优选的实施方案中,微生物聚生体包含90%至99.8%的丙酮丁醇梭菌、0.002%至0.13%的生孢梭菌和/或0.2%至10%的楔形梭菌。
在本发明的方法中,pdo的产生优选通过在包含甘油作为唯一碳源的甘油的培养基中培养上文所述的本发明的重组微生物或微生物聚生体通过厌氧连续发酵来进行,所述培养基是不添加有机氮的基本培养基。
术语“基本培养基”是指包含除了甘油溶液之外的化学成分确定的组成的生物体在其上生长的严格矿物质培养基。这种培养基在本领域中公开,特别是在wo2010/128070和wo2011/042434中,其内容通过引用以其整体并入本文。
在一个优选的实施方案中,根据本发明的方法获得的pdo被进一步纯化。从发酵培养基中回收并最终纯化pdo的方法是技术人员已知的。pdo可以通过蒸馏分离。在大多数实施方案中,pdo从具有副产物如乙酸盐的发酵培养基中蒸馏出来,然后通过已知方法被进一步纯化。特别优选的纯化方法公开于申请wo2009/068110和wo2010/037843中,其内容通过引用以其整体并入本文。
连续发酵过程是本领域技术人员已知的。发酵过程通常是在反应器中进行的,该反应器具有已知确定组成的无机培养基,该无机培养基适应于所使用的细菌,至少含有工业甘油、来自生物柴油产生的副产物,如有必要,还含有用于代谢产物产生的共底物。
本发明的该方法优选地以连续过程实现。本领域技术人员能够管理这些实验条件中的每一个,并根据他的常识来确定根据本发明的微生物的培养条件。特别地,梭状芽胞杆菌在20℃至60℃之间的温度下发酵,对于丙酮丁醇梭菌,优选在25℃至40℃之间的温度下发酵。
在本发明的一个具体实施方案中,使用含有约105g/l或109g/l粗甘油的进料培养基在连续培养中培养丙酮丁醇梭菌菌株dg1pspd5130p型或008p型,稀释率为0.035至0.08h-1,优选为0.07h-1(参见实施例1和2)。所述方法在其不同的实施方案中导致产生至少52g/l的pdo,对于0.7h-1的稀释率,产量为0.4至0.6g/g,产生率大于2.9g/l/h。优选地,产量在0.4至0.5g/g之间,并且产生率大于3.6g/l/h,甚至更优选地大于3.65g/l/h。在某些实施方案中,本发明的方法进一步导致残留甘油水平小于3.7g/l。优选地,残余甘油水平为约3.6g/l。
附图说明
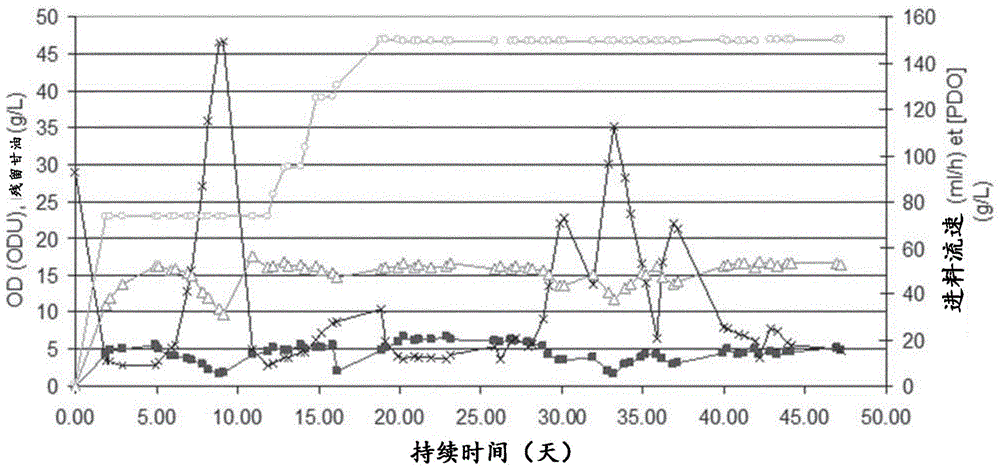
图1:通过使008p型菌株在粗甘油上进行适应,获得丙酮丁醇梭菌dg1pspd5130p型菌株。连续培养物的od620nm(od单位或odu,正方形)、残留甘油(g/l;交叉线)、pdo浓度(g/l;三角形)和进料流速(ml/h;圆圈)的动态显示为培养持续时间的函数(天;d)。
图2:丙酮丁醇梭菌中hcpr和frdx基因的染色体组织。基因hcpr和frdx被组织为双向基因对,两个基因之间有一个123bp的基因间区域。示出了丙酮丁醇梭菌atcc824基因组中的核苷酸位置(ncbi参考序列:nc_003030.1)。
图3:通过定量pcr的hcpr和frdx基因的过表达。通过qrt-pcr测定,与亲代菌株008p比较,hcpr和frdx二者在130p菌株中均过表达。黑条:008p型;灰条:130p型。
实施例
在以下实施例中进一步定义本发明。应该理解的是,这些实施例虽然指示了本发明的优选的实施方案,但是仅以示例的方式给出。从以上公开内容和这些实施例,本领域技术人员可以对本发明进行各种改变,以使其适应各种用途和条件,而无需修改本发明的本质特征。
实施例1:在工业粗甘油上连续培养丙酮丁醇梭菌dg1(pspd5)和获得130p型微生物
细菌菌株:
-008p型:如专利申请wo2010/128070中所述的在高浓度的粗甘油上适应的丙酮丁醇梭菌菌株dg1pspd5
-130p型:如本文所述的,由连续培养形式的丙酮丁醇梭菌dg1pspd5008p型在高浓度的粗工业甘油上连续培养得到的并且过表达hcpr和frdx基因的丙酮丁醇梭菌dg1pspd5型菌株
用于梭状芽胞杆菌分批培养的合成培养基含有,每升自来水:甘油,30g;kh2po4,0.5g;k2hpo4,0.5g;mgso4,7h2o,0.2g;cocl26h2o,0.01g;h2so4,0.1ml;nh4cl,1.5g;生物素,0.16mg;p-氨基苯甲酸,32mg;feso4,7h2o,0.028g。用nh4oh3n将培养基的ph调节至6.3。从sdscarlo_erba购买的商品甘油(纯度为99%)用于分批培养。用于连续培养的进料培养基含有,每升自来水:来自粗甘油的甘油,105g;kh2po4,0.50g;k2hpo4,0.50g;mgso4,7h2o,0.2g;nh4cl,1.5g;cocl26h2o,0.026g;生物素,0.16mg;p-氨基苯甲酸,32mg;feso4,7h2o,0.04g;消泡剂,0,05ml;znso4,7h2o,8mg;cucl2,2h2o,4mg;mnso4,h2o,0.04g;h3bo3,2mg;na2moo4,2h2o,0.8mg。用h2so496%将培养基ph调节至3.5-4。
来自生物柴油的酯交换过程的粗甘油由两个不同的提供者提供,并具有以下组成:
-来自adm(瑞士罗勒)(使用植物油;纯度80.9%;水分12.6%;mong0.39%;灰分6.2%),
-来自greenergy(英国伦敦)(使用食用油;纯度76.5%;水分10.2%;mong7.1%;灰分6.3%)。
任选地,这些甘油通过酸化进行预处理。
纯度和mong组成与甘油对微生物的毒性有关。由于mong浓度高,与英国adm(瑞士罗勒)甘油相比,greenergy(英国伦敦)甘油不但纯度低而且较脏,因此对微生物的毒性更大。确实,greenergy(英国伦敦)甘油中的mong浓度高于5%。
以下实施例显示了使008p型菌株在粗甘油(从adm到greenergy)上进行适应以获得名为130p型的新菌株,该菌株能够在较不精制的工业甘油上生长和产生pdo。此适应是非常有利的,因为较不精制的工业甘油是用于发酵过程的较便宜的原料。
实验装置:
在5ltryton生物反应器(法国pierreguerin)中进行连续培养,工作体积为2000ml。通过自动调节培养物的水平,将培养物的体积保持恒定在2000ml。以200rpm搅拌培养物,将温度设置为35℃,并通过自动添加5.5nnh4oh将ph保持恒定在6.5。为了产生厌氧条件,将容器中的灭菌的培养基在60℃下用无菌的不含o2的氮气冲洗1小时,然后再次冲洗直到达到35℃(在2小时内冲洗)。通过邻苯三酚装置保护生物反应器气体出口免受氧气的侵害(vasconcelos等,1994)。灭菌后,还用无菌的不含o2的氮气冲洗进料培养基,直至达到室温,并保持在氮气下以避免o2进入。
分析程序:
用比浊法在620nm(od620nm)处测量细胞浓度,并将其与直接测定的细胞干重相关联。通过hplc分析测定甘油、pdo、乙醇、乳酸盐、乙酸和丁酸的浓度。在bioradaminexhpx-87h柱上进行分离,并通过折射率进行检测。操作条件如下:流动相硫酸0.5mm;流速0.5ml/min,温度,25℃。
分批和连续培养过程和结果:
在100ml烧瓶中在合成培养基(与上述用于分批培养的相同,但添加乙酸2.2g/l和mops23.03g/l)上生长的在指数生长期结束时获取的培养物用作接种物(5%v/v)。
首先以分批模式培养培养物。在早期指数生长期,我们用进料培养基(与所述用于进料培养的相同)对甘油进行了脉冲处理。在3小时期间以静态流速添加来自粗甘油的甘油(即添加18g/l的甘油)。然后,以分批模式继续生长,并且在指数生长期结束之前,以0.035h-1的进料培养基的稀释率开始连续进料,该进料培养基含有105g/l的来自仅由adm(瑞士罗勒)提供的粗甘油的甘油。如图1所示,在以0.035h-1的稀释率情况下3天后,甘油开始积累,并在6.5停留时间(rt,根据以下所示公式计算)达到46.6g/l,对应于第一个残留甘油峰。此积累与pdo产量的降低(最高达31g/l,而非52g/l)和生物质产量的降低(1.8odu,而非5.6odu)有关。此积累后是快速再次消耗,在d=0.035h-1的稀释率下在9rt后残留甘油降至2.9g/l。此时(接种后12天),稀释率在5天内从0.035h-1增加到0.070h-1。在d=0.07h-1的稀释率下在9rt后,性能稳定在5.5±1.1g/l的甘油和51.6±0.7g/l的pdo。
稳定后(接种后28天(参见图1)),将进料中的粗甘油改为由adm(50%;瑞士罗勒)和由greenergy(50%;英国伦敦))提供的粗甘油的混合物,从而增加mong的水平以及甘油对微生物的毒性。
进料组成的这种变化导致了13天期间的甘油积累(最大35.1g/l)循环和pdo产量下降(最小37.4g/l)。通过关键因素(od,残留甘油和pdo浓度)监控培养物的稳定性,并在第45天鉴定出新的经适应的菌株130p以保藏(见图1)。
在此步骤中,对新菌株进行了测序,并与008p的序列进行了比较。我们鉴定了以下实施例3中所述的基因间突变。
下表2中显示了所得的130p型菌株的性能。
根据稀释率计算停留时间的公式
rt:停留时间(h)
dr:稀释率(h-1)
表2:丙酮丁醇梭菌dg1pspd5菌株130p型的性能。进料培养基中含有105g/l的来自由adm(瑞士罗勒)和greenergy(英国伦敦)提供的粗甘油的甘油,稀释率为0.070h-1。
ypdo:pdo产量(g/g使用的甘油)
qpdo:pdo体积产生率
实施例2:通过用高浓度的粗甘油连续培养在恒化器中的丙酮丁醇梭菌dg1pspd5菌株008p和130p型的pdo产生性能
用于梭状芽胞杆菌分批培养的合成培养基含有,每升自来水:甘油,30g;kh2po4,0.5g;k2hpo4,0.5g;mgso4,7h2o,0.2g;cocl26h2o,0.01g;h2so4,0.1ml;nh4cl,1.5g;生物素,0.16mg;对氨基苯甲酸,32mg以及feso4·7h2o,0.028g。用nh4oh3n将培养基的ph调节至6.3。从sdscarlo_erba购买的商品甘油(纯度为99%)用于分批培养。用于连续培养的进料培养基含有,每升自来水:来自粗甘油的甘油,105g;kh2po4,0.50g;k2hpo4,0.50g;mgso4·7h2o,0.2g;nh4cl,0-1.5g;cocl2·6h2o,0.013-0.026g,生物素,0.08-0.16mg;对氨基苯甲酸,16-32mg;feso4·7h2o,0.04g;消泡剂,0,05ml;znso4·7h2o,8mg;cucl2·2h2o,4mg;mnso4·h2o,0.02g-0.04g;h3bo30-2mg;na2moo4·2h2o,0-0.8mg。用h2so496%将培养基ph调节至3.5-4。
来自生物柴油的酯交换过程的粗甘油获自若干个不同的来源,并具有以下组成:
-novance(compiègne,法国)(使用植物油;纯度82-85%;水分8-13%;mong0.1-0.3%;灰分1.4%)
-adm(瑞士罗勒)(使用植物油;纯度80.9%;水分12.6%;mong0.39%;灰分6.2%),与greenergy甘油混合使用
-greenergy(英国伦敦)(使用食用油;纯度76.5%;水分10.2%;mong7.1%;灰分6.3%),与adm甘油混合使用
任选地,这些甘油通过酸化预处理。
如以上实施例1中所说明的,纯度和mong组成与甘油对微生物的毒性有关。
实验装置如以上实施例1中所述。
分批和连续培养过程:
在100ml烧瓶中在合成培养基(与上述用于分批培养的相同,但添加乙酸2.2g/l和mops23.03g/l)上生长的在指数生长期结束时获得的培养物用作接种物(5%v/v)。
首先以分批模式培养培养物。在早期指数生长期,我们用进料培养基(与所述用于进料培养的相同)对甘油进行了脉冲处理。在3小时期间以静态流速添加来自粗甘油的甘油(即添加18g/l的甘油)。然后,以分批模式继续生长,并且在指数生长期结束之前,以0.035h-1的稀释率开始连续进料。接种生物反应器后五至八天,在5天内稀释率从0.035h-1增加至0.070h-1。此后,培养物稳定,随后使用实施例1中所述的hplc方案在分析程序中分析pdo生产量和甘油消耗。
表3:连续培养中丙酮丁醇梭菌008p型和130p型菌株的性能。
进料培养基含有105g/l的来自粗甘油的甘油,稀释率为0.070h-1。来自8个和17个恒化器的平均数据。每种菌株均标明了培养物中所用甘油的提供者。novance(compiègne,法国)对应的是相对清洁和纯的甘油,而adm(瑞士罗勒)和greenergy(英国伦敦)提供的甘油纯度较差,污染物较多,因此对微生物的毒性更大。
ypdo:pdo产量(g/g使用的甘油)
qpdo:pdo体积产生率
*当使用不同类型的甘油(即novance或adm/greenergy混合物)时,菌株130p型的性能没有明显变化
这些结果表明,带有诱导hcpr和frdx基因过表达的基因间突变(在这种情况下,根据丙酮丁醇梭菌atcc824在染色体上在位置1014234至1014240的核苷酸之间)的130p型菌株令人惊讶地表现出与亲代菌株008p相比pdo产生更好,滴度和产量更高,甘油残留量更少。
这些结果还证明了130p型菌株的优势,130p型菌株生长,且与未携带基因修饰的亲代菌株008p型相比,产生更多pdo(表3)。确实,在使用比008型菌株常用的甘油更大毒性的工业甘油的培养条件下,当与008p相比,130p的所有关键工业参数都得到了改善(较高的pdo滴度和产量,以及更少的残留甘油)。
因此,在hcpr和frdx基因过表达后,丙酮丁醇梭菌dg1pspd5菌株产生更多的pdo,并且更有力,因此更适合工业过程。
实施例3:基因间突变描述
出乎意料的是,在ca_c0884和ca_c0885基因之间的基因间区域(如图2所示,seqidno:15所示)中的单核苷酸插入具有改善pdo的产生和对甘油中存在的杂质mong的抗性。
通过与丙酮丁醇梭菌dg1pspd5130p型的dna相比,使用寡核苷酸基因间区正向引物(seqidno:18)和基因间区反向引物(seqidno:19)在菌株丙酮丁醇梭菌dg1pspd5008p型的dna上扩增的pcr片段的核酸测序,检测核苷酸插入。与亲代型序列(seqidno:16)相比,在seqidno:17中提到的重复“a”核苷酸的区域中,在ca_c0884和ca_c0885基因之间的基因间区域中识别出“a”插入突变。基因ca_c0884hcpr(seqidno:1)编码一氧化氮响应性转录调节因子(seqidno:2),基因ca_c0885frdx(seqidno:3)编码铁氧还蛋白3-样蛋白(seqidno:4)。
实施例4:有或没有基因间核苷酸插入的ca_c0884和ca_c0885基因表达
rna分离
从2ml烧瓶培养物中提取rna,转移到6ml苯酚(5%)/乙醇(95%)的混合物中,并在4℃下于3000g离心5分钟。将片状沉淀在100μl溶菌酶100mg/ml中均质化,在37℃下孵育30分钟,并在maxwellrsc仪器(promega)中使用maxwellrscsimplyrnatissue试剂盒(promega)提取rna。
通过定量逆转录pcr(qrt-pcr)定量特定的核糖核酸序列
由于rna不能用作pcr模板,所以通过qrt-pcr进行基因表达谱分析的第一步是将rna模板逆转录为cdna,然后在pcr反应中进行指数扩增。
用0.2μg总rna进行反转录,并在存在随机引物和oligodt引物的情况下,使用superscriptviloiv(invitrogen)将其反转录为cdna。
逆转录酶反应在总体积20μl中进行。反应完成后,将混合物保持在85℃。
使用ssoadvanceduniversalsybrgreensupermix(bio-radmitrymory,法国)通过定量pcr测定样品中的相对定量。在配备有cfx96tm实时系统(bio-rad)的bio-radc1000tm热循环仪上进行定量pcr。
pcr反应混合物由1×ssoadvanceduniversalsybrgreensupermix(bio-rad)、6μl正向(f)和反向(r)引物(1μm)的混合物、2μl稀释样品和无核酸酶的水组成,达到20μl的最终体积。根据以下热循环程序进行扩增:在98℃初始熔融2分钟(1个循环),然后在98℃熔融40个循环10秒,引物退火,并在60℃延伸30秒。(将熔融曲线从65到95℃,每5秒增加0.5℃)。对于每个实验,使用cfxmanagertm3.1软件(bio-rad)在qpcr反应的指数阶段设置阈值水平(ct)。
通过定量反转录pcr(qrt-pcr)确定每个基因的表达水平。所使用的基于ca_c0884基因的引物是基于ca_c0884基因的正向引物(seqidno:20)和基于ca_c0884基因的反向引物(seqidno:21),并且所使用的基于ca_c0885基因的引物是基于ca_c0885基因的正向引物(seqidno:22)和基于ca_c0885基因的反向引物(seqidno:23)。使用比较阈值循环(ct)方法对每个样品确定每个靶基因相对于管家基因dna促旋酶亚基a(gyra:ca_c1628,使用的引物是基于ca_c1628基因的正向引物(seqidno:24)和基于ca_c1628基因的反向引物(seqidno:25))的量,并使用已知浓度的atcc824基因组dna的连续稀释液作为每个实验的校正剂。为了使用比较ct方法,使用atcc824基因组dna模板的连续稀释液确认引物的效率大致相同。
与携带亲代型基因间区域的008p型菌株相比,携带基因间突变的130p型菌株中的基因ca_c0884和ca_c0885的相对表达水平明显更高(图3)。
这些数据证明,在产生pdo的重组丙酮丁醇梭菌dg1pspd5菌株的ca_c0884和ca_c0885之间的基因间区域中存在的核苷酸插入允许两个所述基因hcpr和frdx的过表达。在存在此突变的情况下,pdo的产生性能特征得到改善,并且该菌株对工业甘油中存在的脏的、高含量的mong化合物具有更强的抵抗力。
参考文献
-altschuls,gishw,millerw,myerse,lipmandj(1990).j.mol.biol;215(3):403–410.
-chambersetal.(1988).gene;68(1):139-49.
-davisjj&olsengj.(2011).mol.biol.evol.;28(1):211-221.
-demll,bojaka,stecks,grafm,wildj,schirmbeckr,wolfh,wagnerr.(2011).
-gonzález-pajuelom,meynial-sallesi,mendesf,andradejc,vasconcelosi,andsoucaillep.2005.metabolicengineering,7:329–336.
-gonzález-pajuelom,meynial-sallesi,mendesf,soucaillep.andvasconcelosi.(2006).appliedandenvironmentalmicrobiology,72:96-101.
-grafm,bojaka,demll,bielerk,wolfh,wagnerr.(2000).j.virol.;74(22):10/22-10826.
-lees,bennettg,papoutsakise,(1992).biotechnologyletters,14(5):427-432.
-papanikolaous,ruiz-sanchezp,parisetb,blanchardfandfickm.(2000),journalofbiotechnology,77:191-2008.
-tummalaetal.(1999).appl.environ.microbiol.,65(9):3793–3799.
-sambrookandrussell,(2001),molecularcloning:3rdedition,coldspringharbor
-vasconcelosi,girball,soucaillep.,(1994),journalofbacteriology,176(5):1443-1450.
序列表
<110>metabolicexplorer
<120>通过在高甘油含量的培养基上发酵来改善1,3-丙二醇产生的微生物和方法
<130>b374950pctd37437
<150>ep17306044.3
<151>2017-08-04
<160>25
<170>patentin版本3.5
<210>1
<211>690
<212>dna
<213>丙酮丁醇梭菌
<220>
<221>misc_特征
<223>ca_c0884基因序列
<400>1
atggataaaaaaattttaaccgtattaaaacattgtatcttattttcaaaaattgaaact60
caggaaattgatcatttattttcatctataaattactcaataaaagaatactataaagat120
gaaactatcgcaattgaaggcgatacgtgcaataaaattggaatagtattaagcggttgt180
gttgaaatacagaaaatttacgaatctggtaaaagtcttacaataacaacactagaagaa240
agcaagatatttggtgaagcaataatattctccaataaaacaagctatccttctacaata300
atagcatgtactaaaagtactataatttttattcccaaagcctcaatatctaaattatgc360
agcgacaattcccttttccttaatggctttatgtctcttctatctaataagatattgatg420
ttgaataaaaaattaaaaaatatgtcttatcatactataagagagaaaatatcaaactat480
atacttgaacagtacgaagctcaaaataacttaacttttaaaatgaataagtcaaaaaaa540
cagttgtctgaaatgcttggaattccaaggccatcattatctagagaattcataaaccta600
cgtgaagaaggaattatagactttgatagaacatctattacaatattagatattaactct660
ttaaaagaaattttagaagctgcagaataa690
<210>2
<211>229
<212>prt
<213>丙酮丁醇梭菌
<220>
<221>misc_特征
<223>ca_c0884蛋白序列:np_347520.1
<400>2
metasplyslysileleuthrvalleulyshiscysileleupheser
151015
lysilegluthrglngluileasphisleupheserserileasntyr
202530
serilelysglutyrtyrlysaspgluthrilealailegluglyasp
354045
thrcysasnlysileglyilevalleuserglycysvalgluilegln
505560
lysiletyrgluserglylysserleuthrilethrthrleugluglu
65707580
serlysilepheglyglualaileilepheserasnlysthrsertyr
859095
proserthrileilealacysthrlysserthrileilepheilepro
100105110
lysalaserileserlysleucysseraspasnserleupheleuasn
115120125
glyphemetserleuleuserasnlysileleumetleuasnlyslys
130135140
leulysasnmetsertyrhisthrileargglulysileserasntyr
145150155160
ileleugluglntyrglualaglnasnasnleuthrphelysmetasn
165170175
lysserlyslysglnleuserglumetleuglyileproargproser
180185190
leuserargglupheileasnleuargglugluglyileileaspphe
195200205
aspargthrserilethrileleuaspileasnserleulysgluile
210215220
leuglualaalaglu
225
<210>3
<211>711
<212>dna
<213>丙酮丁醇梭菌
<220>
<221>misc_特征
<223>ca_c0885基因序列
<400>3
atgaaaagaaaaatagtaaacatagataaagataaatgtaatggatgtggactttgtagt60
gaagcatgtcatgaaaatgcaattgaaataattaatggaaaagcagagcttttatctgat120
gaatattgtgatggtttaggagattgtttacctcattgtccagttgatgcaataactata180
atagagagagaaagtaaggaatatgatgaagaggcagttcagagaagaattgaagaaaag240
aaaaaatcaaagttagctaaaccctgtggatgtccaggagctatggctaaaaaaatagaa300
agagtagctaagcctttagctaaagtaaaggaagataggtcttctgtttcagagttaatg360
cagtggccagttcagttaaggcttgtaagtccaggagctccatattttaaaaatgctaat420
cttttagtagctgcagattgtacagcctatgcttatggtgactttcacaatgattttata480
aagaatcatataacagtaataggatgtccaaaacttgatgatgttacatattacaaagat540
aagttgaaagaaattatagaacttaatgaccttaagagtataacagttgttagaatggag600
gtaccttgctgttcaggcatagtttcagcagtaaagactgctatgcttgaagcaaaagtt660
atagtaccttttagagaagttattataggaactaatggtgaaattagataa711
<210>4
<211>236
<212>prt
<213>丙酮丁醇梭菌
<220>
<221>misc_特征
<223>ca_c0885蛋白序列:np_347521.1
<400>4
metlysarglysilevalasnileasplysasplyscysasnglycys
151015
glyleucysserglualacyshisgluasnalailegluileileasn
202530
glylysalagluleuleuseraspglutyrcysaspglyleuglyasp
354045
cysleuprohiscysprovalaspalailethrileilegluargglu
505560
serlysglutyraspgluglualavalglnargargilegluglulys
65707580
lyslysserlysleualalysprocysglycysproglyalametala
859095
lyslysilegluargvalalalysproleualalysvallysgluasp
100105110
argserservalsergluleumetglntrpprovalglnleuargleu
115120125
valserproglyalaprotyrphelysasnalaasnleuleuvalala
130135140
alaaspcysthralatyralatyrglyaspphehisasnasppheile
145150155160
lysasnhisilethrvalileglycysprolysleuaspaspvalthr
165170175
tyrtyrlysasplysleulysgluileilegluleuasnaspleulys
180185190
serilethrvalvalargmetgluvalprocyscysserglyileval
195200205
seralavallysthralametleuglualalysvalilevalprophe
210215220
arggluvalileileglythrasnglygluilearg
225230235
<210>5
<211>684
<212>dna
<213>巴氏梭菌
<220>
<221>misc_特征
<223>bee63_rs05095基因序列
<400>5
atggataaagaacttttaagtatattaaagaactgtattttattttgcaaaattgatgat60
actaaaattaatgagcttttttcatatataaattattcaataaaaaaatactcaaatgga120
gaaactgtggccattgaaggtgacgagtgcagcagcataggtataattttagacggtttt180
gctgagatacaaaaaatctacgaatccggcaaaagtttaactatagagaccttaaacact240
aacaagatatttggtgaagcaataatattttctaaaaaaaatacttatccggctacaata300
atttcatgtgccaaaacaagtatattattcataccaaaatcctcaataattaaattatgc360
agcgaaaattcaaattttctaaacggctttatgtctcttctatctaataaaatattaatg420
ctaaataaaaagttaaaaaatctttcatataacacaattagagaaaaagtagcaaattac480
ctacttgaagagactataatccaaaaaaatccaaatataaaaatgaataaatcaaaaaaa540
cagctgtctgaattactcggtattccacgtccttctctgtctagagaattaataaagtta600
aggaacgatggtatcatttcctttgatagaaattatattaaaatattaaatatggatgag660
ttaaaaaatatcctagataagtag684
<210>6
<211>227
<212>prt
<213>巴氏梭菌
<220>
<221>misc_特征
<223>bee63_rs05095蛋白序列:wp_066020350
<400>6
metasplysgluleuleuserileleulysasncysileleuphecys
151015
lysileaspaspthrlysileasngluleuphesertyrileasntyr
202530
serilelyslystyrserasnglygluthrvalalailegluglyasp
354045
glucysserserileglyileileleuaspglyphealagluilegln
505560
lysiletyrgluserglylysserleuthrilegluthrleuasnthr
65707580
asnlysilepheglyglualaileilepheserlyslysasnthrtyr
859095
proalathrileilesercysalalysthrserileleupheilepro
100105110
lysserserileilelysleucyssergluasnserasnpheleuasn
115120125
glyphemetserleuleuserasnlysileleumetleuasnlyslys
130135140
leulysasnleusertyrasnthrileargglulysvalalaasntyr
145150155160
leuleuglugluthrileileglnlysasnproasnilelysmetasn
165170175
lysserlyslysglnleusergluleuleuglyileproargproser
180185190
leuserarggluleuilelysleuargasnaspglyileileserphe
195200205
aspargasntyrilelysileleuasnmetaspgluleulysasnile
210215220
leuasplys
225
<210>7
<211>690
<212>dna
<213>丁酸梭菌
<220>
<221>misc_特征
<223>cby_rs14495基因序列
<400>7
atgaaaaatgttaatgagattatagataaaataaagcaaaatgaattttttaaggggata60
gatgataaaaaaatagaaatgattatttcagaattaagtcatatttctaaggaatattca120
aaaggacaggtaattgctaatgagggggaggtttgtaaaaatctaggattagtcgtagat180
ggaattgttgagatacagagaatatactcaagcggaaaacatatagttcttaaacgtatg240
ggagcaggagaagtttttggagaagccataatattttcagataaaaataaatatccagcc300
acaataattgcttcttcggattgtataatatcttatttgaaaaaagaagatattattaaa360
ctttgccttaatgaagaaataatcttaaagaattttataactttattgagcaataaaatt420
tttatattaaatagaaaaataaaaaccatatcttttaaaacaatacgacagaaagttgtt480
aactttatattagaacagtcaaaaagtcagaacaataaaactgtaattttgaaaataagt540
aaagagcagatagcatctttacttggaatacctagaccatcactttcaagagagcttatg600
aagcttagagatgatggacttattgaatttgataggaacaaaataagtattataaatata660
gaaagacttgaagaagaacttttagaatag690
<210>8
<211>229
<212>prt
<213>丁酸梭菌
<220>
<221>misc_特征
<223>cby_rs14495蛋白序列:wp_003410673.1
<400>8
metlysasnvalasngluileileasplysilelysglnasngluphe
151015
phelysglyileaspasplyslysileglumetileilesergluleu
202530
serhisileserlysglutyrserlysglyglnvalilealaasnglu
354045
glygluvalcyslysasnleuglyleuvalvalaspglyilevalglu
505560
ileglnargiletyrserserglylyshisilevalleulysargmet
65707580
glyalaglygluvalpheglyglualaileilepheserasplysasn
859095
lystyrproalathrileilealaserseraspcysileilesertyr
100105110
leulyslysgluaspileilelysleucysleuasnglugluileile
115120125
leulysasnpheilethrleuleuserasnlysilepheileleuasn
130135140
arglysilelysthrileserphelysthrileargglnlysvalval
145150155160
asnpheileleugluglnserlysserglnasnasnlysthrvalile
165170175
leulysileserlysgluglnilealaserleuleuglyileproarg
180185190
proserleuserarggluleumetlysleuargaspaspglyleuile
195200205
glupheaspargasnlysileserileileasnilegluargleuglu
210215220
glugluleuleuglu
225
<210>9
<211>690
<212>dna
<213>丁酸梭菌
<220>
<221>misc_特征
<223>k670_rs19640基因序列
<400>9
atgaaaaatgctaatgagattatagataaaataaagcaaaatgaattttttaaggggata60
gatgataaaaaaatagaaatgattatttcagaattaagtcatatttctaaggaatattca120
aaaggacaggtaattgctaatgagggggaggtttgtaaaaatctaggattagtcgtagat180
ggaattgttgagatacagagaatatactcaagcggaaaacatatagttcttaaacgtatg240
ggagcaggagaagtttttggagaagccataatattttcagataaaaataaatatccagcc300
acaataattgcttcttcggattgtataatatcttatttgaaaaaagaagatattattaaa360
ctttgccttaatgaagaaataattttaaagaattttataactttattgagcaataaaatt420
tttatattaaatagaaaaataaaaaccatatcttttaaaacaatacgacagaaagttgtt480
aactttatattagaacagtcaaaaagtcagaacaataaaactgtaattttgaaaataagt540
aaagagcagatagcatctttacttggaatacctagaccatcactttcaagagagcttatg600
aaacttagagatgatggacttattgaatttgataggaacaaaataagtattataaatata660
gaaagacttgaagaagaacttttagaatag690
<210>10
<211>229
<212>prt
<213>丁酸梭菌
<220>
<221>misc_特征
<223>k670_rs19640蛋白序列:wp_035765453.1
<400>10
metlysasnalaasngluileileasplysilelysglnasngluphe
151015
phelysglyileaspasplyslysileglumetileilesergluleu
202530
serhisileserlysglutyrserlysglyglnvalilealaasnglu
354045
glygluvalcyslysasnleuglyleuvalvalaspglyilevalglu
505560
ileglnargiletyrserserglylyshisilevalleulysargmet
65707580
glyalaglygluvalpheglyglualaileilepheserasplysasn
859095
lystyrproalathrileilealaserseraspcysileilesertyr
100105110
leulyslysgluaspileilelysleucysleuasnglugluileile
115120125
leulysasnpheilethrleuleuserasnlysilepheileleuasn
130135140
arglysilelysthrileserphelysthrileargglnlysvalval
145150155160
asnpheileleugluglnserlysserglnasnasnlysthrvalile
165170175
leulysileserlysgluglnilealaserleuleuglyileproarg
180185190
proserleuserarggluleumetlysleuargaspaspglyleuile
195200205
glupheaspargasnlysileserileileasnilegluargleuglu
210215220
glugluleuleuglu
225
<210>11
<211>690
<212>dna
<213>丁酸梭菌
<220>
<221>misc_特征
<223>at697_rs15885基因序列
<400>11
atgaaaaatgttaatgagattatagataaaataaagcaaaatgaattttttaaggggata60
gatgataaaaaaatagaaatgattatttcagaattaagtcatatttataaggaatattca120
aaaggacaggtaattgctaatgagggggaggtttgtaaaaatctaggattagtcgtagat180
ggaattgttgagatacagagaatatactcaagcggaaaacatatagttcttaaacgtatg240
ggagcaggagaagtttttggagaagccataatattttcagataaaaataaatatccagcc300
acaataattgcttcttcggattgtataatatcttatttgaaaaaagaagatattattaaa360
ctttgccttaatgaagaaataatcttaaagaattttataactttattgagcaataaaatt420
tttatattaaatagaaaaataaaaaccatatcttttaaaacaatacgacagaaagttgtt480
aactttatattagaacagtcaaaaagtcagaacaataaaactgtaattttgaaaataagt540
aaagagcagatagcatctttacttggaatacctagaccatcactttcaagagagcttatg600
aagcttagagatgatggacttattgaatttgataggaacaaaataagtattataaatata660
gaaagacttgaagaagagcttttagaatag690
<210>12
<211>229
<212>prt
<213>丁酸梭菌
<220>
<221>misc_特征
<223>at697_rs15885蛋白序列:wp_046057470.1
<400>12
metlysasnvalasngluileileasplysilelysglnasngluphe
151015
phelysglyileaspasplyslysileglumetileilesergluleu
202530
serhisiletyrlysglutyrserlysglyglnvalilealaasnglu
354045
glygluvalcyslysasnleuglyleuvalvalaspglyilevalglu
505560
ileglnargiletyrserserglylyshisilevalleulysargmet
65707580
glyalaglygluvalpheglyglualaileilepheserasplysasn
859095
lystyrproalathrileilealaserseraspcysileilesertyr
100105110
leulyslysgluaspileilelysleucysleuasnglugluileile
115120125
leulysasnpheilethrleuleuserasnlysilepheileleuasn
130135140
arglysilelysthrileserphelysthrileargglnlysvalval
145150155160
asnpheileleugluglnserlysserglnasnasnlysthrvalile
165170175
leulysileserlysgluglnilealaserleuleuglyileproarg
180185190
proserleuserarggluleumetlysleuargaspaspglyleuile
195200205
glupheaspargasnlysileserileileasnilegluargleuglu
210215220
glugluleuleuglu
225
<210>13
<211>711
<212>dna
<213>巴氏梭菌
<220>
<221>misc_特征
<223>bee63_rs05090基因序列
<400>13
atgaaaagaaaaatagtgaatatagataaagaaaaatgtaatggatgtggactttgtagt60
aaggcatgccatgaaaatgctatagaaataattgatggaaaagcagaacttgtttcggat120
gaatattgtgatggacttggagattgccttccacattgtccagaaaacgctataactata180
atagaaagagaaagtaaagaatacgatgaagaagctgtaaaaagaagaattgaaagtaaa240
gaaaagaaaatgccatgtggatgtccaggttcaatggcaaaaaagataactagagtttct300
aaaaaagtagaagttaaacatgaagatacgggtgcagcatcagaacttatgcagtggcct360
gtacaattgcaattagtaaatccaaatgcaagctattttgaaggcgcaaatcttttagtt420
gcagcggattgtacagcttatgcttatggtaactttcatcaagattttataaaaaatcat480
ataactgtaataggttgtcctaaacttgacgatgttgagtactataagaataaaatcaaa540
gaaattatagaaaataataatcttaaaagtattacagttaccagaatggaagtaccatgc600
tgtggcggtatagtgtcagctgtaaagaatgctatgcttgaggcacaggttatattacct660
tatagagaagtagttataggaactaatggagaagtgaaggaagcgaagtaa711
<210>14
<211>236
<212>prt
<213>巴氏梭菌
<220>
<221>misc_特征
<223>bee63_rs05090蛋白序列:wp_066020349.1
<400>14
metlysarglysilevalasnileasplysglulyscysasnglycys
151015
glyleucysserlysalacyshisgluasnalailegluileileasp
202530
glylysalagluleuvalseraspglutyrcysaspglyleuglyasp
354045
cysleuprohiscysprogluasnalailethrileilegluargglu
505560
serlysglutyraspgluglualavallysargargilegluserlys
65707580
glulyslysmetprocysglycysproglysermetalalyslysile
859095
thrargvalserlyslysvalgluvallyshisgluaspthrglyala
100105110
alasergluleumetglntrpprovalglnleuglnleuvalasnpro
115120125
asnalasertyrphegluglyalaasnleuleuvalalaalaaspcys
130135140
thralatyralatyrglyasnphehisglnasppheilelysasnhis
145150155160
ilethrvalileglycysprolysleuaspaspvalglutyrtyrlys
165170175
asnlysilelysgluileilegluasnasnasnleulysserilethr
180185190
valthrargmetgluvalprocyscysglyglyilevalseralaval
195200205
lysasnalametleuglualaglnvalileleuprotyrarggluval
210215220
valileglythrasnglygluvallysglualalys
225230235
<210>15
<211>1524
<212>dna
<213>丙酮丁醇梭菌
<220>
<221>misc_特征
<223>包含ca_c0884、基因间区域和ca_c0885的全序列
<400>15
ttattctgcagcttctaaaatttcttttaaagagttaatatctaatattgtaatagatgt60
tctatcaaagtctataattccttcttcacgtaggtttatgaattctctagataatgatgg120
ccttggaattccaagcatttcagacaactgtttttttgacttattcattttaaaagttaa180
gttattttgagcttcgtactgttcaagtatatagtttgatattttctctcttatagtatg240
ataagacatattttttaattttttattcaacatcaatatcttattagatagaagagacat300
aaagccattaaggaaaagggaattgtcgctgcataatttagatattgaggctttgggaat360
aaaaattatagtacttttagtacatgctattattgtagaaggatagcttgttttattgga420
gaatattattgcttcaccaaatatcttgctttcttctagtgttgttattgtaagactttt480
accagattcgtaaattttctgtatttcaacacaaccgcttaatactattccaattttatt540
gcacgtatcgccttcaattgcgatagtttcatctttatagtattcttttattgagtaatt600
tatagatgaaaataaatgatcaatttcctgagtttcaatttttgaaaataagatacaatg660
ttttaatacggttaaaatttttttatccatgtttttctcccaatacttttttgtgtaaca720
tatgttactgatatatttatataattttaatataatttaatcataaagacaagtaatttt780
attttgcggaactttatatttaggaggaaaaaaatgaaaagaaaaatagtaaacatagat840
aaagataaatgtaatggatgtggactttgtagtgaagcatgtcatgaaaatgcaattgaa900
ataattaatggaaaagcagagcttttatctgatgaatattgtgatggtttaggagattgt960
ttacctcattgtccagttgatgcaataactataatagagagagaaagtaaggaatatgat1020
gaagaggcagttcagagaagaattgaagaaaagaaaaaatcaaagttagctaaaccctgt1080
ggatgtccaggagctatggctaaaaaaatagaaagagtagctaagcctttagctaaagta1140
aaggaagataggtcttctgtttcagagttaatgcagtggccagttcagttaaggcttgta1200
agtccaggagctccatattttaaaaatgctaatcttttagtagctgcagattgtacagcc1260
tatgcttatggtgactttcacaatgattttataaagaatcatataacagtaataggatgt1320
ccaaaacttgatgatgttacatattacaaagataagttgaaagaaattatagaacttaat1380
gaccttaagagtataacagttgttagaatggaggtaccttgctgttcaggcatagtttca1440
gcagtaaagactgctatgcttgaagcaaaagttatagtaccttttagagaagttattata1500
ggaactaatggtgaaattagataa1524
<210>16
<211>123
<212>dna
<213>丙酮丁醇梭菌
<220>
<221>misc_特征
<223>丙酮丁醇梭菌atcc824基因组的位置1014117和1014239之间的基因间区域
<400>16
gtttttctcccaatacttttttgtgtaacatatgttactgatatatttatataattttaa60
tataatttaatcataaagacaagtaattttattttgcggaactttatatttaggaggaaa120
aaa123
<210>17
<211>124
<212>dna
<213>人工
<220>
<223>包含单个核苷酸插入的基因间区域
<400>17
gtttttctcccaatacttttttgtgtaacatatgttactgatatatttatataattttaa60
tataatttaatcataaagacaagtaattttattttgcggaactttatatttaggaggaaa120
aaaa124
<210>18
<211>17
<212>dna
<213>人工
<220>
<223>基因间区域正向引物
<400>18
gccattaaggaaaaggg17
<210>19
<211>19
<212>dna
<213>人工
<220>
<223>基因间区域反向引物
<400>19
gctcctggacttacaagcc19
<210>20
<211>20
<212>dna
<213>人工
<220>
<223>基于ca_c0884基因的正向引物
<400>20
gaagagacataaagccatta20
<210>21
<211>21
<212>dna
<213>人工
<220>
<223>基于ca_c0884基因的反向引物
<400>21
acaataacaacactagaagaa21
<210>22
<211>19
<212>dna
<213>人工
<220>
<223>基于ca_c0885基因的正向引物
<400>22
gctaaaccctgtggatgtc19
<210>23
<211>22
<212>dna
<213>人工
<220>
<223>基于ca_c0885基因的反向引物
<400>23
ggacttacaagccttaactgaa22
<210>24
<211>18
<212>dna
<213>人工
<220>
<223>基于ca_c1628基因的正向引物
<400>24
cccttagaggaggctatg18
<210>25
<211>18
<212>dna
<213>人工
<220>
<223>基于ca_c1628基因的反向引物
<400>25
gccatctcttacatctgg18
- 还没有人留言评论。精彩留言会获得点赞!