用于向细胞外囊泡装载化学和生物试剂/分子的组合物和方法与流程
相关申请的交叉引用
本申请要求2017年5月11日提交的美国临时申请号62/504,941和2018年2月15日提交的美国临时申请号62/631,060的优先权,其公开内容因此通过引用并入。
政府基金
本发明是在由美国国立卫生研究院(nationalinstitutesofhealth)授予的基金号hl126082、eb021454和eb023262的政府支持下完成的。政府拥有本发明的某些权利。
本公开内容总体上涉及用于向外泌体/细胞外囊泡装载化学和生物试剂和/或用于调节外泌体/细胞外囊泡的运载物(cargo)分选的组合物和方法。
背景技术:
在1毫升的人血液中,存在大约十亿个外泌体:包含蛋白质、mirna和mrna的脂质囊泡。细胞将外泌体释放到细胞外环境中,在那里它们参与许多生理和病理生理过程。外泌体起源于多囊泡体(multivesicularbodie,mvb),其在释放到细胞外环境之前与浆细胞膜融合。释放时,外泌体可与邻近的细胞融合以转移其运载物。现已知道,外泌体并不仅仅是不需要的细胞蛋白质和“垃圾rna”的运载体(vehicle),而且细胞主动分泌外泌体以调节其微环境。
外泌体参与多种疾病的发病机理,包括癌症、神经变性、自身免疫和肝脏疾病。多项研究已经检查了外泌体在癌症生长和转移中的作用[costa-silvab等人natcellbiol.2015;17(6):816-826;grangec等人cancerresearch.2011;71(15):5346-5356;hoodjl等人cancerresearch.2011;71(11):3792-3801;kucharzewskap等人proceedingsofthenationalacademyofsciencesoftheunitedstatesofamerica.2013;110(18):7312-7317;peinadoh等人natmed.2012;18(6):883-891]。由于来源于肿瘤细胞的细胞外囊泡(例如外泌体)有将脂肪来源的间充质干细胞转化为肿瘤相关的肌成纤维细胞潜力,因此已经提出这些外泌体可通过产生肿瘤基质而促进肿瘤进展和恶性表型[choja等人intjoncol.2012;40(1):130-138]。外泌体在诱导转移中也发挥着作用,最终导致超过90%的癌症相关死亡:转移性疾病的治疗仍然是临床挑战。癌细胞来源的外泌体有可能将其直接微环境中的健康细胞转化为肿瘤形成细胞:由癌细胞释放的外泌体可以将癌基因(主要通过致癌小rna)转移至受体细胞,诱导癌细胞迁移并促进血管生成,这是重要的癌症“标志”[meehank等人critrevclinlabsci.2015:1-11]。由于其固有的全身扩散能力,它们还可以通过准备转移前微环境(pre-metastaticniche)来在远处位点引发新的肿瘤生长。特别地,已经显示出来自肺向性4175-lut癌细胞(mda-mb-231乳腺癌子系)的外泌体是如何在全身性施用后特异性地定位至肺部并且被肺部驻留的成纤维细胞摄取。这些外泌体不仅能够使骨向性肿瘤细胞的迁移从骨部位定向至肺,还能使那些细胞在肺中的转移能力提高10,000倍[hoshinoa等人nature.2015;527(7578):329-335]。这些数据表明,循环的外泌体为将来的转移性肿瘤准备了离散的位点,符合种子-土壤假说。外泌体和/或细胞外囊泡在生理过程中也发挥作用,并且可具有再生作用。例如,已经显示来源于间充质干细胞(msc)的外泌体/细胞外囊泡在心肌梗死后通过调节受损的组织环境、诱导血管生成并诱导细胞增殖和分化来介导心脏组织修复[barilel等人cardiovascres.2014;103(4):530-541;lairc等人stemcellresearch.2010;4(3):214-222]。
因此,对可用于通过将外泌体重编程以阻止和/或逆转疾病进展来阻断不希望的细胞通讯并且可用于将外泌体重编程或装载化学和生物试剂使得其可以将其它期望运载物递送至靶细胞的组合物和方法存在着持续且未被满足的需求。本公开内容与这些需求有关。
技术实现要素:
本公开内容涉及rna多核苷酸序列(在本文中不时称为“exo-code”),其能够a)被选择性地分选至细胞外囊泡(例如外泌体),并且b)递送多种运载物类型以对细胞外囊泡进行编程或重编程。尽管使用外泌体说明了本公开内容的非限制性实施例,但是本公开内容包括使用exo-code将含有exo-code的多核苷酸分选至任何分泌的膜结构,包括但不限于外泌体、囊泡、微囊泡、微颗粒、内体来源的囊泡、多囊泡体、凋亡小体及其组合。
在某些方面,本公开内容包括含有exo-code的试剂,其包含rna片段,其中所述rna片段包含exo-code序列,并且其中所述试剂还包含运载物部分。所述运载物部分可选自多核苷酸、肽、多肽、蛋白质、荧光团和小(药物)分子。
本公开内容包括将任何经修饰的多核苷酸和/或经修饰的外泌体和/或经修饰的细胞组合物的作用与合适的参考进行比较。参考可以包括经修饰的多核苷酸和/或经修饰的外泌体和/或经修饰的细胞组合物的功能的任何合适的对照值或者测量量,例如标准曲线、滴定、曲线下面积,或者与天然存在的组合物或过程的能力,包括但不限于未经修饰的或其它对照系统中rna外泌体分选的效率、动力学、量等的比较。
本公开内容包括组合物,包括但不限于适用于人和/或兽医用途的药物组合物,其中所述组合物包含含有exo-code的多核苷酸。药物组合物通常包含至少一种药学上可接受的赋形剂、载体、稀释剂等。本公开内容包括经修饰以产生包含含有exo-code的多核苷酸的外泌体的细胞培养物,包含所述外泌体的细胞培养基以及分离和/或纯化的外泌体群体,其中所述群体中的至少一些外泌体包含含有exo-code的多核苷酸。本公开内容包括包含外泌体的药物组合物,所述外泌体包含含有exo-code的多核苷酸。本公开内容包括制备含有exo-code的多核苷酸的方法,例如通过化学合成或者体外或体内转录。还包括含有exo-code的不同多核苷酸的组合,其中所述组合具有大于叠加的或协同的对至少一种exo-code/外泌体分选特性的作用和/或对引入了本公开内容的经修饰的外泌体的细胞的作用。本公开内容的多核苷酸可包含一个或多于一个exo-code序列,并且可包含多于一个相同的exo-code序列或不同的exo-code序列。
更详细地,多价是设计两个部分之间更高亲和力相互作用的良好建立的方法。可以将基本上无限数量的exo-code和不同exo-code的组合引入任何单个多核苷酸中,以用于实现增强的外泌体递送的潜力。但是,随着引入更多的exo-code,在增加的亲和力与多核苷酸的尺寸之间可能存在反比关系。因此,在一些非限制性实施例中,本公开内容的多核苷酸包含1、2、3或更多个exo-code。此外,尽管当直接电穿孔到活细胞中以进行有效的外泌体包装时可以实现exo-code的优势,但在其不可行的应用中,可以使用直接外泌体装载。出于体内递送的目的,可以通过良好建立的电穿孔方案将exo-code直接装载到分离的患者来源外泌体中。其也可以引入到能够将exo-code递送至受体细胞的细胞质的任何合成脂质递送运载体(例如脂质体、阳离子脂质复合物(cationiclipoplexes))、其它聚合物递送运载体或任何宿主递送运载体。这些细胞然后可以将exo-code包装到外泌体中,用于体内外泌体编程或其它目的。
本公开内容包括用于制备、筛选和使用exo-code和包含它们的多核苷酸的试剂盒,其用于多种研究、治疗和预防方法,鉴于本公开内容的益处,这对于本领域技术人员而言将是显而易见的。试剂盒可以包含用于制备和筛选外泌体的试剂,并且可以包含含有exo-code的多核苷酸,并且可以包含可用于将这样的多核苷酸引入细胞的试剂。
附图说明
为了更全面地理解本公开内容的本质和目的,应当参考结合附图的以下详细描述。
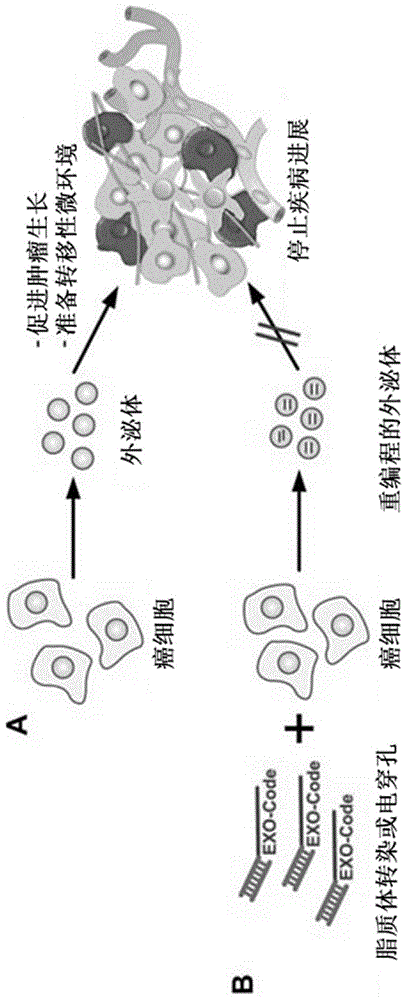
图1提供了使用含有exo-code的多核苷酸对从癌细胞释放的外泌体进行重编程,从而停止疾病进展的非限制性说明。(a)肿瘤分泌的外泌体已经显示出在远处的器官中准备转移性微环境。(b)描绘了exo-code缀合至治疗性运载物以对肿瘤来源的外泌体进行重编程以停止疾病进展的示意图。
图2提供了说明利用外泌体生物发生来鉴定在外泌体中特异性富集的仿生exo-code的示意图。在外泌体生物发生过程中,非编码rna/mrna可以在mvb膜向内出芽的过程中装载到外泌体中。核酸的装载可以通过以下方式发生:(a)被动机制,(b)/(c)/(d)经通过序列特异性序列的主动分选机制。(e&f)外泌体通过与细胞膜融合而释放到细胞外环境中。(d)我们调整了该过程以鉴定本文所述的exo-code。
图3提供了用于鉴定本公开内容的exo-code的rna文库筛选方法的示意图。用大的rna序列库(1012多样性)对细胞进行电穿孔。收集外泌体并提取rna序列。在转化为cdna之后,将pcr扩增的dna转录为rna,以产生用于下一个选择回合的rna库。利用递增的选择压力(通过减少输入rna的量)将选择回合重复6至10次。已对mda-mb-231细胞、pc-3细胞、hek293细胞、间充质干细胞和raw264.7细胞中的选择回合3至7进行了illumina下一代测序。
图4示出了使用本公开内容的方法来鉴定exo-code序列的说明性概念验证示例。(a)在三个选择回合之后鉴定exo-code基序。e值:9.5e-018,(b)对于非特异性对照,在高度保守的位置将点突变引入exo-code。(c)这导致外泌体富集的几乎完全消除,并强烈表明了我们的新exo-code的特异性。数据为平均值±标准偏差。通过双尾学生t检验,**p<0.01。
图5显示了(a)rna文库的多样性随着每一个选择回合而降低。这表明我们的选择程序导致了特异性序列的富集。(b)在mda-mb-231细胞中10个选择回合后,前15个富集的exo-code。
图6显示了在mda-mb-231细胞、raw264.7细胞、间充质干细胞和hek293细胞中经过数个选择回合之后rna序列的富集。分析选择回合3、5、7和/或10中存在的每个序列的每百万读读段(readspermillion)。
图7显示了本公开内容的iupac核苷酸代码和iupac核苷酸代码的图例。
图8显示了(a和b)归一化的计算机对接评分,其显示与实验确定的rna序列与hnrnpl的结合亲和力具有高度相关性(r2=0.82)。exo-code中的序列与hnrnpl结合,预测的结合亲和力为3.23nm(ccccaca)和223.2nm(cacagca)。(c)与hnrnpl的rna识别基序(rrm)结合的ccccaca(白色)的表面渲染图。(d)计算的静电表面电势显示ccccaca与hnrnpl的相互作用(显示阴离子和阳离子电荷)。(e)通过计算机对接预测的与hnrnpl的主要结合基序是“caca”。如在我们的基序(cccccaca)中可以发现,以“ccc”开头的“caca”似乎没有引起空间位阻,因为计算机对接评分仍在低纳摩尔范围内对基序排名。cccccaca与hnrnpl的结合由分子间氢键(虚线)介导。该结合通过c4和arg102之间的静电相互作用以及a5和tyr168之间的堆叠相互作用进一步稳定。该数据与hnrnpl和ca重复序列(例如,cacaca)或通过间隔区分隔的重复序列(例如canrca和canrca)的结合是一致的。
图9显示了在电穿孔进入其亲本细胞系(mda-mb-231细胞、间充质干细胞、raw264.7细胞和hek293细胞)之后,exo-code的相对外泌体富集。
图10显示了(a、b和c)rna沉降测定(rna-pulldownassay)。
图11显示了使用rnaimax在12孔板中用对照sirna(有义:ucacaagggagagaaagagaggaagga(seqidno:61))或两种hnrnpmsirna(有义1:gcauaggauuuggaauaaa(seqidno:62))有义2:ggaauggaaggcauaggauuu(seqidno:63))以30pmol的总sirna处理mda-mb-231细胞。a)在转染后48小时收集来自每种条件的3个孔,并进行western印记以评估hnrnpm敲低(origeneta301557)。b)使用imagej软件评估归一化至gapdh的敲低百分比。在sirna处理后48小时,用1μgexo-codem1rna或m8对来自每种条件的另外3个孔进行电穿孔。在电穿孔后24小时,收集外泌体/ev并提取rna。由qpcr确定相对m1和m8丰度。耗尽约75%的hnrnpm的细胞在装载m8exo-code方面表现出约60%降低,并且在将m1exo-code装载到外泌体/ev方面表现出约90%降低。hnrnpm敲低不影响随机对照(rc)序列的分选。
图12显示(a)exo-code/pre-mir-31嵌合体的示意图。(b)用嵌合exo-code/pre-mir-31重编程的外泌体使细胞迁移抑制75%。相比之下,来自用ns-ctrl/pre-mir-31嵌合体处理细胞的外泌体仅显示对mda-mb-231细胞迁移的31%抑制。与仅pre-mir-31对照相比,抑制不是统计学显著的。(c)划痕测定的代表性图像,其显示当细胞与来源于ns-ctrl/pre-mir-31和pre-mir-31ctrl处理的细胞的外泌体一起孵育时,间隙完全闭合。对于用经exo-code/pre-mir-31重编程的外泌体处理的mda-mb-231细胞,24小时后大量间隙依然可见。比例尺:200μm。数据为平均值±sd。通过单因素anova和事后tukey,**p<0.01,*p<0.05。
图13显示了(a)将7个选择回合后来自干细胞的6号序列exo-codes6添加到用“a”接头间隔开的mir-132的互补序列,以形成顶链rna:gcuggucaggaugcgggggcaaaguaacaaucgaaagccacgguuuu(seqidno:66);然后将该rna与合成的mir-132:aaccguggcuuucgauuguuacuu(seqidno:67)双链体化。(b)用等摩尔量的单独的合成mir-132或附着于s6exo-code的mir-132一式三份对间充质干细胞进行电穿孔。收集外泌体并对mir-132进行定量,电穿孔的mir-132使间充质干细胞外泌体中此mirna的量增加了10倍。利用exo-codes6可以使外泌体丰度再增加6倍,从而使msc外泌体的mir-132效力提高60倍。(c)人脐静脉内皮细胞(huvec)的血管生成测定。用从用exo-code-msc6/mir-132或未经修饰的mir-132电穿孔的人间充质干细胞收集的外泌体处理huvec。在所有显示的参数中,来自用exo-code-msc6/mir-132处理的间充质干细胞的外泌体在节点数、连接数、总长度、总分支长度和分支数方面显示出统计学上的显著增加。用单因素anova和事后tukey分析数据(n=3),****p<0.0001,***p<0.001,**p<0.01,*p<0.5。
图14显示了示出对exo-code将不同尺寸的运载物递送到外泌体的能力的评估的示意图。可以通过(a)电穿孔和(b)通过脂质体、聚合物和肽转染以及通过其它合适的方法将包含多价exo-code的mirna/sirna和mrna递送至mda-mb-231、pc-3和其它细胞。可以通过茎环rt-pcr(对于mirna/sirna)和rt-pcr(对于mrna)对分选至外泌体的mirna/sirna和mrna的量进行定量。
图15显示了使用经修饰的核苷酸,干细胞和mda细胞中snailsirna的增加的装载。
图16在(a)中显示:分析从最新选择回合到meme基序算法的前1,000个序列,证明了高统计学显著性基序(显示的最低e值基序)。来自raw和msc细胞的最高显著性基序显示出高度相似性。(b)来自pc-3和raw细胞的较低统计学显著性基序显示出高度相似性。
图17显示了外泌体/ev表征研究(a)外泌体/ev群体的纳米颗粒追踪分析(nanoparticletrackinganalysis,nta)。(b)透射电子显微镜(tem)。nta和tem显示,大多数外泌体/ev落在30-150nm的预期尺寸范围内。(c)nta颗粒计数也与蛋白质含量相关,作为囊泡计数的次要手段。
图18显示了具有相似序列元件的基序在视觉上进行了比对和分组;箭头指示细胞系之间的相似基序。
图19显示了msc和mda-mb-231细胞的其它序列。具体地,在由三个腺嘌呤分隔的连续链中合成计算机方法鉴定的基序和snailsirna的反义链。将这些构建体与cy5标记的snail有义链rna退火,并一式三份以每120,000msc或300,000mda-mb-231细胞5ug的有义荧光报告基因sirna进行电穿孔。将细胞在无外泌体的培养基中培养24小时,并通过总外泌体分离试剂法分离外泌体/ev。然后将外泌体/ev重悬在pbs中,并在4℃下与cd63珠结合过夜。洗涤与外泌体/ev结合的珠,并通过流式细胞术进行分析。绘制了平均荧光强度相对于背景的变化倍数。背景定义为与cy5标记的snail结合的随机对照减去没有将cy5标记的snail电穿孔到繁殖细胞中的外泌体结合珠。多个计算机预测的基序显示出比对照显著更高的富集。通过单因素anova然后dunnett事后检验进行统计分析。
图20显示与未经修饰的mcherrymrna相比,exo-code使mcherrymrna进入mda-mb-231细胞的外泌体的装载增加了约7倍。
图21显示了用杂交体exo-code标记的snailsirna分选至来源于mda-mb-231细胞的外泌体。将突变引入杂交体exo-code中以评估对分选的影响。
图22显示了用m8exo-code标记的snailsirna分选至来源于mda-mb-231细胞的外泌体。将突变引入m8exo-code中以评估对分选的影响。
图23显示了用s6exo-code标记的snailsirna分选至来源于msc的外泌体。将突变引入s6exo-code中以评估对分选的影响。
具体实施方式
除非本文另外定义,否则本公开内容中使用的所有技术和科学术语具有与本公开内容所属领域的普通技术人员通常理解的相同含义。
在整个说明书中给出的每个数值范围包括其上限值和下限值,以及落入该范围内的每个较窄的数值范围,如同这些较窄的数值范围均在本文中明确写出。
本公开内容包括本文所述的所有多核苷酸和氨基酸序列。每个rna序列包括其dna等同物,并且每个dna序列包括其rna等同物。包括互补和反平行多核苷酸序列。本公开内容涵盖编码本文公开的多肽的每个dna和rna序列。本公开内容包括多核苷酸共有序列和基序。
本公开内容涉及rna多核苷酸序列(在本文中不时称为“exo-code”),其能够a)被选择性地分选至细胞外囊泡(例如外泌体),以及b)递送多种运载物类型以对细胞外囊泡进行编程或重编程。尽管使用外泌体说明了本公开内容的非限制性实施例,但是本公开内容包括使用exo-code将含有exo-code的多核苷酸分选至任何分泌的膜结构,包括但不限于外泌体、囊泡、微囊泡、微颗粒、内体来源的囊泡、多囊泡体、凋亡小体及其组合。
关于一个实施例,并且本领域技术人员将认识到,外泌体是细胞分泌的膜结合囊泡。它们属于细胞外囊泡的组,后者共同包括外泌体、微囊泡、凋亡小体和其它外囊泡群体,例如核外颗粒体(ectosome)。它们的尺寸在30-150纳米的范围内。外泌体中富集的蛋白质包括但不限于内体和跨膜标记物,例如四跨膜蛋白(cd63、cd9、cd81)、整联蛋白、细胞粘附分子(epcam)、生长因子受体、异三聚体g蛋白和磷脂酰丝氨酸结合mfg-e8/乳凝集素。此外,外泌体可能富含内体或膜结合蛋白(tsg101)、膜联蛋白、rab和信号转导或支架蛋白。但是,不需要任何这样的蛋白质成分的存在就可以将释放的囊泡视为外泌体。特别地,外泌体膜组成是变化的,并且可能包含不同比例的磷脂(例如磷脂酰肌醇、磷脂酰乙醇胺、磷脂酰胆碱和磷脂酰丝氨酸),以及它们的“-lyso”和“甘油”衍生物,鞘磷脂、糖鞘脂、神经酰胺、胆固醇(和酯化胆固醇),以及溶血磷脂酸(lysobisphosphatidicacid,lbpa)等其它脂质。用于阐明本公开内容的实施例的使用本文进一步描述的exo-code修饰的外泌体可以将运载物递送至靶细胞。在某些方法中,本公开内容包括对病理性外泌体进行重编程。在其它方法中,本公开内容包括调节分选至外泌体的核酸或运载物。在一些实施例中,外泌体或其它分泌的膜结构包含本文所述的rna多核苷酸。当rna多核苷酸引入到外泌体中时,外泌体“包含”rna多核苷酸。因此,rna多核苷酸可以包含在外泌体的内部,例如在其中所含的水溶液或混合物之内,例如作为外泌体的组分分泌的胞质溶胶或其它细胞液体,或合成溶液。在一些实施例中,rna多核苷酸可以至少部分地嵌入膜结构中。在一些实施例中,rna多核苷酸的运载物部分也可以包含在外泌体内,或者运载物可以至少部分地嵌入膜中,或者运载物可以从膜(例如双层外泌体膜)向外突出。
本公开内容包括将任何经修饰的多核苷酸和/或经修饰的外泌体和/或经修饰的细胞组合物的作用与合适的参考进行比较。参考可以包括经修饰的多核苷酸和/或经修饰的外泌体和/或经修饰的细胞组合物的功能的任何合适的对照值或者测量量,例如标准曲线、滴定、曲线下面积,或者与天然存在的组合物或过程的能力(包括但不限于未经修饰的或其它对照系统中rna外泌体分选的效率、动力学、量等)的比较。在一些实施例中,外泌体或其它膜结构,其中本公开内容的含有exo-code序列的rna多核苷酸优先分选到由细胞分泌的外泌体中,因此,外泌体相对于对照值包含更多的rna多核苷酸。鉴于本公开内容的益处,合适的对照值对于本领域技术人员将是明显的,并且可以包括例如一定量的分选到外泌体中的含有非exo-code(non-exo-code)的rna,和/或一定量的分选至外泌体的混杂rna序列(scrambledrnasequence)(即完全或部分随机产生的序列)。
在一些实施例中,本公开内容的任一个rnaexo-code序列可以包含以下或由以下组成:本发明的说明书中、本公开内容的表格中和本公开内容的图中给出的序列或序列的片段。在一些实施例中,序列可以包含一个或多个插入、缺失和突变,只要它们保持其被优先分选至外泌体或其它膜结构中的能力即可。在某些实施例中,exo-code序列包含3-70(包括端值,并且包括其间的所有整数和整数的范围)个核苷酸或由其组成。在一些实施例中,rna多核苷酸包含至少一个本文所述的exo-code序列。在一些实施例中,rna多核苷酸包含2、3、4、5或超过5个本文所述的exo-code序列。
根据本公开内容修饰的外泌体通常将具有30-150nm的直径和约10-70nm的内部半径,因此一个外泌体的内部体积(4/3πr3)为约4×10-24–1.5×10-21m3。一个平均50kda蛋白质或100ntrna分子的体积为约6×10-26m3。因此,预计每个外泌体能够容纳约70–25000个小rna或蛋白质分子(参见例如li等人analysisofthernacontentoftheexosomesderivedfrombloodserumandurineanditspotentialasbiomarkers,phil.trans.r.soc.b369:20130502,dx.doi.org/10.1098/rstb.2013,其描述通过引用并入本文)。因此,如果提供有效包装设备,那么即使非常大的rna也可以引入外泌体。
在某些方面,本公开内容包括含有exo-code的试剂,其包含rna片段,其中所述rna片段包含exo-code序列,并且其中所述试剂还包含运载物部分。所述运载物部分可选自多核苷酸、肽、多肽、蛋白质、荧光团和小(药物)分子。
含有exo-code的多核苷酸可包含修饰以改善其功能、生物利用度、稳定性等。例如,含有exo-code的多核苷酸可包含经修饰的核苷酸和/或经修饰的核苷酸键。合适的修饰及其制备方法是本领域周知的。一些实例包括但不限于包含经修饰的核糖核苷酸或脱氧核糖核苷酸的多核苷酸。例如,经修饰的核糖核苷酸可包含核糖部分的2’位的甲基化和/或取代有以下基团:包含1-6个饱和或不饱和碳原子的--o--低级烷基,或具有2-6个碳原子的--o-芳基,其中这样的烷基或芳基可以未被取代或者可以取代有例如卤素、羟基、三氟甲基、氰基、硝基、酰基、酰氧基、烷氧基、羧基、烷氧羰基或氨基;或者取代有羟基、氨基或卤素基团。在一些实施例中,经修饰的核苷酸包含甲基胞苷和/或假尿苷。核苷酸可以通过磷酸二酯键或通过合成键(即磷酸二酯键以外的键)连接。可以在本公开内容中使用的多核苷酸试剂中核苷之间的键的实例包括但不限于磷酸二酯、烷基膦酸酯、硫代磷酸酯、二硫代磷酸酯、磷酸酯、烷基硫代磷酸酯、氨基磷酸酯、氨基甲酸酯、碳酸酯、吗啉代、磷酸三酯、乙酰胺(acetamidate)、羧甲基酯或其组合。
在某些方面,例如为了调节靶细胞中基因的表达或为了调整外泌体的性质或功能,和/或为了对外泌体进行重编程,含有exo-code的多核苷酸可包含功能性多核苷酸片段。“功能性”片段是指多核苷酸的片段,其能够对引入其的细胞施加作用,特别是当作为外泌体或其它膜结构的组分或通过与之融合而引入时。在一些实施例中,施加于细胞的作用包括抑制rna转录和/或蛋白质翻译、编辑染色体、干扰酶的功能和/或引起或参与引起细胞表型的改变,例如从病理表型转变为非病理表型,其非限制性实例是从恶性表型转变为非恶性表型。在一个实施例中,对受体细胞进行重编程,以产生在接受含有exo-code的多核苷酸之前其不产生的蛋白质。功能性片段还可以引起或参与引起细胞生长的抑制,包括但不限于细胞死亡。
在一些实施例中,施加于细胞的作用包括表达由运载物rna编码的蛋白质,所述运载物rna是本文所述的含有exo-code的多核苷酸的组分。在一些非限制性实施例中,含有exo-code的多核苷酸可适用于rna干扰(rnai)介导的靶mrna的沉默或下调,或者可适用于递送微小rna(mirna)或其它非编码rna(ncrna)。这可以例如通过将exo-code序列与至少一种rnai剂连接来实现。rnai剂可以在细胞中表达为短发夹rna(shrna)。shrna是包含有义链、反义链以及在有义片段和反义片段之间的短环序列的rna分子。shrna被输出到细胞质中,继而在细胞质中通过dicer加工成短干扰rna(sirna)。sirna是21-23个核苷酸的双链rna分子,其被rna诱导的沉默复合物(rna-inducedsilencingcomplex,risc)识别。一旦引入risc中,sirna促进靶mrna的切割和降解。因此,为了用于rnai介导的靶rna的沉默或下调,多核苷酸组分可以是sirna、shrna或mirna。在一些实施例中,功能性rna片段与exo-code序列顺式转录。在一些实施例中,功能性rna片段可以化学附接到含有exo-code的序列。在一些实施例中,可以通过使用核酶作为运载物来实现通过功能性rna片段靶向rna。在一些实施例中,核酶包括锤头状核酶、发夹状核酶或肝炎三角洲病毒核酶。
在一些实施例中,含有exo-code的多核苷酸的片段包含可以参与crispr(成簇的规则间隔的短回文重复序列)dna或rna编辑的功能性rna片段。在一些实施例中,运载物rna可以是crisprrna(crrna)或指导rna,例如sgrna。因此,运载物rna的序列可以包含与靶多核苷酸中的任何crispr位点相同或互补的片段,其还可以包含前间隔序列相邻基序(protospaceradjacentmotif,pam),前间隔序列相邻基序(pam)通常为2-6个碱基对的dna序列,并且紧接可以催化单链或双链dna断裂的核酸酶靶向的dna序列之后。在一些实施例中,可以参与crisprdna或rna编辑的运载物rna片段将包含长度为12-20个核苷酸的片段或由其组成,并且可以与靶标中的所谓间隔区序列相同或互补。指向间隔序列的12-20个核苷酸可以存在于运载物rna中,而不管靶向rna是crrna还是指导rna。在一些实施例中,可以使用单独的反式激活crrna(tracrrna)来辅助crrna的成熟。可以靶向任何rna或mrna或dna。在一些非限制性实施例中,靶向周知的snail或slugmrna、或s100a4mrna、或其组合。
含有exo-code的多核苷酸可编码或不编码蛋白质,包括但不限于旨在促进rna和/或外泌体定位和/或可视化的蛋白质,或能够在外泌体和/或靶细胞中发挥功能的蛋白质。在一些实施例中,含有exo-code的多核苷酸可包含内部核糖体进入序列(internalribosomeentrysequence,ires)。在一些非限制性实施例中,ires是野生型或经修饰的脑心肌炎病毒ires或丙型肝炎病毒ires。在一些实施例中,邻近ires(例如ires的3’)的序列包含开放阅读框。在一些实施例中,ires的3’的序列包含翻译终止密码子,使得可以停止开放阅读框的翻译。在一些实施例中,在本公开内容的含有exo-code的多核苷酸上可以存在聚a尾。在一些实施例中,本公开内容的含有exo-code的多核苷酸可具有5’帽。因此,本公开内容的实施例包括含有exo-code的多核苷酸,其至少一部分可以充当mrna,因此包含翻译开放阅读框。在一些实施例中,含有exo-code的多核苷酸可包含编码一种或多种核酸酶的序列,所述核酸酶包括但不限于cas核酸酶,例如cas9核酸酶。在一些实施例中,含有exo-code的多核苷酸可编码其它核酸酶,例如转录激活因子样效应物核酸酶(transcriptionactivator-likeeffectornuclease,talen)或锌指核酸酶(zinc-fingernuclease,zfn)。exo-code多核苷酸可编码或包含核定位信号。
在某些方面,含有exo-code的多核苷酸可编码和/或被修饰以附着于产生可检测信号的蛋白质,所述可检测信号包括但不必然限于视觉上可检测的信号、荧光信号等。在某些方法中,含有exo-code的多核苷酸可与任何肽或多肽共价连接。除外外泌体或其它分泌的膜结构的尺寸限制,这些部分的类型、序列和功能没有特别限制。在某些方法中,含有exo-code的多核苷酸可与功能性蛋白质或其片段连接。在某些实施例中,蛋白质选自酶(包括但不限于上述酶)或者受体配体、转录因子、生长因子、抗体或其抗原结合片段、可用于刺激免疫应答的肽或蛋白质免疫原(即疫苗)、基于蛋白质的化学治疗剂和毒素。在某些实施例中,连接的蛋白质包括胰岛素、生长激素或生长激素释放因子、血小板来源生长因子、表皮生长因子、任何胰岛素样生长因子、凝血因子、干扰素、任何白介素、淋巴毒素等。在一些实施例中,连接的蛋白质包含基于蛋白质的毒素,例如酶促活性毒素,其包括但不限于白喉a链、白喉毒素的非结合活性片段、外毒素a链(来自铜绿假单胞菌(pseudomonasaeruginosa))、蓖麻毒蛋白a链、相思豆毒蛋白a链、蒴莲根毒蛋白a链、α帚曲毒蛋白(sarcin)、油桐(aleuritesfordii)蛋白、香石竹毒蛋白和美洲商陆(phytolacaamericana)蛋白(papi、papii和pap-s)。通常,预期体积为1.5×10-21m3的蛋白质能够存在于直径为30-150nm的外泌体内部。特别地,直径为30-150nm的外泌体的内部半径为约10–70nm,因此一个外泌体的内部体积(4/3πr3)为约4×10-24–1.5×10-21m3。一个平均50kda蛋白质或100ntrna分子的体积为约6×10-26m3。在一些非限制性实施例中,每个外泌体可容纳任何体积小于1.5×10-21m3的蛋白质。在exo-code介导分选至较大细胞外囊泡的情况下,可以容纳较大的蛋白质。
在一些实施例中,表达本文所述的含有exo-code的多核苷酸的细胞分泌含有该多核苷酸的外泌体,并且这样的外泌体被不同细胞摄取。“不同细胞”是指在通过外泌体摄取最初暴露于含有exo-code的多核苷酸之前不包含含有exo-code的多核苷酸的细胞。因此,含有exo-code的多核苷酸可以通过作为含有exo-code的多核苷酸的组分的运载物对不同细胞产生作用。
在一些实施例中,exo-code序列不包含序列5'uagggaagagaaggacauaugau(seqidno:1)和/或不包含序列5’uugacuaguacaugaccacuuga3’(seqidno:2)。在某些实施例中,exo-code序列不包含以下序列:acccugccgccuggacuccgccugu(seqidno:3)。在某些实施例中,exo-code序列不包含ggag基序。
为了使含有exo-code的多核苷酸发挥其功能,可使用多种方法中的任何一种将它们引入外泌体或其它膜结构中。在一些实施例中,通过细胞加工将含有exo-code的多核苷酸引入外泌体。因此,可以将含有exo-code的多核苷酸引入到在原生细胞环境和外泌体加工机制中将含有exo-code的多核苷酸引入外泌体的细胞中,或者可以将它们引入个体,从而它们进入进到个体中的细胞中,并且随后引入外泌体。通过使用任何合适的技术将含有exo-code的多核苷酸引入细胞,所述技术的实例包括但不限于电穿孔、孵育、细胞活化和转染、脂质转染、脂质递送、脂质体递送、聚合物转染、聚合物递送、通过肽递送(即但不限于阳离子肽、两亲性肽、细胞穿透肽)、钙或镁沉淀和离子沉淀(也称为dna-磷酸钙沉淀)。因此,本公开内容包括体外细胞培养物,其包含一种或多种含有exo-code的多核苷酸和一种或多种促进含有exo-code的多核苷酸进入细胞的试剂。本公开内容包括编码含有exo-code的多核苷酸的转录模板,例如被配置为表达含有exo-code的多核苷酸的表达载体。可以通过使用任何合适的技术将含有exo-code的多核苷酸直接引入本文所述的外泌体或其它囊泡结构中,所述技术的实例包括但不限于电穿孔、孵育、细胞活化和转染、脂质转染、脂质递送、脂质体递送、聚合物转染、聚合物递送、通过肽递送(即但不限于阳离子肽、两亲性肽、细胞穿透肽)、钙或镁沉淀和离子沉淀(也称为dna-磷酸钙沉淀)。
本公开内容包括组合物,包括但不限于适用于人和/或兽医用途的药物组合物,其中所述组合物包含含有exo-code的多核苷酸。药物组合物通常包含至少一种药学上可接受的赋形剂、载体、稀释剂等。本公开内容包括经修饰以产生包含含有exo-code的多核苷酸的外泌体的细胞培养物,包含所述外泌体的细胞培养基以及分离和/或纯化的外泌体群体,其中群体中的至少一些外泌体包含含有exo-code的多核苷酸。本公开内容包括包含外泌体的药物组合物,所述外泌体包含含有exo-code的多核苷酸。本公开内容包括例如通过化学合成或者体外或体内转录来制备含有exo-code的多核苷酸的方法。还包括包含exo-code的不同多核苷酸的组合,其中所述组合具有大于叠加的或协同的对至少一种exo-code/外泌体分选特性的作用或者对引入了本公开内容的经修饰的外泌体的细胞的作用。本公开内容的多核苷酸可包含一个或多于一个exo-code序列,并且可包含多于一个相同的exo-code序列或不同的exo-code序列。更详细地,多价是设计两个部分之间更高亲和力相互作用的良好建立的方法。可以将基本上无限数量的exo-code和不同exo-code的组合引入任何单个多核苷酸中,以用于实现增强的外泌体递送的潜力。但是,随着引入更多的exo-code,在增加的亲和力与多核苷酸的尺寸之间可能存在反比关系。因此,在一些非限制性实施例中,本公开内容的多核苷酸包含1、2、3或更多个exo-code。此外,尽管当直接电穿孔到活细胞中以进行有效的外泌体包装时可以实现exo-code的优势,但在其不可行的应用中,可以使用直接外泌体装载。出于体内递送的目的,可以通过良好建立的电穿孔方案将exo-code直接装载到分离的患者来源外泌体中。也可以将它们引入到能够将exo-code递送至受体细胞的细胞质的任何合成脂质递送运载体(例如脂质体、阳离子脂质复合物(cationiclipoplexes))、其它聚合物递送运载体或任何宿主递送运载体。这些细胞然后可以将exo-code包装到外泌体中,用于体内外泌体编程或其它目的。在一些实施例中,分泌本文所述的外泌体的细胞可以是体外细胞组合物,例如任何来源的真核细胞、真核细胞系等。
可以使用任何合适的技术方法和方法将含有exo-code的多核苷酸引入个体。可以将含有exo-code的多核苷酸作为经修饰的或未经修饰的rna引入个体,或者通过使用例如可以表达多核苷酸的重组病毒载体,包括但不必然限于慢病毒载体、腺病毒载体和腺相关病毒载体。在某些方法中,将含有exo-code的多核苷酸作为一种或多种细胞的组分引入个体,所述细胞可以是自体细胞或异源细胞,包括但不必然限于多种类型和发育阶段的干细胞。在一个实施例中,将包含含有exo-code的多核苷酸或编码该exo-code的多核苷酸的组合物引入需要其的个体。在一些实施例中,个体需要预防和/或治疗一种或多种癌症、神经变性疾病、自身免疫疾病或其中重编程的外泌体可能对该个体具有治疗或预防益处的任何其它疾病。在一些实施例中,将有效量的含有exo-code的多核苷酸施用于个体或在个体中表达,使得至少一种rna的表达被抑制或消除,或者表达的rna(例如mrna)被降解,或者mrna的翻译被抑制。在一些实施例中,这样的施用导致个体的疾病或病症的一种或多种症状的改善。在某些方面,本公开内容的组合物和方法用于抑制转移,例如通过对癌细胞来源的外泌体进行重编程以降低或消除其在所处直接微环境中将健康细胞转化为肿瘤形成细胞的能力,和/或降低或消除致癌因子(例如致癌小rna)向受体细胞的转移。在其它某些方面,本公开内容的组合物和方法与组织再生结合使用,例如在伤口愈合的情况下,包括但不必然限于在医疗程序期间发生的伤口以及在医疗机构以外发生的伤口,包括但不限于由于任何非医疗原因而引起的组织或骨骼创伤。因此,在一些实施例中,本公开内容涉及例如紧急和非紧急医疗护理。在一些实施例中,本公开内容涉及促进血管生成,例如在血管生成将是伤口愈合的期望成分的任何情况下。
本领域技术人员将理解如何分离根据本公开内容制备的经修饰的外泌体。通常,从细胞上清液中收集外泌体,并且可以根据周知的方案通过差速离心或密度离心来分离。可以将包含多核苷酸的外泌体与不包含多核苷酸的外泌体分离,并且可以获得外泌体的亚群。可以使用标准方法从外泌体中分离含有exo-code的多核苷酸。可以使用标准方法来确定exo-code的序列,所述标准方法包括但不必然限于制备和扩增cdna,并且使用已知的测序技术和设备(包括但不限于高通量方法)来确定cdna序列。因此,本公开内容包括制备、筛选和鉴定如本文进一步描述的exo-code序列的方法。通常,用于鉴定exo-code的方法包括使具有不同序列的多个exo-code经受原生细胞内环境,该环境利用内源细胞运输机制将exo-code分选至外泌体。这种方法称为postal:通过适配体文库的细胞器特异性靶向程序(procedurefororganelle-specifictargetingbyaptamerlibraries)。通过化学合成高度多样性的rna序列,我们引入了在分选至外泌体的能力方面具有潜力胜过天然mirna和mrna的序列。因此,与仅在内源mirna内寻找基序的现有方法相比,该方法具有潜力选择显示出改善的外泌体富集的序列。在某些实施例中,相对于内源分选至外泌体的天然存在的rna多核苷酸,本公开内容的含有exo-code的多核苷酸显示出在外泌体中的优先富集。在某些实施例中,本公开内容的含有exo-code的多核苷酸表现出至少2-1000倍(包括端值,并且包括其间的所有整数和整数的范围)的富集。外泌体中的富集可通过与使用野生型或内源存在的序列确定富集而获得的值进行比较来确定。
本公开内容包括用于制备、筛选和使用exo-code和包含它们的多核苷酸的试剂盒,其用于多种研究、治疗和预防方法,鉴于本公开内容的益处,这对于本领域技术人员而言将是显而易见的。试剂盒可以包含用于制备和筛选外泌体的试剂,并且可以包含含有exo-code的多核苷酸,并且可以包含用于将这样的多核苷酸引入细胞的试剂。
在一方面,本公开内容包括多个集合文库等,其包含至少两个不同的多核苷酸,所述至少两个不同的多核苷酸包含本文所述的不同exo-code序列。因此,该文库可包含2至1000个不同的exo-code序列。因此,本公开内容包括包含2至10000个不同的exo-code序列的文库,其中至少一个exo-code序列在本文中描述。
在一种方法中,本公开内容包括选择细胞或细胞类型,以用于通过使其与本公开内容的经修饰的外泌体接触来进行修饰。该方法通常包括将细胞组合物与经修饰的外泌体混合,从而使外泌体被细胞摄取,并且包含exo-code和利用其对多核苷酸进行修饰的任何特定运载物的经修饰的多核苷酸被释放到细胞质中。随后,包含exo-code的经修饰的多核苷酸可在靶细胞中发挥其作用。这些方法可以在体外使用,并且预期适合在体内使用。因此,本公开内容包括选择需要对外泌体进行修饰和/或重编程的个体,并且向该个体施用本文涵盖的含有exo-code的多核苷酸。
提供以下实例以说明本发明的某些实施例,但其并未旨在以任何方式进行限制。
实施例1
图1提供了使用含有exo-code的多核苷酸对从癌细胞释放的外泌体进行重编程,从而停止疾病进展的非限制性说明。(a)肿瘤分泌的外泌体已经显示出在远处的器官中准备转移性微环境。(b)描绘了exo-code缀合至治疗性运载物以对肿瘤来源的外泌体进行重编程以停止疾病进展的示意图。
图2提供了说明利用外泌体生物发生来鉴定在外泌体中特异性富集的仿生exo-code的示意图。在外泌体生物发生过程中,非编码rna/mrna可以在mvb膜向内出芽的过程中装载到外泌体中。核酸的装载可以通过以下方式发生:(a)被动机制,(b)/(c)/(d)经通过序列特异性序列的主动分选机制。(e&f)外泌体通过与细胞膜融合而释放到细胞外环境中。(d)我们调整了该过程以鉴定本文所述的exo-code。
图3提供了用于鉴定本公开内容的exo-code的rna文库筛选方法的示意图。用大的rna序列库(1012多样性)对细胞进行电穿孔。收集外泌体并提取rna序列。在转化为cdna之后,将pcr扩增的dna转录为rna,以产生用于下一个选择回合的rna库。利用递增的选择压力(通过减少输入rna的量)将选择回合重复10次。已对mda-mb-231细胞、pc-3细胞、hek293细胞、间充质干细胞和raw264.7细胞中的选择回合3至7进行了illumina下一代测序。
图4示出了使用本公开内容的方法来鉴定exo-code序列的说明性概念验证示例。我们在转移性mda-mb-231乳腺癌细胞系中筛选rna文库,其导致鉴定了exo-code候选物。特别地,通过电穿孔将大的rna文库(两侧分别连接pcr引物位点的中央20个碱基的随机结构域的1012多样性物质)引入mda-mb-231细胞中。为了使rna能够分选至外泌体,使细胞生长48-72小时,然后收集分泌的外泌体。从外泌体提取富集的rna序列,并进行另外两个选择回合。为了增加选择压力,在数个选择回合中将输入rna的量从50μgrna减少至10μgrna。通过下一代测序(illuminamiseq2000,genomicsandbioinformaticscore,universityatbuffalo)分析了富集的rnaexo-code读段。三个选择回合使得可以鉴定出图4所示的代表性且非限制性exo-code。从图4中可以看出,在三个选择回合之后鉴定出了exo-code基序。e值:9.5e-018,(b)对于非特异性对照,在高度保守的位置将点突变引入exo-code。(c)这导致外泌体富集几乎完全消除,并强烈表明了我们的新exo-code的特异性。数据为平均值±标准偏差。通过双尾学生t检验,**p<0.01。
当引入到mda-mb-231细胞中时,图4所示的exo-code表现出的相对于本体细胞裂解物(bulkcelllysate)在外泌体中22倍的富集。为了评估外泌体富集的特异性,将点突变引入exo-code中,得到非特异性对照acaguagagccuucaccggc(seqidno:4),其在外泌体中仅显示出背景富集(~1.25倍富集外泌体/细胞比)。因此,数据表明该exo-code对外泌体是高度特异性的。接下来,我们进行了另一个选择回合,并鉴定了在外泌体中富集的另外的rna序列。针对mda-mb-231细胞已经分析了第3、5、7和10选择回合。十个选择回合后的15个最具代表性的序列如图5b所示。这些序列的外泌体富集的宽范围(从185至806倍的任何值)为阐明结构-活性关系提供了基础。
从图5(a)中可以看出,rna库的多样性随着每个选择回合而降低。这表明我们的选择过程导致了特异性序列的富集。5(b)在mda-mb-231细胞中10个选择回合之后,前15个富集的exo-code。在10个选择回合之后,我们能够鉴定出可以用作exo-code的额外序列。图6显示了在mda-mb-231细胞、raw264.7细胞、间充质干细胞和hek293细胞中经过数个选择回合之后rna序列的富集。分析选择回合3、5、7和/或10中存在的每个序列的每百万读读段。使用富集分布的描述性统计,基于标准偏差设置了6个富集水平,最低水平包含高至大于平均rpm一个标准偏差,最高富集水平保留给落入高于平均值32σ以上的最富集序列。此分箱方法用于最早顺序的选择回合,并绘制了每个簇的平均rpm。然后将每个簇相对于随后选择回合的那些水平绘图,允许分支到进一步富集或有时耗尽的序列级分。以这种通过选择回合的方式检查序列富集轨迹表明,大多数序列并未变得显著富集或被选择。相反,对于作为潜在exo-code审查的前10个序列,与背景读段相比存在显著且持续的富集。
我们的鉴定外泌体富集序列的方案包括导致无偏且相关的库富集的选择回合。优化通过电穿孔将大的多样的rna文库(1012多样性)递送到mda-mb-231细胞、hek293细胞、msc、raw264.7细胞和其它细胞类型中,导致将rna文库递送到细胞质中,在那里序列被分选至外泌体。文库包含两侧分别连接用于逆转录和pcr扩增(trilinkbiotechnologies)的恒定区的长度为20个核苷酸碱基的单链rna序列。培养24至72小时后可以收集外泌体。使用qiazol裂解试剂从外泌体中提取rna,逆转录,并使用以下引物进行pcr扩增:fp5'-ttcaggtaatacgactcactatagggaagaga-aggacatatgat-3'(seqidno:5)和rp5'-tcaagtggtcatgtac-tagtcaa-3'(seqidno:6)。将t7启动子引入正向引物中以允许转录成rna。分离正确大小的pcr产物并使用无细胞转录系统(来自ambion的megashortscriptt7)转录为rna。
高通量rna测序和分析。对于illuminamiseq测序,使用逆转录酶和illumina扩增子引物对选择回合3、5、7和10的rna序列进行逆转录,该引物包含用于我们rna文库的接头连接和基因特异性引物序列。使用以下正向和反向引物:fp5'-tcgtcggcagcgtcagatgtgtataagagacagtagggaagagaaggacatatgat-3'(seqidno:7)和rp5'-gtctcgtgggctcggagatgtgtataagagacagtcagtggtcatg-tactagtcaa-3'(seqidno:8)。以有限数量的pcr循环扩增所得产物,以避免文库过度扩增和潜在的偏差。然后对pcr产物(每个序列包含用于与芯片退火的接头连接)进行测序。如对mda-mb-231细胞所做的那样,在修剪接头序列和pcr引物位点并消除错误长度的读段后,使用“fastaptamer”分析新生成的数据。获得了唯一读段(uniqueread)数量的减少和在每个选择回合中显示多重读段(muitpleread)的序列数量的增加,强烈表明了外泌体特异性序列的富集(图5a)。
评估外泌体中exo-code的富集并确定富集动力学。如图4中对于前导exo-code所示,可以评估图5b的exo-code和本文所述的其它exo-code的外泌体富集。简单地说,使用改良茎环rt-pcr方案确定新鉴定的exo-code的富集。首先,先将茎环rt引物与细胞中存在的rna物质的3’部分杂交,然后对外泌体裂解物进行逆转录(appliedbiosystems)。rt产物将通过taqman实时pcr进行定量。此外,对所有鉴定的exo-code的外泌体分选动力学进行定量。将exo-code转染到相关细胞系中,并在施用后6小时、12小时、18小时、24小时、48小时、60小时和72小时收集外泌体。通过rt-pcr定量外泌体exo-code,并计算分选动力学以确定序列的结构-活性关系。
竞争性分选测定。在一种方法中,对每种细胞系的五个最高富集的exo-code进行竞争性外泌体分选测定。在将所有五个序列电穿孔到每个细胞系中之后,进行分析以确定多个序列是否可以同时分选至外泌体,以及是否任何序列显示出优先和/或更快地分选至外泌体。
本公开内容包括被认为是系统地编译和分析关于外泌体富集的mirna的公开数据以鉴定负责主动分选的保守基序的首次尝试。为了鉴定天然存在序列中的外泌体富集基序,我们对来自报道细胞和外泌体中mirna的相对丰度的出版物的mirna数据进行了回顾性计算机分析。仅选择与全细胞裂解物相比在外泌体中富集的mirna进行进一步检查。这得到总共253个mirna序列。利用零阶马尔可夫背景模型(zero-ordermarkovbackgroundmodel)和每序列基序数的零或无的假设以及限制在4至12个核苷酸的基序长度,使用meme基序查找器套件分析序列。这得到了3个统计学显著性基序(图7)。整体基序a的e值为1.0e-9,基序b为4.0e-5,c值为2.4e-4。
显示显著的外泌体富集的exo-code。本公开内容包括但不限于从实验和计算机分析两者生成的exo-code序列(图5、6和7)。可以建立这些序列的结构-活性关系及其介导分选的能力。
不希望受任何特定理论的约束,认为exo-code的外泌体分选受细胞蛋白质及其表达水平的相互作用的调节。本公开内容的exo-code可以考虑这样的相互作用。特别是,我们使用rna结合蛋白图(rna-bindingproteinsmap,rbpmap)和sybyl计算机对接的分析表明,上文鉴定的一种exo-code序列可能与hnrnpl和g3bp1结合,这两者均存在于mda-mb-231细胞的外泌体中。hnrnp(异质核核糖核蛋白)蛋白是重要的rna结合蛋白家族,长期以来就知道其是rna生物发生和表达、细胞质输出和mrna翻译中的关键角色。数据表明,它们在mirna调节和衰变中具有更广泛的功能。具体地,已显示hnrnpl调节多种癌细胞的致瘤能力,并且因此,其本身可能是致癌的。令人惊讶的是,hnrnpl是存在于癌症外泌体中的蛋白质。基于计算机预测,本文所述的我们的exo-code序列内的基序“ccccaca”表现出低纳摩尔亲和力,而基序“cacagca”可在高纳摩尔范围内结合。使用hnrnpl的模型蛋白质-rna晶体结构(rcsbpdbid:2mqo)。使用原生rna物质(cacaca)产生protomol对接区,然后进行突变以解决任何序列变异。当求解20个姿态(pose)并将最佳姿态选择为最高“总评分”且“共有序列评分”为至少4时,获得了这8个rna的结合的排名顺序。归一化的计算机对接得分与从已知参考文献中提取的实验确定的结合亲和力相关。如图8b所示,这显示出强的对数-对数关系(r2=0.82)。如图8a所示,“caca”基序中的核苷酸突变降低了与hnrnpl的结合亲和力。图8a是计算机演示,并不意味着限制。特别是,图8(a&b)显示了归一化的计算机对接评分,其显示与实验确定的rna序列与hnrnpl的结合亲和力(圆圈)显示出高度相关性(r2=0.82)。exo-code中的序列与hnrnpl结合,预测的结合亲和力为3.23nm(ccccaca)和223.2nm(cacagca)。(c)与hnrnpl的rna识别基序(rrm)结合的ccccaca(白色)的表面渲染图。(d)计算的静电表面电势显示ccccaca与hnrnpl的相互作用(显示阴离子和阳离子电荷)。(e)通过计算机对接预测的与hnrnpl的主要结合基序是“caca”。如在我们的基序(cccccaca)中可以发现,以“ccc”开头的“caca”似乎没有引起空间位阻,因为计算机对接评分仍在低纳摩尔范围内对基序排名。cccccaca与hnrnpl的结合由分子间氢键(虚线)介导。该结合通过c4和arg102之间的静电相互作用以及a5和tyr168之间的堆叠相互作用进一步稳定。该数据与hnrnpl和ca重复序列(例如,cacaca)或通过间隔分隔的重复序列(例如canrca和canrca)的结合是一致的。具体而言,如图8e所示,本文所述的一种exo-code中的“ccccaca”与hnrnpl的结合是通过分子间氢键介导的,如黄色虚线所示(a5/prol90,c6/tyr168/thr170/ser169,a7/ser175)。通过c4磷酸基团和arg102之间的静电相互作用以及a5和tyr168之间的堆叠相互作用,可以进一步稳定结合。此外,根据其序列,可以预测前导exo-codeugugugccccacagcagug(seqidno:9)以通过cccacagcag(seqidno:10)与g3bp1结合。与hnrnpl相似,g3bp1(gtpase激活蛋白(sh3结构域)结合蛋白1)属于rna结合蛋白家族。g3bp1在mda-mb-231和其它乳腺癌细胞系的增殖中起重要作用。
作为对计算机预测的补充,本公开内容包括使用沉降测定法和质谱法对新的exo-code结合蛋白进行的实验室确定。一旦鉴定了结合的蛋白,就进行计算机对接分析,以预测和分析与已鉴定的蛋白质结合的rna基序,并阐明exo-code及其蛋白伴侣的结构-活性关系。我们已经成功地表明,我们的计算机方法可以正确预测rna基序与hnrnpl的排名顺序的结合亲和力(图8)。使用sybyl生成的归一化计算机“对接评分”(结合亲和力的测量量)显示与实验确定的结合亲和力强相关性。
表1.
表1:在mda-mb-231细胞、间充质干细胞、raw264.7细胞、hek293细胞的选择回合7或10时通过读段计数的前10个序列(从高至低)
图9描述了电穿孔进入其亲本细胞系(mda-mb-231细胞、间充质干细胞、raw264.7细胞和hek293细胞)后,exo-code的相对外泌体富集。以下序列用作对照序列:随机20n:acaguagagccuucaccggc(seqidno:51),mda混杂20n:gcucucggaaggcuugggcu(seqidno:52),raw混杂20n:agucgggguaugccuggaug(seqidno:53),文献exo-motifvb1:uggucuaggauuguu-ggaggag(seqidno:54),文献exo-motifvb2:gguccagaggggagauagguuc(seqidno:55)[来自villarroya-beltri等人natcommun.2013;4:2980.doi:10.1038/ncomms3980],和文献exo-motifb:acccugccgccuggacuccgccugu(seqidno:56)[bolukbasi等人molthernucleicacids.2012feb7;1:e10.doi:10.1038/mtna.2011.2.]。在100μl吸头中使用每个细胞系的优化的neon设置,将10μg的每种rna电穿孔到以下中:对于mda的6e5细胞;对于raw的1.5e6细胞;对于hek的1e6细胞;和对于msc的3e5胞。将细胞分配到dmem中并洗涤3次。将每种rna的细胞悬液分到12孔板的3个孔中,并用exo培养基补充至500μl。将细胞培养72小时至接近汇合,然后收集外泌体/ev。将外泌体/ev重悬于100μlpbs中,将1μl用于外泌体/ev定量,而其余样品用rnase处理,然后用qiazol处理以用于rna提取。将30ng秀丽隐杆线虫(c.elegans)mirna39(ucaccggguguaaaucagcuug(seqidno:57))和54(uacccguaa-ucuucauaauccgag(seqidno:58))在该步骤中导入(spike)到每个样品中。将总rna在mirneasy柱上纯化,并使用来自quanta的qscript微小rnacdna合成试剂盒合成cdna。使用来自quanta的sybrlowrox在pcr反应中对rna进行定量。从每个rna的标准曲线获得rna拷贝,并校正来自两个导入的rna的cdna输入量和平均回收率。通过每个样品回收的外泌体/ev的量将该数字归一化。b)将随机20n设置为1证明,对于最佳性能的exo-code,每个ev的15-400相对rna拷贝的富集倍数。c)将来自相同样品的一组定量内源mirna设置为1证明,使用exo-code技术显著提高了rna效能。与内源rna相比,exo-code在外泌体/ev中富集1e5至3e6倍。
图10a描绘了使用在mda-mb-231细胞的回合10中鉴定的前两个序列的rna沉降测定法。泳道4和5:ucguaggucaggaagcggcg(seqidno:59),泳道6和7:gcgauaggcggucguugguc(seqidno:60)。泳道4-7中箭头所示的蛋白质被鉴定为hnrnpm。图10b:显示了与nono(不含pou结构域的八聚体结合蛋白,non-poudomain-containingoctamer-bindingprotein)和sfpq(剪接因子,富含脯氨酸和谷氨酰胺)rna结合蛋白结合的对照rna和m8exo-coderna。但是,在沉降测定中,m8exo-code与hnrnpm的结合比对照rna结合多得多(用箭头指示)。在图10c中,在raw细胞(r2)中最富集的exo-code的沉降测定中,与对照rna相比,生物素化的rna与多种蛋白质的更多地结合。质谱鉴定出最大差异结合的蛋白质是鼠ascc3、dock2、ddx17、ddx5、hnrnpf和hnrnph1/h2。
图10a的rna沉降测定显示了对于在mda细胞的回合10中鉴定的前两个序列(泳道4-7)存在70和100kda的两个条带(由箭头指示),其不存在于混杂对照序列沉降(泳道8-9)中。从泳道6和7中切下两个条带,并通过质谱分析对泳道8和9中的相应对照区域进行分析。两条带被鉴定为异质核核糖核蛋白m(hnrnpm),预测kda72/74。泳道4、6和8与磁性链霉亲和素珠粒一起加载到孔中,而泳道5、7和9与相同沉降的剩余裂解物在没有磁性链霉亲和素珠粒的情况下加载。在图10b和c中,示出了用mda回合10的#8序列和raw264.7细胞回合10的#2序列进行的沉降实验的结果。为了获得图10中的结果,对生物素化的rna序列进行了3’-生物素修饰。将20ugrna与mda细胞的预澄清化细胞裂解物(来自3x107细胞的1ml1mg/ml细胞裂解物)孵育1小时。然后使用promega磁性链霉亲和素珠粒捕获结合蛋白(孵育45分钟),并通过5次洗涤去除未结合的蛋白质。所有步骤均使用蛋白酶和rnase处理的缓冲液在4℃进行。在先前的研究中已知hnrnpm存在于来自mda细胞的外泌体中,并且显示在mda-mb-231细胞的转移表型中是重要的。还显示hnrnpm对于乳腺癌细胞在体内有效转移至肺是必需的(xuyet.genesdev.2014jun1;28(11):1191-203)。
可以使用沉降测定法和质谱法。将涂有(a)生物素化的exo-code或(b)作为阴性对照的生物素化的聚(a)rna序列的磁性链霉亲和素珠粒与来自mda-mb-231、raw264.7细胞和其它细胞类型的细胞和外泌体提取物一起孵育。使用高灵敏度和全面的基于液相色谱-质谱(lc-ms)的蛋白质组学方法,分析与exo-code结合的蛋白质。将3’-生物素化的exo-code加热至65℃,然后经过30分钟冷却至4℃,以确保正确折叠。然后将3’-生物素化的rna加入到外泌体裂解物或细胞裂解物中,并在4℃旋转孵育1小时。将酪蛋白封闭的磁性链霉亲和素珠粒添加到混合物中,并孵育30分钟。通过磁性分离洗涤珠粒。最后一次洗涤后将珠粒重悬于水中,并制备在降低lds的缓冲液中,然后在nupage4-12%bis/tris凝胶上跑胶。必要时,将凝胶用coomassie(biorad)或silverquest(invitrogen)染色。切下exo-coderna特有的但不存在于对照rna上的蛋白质条带,并在qexactiveplus上通过质谱进行分析。使用mascot搜索引擎swissprotfasta人类数据库在proteomediscoverer1.4中分析数据。胰蛋白酶用作酶,同时允许1次错位切割。氧化的甲硫氨酸设置为可变修饰,而脲甲基(carbamidomethyl)设置为静态修饰。
最小序列要求和结合亲和力。将计算机对接信息点突变(insilicodocking-informedpointmutation)引入鉴定的exo-code序列中,以阐明最低序列要求。使用sybyl平台上的surflex对接工具包,我们能够根据其与hnrnpl的结合亲和力对rna序列及其突变进行排名。该方法可以扩展到本文中发现或描述的任何其它exo-code,以及从沉降质谱实验中鉴定出从头合成的exo-code结合蛋白靶标。
蛋白-核酸相互作用。将鉴定的蛋白质从大肠杆菌中过表达和纯化,并使用纯化的蛋白质、我们的新exo-code和对照聚(a)rna序列进行电泳迁移率测定。使用放射性标记的rna底物进行实验。将32p-放射性标记的t7转录的rna底物与递增浓度的鉴定的蛋白质一起孵育,然后在4.5%天然聚丙烯酰胺凝胶上进行解析。将凝胶用phosphorimagersi(amersham)成像。蛋白质结合的rna比游离rna迁移得慢。
表面等离振子共振可用于确定exo-code及其突变与鉴定的蛋白质的结合亲和力。用reichertsr7000dcspr仪器进行结合分析。使用标准胺偶联化学方法将鉴定的蛋白质固定在羧甲基葡聚糖水凝胶金芯片的表面上。使含有rna序列的样品流过芯片,以确定结合亲和力。使用10mm甘氨酸溶液ph2除去结合的分析物来进行金芯片的再生。结合亲和力数据与计算机预测以及exo-code的分选效率和动力学相关。
功能丧失研究:鉴定的蛋白质的沉默和过表达。可通过mda-mb-231、pc-3、raw264.7、hek293和间充质干细胞中鉴定的候选物的基因沉默和过表达来进行功能丧失研究以评估各蛋白质对exo-code分选的影响。可通过细胞和外泌体样品的rt-pcr(在mrna水平上)和western印迹(在蛋白质水平上)来确认蛋白质的沉默和过表达。通过rt-pcr监测外泌体中的rna富集。
在图11a)中,使用rnaimax在12孔板中用对照sirna(有义:ucacaagggagagaaagagaggaagga(seqidno:61))或两种hnrnpmsirna(有义1:gcauaggauuuggaauaaa(seqidno:62))有义2:ggaauggaaggcauaggauuu(seqidno:63))以30pmol的总sirna处理mda-mb-231细胞。a)在转染后48小时收集来自每种条件的3个孔,并进行western印记以评估hnrnpm敲低(origeneta301557)。b)使用imagej软件评估归一化至gapdh的敲低百分比。在sirna处理后48小时,用1μgexo-codem1rna或m8对来自每种条件的另外3个孔进行电穿孔。在电穿孔后24小时,收集外泌体/ev并提取rna。由qpcr确定相对m1和m8丰度。耗尽约75%的hnrnpm的细胞在装载m8exo-code方面表现出约60%降低,并且在将m1exo-code装载到外泌体/ev方面表现出约90%降低。hnrnpm敲低不影响随机对照(rc)序列的分选。
本公开内容包括评估exo-code将治疗性运载物递送至外泌体的能力。我们已经鉴定出在外泌体中富集的exo-code。预期这些exo编码用于递送治疗性运载物以对病理性外泌体重编程并发挥表型作用。可以确定rna尺寸-效率关系和rna尺寸-动力学关系。可以在迁移和侵袭测定中评估exo-code对肿瘤来源的外泌体重编程的能力。
在本公开内容的实施例的一个非限制性说明中,我们将pre-mir-31附接至上文鉴定的exo-code,以对来自mda-mb-231细胞的外泌体进行重编程。mir-31是充当肿瘤抑制因子的抗转移性mirna,并抑制癌细胞的增殖、迁移和转移。
图12(a)提供了exo-code/pre-mir-31嵌合体的示意图。(12b)用嵌合exo-code/pre-mir-31重编程的外泌体使细胞迁移抑制75%。相比之下,来自用ns-ctrl/pre-mir-31嵌合体处理细胞的外泌体仅显示对mda-mb-231细胞迁移的31%抑制。与仅pre-mir-31ctrl相比,抑制不是统计学显著的。(12c)划痕测定的代表性图像,其显示当细胞与来源于ns-ctrl/pre-mir-31和pre-mir-31ctrl处理的细胞的外泌体一起孵育时,间隙完全闭合。对于用经exo-code/pre-mir-31重编程的外泌体处理的mda-mb-231细胞,24小时后大量间隙依然可见。比例尺:200μm。数据为平均值±sd。通过单因素anova和事后tukey,**p<0.01,*p<0.05。
对于外泌体重编程,将mda-mb-231细胞用exo-code/pre-mir-31嵌合体、仅pre-mir-31和非特异性ctrl/pre-mir-31嵌合体进行电穿孔,并在耗尽外泌体的培养基中培养48小时。为了获得如图9a所示的exo-code/pre-mir-31嵌合体,用接头区域合成exo-code,产生以下序列:5’guacauucuagauagccaugau-uguguguccccacagcacuguug-3’(seqidno:64)。该接头区域与连接至pre-mir-31的互补接头退火:5’ggcuaucuagaauguacaaaggcaagagcuggcauagcuguugaacugggaacugcuaugcca-acaua-uugccau-3’(seqidno:65)。斜体字表示互补的接头区域,黑体字表示exo-code,成熟的mir-31用下划线标出。收集培养基,并使用总外泌体分离试剂(invitrogen)沉淀外泌体。将外泌体重悬于pbs中,并施用于铺板的mda-mb-231细胞,以进行体外划痕测定,这是评估细胞迁移和细胞侵袭的良好建立的方法[64]。简单地说,为了获得无细胞间隙,使用移液器吸头在孔的中心划痕。间隙封闭用作细胞迁移的标志。来自用exo-code/pre-mir-31处理的细胞的重编程外泌体将细胞迁移抑制75%(图9b和c)。相比之下,来自ns-ctrl/pre-mir-31嵌合体处理的细胞的外泌体显示使mda-mb-231细胞的迁移抑制仅31%。
递送不同尺寸的运载物和确定外泌体装载能力。为了优化运载物装载,评估了exo-code装载不同尺寸运载物的能力。通过rt-pcr监测mirna、sirna、dna和更大的核酸(例如mrna或质粒dna)向外泌体的装载。可以确定rna尺寸和装载效率以及rna尺寸和装载动力学之间的关系。用sirna或mirna稳定转染细胞以间接装载外泌体的被动策略在文献中很普遍。然而,这些方法非常低效,需要大量的rna,并且不适合直接体内重编程以及装载较大的核酸,例如mrna和pdna,尤其是使用常规方法的挑战(装载效率约0.3%)。认为mrna通过序列特异性分选装载到外泌体中。通过将exo-code附接于mrna,本公开内容模仿了内源分选策略以有效地将mrna装载至外泌体中。绿色荧光蛋白(gfp)可用作mrna递送的模型蛋白。使用我们的脂质、聚合物或肽通过(a)电穿孔和(b)通过脂质体转染,将附接至exo-code的mirna/sirna和mrna递送至mda-mb-231、间充质干细胞和其它细胞。可以通过茎环rt-pcr(对于mirna/sirna)和rt-pcr(对mrna)对分选至外泌体的mirna/sirna和mrna的量进行定量。
将能够介导组织再生的运载物装载到外泌体中。在图13中,显示了将mirna装载到来源于人间充质干细胞(msc)的外泌体中的过程。与仅将mir-132电穿孔入人msc相比,exo-code-msc6介导mir-132向外泌体的显著更高的富集。为了测试装载的外泌体对血管生成的影响,将人脐静脉内皮细胞(huvec)用来源于仅用mir-132或exo-code-msc6/mir-132嵌合体电穿孔的细胞的外泌体进行处理,然后铺在涂有geltrex的96孔板中。17小时后,将细胞用钙黄绿素染色并用spectramaxminimax300成像细胞仪在em/ex495/515成像。与未经修饰的外泌体和来源于仅用mir-132电穿孔的msc的外泌体相比,含有exo-code-msc6-mir-132的外泌体介导显著更高的对血管生成的影响。血管生成测定法通常用作替代测定法以评估新血管的形成,这与涉及伤口愈合和组织再生的过程有关。
图13显示了获自进入人间充质干细胞外泌体的mirna装载和对血管生成的影响的数据。图13:(a)将7个选择回合后来自干细胞的6号序列exo-codes6添加到用“a”接头间隔开的mir-132的互补序列,以形成顶链rna:gcuggucaggaugcgggggcaaaguaacaaucgaaagccacgguuuu(seqidno:66);然后将该rna与合成的mir-132:aaccguggcuuucgauuguuacuu(seqidno:67)双链体化。(b)用等摩尔量的单独的合成mir-132或附着于s6exo-code的mir-132对间充质干细胞进行电穿孔,一式三份。收集外泌体并定量mir-132,电穿孔的mir-132使间充质干细胞外泌体中此mirna的量增加了10倍。利用exo-codes6可以使外泌体丰度再增加6倍,从而使msc外泌体的mir-132效力提高60倍。(c)人脐静脉内皮细胞(huvec)的血管生成测定。用从用exo-code-msc6/mir-132或未经修饰的mir-132电穿孔的人间充质干细胞收集的外泌体处理huvec。在所有显示的参数中,来自用exo-code-msc6/mir-132处理的间充质干细胞的外泌体在节点数、连接数、总长度、总分支长度和分支数方面显示出统计学上的显著增加。用单因素anova和事后tukey分析数据(n=3),****p<0.0001,***p<0.001,**p<0.01,*p<0.5。
可以评估多价对外泌体分选的动力学和效率的影响。exo-code可以以不同的化合价(n=1-3)附接至运载物(mirna和mrna)。在这方面,图14提供了示出对exo-code将不同大小的运载物递送到外泌体的能力的评估的示意图。可以通过(a)电穿孔和(b)通过脂质体、聚合物和肽转染以及通过其它合适的方法将包含多价exo-code的mirna/sirna和mrna递送至mda-mb-231、pc-3和其它细胞。可以通过茎环rt-pcr(对于mirna/sirna)和rt-pcr(对于mrna)对分选至外泌体的mirna/sirna和mrna的量进行定量。
转移抑制性sirna/mirna的递送。为了对病理性转移起始外泌体进行重编程,在某些实施例中,靶向snaii和slug。snaii和slug在多种癌症类型的肿瘤生长和远传转移的诱导中起主要作用。它们属于参与癌细胞的‘上皮间质转化’(epithelialtomesenchymaltransition,emt)的锌指转录因子家族,emt在肿瘤转移中起重要作用,是上皮细胞获得具有增强的迁移能力和显著更高侵袭性的间质表型的机制。已经显示snail和slug的沉默显著降低乳腺癌和前列腺癌细胞的肿瘤生长和淋巴结转移。本公开内容包括递送嵌合的exo-code/针对snaili和slug的sirna作为癌细胞中外泌体重编程的靶标。为了获得嵌合的exo-code/sirna构建体,将有义链与exo-code共价连接,并且反义链与有义链退火。将使用以下sirna序列:snail:5-ccacagaaauggccaugggaaggccuc-3(seqidno:68),slug:5-uccgaauaugcaucuucagggcgccca-3(seqidno:69),阴性对照5-ucacaagggagagaaagagaggaagga-3(seqidno:70)。
使用体外划痕测定的迁移测定。exo-code可与已知充当细胞迁移、侵袭和转移的抑制剂的抗转移mirna(mir-31)、抗snail和抗slugsirna嵌合。预期这些exo-code/mirna和exo-code/sirna能够递送至肿瘤细胞用于外泌体重编程。可以使用划痕测定来测定重编程外泌体抑制细胞迁移的能力(图12)。在孵育后24小时和48小时,通过显微镜监测接受重编程外泌体的mda-mb-231、pc-3细胞和其它癌细胞的迁移。使用imagej软件对划痕区域和已迁移到划痕区域的细胞数量进行定量。可通过相应mrna的rt-pcr证实重编程外泌体对细胞迁移的影响,以排除非特异性抑制机制。
侵袭测定。改良的基质胶涂覆的博伊登室测定法可用于评估用重编程外泌体处理的mda-mb-231细胞、pc-3细胞和其它癌细胞的侵袭性。将细胞铺板在transwell的顶端部位,并用重编程外泌体处理。24和48小时后,对迁移到基底侧的细胞数量进行计数。对最小细胞数为10的每个膜的10个视野进行计数。
剂量-响应关系。为了建立剂量响应关系,可以用递增浓度的重编程外泌体处理细胞,并确定其对细胞迁移和侵袭的影响。此外,可以确定重编程外泌体进入mda-mb-231细胞、pc-3细胞和hek293、间充质干细胞、raw细胞、心肌细胞、内皮细胞和其它细胞类型的细胞摄取。外泌体可以用pkh26膜染料(sigmaaldrich)进行荧光标记。通过流式细胞仪对摄取到细胞中的外泌体的量进行定量。
为了产生化学修饰的rna以用于增加exo-code稳定性,在体外反应中使用甲基ctp和假utp将dna模板翻译成rna。在两个实例中(图15),相对于随机对照和/或未经修饰的exo-code,经化学修饰的exo-code显示出显著增加的向外泌体的富集。
图15证明了使用经修饰的核苷酸,干细胞和mda细胞中snailsirna的装载增加。特别地,合成了在snailsirna反义序列的5’上游的用3-腺苷接头隔开的exo-codes6、随机对照、经化学修饰的exo-codemod-s6、exo-codem1、具有经修饰的核苷酸(甲基胞苷和假尿苷,以赋予增强的稳定性)的经化学修饰的exo-codemod-m1。然后snailsirna有义链通过碱基配对缀合,并将这些构建体电穿孔到mda-mb-231细胞和间充质干细胞(n=3-4)中。24小时后,收集外泌体,并根据mirneasy方案(qiagen)提取rna。使用mirscriptcdna合成试剂盒产生cdna,并且使用针对snail有义链的sybrlowrox(quanta)进行qpcr。通过δct方法+平均值的标准误差,将数据绘制为相对于随机对照的装载的倍数改善。使用anova和事后tukey评估装载的倍数改善的统计学显著性(*=p<0.05,**=p<0.001,***=p<0.0001)。在mda-mb-231细胞中,经化学修饰的exo-codemod-m1显示出相对于随机对照约118倍的改善,而未经修饰的exo-codem1显示出相对于随机对照约8倍的改善。在msc中,经化学修饰的exo-codemod-s6显示出相对于随机对照约21倍的改善,而未经修饰的exo-codes6显示出相对于随机对照约9倍的改善。
对于图12,将供体mda细胞用exo-code-pre-mirna嵌合体(n=3)、exo-code和仅pre-mirna的对照(n=3,n=2)以及无rna的生长对照(n=5)进行电穿孔。然后将来自这些细胞的外泌体铺在玻璃底96孔板上的已经做出划痕的受体mda细胞上。在接种用于做出划痕的具有外泌体的细胞之前6至10小时的伤口闭合已经相对于无rna的生长对照进行归一化。通过编程的蔡司显微镜采集获得伤口面积,并使用axiovision软件进行定量。用anova事后tukey进行处理组的比较,在0.01的α下用富集序列(e-31)靶向mir-31比未靶向的mir-31更好,仅mir-31vse-31的p值=0.0011。在0.05的α下混杂对照(s-31)在统计学上不同于s-31和“仅mir-31”,分别p=0.0308和0.0352。使用anova和事后tukey,在生长对照和“仅mir-31”对照之间以及在仅富集或混杂序列适配体对照之间,没有统计学显著差异。为了提高exo-code的稳定性,在体外反应中使用甲基ctp和假utp将dna模板翻译为rna。
可在本公开内容的实施例中单独使用或以任何组合使用的代表性和非限制性exo-code序列以及相关信息显示在图16中,其提供了基序和共有序列分析。特别地,图16在a)中显示:分析从最新选择回合到meme基序算法前1,000个序列,证明了高度统计学显著性基序(显示的最低e值基序)。来自raw和msc细胞的最高显著性基序显示出高度相似性。16b)来自pc-3和raw细胞的较低统计学显著性基序显示出高度相似性。
表2:最终选择回合后前10个的共同序列
表2:在测试的前十个exo-code序列中,有两个细胞系中存在6个。1)exo-coderaw#4和msc#9,2)exo-coderaw#6和msc#8,3)exo-codehek#4和raw#8,4)exo-codemsc#1和raw#2,5)exo-codemsc#2和msc#5,6)exo-codemsc#1和msc#3。
图17:外泌体/ev表征。特别地,图17显示:外泌体/ev表征研究a)外泌体/ev群的纳米颗粒追踪分析(nta)。b)透射电子显微镜(tem)。nta和tem显示,大多数外泌体/ev落在30-150nm的预期尺寸范围内。c)nta颗粒计数也与蛋白质含量相关,作为囊泡计数的次要手段。
本公开内容提供的代表性和非限制性外泌体序列包括但不限于以下:
rawrd5前10序列
rawrd7前10序列
rawrd10前10序列
mda-mb-231细胞,rd5前10序列
回合5
mda-mb-231细胞,rd7前10序列
mda-mb-231细胞,rd10前10序列
pc-3细胞,回合5,前10序列
pc-3细胞,回合7,前10序列
pc-3细胞,回合10,前10序列
间充质干细胞,回合5,前10序列
间充质干细胞,回合7,前10序列
hek,回合5,前10序列
hek,回合7,前10序列
图18、19和20包括由通过图3中概述的方法鉴定和筛选的序列预测的exo-code的另外的和非限制性的实施例。特别是,为了鉴定保守的基序,我们在3、5和7个选择回合之后分析了序列,显示了高于各回合平均序列读段的16个标准差。合并这些序列,并使用meme套件进行分析。搜索条件设置为包括可以在序列中以任意数量的重复出现的至多12个核苷酸的基序。mda-mb-231细胞有8个具有高统计学显著性的基序,间充质干细胞有10个具有高统计学显著性的基序,和raw264.7细胞有8个具有高统计学显著性的基序。将具有相似序列元件的基序在视觉上进行比对和分组;箭头指示细胞系之间的相似基序,如图18所示。meme将基序表示为位置依赖性字母概率矩阵。每个序列标识/基序由核苷酸符号堆叠组成,其中一个堆叠代表序列中的每个位置。堆叠的总高度指示在该位置处的序列保守性,而堆叠中核苷酸的高度指示在该位置处的各个核苷酸的相对频率。另外,示出了各个基序的iupac核苷酸代码。
图19显示了msc和mda-mb-231细胞的其它序列。具体地,在由三个腺嘌呤分隔的连续链中合成计算机方法鉴定的基序和snailsirna的反义链。将这些构建体与cy5标记的snail有义链rna退火,并一式三份以每120,000msc或300,000mda-mb-231细胞5ug的有义荧光报告基因sirna进行电穿孔。将细胞在无外泌体的培养基中培养24小时,并通过总外泌体分离试剂法分离外泌体/ev。然后将外泌体/ev重悬在pbs中,并在4℃下与cd63珠粒结合过夜。洗涤与外泌体/ev结合的珠粒,并通过流式细胞术进行分析。绘制了平均荧光强度相对于背景的变化倍数。背景定义为与cy5标记的snail结合的随机对照减去没有将cy5标记的snail电穿孔到繁殖细胞中的外泌体结合珠粒。多个计算机预测的基序显示出比对照显著更高的富集。通过单因素anova然后dunnett事后检验进行统计分析。
图20呈现了用mcherry转录物的3’-utr中的m8exo-codemcherry(包含序列aucuugugguc(seqidno:207))获得的数据。m8-exo-codemcherry(包含mcherry转录物的3’-utr中的序列aucuugugguc)进入mda-mb-231细胞外泌体的装载能力是对照mcherryrna的约7倍。使用neon电穿孔仪在三个剂量水平0.1ug、1ug和10ug用mrna构建体对500,000个mda细胞进行电穿孔,一式三份。将细胞在dmem中洗涤3次,并铺在dmem+10%外泌体耗尽的fbs中。将细胞培养24小时,并使用来自cellmedia(thermofisher)的总外泌体分离试剂分离外泌体。将外泌体以13,000rpm沉淀1小时。完全除去上清液,并将外泌体重悬于无菌pbs(20ul)中。将外泌体样品在37℃下用1ulrnasea/t1处理1小时。用500ulqiazol裂解外泌体样品,并根据qiagenmirneasy方案提取rna,并在20ul无rnase的水中洗脱rna。使用nebprotoscriptii试剂盒,使用6ul外泌体rna的最大输入产生来自每个样品的cdna。使用quantasybrmastermix(fp:cctgtcccctcagttcatgt;rp:cccatggtcttcttctgcat)通过qpcr对mcherry进行定量。与未经修饰的mcherrymrna相比,m8exo-codemcherry(包含序列aucuugugguc(seqidno:207))将mcherrymrna装载到mda-mb-231细胞外泌体的能力提高了约7倍。
实施例2
为了评估特定核苷酸对已鉴定的exo-code基序的分选潜力的重要性,合成了包含单个突变的突变体构建体。在每种情况下,c突变为g并且反之亦然,a突变为u并且反之亦然,只有共有序列突变体4除外,其中c突变为a,因为在基序中此位置更通常是c、g或u而不是a。合成了在snailsirna序列的5’上游的用4个a核苷酸间隔开的基序和基序杂交体。对于杂交体序列(5’-aaguggcgug-3’)(seqidno:208),基序与反义snail序列连续,并缀合至apc荧光标记的有义链。对于m8(5’-aucuugugguc-3’)(seqidno:207)和s6(5’-augcgggggc-3’)(seqidno:209)基序二者,交换了该方向,以便活性反义链是被缀合的荧光报告分子。将mda或msc用每种构建体一式三份电穿孔,并在铺板前洗涤5次。24小时后收集细胞培养基,分离外泌体并重悬于200ulpbs中。通过用50ulpbs中的10%triton裂解100ul外泌体并使用spectramax读板仪在633/660nm处进行分析来获得总荧光信号。按照制造商对于外泌体分析的设置,还使用macsquant流式细胞仪获得每个外泌体的平均apc信号。
对于杂交体序列(aaguggcgug)(seqidno:208),基序与反义snail序列连续,并且与apc荧光标记的有义链退火。snailsirna的序列如下:apc-snail(反义=guggcuucggaugugcaucuu(seqidno:210),有义=gaugcacauccgaagccacuu(seqidno:211))。对于该实验,将mda-mb-231细胞用构建体电穿孔并洗涤3次以除去未引入的rna。将细胞培养24小时,然后收集培养基并以2,000g离心30分钟。将培养基上清液与0.5体积的来自cellmedia(thermofisher)的总外泌体分离试剂合并,并在4℃下保持过夜。将外泌体以13,000rpm沉淀1小时。完全除去上清液,并将外泌体重悬于无菌pbs(200ul)中。通过用50ulpbs中的10%triton裂解100ul外泌体并在spectramax读板仪上以633/660nm读取来获得总荧光信号。还根据制造商对外泌体的说明在macsquant流式细胞仪上分析了50ul的每个外泌体样品。
如图中所示,每个突变导致snailsirna进入外泌体的装载减少。分选的降低是显著的,与杂交体序列相比,hmut1的p<0.001,与杂交基序相比,hmut2和hmut4的p<0.05。
对于m8基序(5’aucuugugguc)(seqidno:207)、m8全长(full)和包含突变的各序列,exo-code序列是具有通过四个a核苷酸间隔区连接的有义snailsirna的连续链。将荧光标记的snail反义与exo-code/snail有义链rna退火。snailsirna的序列如下:apc-snail(反义=guggcuucggaugugcaucuu(seqidno:210),有义=gaugcacauccgaagccacuu(seqidno:211))。对于该实验,将mda-mb-231细胞用构建体电穿孔并洗涤3次以除去未引入的rna。将细胞培养24小时,然后收集培养基并以2,000g离心30分钟。将培养基上清液与0.5体积的来自cellmedia(thermofisher)的总外泌体分离试剂合并,并在4℃下保持过夜。将外泌体以13,000rpm沉淀1小时。完全除去上清液,并将外泌体重悬于无菌pbs(200ul)中。通过用50ulpbs中的10%triton裂解100ul外泌体并在spectramax读板仪上以633/660nm读取来获得总荧光信号。还根据制造商对外泌体的说明在macsquant流式细胞仪上分析了50ul的每个外泌体样品。
如图中所示,每个突变导致snailsirna进入来源于mda-mb-231细胞的外泌体的装载减少。分选的降低是显著的,与m8全长序列相比,m8基序、mut1、mut2、mut3、mut4、mut5、mut6、混杂序列的p<0.0001;与m8基序相比,mut1、mut2的p<0.001;与m8基序相比,mut3、mut4、mut5、混杂序列的p<0.0001;并且与m8基序相比,mut6的p<0.01。
对于s6(5’-augcgggggc-3’)(seqidno:209)和包含突变的各序列,exo-code序列是具有通过四个a核苷酸间隔区连接的有义snailsirna的连续链。s6基序是msc6序列的缩短基序:5’-gcuggucaggaugcgggggc-3’(seqidno:212)。将荧光标记的snail反义与exo-code/snail有义链rna退火。snailsirna的序列如下:apc-snail(反义=guggcuucggaugugcaucuu(seqidno:210)有义=gaugcacauccgaagccacuu(seqidno:211))。对于该实验,将间充质干细胞(msc)用构建物电穿孔并洗涤3次以除去未引入的rna。将细胞培养24小时,然后收集培养基并以2,000g离心30分钟。将培养基上清液与0.5体积的来自cellmedia(thermofisher)的总外泌体分离试剂合并,并在4℃下保持过夜。将外泌体以13,000rpm沉淀1小时。完全除去上清液,并将外泌体重悬于无菌pbs(200ul)中。通过用50ulpbs中的10%triton裂解100ul外泌体并在spectramax读板仪上以633/660nm读取来获得总荧光信号。还根据制造商对外泌体的说明在macsquant流式细胞仪上分析了50ul的每个外泌体样品。
如图所示,每个突变导致snailsirna进入来源于msc的外泌体的装载减少。分选的降低是统计学显著的,与s6-exo-code/sirna构建体相比,sirna(未经修饰的sirna)、混杂、s6、s-mut1、s-mut2、s-mut3、s-mut4、s-mut5、s-mut6、s-mut7和s-mut8的p<0.0001。
尽管已经通过具体实施例描述了本发明,但是常规修改对于本领域技术人员而言将是显而易见的,并且这样的修改旨在落入本发明的范围内。
序列表
<110>纽约州立大学研究基金会
<120>用于向细胞外囊泡装载化学和生物试剂/分子的组合物和方法
<130>011520.01328
<140>pct/us18/32226
<141>2018-05-11
<150>62/501,461
<151>2017-05-04
<150>62/631,060
<151>2018-02-15
<160>313
<170>patentinversion3.5
<210>1
<211>23
<212>rna
<213>人工序列
<220>
<223>排除的exo-code
<400>1
uagggaagagaaggacauaugau23
<210>2
<211>23
<212>rna
<213>人工序列
<220>
<223>排除的exo-code
<400>2
uugacuaguacaugaccacuuga23
<210>3
<211>25
<212>rna
<213>人工序列
<220>
<223>排除的exo-code
<400>3
acccugccgccuggacuccgccugu25
<210>4
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>4
acaguagagccuucaccggc20
<210>5
<211>44
<212>dna
<213>人工序列
<220>
<223>引物
<400>5
ttcaggtaatacgactcactatagggaagagaaggacatatgat44
<210>6
<211>23
<212>dna
<213>人工序列
<220>
<223>引物
<400>6
tcaagtggtcatgtactagtcaa23
<210>7
<211>56
<212>dna
<213>人工序列
<220>
<223>引物
<400>7
tcgtcggcagcgtcagatgtgtataagagacagtagggaagagaaggacatatgat56
<210>8
<211>56
<212>dna
<213>人工序列
<220>
<223>引物
<400>8
gtctcgtgggctcggagatgtgtataagagacagtcagtggtcatgtactagtcaa56
<210>9
<211>19
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>9
ugugugccccacagcagug19
<210>10
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>10
cccacagcag10
<210>11
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>11
ucguaggucaggaagcggcg20
<210>12
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>12
gcgauaggcggucguugguc20
<210>13
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>13
cccgcuucuuccucgggugg20
<210>14
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>14
gucacucggccuagugcguc20
<210>15
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>15
ccugaccuuacucggguggc20
<210>16
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>16
guggucgauaagcguacgcg20
<210>17
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>17
agggcguuugcucgcggguc20
<210>18
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>18
ccgggcguaucuuguggucg20
<210>19
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>19
cagacgguacucaggugugc20
<210>20
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>20
ggccuuggucgcgugcuggc20
<210>21
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>21
uggaacgucgaugugggccg20
<210>22
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>22
gguucagggugugggccgcc20
<210>23
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>23
ucguaggucaggaagcggcg20
<210>24
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>24
aaggccggugcuaguaguga20
<210>25
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>25
gcgauaggcggucguugguc20
<210>26
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>26
gcuggucaggaugcgggggc20
<210>27
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>27
gcugcgaaguggggcagguc20
<210>28
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>28
ggucgagugaugcggggcgc20
<210>29
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>29
cguggcugguucgugcgggg20
<210>30
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>30
gggcgaaauuggcauggccg20
<210>31
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>31
gaugguaaguuggaaagcga20
<210>32
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>32
uggaacgucgaugugggccg20
<210>33
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>33
gaugguauguuggaaagcga20
<210>34
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>34
cguggcugguucgugcgggg20
<210>35
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>35
uucguguaucuagugcaguc20
<210>36
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>36
ggucgagugaugcggggcgc20
<210>37
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>37
uuucguguauccuaguugcu20
<210>38
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>38
ugauguauuugguuugcaag20
<210>39
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>39
uucguguucugguugagugu20
<210>40
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>40
gcgcgggaaggguggcaugg20
<210>41
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>41
ugauguauugguaaguuucg20
<210>42
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>42
uauagaugugcuaguuugca20
<210>43
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>43
uuucguguuuagcguuugac20
<210>44
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>44
ugauguauuugguuugcaag20
<210>45
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>45
acguguauuacuauugacua20
<210>46
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>46
gaggccugugcuaguaguga20
<210>47
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>47
uuagcguuguauuaguugca20
<210>48
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>48
ucauugugcugaauugacua20
<210>49
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>49
aauuacacugugcuaggaug20
<210>50
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>50
aucgauguaugagucauaua20
<210>51
<211>20
<212>rna
<213>人工序列
<220>
<223>对照
<400>51
acaguagagccuucaccggc20
<210>52
<211>20
<212>rna
<213>人工序列
<220>
<223>对照
<400>52
gcucucggaaggcuugggcu20
<210>53
<211>20
<212>rna
<213>人工序列
<220>
<223>对照
<400>53
agucgggguaugccuggaug20
<210>54
<211>22
<212>rna
<213>人工序列
<220>
<223>对照
<400>54
uggucuaggauuguuggaggag22
<210>55
<211>22
<212>rna
<213>人工序列
<220>
<223>对照
<400>55
gguccagaggggagauagguuc22
<210>56
<211>25
<212>rna
<213>人工序列
<220>
<223>对照
<400>56
acccugccgccuggacuccgccugu25
<210>57
<211>22
<212>rna
<213>秀丽隐杆线虫(caenorhabditiselegans)
<400>57
ucaccggguguaaaucagcuug22
<210>58
<211>24
<212>rna
<213>秀丽隐杆线虫
<400>58
uacccguaaucuucauaauccgag24
<210>59
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>59
ucguaggucaggaagcggcg20
<210>60
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>60
gcgauaggcggucguugguc20
<210>61
<211>27
<212>rna
<213>人工序列
<220>
<223>对照sirna有义链
<400>61
ucacaagggagagaaagagaggaagga27
<210>62
<211>19
<212>rna
<213>人工序列
<220>
<223>hnrnpmsirna有义链1
<400>62
gcauaggauuuggaauaaa19
<210>63
<211>21
<212>rna
<213>人工序列
<220>
<223>hnrnpmsirna有义链2
<400>63
ggaauggaaggcauaggauuu21
<210>64
<211>45
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>64
guacauucuagauagccaugauuguguguccccacagcacuguug45
<210>65
<211>75
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>65
ggcuaucuagaauguacaaaggcaagagcuggcauagcuguugaacugggaacugcuaug60
ccaacauauugccau75
<210>66
<211>47
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>66
gcuggucaggaugcgggggcaaaguaacaaucgaaagccacgguuuu47
<210>67
<211>24
<212>rna
<213>人工序列
<220>
<223>合成mir-132
<400>67
aaccguggcuuucgauuguuacuu24
<210>68
<211>27
<212>rna
<213>人工序列
<220>
<223>snailsirna
<400>68
ccacagaaauggccaugggaaggccuc27
<210>69
<211>27
<212>rna
<213>人工序列
<220>
<223>slugsirna
<400>69
uccgaauaugcaucuucagggcgccca27
<210>70
<211>27
<212>rna
<213>人工序列
<220>
<223>阴性对照
<400>70
ucacaagggagagaaagagaggaagga27
<210>71
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>71
cguggcugguucgugcgggg20
<210>72
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>72
ggucgagugaugcggggcgc20
<210>73
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>73
ugauguauuugguuugcaag20
<210>74
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>74
uggaacgucgaugugggccg20
<210>75
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>75
gcgauaggcggucguugguc20
<210>76
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>76
ucguaggucaggaagcggcg20
<210>77
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>77
gaaugaucuugguugucaac20
<210>78
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>78
uggaacgucgaugugggccg20
<210>79
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>79
agcgagguggaguggcgugg20
<210>80
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>80
cguggcugguucgugcgggg20
<210>81
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>81
gcgcgggaaggguggcaugg20
<210>82
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>82
gaugguauguuggaaagcga20
<210>83
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>83
ggucgagugaugcggggcgc20
<210>84
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>84
gaugguaaguuggaaagcga20
<210>85
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>85
caguauuagcaagggagggg20
<210>86
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>86
gagggggaggagggcaugcc20
<210>87
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>87
uggaacgucgaugugggccg20
<210>88
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>88
ggucgagugaugcggggcgc20
<210>89
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>89
gaugguaaguuggaaagcga20
<210>90
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>90
cguggcugguucgugcgggg20
<210>91
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>91
gaugguauguuggaaagcga20
<210>92
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>92
gcuggucaggaugcgggggc20
<210>93
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>93
caaccuaggugcggggcggg20
<210>94
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>94
ggaagaucaaggugcggggc20
<210>95
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>95
gguucagggugugggccgcc20
<210>96
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>96
gcgcgggaaggguggcaugg20
<210>97
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>97
gaugguaaguuggaaagcga20
<210>98
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>98
uggaacgucgaugugggccg20
<210>99
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>99
gaugguauguuggaaagcga20
<210>100
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>100
cguggcugguucgugcgggg20
<210>101
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>101
uucguguaucuagugcaguc20
<210>102
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>102
ggucgagugaugcggggcgc20
<210>103
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>103
uuucguguauccuaguugcu20
<210>104
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>104
ugauguauuugguuugcaag20
<210>105
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>105
uucguguucugguugagugu20
<210>106
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>106
gcgcgggaaggguggcaugg20
<210>107
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>107
gagucuuuccgucuugcgug20
<210>108
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>108
agcgaguacgcugcgauugc20
<210>109
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>109
gcgauguaugguuuucggug20
<210>110
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>110
ccagaggugauggcggucgc20
<210>111
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>111
ggcgaucggaaggcaugcac20
<210>112
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>112
cacucgagaugccgcguagg20
<210>113
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>113
cacgcaggcgagguggcagc20
<210>114
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>114
ccuuugugucugcaggcucu20
<210>115
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>115
ccgggcguaucuuguggucg20
<210>116
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>116
caucugcucucgggggcaca20
<210>117
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>117
ucguaggucaggaagcggcg20
<210>118
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>118
ccgggcguaucuuguggucg20
<210>119
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>119
guggucgauaagcguacgcg20
<210>120
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>120
ugcgacgucguuagugguac20
<210>121
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>121
ggccuuggucgcgugcuggc20
<210>122
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>122
ccagaggugauggcggucgc20
<210>123
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>123
ccgcgugguuugugcgggug20
<210>124
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>124
ccagaacuugugguaccugc20
<210>125
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>125
gcgauguaugguuuucggug20
<210>126
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>126
caucugcucucgggggcaca20
<210>127
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>127
ucguaggucaggaagcggcg20
<210>128
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>128
gcgauaggcggucguugguc20
<210>129
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>129
cccgcuucuuccucgggugg20
<210>130
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>130
gucacucggccuagugcguc20
<210>131
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>131
ccugaccuuacucggguggc20
<210>132
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>132
guggucgauaagcguacgcg20
<210>133
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>133
agggcguuugcucgcggguc20
<210>134
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>134
ccgggcguaucuuguggucg20
<210>135
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>135
cagacgguacucaggugugc20
<210>136
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>136
ggccuuggucgcgugcuggc20
<210>137
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>137
gucggugaacgacugguggc20
<210>138
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>138
gguuaauuuuaugugucaac20
<210>139
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>139
gggcuuucggcaggcggucg20
<210>140
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>140
guggauguagggggacgggc20
<210>141
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>141
ugcgauguugugaguggccc20
<210>142
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>142
gcauaugggcgcuugugugg20
<210>143
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>143
gggugcggaagucagugugg20
<210>144
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>144
gagcugcauguggcguuggg20
<210>145
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>145
gugguaagguuggaaggugu20
<210>146
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>146
gggccuugaucacguggugc20
<210>147
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>147
gaggccugugcuaguaguga20
<210>148
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>148
ugguagguuggaaggucggg20
<210>149
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>149
cgugguaugguuggaacggu20
<210>150
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>150
aaggguaaguuggaaagucg20
<210>151
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>151
gugguagguuggaaggccgg20
<210>152
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>152
gugguagguuggaaaggcug20
<210>153
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>153
ggugguauguuggaagguug20
<210>154
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>154
ugcgauguugugaguggccc20
<210>155
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>155
agugguaaguuggaaugcac20
<210>156
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>156
gggccuugaucacguggugc20
<210>157
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>157
aaggccggugcuaguaguga20
<210>158
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>158
gugguagguuggaucgucgg20
<210>159
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>159
ugugguaucguuggaaagcg20
<210>160
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>160
ggugguagguuggaacggcg20
<210>161
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>161
agguaugggcgcuucguggc20
<210>162
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>162
gucgugguauguuggagggc20
<210>163
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>163
gggguauuguuggaacggcg20
<210>164
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>164
cggguauaguuggaacgucg20
<210>165
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>165
gugguagguuggaaaggcug20
<210>166
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>166
gugguaugguuggaacgcag20
<210>167
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>167
uggaacgucgaugugggccg20
<210>168
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>168
gguucagggugugggccgcc20
<210>169
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>169
gcuggucaggaugcgggggc20
<210>170
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>170
cguggcugguucgugcgggg20
<210>171
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>171
ggucgagugaugcggggcgc20
<210>172
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>172
cuagcgacggugcgggggug20
<210>173
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>173
uggggauaaguauguugcgg20
<210>174
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>174
ucguaggucaggaagcggcg20
<210>175
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>175
ugcgauguugugaguggccc20
<210>176
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>176
gcgcgggaaggguggcaugg20
<210>177
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>177
uggaacgucgaugugggccg20
<210>178
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>178
gguucagggugugggccgcc20
<210>179
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>179
ucguaggucaggaagcggcg20
<210>180
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>180
aaggccggugcuaguaguga20
<210>181
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>181
gcgauaggcggucguugguc20
<210>182
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>182
gcuggucaggaugcgggggc20
<210>183
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>183
gcugcgaaguggggcagguc20
<210>184
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>184
ggucgagugaugcggggcgc20
<210>185
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>185
cguggcugguucgugcgggg20
<210>186
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>186
gggcgaaauuggcauggccg20
<210>187
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>187
aaggccggugcuaguaguga20
<210>188
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>188
ugauguauugguaaguuucg20
<210>189
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>189
uauagaugugcuaguuugca20
<210>190
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>190
uuagcguuguauuaguugca20
<210>191
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>191
aucgauguaugagucauaua20
<210>192
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>192
aauuacacugugcuaggaug20
<210>193
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>193
uugaaguguacauugucgua20
<210>194
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>194
ugauguguuaguuugaaugu20
<210>195
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>195
ugaaaugagacugguuuugc20
<210>196
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>196
ugauguauuugguuugcaag20
<210>197
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>197
ugauguauugguaaguuucg20
<210>198
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>198
uauagaugugcuaguuugca20
<210>199
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>199
uuucguguuuagcguuugac20
<210>200
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>200
ugauguauuugguuugcaag20
<210>201
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>201
acguguauuacuauugacua20
<210>202
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>202
gaggccugugcuaguaguga20
<210>203
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>203
uuagcguuguauuaguugca20
<210>204
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>204
ucauugugcugaauugacua20
<210>205
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>205
aauuacacugugcuaggaug20
<210>206
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>206
aucgauguaugagucauaua20
<210>207
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>207
aucuugugguc11
<210>208
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>208
aaguggcgug10
<210>209
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>209
augcgggggc10
<210>210
<211>21
<212>rna
<213>人工序列
<220>
<223>snailsirna反义
<400>210
guggcuucggaugugcaucuu21
<210>211
<211>21
<212>rna
<213>人工序列
<220>
<223>snailsirna有义
<400>211
gaugcacauccgaagccacuu21
<210>212
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>212
gcuggucaggaugcgggggc20
<210>213
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>213
cctgtcccctcagttcatgt20
<210>214
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<400>214
cccatggtcttcttctgcat20
<210>215
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>215
uguguguccccacagcagug20
<210>216
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>216
tcgtaggtcaggaagcggcg20
<210>217
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>217
gcgataggcggtcgttggtc20
<210>218
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>218
cccgcttcttcctcgggtgg20
<210>219
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>219
gtcactcggcctagtgcgtc20
<210>220
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>220
cctgaccttactcgggtggc20
<210>221
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>221
gtggtcgataagcgtacgcg20
<210>222
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>222
agggcgtttgctcgcgggtc20
<210>223
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>223
ccgggcgtatcttgtggtcg20
<210>224
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>224
cagacggtactcaggtgtgc20
<210>225
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>225
ggccttggtcgcgtgctggc20
<210>226
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>226
ccatgctcgcgcggagtggc20
<210>227
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>227
tgcgatgttgtgagtggccc20
<210>228
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>228
gcgacgtcaggtgggcttgg20
<210>229
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>229
tagcgggtgcgatgtggtgc20
<210>230
<211>20
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>230
agcgatttcgcaggtggtgc20
<210>231
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>231
wwmgygcwymyw12
<210>232
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(4)..(4)
<223>n是a、c、g或u
<400>232
ccynkvrahcc11
<210>233
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>233
cygsryukrgrg12
<210>234
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>234
ucguaggucaggaagcggcg20
<210>235
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>235
gcgauaggcggucguugguc20
<210>236
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>236
acaguagagccuucaccggc20
<210>237
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>237
ccgggcguaucuuguggucg20
<210>238
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>238
uggaacgucgaugugggccg20
<210>239
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>239
gcuggucaggaugcgggggc20
<210>240
<211>21
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>240
gaugcacauccgaagccacuu21
<210>241
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>241
ddrgkuagggk11
<210>242
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(6)..(7)
<223>n是a、c、g或u
<220>
<221>misc_feature
<222>(10)..(10)
<223>n是a、c、g或u
<400>242
dvvrvnnvdnvdbdbbgugg20
<210>243
<211>15
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(1)..(2)
<223>n是a、c、g或u
<220>
<221>misc_feature
<222>(4)..(4)
<223>n是a、c、g或u
<220>
<221>misc_feature
<222>(6)..(7)
<223>n是a、c、g或u
<220>
<221>misc_feature
<222>(10)..(11)
<223>n是a、c、g或u
<400>243
nnvndnnbbnnrykk15
<210>244
<211>15
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(4)..(5)
<223>n是a、c、g或u
<400>244
dbdnngkskbbdbgb15
<210>245
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>245
wrvuguryydgbddddydvb20
<210>246
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(4)..(4)
<223>n是a、c、g或u
<400>246
dwwnguuggrd11
<210>247
<211>14
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(5)..(5)
<223>n是a、c、g或u
<400>247
kdddnguuggrwhb14
<210>248
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>248
ssscaaguggc11
<210>249
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>249
acwcgagwkgc11
<210>250
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>250
uggckuguggcu12
<210>251
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>251
awcuugugguc11
<210>252
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(7)..(7)
<223>n是a、c、g或u
<400>252
ddguggnygugc12
<210>253
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>253
vgugkgkugugc12
<210>254
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>254
wgcgasgusgyg12
<210>255
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>255
cmycacakcmgy12
<210>256
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(2)..(2)
<223>n是a、c、g或u
<400>256
rnrdguggcrug12
<210>257
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>257
skmdrguggcy11
<210>258
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>258
gsgcgkhguggy12
<210>259
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>259
agcgaygkugyg12
<210>260
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>260
uagcgrgugcg11
<210>261
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>261
agmgryggucgu12
<210>262
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>262
gcguadkkbgbg12
<210>263
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>263
uasguuggramg12
<210>264
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>264
augcgggggc10
<210>265
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>265
arabmdaggygc12
<210>266
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(9)..(9)
<223>n是a、c、g或u
<400>266
aguuggaangcg12
<210>267
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(2)..(2)
<223>n是a、c、g或u
<400>267
gnrwgcggkggc12
<210>268
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(7)..(7)
<223>n是a、c、g或u
<220>
<221>misc_feature
<222>(9)..(9)
<223>n是a、c、g或u
<400>268
ugurcwnknww11
<210>269
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>269
hggrgsagugg11
<210>270
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(2)..(3)
<223>n是a、c、g或u
<400>270
rnnrrwgcgggg12
<210>271
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(10)..(10)
<223>n是a、c、g或u
<400>271
kmgurwrucnw11
<210>272
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<220>
<221>misc_feature
<222>(4)..(4)
<223>n是a、c、g或u
<400>272
krdnwgguggch12
<210>273
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>273
gggggcruaug11
<210>274
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>274
auucgucgaguacagaccug20
<210>275
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>275
gcgcagggagggcuuggguc20
<210>276
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>276
agcgacguugcg12
<210>277
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>277
gcguauggcgug12
<210>278
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>278
uagguuggaaag12
<210>279
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>279
gggcgucguggc12
<210>280
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>280
augcgggggc10
<210>281
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>281
aaguggcgug10
<210>282
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>282
ccgcaaguggc11
<210>283
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>283
aucuugugguc11
<210>284
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>284
gugguugugc10
<210>285
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>285
guguggugugc11
<210>286
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>286
acucgaguggc11
<210>287
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>287
ccccacagcagu12
<210>288
<211>12
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>288
uggcguguggcu12
<210>289
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>289
aaguggcgug10
<210>290
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>290
aaguggcgug10
<210>291
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>291
aagaggcgug10
<210>292
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>292
aagucgcgug10
<210>293
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>293
aagugccgug10
<210>294
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>294
aaguggagug10
<210>295
<211>20
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>295
ccgggcguaucuuguggucg20
<210>296
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>296
aucuugugguc11
<210>297
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>297
gugcuucugua11
<210>298
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>298
auguugugguc11
<210>299
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>299
aucuucugguc11
<210>300
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>300
aucuugagguc11
<210>301
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>301
aucuugucguc11
<210>302
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>302
aucuugugcuc11
<210>303
<211>11
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>303
aucuuguggac11
<210>304
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>304
augcgggggc10
<210>305
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>305
ggucggacgg10
<210>306
<211>10
<212>rna
<213>人工序列
<220>
<223>seqidno:307
<400>306
uugcgggggc10
<210>307
<211>10
<212>dna
<213>人工序列
<220>
<223>exo-code
<400>307
aagcgggggc10
<210>308
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>308
auccgggggc10
<210>309
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>309
augggggggc10
<210>310
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>310
augccggggc10
<210>311
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>311
augcgcgggc10
<210>312
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>312
augcggcggc10
<210>313
<211>10
<212>rna
<213>人工序列
<220>
<223>exo-code
<400>313
augcgggcgc10
- 还没有人留言评论。精彩留言会获得点赞!