一种鼠伤寒沙门菌MLVA分型的检测引物组、试剂盒及其应用的制作方法
本发明属于微生物技术领域,具体涉及一种鼠伤寒沙门菌mlva分型的检测引物组、试剂盒及其应用。
背景技术:
沙门菌(salmonellaspp)是公共卫生学上具有重要意义的人畜共患病病原菌,可引起人和动物的伤寒、副伤寒、胃肠炎和败血症等许多严重疾病,使全球遭受巨大的经济损失。目前,全世界已发现约2610种沙门菌血清型。其中,鼠伤寒沙门菌(salmonellaentericasubsp.entericaserovartyphimurium,stm)是存在于畜禽及食品中的优势血清型,也是引起人类感染的主要血清型。
采用有效的分型方法对不同来源、不同血清型甚至不同致病力的沙门菌加以区分,对沙门菌危害溯源、风险预警和疫情监测具有重要意义。多位点可变数目串联重复序列分析(multiple-locusvariable-numbertandem-repeatanalysis,mlva)分型方法是利用不同微生物基因组中不同位点串联重复序列(variable-numbertandem-repeat,vntr)数目的不同,对其进行分型,被广泛用于微生物,尤其是细菌的研究。mlva分型方法操作简单、快速、分辨率高,得到明确的数字化的结果,重复性好,利于各实验室间交流和共享数据、比较结果。2004年lindstedt等利用鼠伤寒沙门菌的5个vntr位点-sttr9、sttr5、sttr6、sttr10和sttr3成功建立了用于鼠伤寒沙门菌的mlva-5位点分型方法,该方法大大提高了对鼠伤寒沙门菌多态性的分辨能力。该方法用两个复合pcr体系分别扩增5个vntr位点,然后通过毛细管电泳法检测产物,获得vntr位点重复序列数目。随着测序技术的发展,mlva中vntr位点重复序列数目的确定,逐渐发展为更加方便、精确的对vntr位点序列信息的分析。在对大量样本的实验验证中发现,原mlva-5位点分型方法中sttr9、sttr5、sttr6、sttr10位点的分辨率高,重复序列短(6-9bp),但sttr3位点的重复序列却很长,而且有2种,分别是27bp和33bp,长序列的重复片段影响了序列分析的准确性,从而影响了结果的可靠性,而且研究中发现sttr3位点的多态性并不能提高整个方法的分辨率。同时,该mlva-5位点方法中各vntr位点扩增过程不能在同一反应体系中完成。
技术实现要素:
本发明的目的是为了克服现有技术中的缺陷,提供一种更有效、更简便、高分辨率的鼠伤寒沙门菌mlva分型的检测引物组、试剂盒及其应用。
本发明针对鼠伤寒沙门菌具有较好分辨能力的vntr位点——mlva1(即sttr9)、mlva2(即sttr5)、mlva3(即sttr6)、mlva4(即sttr10),分别设计可用于多重pcr扩增的特异性引物序列,建立多重pcr扩增检测方法;在结果判定中,采用简便、直观的本地blast序列分析技术确定4个vntr区的重复序列数目组合,以对鼠伤寒沙门菌进行mlva分型。克服原有鼠伤寒沙门菌mlva分型方法的弊端,采用更少的vntr位点、更有效的一次多重pcr扩增检测,以及利用本地blast序列分析的更直观、方便的结果判定方法,建立一种鼠伤寒沙门菌mlva分型方法。
本发明的第一个目的是提供一种鼠伤寒沙门菌mlva分型的检测引物组,由下列引物组成:
针对mlva1位点:
mlva1-f:5’-ccggtactgtcagggaaaga-3’(如seqidno.5所示),mlva1-r:5’-agcataccaaacgaccgcta-3’(如seqidno.6所示);
针对mlva2位点:
mlva2-f:5’-gctggatgttctggcggac-3’(如seqidno.7所示),mlva2-r:5’-gaatatcggcagcatggg-3’(如seqidno.8所示);
针对mlva3位点:
mlva3-f:5’-cgtttattcgggcatgcg-3’(如seqidno.9所示),mlva3-r:5’-tctggtggggagaatgactg-3’(如seqidno.10所示);
针对mlva4位点:
mlva4-f:5’-aagaatgccccggaatatgc-3’,mlva4-r:5’-agcatgacagtcccggttc-3’(如seqidno.12所示)。
本发明的第二个目的是提供一种鼠伤寒沙门菌mlva分型试剂盒,包括多重pcr扩增试剂和检测引物组,所述的检测引物组由下列引物组成:
针对mlva1位点:
mlva1-f:5’-ccggtactgtcagggaaaga-3’(如seqidno.5所示),mlva1-r:5’-agcataccaaacgaccgcta-3’(如seqidno.6所示);
针对mlva2位点:
mlva2-f:5’-gctggatgttctggcggac-3’(如seqidno.7所示),mlva2-r:5’-gaatatcggcagcatggg-3’(如seqidno.8所示);
针对mlva3位点:
mlva3-f:5’-cgtttattcgggcatgcg-3’(如seqidno.9所示),mlva3-r:5’-tctggtggggagaatgactg-3’(如seqidno.10所示);
针对mlva4位点:
mlva4-f:5’-aagaatgccccggaatatgc-3’,mlva4-r:5’-agcatgacagtcccggttc-3’(如seqidno.12所示)。
本发明的第三个目的是提供一种非疾病的诊断和治疗目的的鼠伤寒沙门菌mlva分型方法,包括以下步骤:提取待测鼠伤寒沙门菌的基因组dna作为模板,以所述的mlva1-f/mlva1-r、mlva2-f/mlva2-r、mlva3-f/mlva3-r和mlva4-f/mlva4-r作为扩增引物进行多重pcr扩增,对多重pcr扩增产物进行测序,根据测序结果对待测鼠伤寒沙门菌进行mlva分型。
所述的多重pcr扩增的反应体系优选为25μl,包括10×pcrbuffer2.5μl,3.0mmol/lmgcl2,250μmol/ldntp,引物mlva1-f/mlva1-r、mlva2-f/mlva2-r、mlva3-f/mlva3-r和mlva4-f/mlva4-r各120nmol/l,1.5utaq酶,模板2μl,余量为ddh2o。
所述的多重pcr扩增的反应条件优选为95℃预变性5min;95℃变性45s、58℃退火45s、72℃延伸45s,共进行30个循环;72℃延伸10min。
所述的根据测序结果对待测鼠伤寒沙门菌进行mlva分型具体为根据测序结果采用本地blast分析法对待测鼠伤寒沙门菌mlva1、mlva2、mlva3、mlva4位点重复数结果判定,根据4个位点的重复数对待测鼠伤寒沙门菌进行mlva分型。mlva1、mlva2、mlva3、mlva4位点的重复序列短,分别为9bp、6bp、6bp、6bp,利用本地blast分析vntr位点的重复序列数目进行更方便、直观的结果判定。
根据测序结果对待测鼠伤寒沙门菌进行mlva分型可以采用公式法或本地blast分析法。
采用公式法:根据测序结果,重复序列的数目(repeartnumber)={产物大小(lengthofproduct)-重复序列周围的片段大小(flankingsize)}/每个重复序列的大小(lengthofeachrepeat)。
采用本地blast分析法:将同一vntr位点的不同样本的测序序列信息组合成一个文档,采用命令“makeblastdb-inall.ffn-dbtypenucl-parse_seqids-outdb.nt”以此文档构建本地数据库db.nt(all.ffn为测序序列信息组合成的文档名,db.nt为构建的数据库名),再以目标vntr位点的序列信息文档mlva1为目标(以分析mlva1位点为例,mlva1为vntr位点mlva1的序列信息文档),以命令“blastn-dbdb.nt-evalue1e-5-outfmt0-num_descriptions10-num_threads64-querymlva1-outmlva1result”对本地数据库db.nt进行mlva1位点的blast分析(mlva1result为输出结果的命名)。
本发明的第四个目的是提供所述的检测引物组在鼠伤寒沙门菌mlva分型中的应用。
本研究的关键是:1.去除原方法中重复片段长且影响分析的vntr位点(sttr3),针对鼠伤寒沙门菌的4个具有较好分辨能力的vntr位点设计特异性引物,应用于鼠伤寒沙门菌mlva分型检测。2.4对引物序列在同一pcr反应体系中不会出现相互交叉反应,可以同时扩增出4条特异性核苷酸序列片段。3.原mlva-5位点分型方法中sttr9、sttr5、sttr6、sttr10位点的重复序列短,分别为9bp、6bp、6bp、6bp,可以利用本地blast分析vntr位点的重复序列数目进行更方便、直观的结果判定,但sttr3位点的重复序列却很长,而且有2种(27bp和33bp),长序列的重复片段在blast分析中会产生较大缺口,影响了序列分析的准确性。
本发明根据已知的鼠伤寒沙门菌标准菌株lt2vntr位点序列,针对4个具有较好分辨能力的vntr位点设计特异性引物,建立多重pcr扩增检测方法,对各位点pcr扩增产物片段进行测序。采用本地blast技术对测序结果进行分析,判断不同vntr位点的重复序列数目。获得4个位点重复序列数目组合,结果表示为n-n-n-n。经过大量鼠伤寒沙门菌检测验证,此方法可对鼠伤寒沙门菌进行稳定的分型检测,仅分析4个vntr位点、一次多重pcr扩增检测、直观方便的本地blast分析获得vntr重复序列数目,其分辨率与原mlva-5点分型方法相同,但分型效率高于原有mlva-5位点法,更简单、方便。
附图说明:
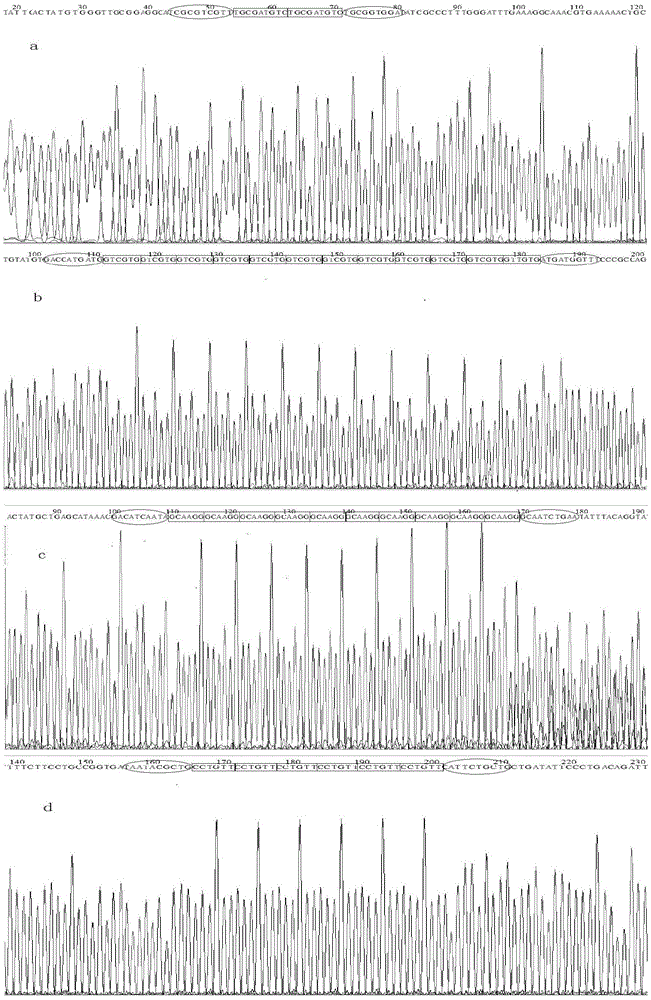
图1是鼠伤寒沙门菌菌株54-13的vntr位点多重pcr扩增产物测序验证结果;其中a为鼠伤寒沙门菌菌株54-13的vntr位点mlva1特异性引物pcr反应产物测序验证结果,b为鼠伤寒沙门菌菌株54-13的vntr位点mlva2特异性引物pcr反应产物测序验证结果,c为鼠伤寒沙门菌菌株54-13的vntr位点mlva3特异性引物pcr反应产物测序验证结果,d为鼠伤寒沙门菌菌株54-13的vntr位点mlva4特异性引物pcr反应产物测序验证结果;其重复序列数目分别为mlva1:重复序列数rep=2;mlva2:重复序列数rep=12;mlva3:重复序列数rep=10;mlva4:重复序列数rep=6;鼠伤寒沙门菌菌株54-13的mlva型别表示为2-12-10-6。
图2是鼠伤寒沙门菌菌株54-13的vntr位点序列信息blast比对结果;其中a为鼠伤寒沙门菌菌株54-13的vntr位点mlva1序列信息blast比对结果,b为鼠伤寒沙门菌菌株54-13的vntr位点mlva2序列信息blast比对结果,c为鼠伤寒沙门菌菌株54-13的vntr位点mlva3序列信息blast比对结果,d为鼠伤寒沙门菌菌株54-13的vntr位点mlva4序列信息blast比对结果;其重复序列数目分别为mlva1:重复序列数rep=2;mlva2:重复序列数rep=12;mlva3:重复序列数rep=10;mlva4:重复序列数rep=6;鼠伤寒沙门菌菌株54-13的mlva型别表示为2-12-10-6。
具体实施方式:
以下实施例是对本发明的进一步说明,而不是对本发明的限制。
实施例1:
1、特异性引物序列的设计:
分析已知的鼠伤寒沙门菌标准菌株lt2序列信息,针对4个具有较好分辨能力的vntr位点——mlva1、mlva2、mlva3、mlva4,分别设计特异性引物序列。利用primerpremier5.0软件在4个vntr区两侧的保守区分别设计引物,且尽量使得目的片段长度具有较大的差异,便于结果判断。保证各引物序列具有相近的tm值,引物自身避免形成二级结构发夹结构,在同一反应体系中引物之间无交叉反应。优化设计后,4对特异性引物序列见表1。
表1.vntr位点扩增引物
以鼠伤寒沙门菌标准菌株lt2为例,各vntr位点引物及扩增序列:
1)mlva1引物及扩增序列248bp(其核苷酸序列如seqidno.1所示,下划线部分序列分别为mlva1-f序列及mlva1-r的碱基互补序列,加粗区域为侧翼序列,加粗区域的中间为重复单位也称为核心序列)
ccggtactgtcagggaaagaggcgctgcgattgacgatatctatgactttatggaatagcatattcactatgtgggttgcggaggcatcgcgtcgtttgcgatgtctgcgatgtctgcgatgtctgcgatgtctgcggtggatatcgccctttgggatttgaaaggcaaacgtgaaaaactgccgctgtggaaaatggctggttgaaggcggcgcgtctgtcgacgaatagcggtcgtttggtatgct
2)mlva2引物及扩增序列219bp(其核苷酸序列如seqidno.2所示,下划线部分序列分别为mlva2-f序列及mlva2-r的碱基互补序列,加粗区域为侧翼序列,加粗区域的中间为重复单位也称为核心序列)
gctggatgttctggcggacatggcgaggcgagcagcagtggctggcgggaaaccaccatcacgaccacgaccacgaccacgaccacgaccacgaccacgaccacgaccacgaccacgaccacgaccacgaccacgaccatcatggtcacatacatccggaaggtgcaacgtcaaaagcgtatcaggatgcccatgaacgcgcccatgctgccgatattc
3)mlva3引物及扩增序列350bp(其核苷酸序列如seqidno.3所示,下划线部分序列分别为mlva3-f序列及mlva3-r的碱基互补序列,加粗区域为侧翼序列,加粗区域的中间为重复单位也称为核心序列)
cgtttattcgggcatgcgttgaaaacctgcaaaaaaagaaaaatatcgatgcttattattttttctttaattaaattttcgctcaacaaacttaattattaatccaatgacagtgaagtgtgaactatgctgagcataaacgacatcaatagcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaatctgagtatttacaggtgaaattatgaaaaatagtatatttttaccattgcttctggtgctgtcggcaacgaccttttcggcattggtgatggctgccagtcattctccccaccaga
4)mlva4引物及扩增序列296bp(其核苷酸序列如seqidno.4所示,下划线部分序列分别为mlva4-f序列及mlva4-r的碱基互补序列,加粗区域为侧翼序列,加粗区域的中间为重复单位也称为核心序列)
aagaatgccccggaatatgccagaaaagaggatgaggttaaaaaatagcgccattcctgatgcattctgcctgtcgctggccggattttacgcatgccgggtgatgtttttttctggccggggtgtgtgattttcttcctgccggtgataatacgctgcctgttcctgttcctgttcctgttcctgttcctgttcctgttcctgttcctgttcctgttcattctgctgctgatattccctgacagatttcgctgacgccgggcgacttccctgacgggaaccgggactgtcatgct2、利用上述引物对鼠伤寒沙门菌菌株54-13的mlva分型检测应用,具体步骤:
(1)细菌dna提取
可采用自有方法或者商用细菌dna提取试剂盒提取所测菌株的dna。
(2)多重pcr扩增反应体系及反应条件
多重pcr扩增反应体系为25μl:包括10×pcrbuffer2.5μl,3.0mmol/lmgcl2,250μmol/ldntpeach,引物mlva1-f/mlva1-r、mlva2-f/mlva2-r、mlva3-f/mlva3-r和mlva4-f/mlva4-r各120nmol/l,1.5utaq酶,dna模板2μl,双蒸水补齐至总体积25μl。
多重pcr扩增反应条件:95℃预变性5min;95℃变性45s、58℃退火45s、72℃延伸45s,共进行30个循环;72℃延伸10min。同时设立阳性对照(鼠伤寒沙门菌标准菌株lt2基因组dna)和空白对照(ddh2o)。
(3)测序分析
将扩增片段进行纯化和测序分析,获得目标vntr位点的序列信息。
(4)结果判定:
测序结果如图1所示:
采用本地blast分析法:将同一vntr位点的不同样本的测序序列信息组合成一个文档,采用命令“makeblastdb-inall.ffn-dbtypenucl-parse_seqids-outdb.nt”以此文档构建本地数据库db.nt(all.ffn为测序序列信息组合成的文档名,db.nt为构建的数据库名),再以目标vntr位点的序列信息文档mlva1为目标(以分析mlva1位点为例,序列信息文档mlva1为vntr位点mlva1的序列信息文档(即以mlva1f/mlva1r为引物扩增得到的目的片段的核苷酸序列)),以命令“blastn-dbdb.nt-evalue1e-5-outfmt0-num_descriptions10-num_threads64-querymlva1-outmlva1result”对本地数据库db.nt进行mlva1位点的blast分析(mlva1result为输出结果的命名),分别对mlva1~4位点分析,blast分析见图2。
鼠伤寒沙门菌菌株54-13的4个vntr位点重复序列数目分别为mlva1:重复序列数rep=2;mlva2:重复序列数rep=12;mlva3:重复序列数rep=10;mlva4:重复序列数rep=6。
(5)结果表示
根据各位点pcr扩增片段测序结果中每一个位点重复序列的数目,结果以n-n-n-n表示,鼠伤寒沙门菌菌株54-13的mlva型别表示为2-12-10-6。
3、利用表1的引物对实验室保藏的67株鼠伤寒沙门菌菌株进行mlva分型检测的应用,具体步骤同步骤2:
由对实验室保藏的67株鼠伤寒沙门菌菌株进行mlva分型检测的应用可知,该方法对67株鼠伤寒沙门菌菌株的分辨率与原mlva-5点法相同,67株鼠伤寒沙门菌菌株分成56种型,有4种型别的菌株各有2株菌,有1个型别的菌株为4株菌,有1个型别的菌株为5株菌。结果显示,该mlva方法无法区分的菌株(表中加粗部分),原mlva-5点方法中的sttr3也均相同,无法区分。
表2.67株鼠伤寒沙门菌菌株的mlva分型结果
序列表
<110>广东省微生物研究所(广东省微生物分析检测中心)
<120>一种鼠伤寒沙门菌mlva分型的检测引物组、试剂盒及应用
<160>12
<170>siposequencelisting1.0
<210>1
<211>248
<212>dna
<213>鼠伤寒沙门菌lt2(salmonellatyphimuriumlt2)
<400>1
ccggtactgtcagggaaagaggcgctgcgattgacgatatctatgactttatggaatagc60
atattcactatgtgggttgcggaggcatcgcgtcgtttgcgatgtctgcgatgtctgcga120
tgtctgcgatgtctgcggtggatatcgccctttgggatttgaaaggcaaacgtgaaaaac180
tgccgctgtggaaaatggctggttgaaggcggcgcgtctgtcgacgaatagcggtcgttt240
ggtatgct248
<210>2
<211>219
<212>dna
<213>鼠伤寒沙门菌lt2(salmonellatyphimuriumlt2)
<400>2
gctggatgttctggcggacatggcgaggcgagcagcagtggctggcgggaaaccaccatc60
acgaccacgaccacgaccacgaccacgaccacgaccacgaccacgaccacgaccacgacc120
acgaccacgaccacgaccatcatggtcacatacatccggaaggtgcaacgtcaaaagcgt180
atcaggatgcccatgaacgcgcccatgctgccgatattc219
<210>3
<211>350
<212>dna
<213>鼠伤寒沙门菌lt2(salmonellatyphimuriumlt2)
<400>3
cgtttattcgggcatgcgttgaaaacctgcaaaaaaagaaaaatatcgatgcttattatt60
ttttctttaattaaattttcgctcaacaaacttaattattaatccaatgacagtgaagtg120
tgaactatgctgagcataaacgacatcaatagcaagggcaagggcaagggcaagggcaag180
ggcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaagggcaatctgagt240
atttacaggtgaaattatgaaaaatagtatatttttaccattgcttctggtgctgtcggc300
aacgaccttttcggcattggtgatggctgccagtcattctccccaccaga350
<210>4
<211>296
<212>dna
<213>鼠伤寒沙门菌lt2(salmonellatyphimuriumlt2)
<400>4
aagaatgccccggaatatgccagaaaagaggatgaggttaaaaaatagcgccattcctga60
tgcattctgcctgtcgctggccggattttacgcatgccgggtgatgtttttttctggccg120
gggtgtgtgattttcttcctgccggtgataatacgctgcctgttcctgttcctgttcctg180
ttcctgttcctgttcctgttcctgttcctgttcctgttcattctgctgctgatattccct240
gacagatttcgctgacgccgggcgacttccctgacgggaaccgggactgtcatgct296
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
ccggtactgtcagggaaaga20
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
agcataccaaacgaccgcta20
<210>7
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>7
gctggatgttctggcggac19
<210>8
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>8
gaatatcggcagcatggg18
<210>9
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>9
cgtttattcgggcatgcg18
<210>10
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>10
tctggtggggagaatgactg20
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
aagaatgccccggaatatgc20
<210>12
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>12
agcatgacagtcccggttc19
- 还没有人留言评论。精彩留言会获得点赞!