一种筛选具有不同生育时期的大豆品种的方法及其专用试剂盒与流程
本发明属于植物育种领域,具体涉及一种筛选具有不同生育时期的大豆品种的方法及其专用试剂盒。
背景技术:
大豆是研究光周期现象的重要模式植物,许多光周期现象的发现均是以大豆为实验材料。研究表明大豆在全生育期(出苗至成熟)都具有较强的感光性。大豆是短日植物且具有短日诱导累积效应,光照不仅影响大豆的开花时间还影响大豆的品质和其它的农艺性状。随着分子生物技术的发展,重要基因的功能不断被揭示,如何将优异基因用于大豆新品种的选育,成为分子生物学家、遗传学家和大豆育种工作者共同关注的焦点。从大豆种质资源中发掘与生育期等农艺性状相关的等位基因,然后通过传统杂交和分子标记辅助选择相结合加以利用,是最直接有效且最易被接受的大豆品种改良方法。
大豆gmrav基因受生物钟调控,且在短日照(sd)条件的mrna转录物丰度显著高于长日照(ld)条件,进一步地,gmrav干涉对植株的生长有促进作用并且提早开花(luq,zhaol,lid,etal.agmravorthologisinvolvedinphotoperiodandsucrosecontroloffloweringtimeinsoybean.[j].plosone,2014,9(2):e89145.)。因此gmrav基因是与大豆花期相关的优异基因资源,然而目前尚未开发出与gmrav基因相关的分子标记。
技术实现要素:
本发明的目的是筛选具有不同生育时期的大豆品种。
本发明首先保护与大豆始花期、盛花期、始荚期、盛荚期、始粒期、鼓粒期和初熟期相关的多态性位点组合,可为如下(x1)或(x2):
(x1)包括下表所示的indel_-1120、indel_-904~-866、indel_-655、indel_-557、snp_-293和indel_-201~-200;
(x2)由所述indel_-1120、所述indel_-904~-866、所述indel_-655、所述indel_-557、所述snp_-293和所述indel_-201~-200组成;
本发明还保护成套引物,可包括下表所示的引物对甲、引物对乙和引物对丙;
所述成套引物具体可由所述引物对甲、所述引物对乙和所述引物对丙组成。
本发明还保护所述成套引物的应用,可为a-1)至a-16)中的任一种:
a-1)制备用于鉴定待测大豆始花期长短的试剂盒;
a-2)制备用于鉴定待测大豆盛花期长短的试剂盒;
a-3)制备用于鉴定待测大豆始荚期长短的试剂盒;
a-4)制备用于鉴定待测大豆盛荚期长短的试剂盒;
a-5)制备用于鉴定待测大豆始粒期长短的试剂盒;
a-6)制备用于鉴定待测大豆鼓粒期长短的试剂盒;
a-7)制备用于鉴定待测大豆初熟期长短的试剂盒;
a-8)制备用于鉴定待测大豆生育时期长短的试剂盒;
a-9)筛选具有不同始花期的大豆品种;
a-10)筛选具有不同盛花期的大豆品种;
a-11)筛选具有不同始荚期的大豆品种;
a-12)筛选具有不同盛荚期的大豆品种;
a-13)筛选具有不同始粒期的大豆品种;
a-14)筛选具有不同鼓粒期的大豆品种;
a-15)筛选具有不同初熟期的大豆品种;
a-16)筛选具有不同生育时期的大豆品种。
本发明还保护含有所述成套引物的试剂盒;所述试剂盒的用途可为a-9)至a-16)中的任一种:
a-9)筛选具有不同始花期的大豆品种;
a-10)筛选具有不同盛花期的大豆品种;
a-11)筛选具有不同始荚期的大豆品种;
a-12)筛选具有不同盛荚期的大豆品种;
a-13)筛选具有不同始粒期的大豆品种;
a-14)筛选具有不同鼓粒期的大豆品种;
a-15)筛选具有不同初熟期的大豆品种;
a-16)筛选具有不同生育时期的大豆品种。
所述试剂盒的制备方法也属于本发明的保护范围。所述试剂盒的制备方法可包括将各条引物单独包装的步骤。
本发明还保护一种筛选不同生育时期大豆品种的方法,可包括如下步骤:检测待测大豆品种基于上述任一所述多态性位点组合的基因型,“所述indel_-1120无缺失和/或所述indel_-904~-866无缺失和/或所述indel_-655无缺失和/或所述indel_-557无缺失和/或所述snp_-293为cc纯合型和/或所述indel_-201~-200无缺失”的待测大豆品种的生育时期短于“所述indel_-1120有缺失和/或所述indel_-904~-866有缺失和/或indel_-655有缺失和/或所述indel_-557有缺失和/或所述snp_-293为aa纯合型和/或所述indel_-201~-200有缺失”的待测大豆品种。
上述方法中,所述生育时期可为始花期、盛花期、始荚期、盛荚期、始粒期、鼓粒期和初熟期中的至少一种。
上述方法中,所述“短于”可为在统计学上的短于(即具有统计学意义)。
上述任一所述方法中,所述“检测待测大豆品种基于所述多态性位点组合的基因型”的方法如下:以待测大豆品种的基因组dna为模板,采用所述引物对甲、所述引物对乙或所述引物对丙进行pcr扩增,得到pcr扩增产物;将所述pcr扩增产物进行测序,从而评判待测大豆品种基于所述多态性位点组合的基因型。
所述多态性位点组合、或、所述成套引物、或、所述试剂盒、或、上述任一所述的方法在大豆育种中的应用也属于本发明的保护范围。
上文中,所述indel_-1120有缺失可为gmrav基因的第-1120位的t缺失。所述indel_-904~-866有缺失可为gmrav基因的第-904位和第-866位的tatttataataaatattattataagtcttttttatcttt缺失。所述indel_-655有缺失可为gmrav基因的第-655位的t缺失。所述indel_-557有缺失可为gmrav基因的第-557位的t缺失。所述indel_-201~-200有缺失可为gmrav基因的第-201位和第-200位的ta缺失。
上文中,含有“-”的多态性位点(如所述indel_-1120、所述indel_-904~-866、所述indel_-655、所述indel_-557、所述snp_-293、所述indel_-201~-200)表示该多态性位点位于gmrav基因的转录起始位点(即atg)之前。
上文中,以待测大豆品种的基因组dna为模板,采用所述引物对甲进行pcr扩增,得到的pcr扩增产物的核苷酸序列如序列表中序列7或序列8所示。以待测大豆品种的基因组dna为模板,采用所述引物对乙进行pcr扩增,得到的pcr扩增产物的核苷酸序列如序列表中序列9或序列10所示。以待测大豆品种的基因组dna为模板,采用所述引物对丙进行pcr扩增,得到的pcr扩增产物的核苷酸序列如序列表中序列11或序列12所示。
实验证明,利用本发明提供的方法筛选不同生育时期的大豆品种,操作简单且准确率高;通过检测大豆品种的基因组dna即可预测大豆品种的生育时期,具有重要的应用价值。
附图说明
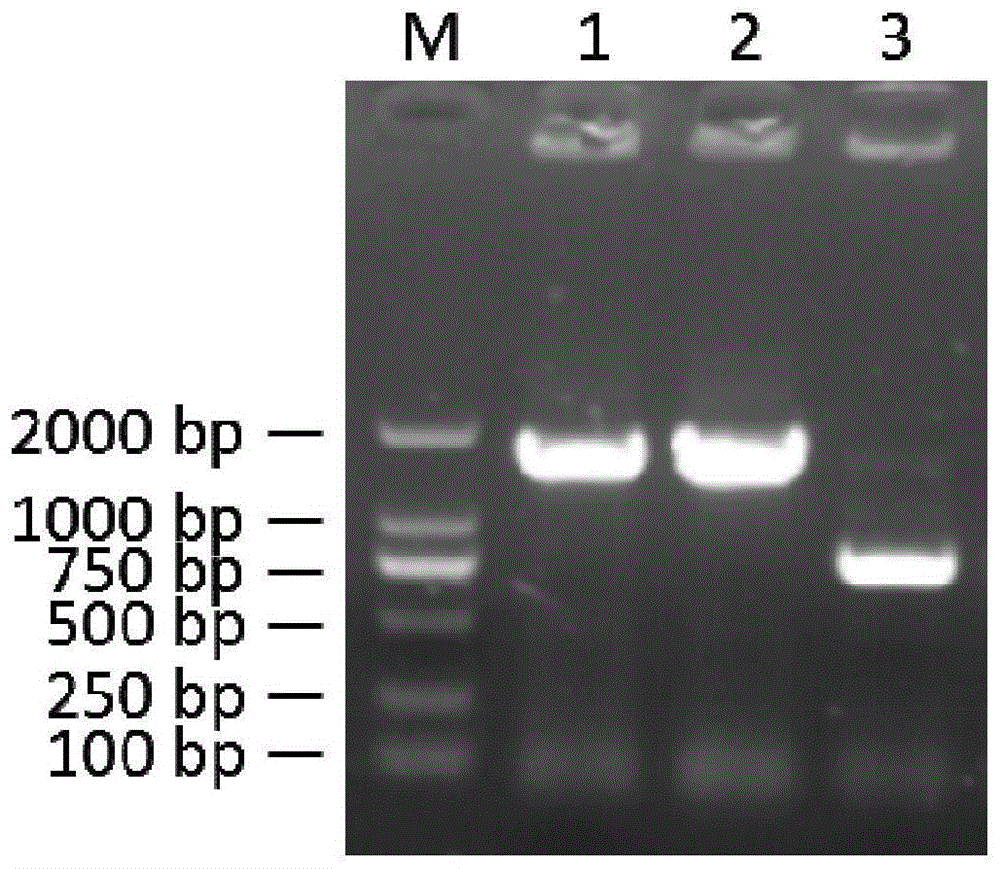
图1为实施例1步骤二中3的部分实验结果。
图2实施例2步骤三中2的部分实验结果。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。
下述实施例中,引物合成和测序均由华大基因进行。
实施例1、与大豆生育时期相关的6个多态性位点的发现
一、试验材料的种植和目标农艺性状的测定
试验材料为具有不同纬度适应性(20°13′n-50°15′n)的249个大豆品种。249个大豆品种(记载于如下文献中:han,y.,zhao,x.,cao,g.,wang,y.,li,y.,liu,d.,teng,w.,zhang,z.,li,d.andqiu,l.(2015)geneticcharacteristicsofsoybeanresistancetohgtype0andhgtype1.2.3.5.7ofthecystnematodeanalyzedbygenome-wideassociationmapping.bmcgenomics,16,1-11.)的名称和来源地详见表1。
表1
1、分别于2015年和2016年,在试验点(哈尔滨试验点、长春试验点或沈阳试验点)种植249个大豆品种,正常水肥管理。调查各个品种在6个环境中的农艺性状(如始花期、盛花期、始荚期、盛荚期、始粒期、鼓粒期、初熟期)。
部分大豆品种在6个环境中的农艺性状见表2。
表2
2、完成步骤1后,将同一环境中的各个农艺性状取平均值。结果见表3。
表3
二、筛选多态性位点与农艺性状关联分析
1、根据大豆品种willianms82的gmrav基因的核苷酸序列(genebank号为:fj794268.1),设计并合成3个引物对gmrav-1、gmrav-2和gmrav-3(见表4),用于检测gmrav基因。
表4
2、完成步骤1后,分别以249个大豆品种幼嫩叶片的基因组dna为模板,采用gmrav-1、gmrav-2或gmrav-3进行pcr扩增,得到pcr扩增产物。
反应程序:94℃10min;94℃30s,55℃30s,72℃1min30s,35个循环;72℃10min。
反应体系共50μl,由1μl大豆品种幼嫩叶片的基因组dna(含50-100ng基因组dna)、1μl上游引物(浓度为10μmol/μl)、1μl对应的下游引物(浓度为10μmol/μl)、5μl10×lapcrbufferii(mg2+)、8μldntpmixture、0.5μllataq和33.5μlddh2o组成。
lataq为takara公司的产品。10×lapcrbufferii(mg2+)为lataq中的组件。dntpmixture为生工生物工程(上海)股份有限公司的产品。
3、完成步骤2后,取pcr扩增产物,进行琼脂糖凝胶电泳。
大豆品种东农42的琼脂糖凝胶电泳的结果见图1(m为dnamarker2000,1为gmrav-1,2为gmrav-2,3为gmrav-3)。结果表明,采用gmrav-1、gmrav-2和gmrav-3进行pcr扩增,得到的pcr扩增产物大小依次为1610bp、1661bp和754bp。
4、完成步骤3后,取步骤2得到的pcr扩增产物,纯化后测序。
5、完成步骤4后,使用clustalx软件(http://www.clustal.org/)进行序列拼接,然后比对,筛选得到11个次级等位基因频率(maf)大于4%的多态性位点。用tassel软件(https://tassel.bitbucket.io/)mlm模型将各个多态性位点与农艺性状的表型进行关联分析。部分实验结果见表5和表6。结果表明,indel_-1120、indel_-904~-866、indel_-655、indel-557、snp_-293和indel_-201~-200为6个与大豆生育时期(如始花期、盛花期、始荚期、盛荚期、始粒期、鼓粒期、初熟期)性状显著相关的多态性位点(-log10p-value>1.30)。其中,indel_-557和indel_-201~-200与2个环境中的始花期显著相关;indel_-655、indel_-557和indel_-201~-200与至少1个环境中的盛花期显著相关;indel_-655、
indel_-557和indel_-201~-200与1个环境中的始荚期显著相关;indel_-557与1个环境中的盛荚期显著相关;indel_-904~-866与2个环境中的始粒期显著相关;indel_-904~-866和snp_-293与至少2个环境中的鼓粒期显著相关;indel_-1120、indel_-904~-866、
indel_-655、indel-557和snp_-293与至少2个环境中的初熟期显著相关。
表5关联分析获得与表型相关的多态性位点的-log10p值
表6.关联分析获得与表型相关的gmrav多态性位点的信息
三、249个大豆品种中6个多态性位点的相关性状统计分析
统计249个大豆品种基于上述6个多态性位点的基因型,然后与6个环境中的始花期、盛花期、始荚期、盛荚期、始粒期、鼓粒期和初熟期进行统计分析。
结果表明(表7),gmrav基因中“所述indel_-1120无缺失和/或所述indel_-904~-866无缺失和/或所述indel_-655无缺失和/或所述indel_-557无缺失和/或所述snp_-293为cc纯合型和/或所述indel_-201~-200无缺失”的待测大豆的性状的表型值低于“所述indel_-1120有缺失和/或所述indel_-904~-866有缺失和/或indel_-655有缺失和/或所述indel_-557有缺失和/或所述snp_-293为aa纯合型和/或所述indel_-201~-200有缺失”的待测大豆;性状为始花期、盛花期、始荚期、盛荚期、始粒期、鼓粒期和初熟期中的至少一种。
表7.249个大豆品种中6个多态性位点的相关性状统计分析结果
注:v表示tatttataataaatattattataagtcttttttatcttt。
实施例2、用于鉴定待测大豆的农艺性状的试剂盒的制备
一、正向引物和反向引物的合成
根据实施例1发现的6个多态性位点在gmrav基因上的位置,设计并合成正向引物和反向引物。正向引物的核苷酸序列见表8中第3列。反向引物的核苷酸序列见表8中第4列。引物对的名称见表8中第2列。靶序列的相关信息见表9,靶序列即采用正向引物和反向引物扩增获得的pcr扩增产物。
表8.引物对的信息
表9.采用引物对扩增的pcr扩增产物的信息
二、试剂盒的制备
试剂盒由引物对甲、引物对乙和引物对丙组成。其中引物对甲用于鉴定indel_-1120和indel_-904~-866的基因型,引物对乙用于鉴定indel_-655和indel_-557的基因型,引物对丙用于鉴定snp_-293和indel_-201~-200的基因型。
三、试剂盒在鉴定待测大豆的农艺性状中的应用
待测大豆为实施例1所述的249个大豆品种。
1、分别以待测大豆幼嫩叶片的基因组dna为模板,采用引物对甲、引物对乙或引物对丙进行pcr扩增,得到pcr扩增产物。
反应程序:94℃10min;94℃30s,55℃30s,72℃1min30s,35个循环;72℃10min。
反应体系共50μl,由1μl待测大豆品种幼嫩叶片的基因组dna(含50-100ng基因组dna)、1μl正向引物(浓度为10μmol/μl)、1μl对应的反向引物(浓度为10μmol/μl)、5μl10×lapcrbufferii(mg2+)、8μldntpmixture、0.5μllataq和33.5μlddh2o组成。
2、完成步骤1后,取pcr扩增产物,进行琼脂糖凝胶电泳。
大豆品种东农42的琼脂糖凝胶电泳的结果见图2(m为dnamarker2000,1为引物对甲,2为引物对乙,3为引物对丙)。
3、完成步骤2后,取pcr扩增产物,纯化后测序。
根据测序结果,确定待测大豆在上述6个多态性位点的基因型。“所述indel_-1120无缺失和/或所述indel_-904~-866无缺失和/或所述indel_-655无缺失和/或所述indel_-557无缺失和/或所述snp_-293为cc纯合型和/或所述indel_-201~-200无缺失”的待测大豆的性状的表型值低于“所述indel_-1120有缺失和/或所述indel_-904~-866有缺失和/或indel_-655有缺失和/或所述indel_-557有缺失和/或所述snp_-293为aa纯合型和/或所述indel_-201~-200有缺失”的待测大豆;性状为始花期、盛花期、始荚期、盛荚期、始粒期、鼓粒期和初熟期中的至少一种。
上述实验结果与实施例1中统计的农艺性状结果完全一致。因此,步骤二制备的试剂盒可用于鉴定待测大豆的始花期和/或盛花期和/或始荚期和/或盛荚期和/或始粒期和/或鼓粒期和/或初熟期。
<110>东北农业大学
<120>一种筛选具有不同生育时期的大豆品种的方法及其专用试剂盒
<160>12
<170>patentinversion3.5
<210>1
<211>22
<212>dna
<213>artificialsequence
<400>1
aaaatgaggtggatgataaaga22
<210>2
<211>22
<212>dna
<213>artificialsequence
<400>2
ggtcaactattggagacggagg22
<210>3
<211>22
<212>dna
<213>artificialsequence
<400>3
ccaaagggtgtcaatgtgattt22
<210>4
<211>22
<212>dna
<213>artificialsequence
<400>4
ggtcaactattggagacggagg22
<210>5
<211>22
<212>dna
<213>artificialsequence
<400>5
aagggtgtcaatgtgatttaga22
<210>6
<211>22
<212>dna
<213>artificialsequence
<400>6
gagaataggtatggtttggttt22
<210>7
<211>948
<212>dna
<213>artificialsequence
<220>
<221>misc_feature
<222>(136)
<223>kistordetetion
<400>7
aaaatgaggtggatgataaagaaaagagaggtaaaaaacagaaataaaaaaattacacta60
atctttcctaagtctttttgatgacataattttaaacaagcacttacctcctgcatattt120
attgtaacacacaccktttttttttatttaatacagtgacacacaccctttctttaatgt180
tatttagaagagatgagtgttttctaaaaaaagccaaagggtgtcaatgtgatttagaaa240
gacctcaattgaattagggtcattctcctaataagaaaaagagagaggtaaaataataga300
tataactaatattcctttttcataaaaaaaatattaaaaaaagttaacgaatatttataa360
taaatattattataagtcttttttatctttctcttcacatcacatcattatcaaatttat420
cattttttttctttgttgttcactatttttctcttctatttatccacacatcccaaatat480
gtaaaaataataatattcaattcattcatatattttattttacatctatttttatctctt540
tttttttctctttttcaccttcaacgtcacatcatatatcgcatttattattatttctga600
ttttttttttaataagaaataagttatagtatgatattttatattgtaacttatttctta660
taatttgattattataaataaaaaggaagataatattatttttttttctctatctattca720
tttccttcatcccattccgcctaaaaataaggtgaacgttcacaatttccgaatttataa780
tgtatgaatatatgactgccctgcccttatatttgtattgttagctcattttatgaaatt840
caaattgtcaagtgacccacatctagtgatcttatcctttttcctaatttcccattgaaa900
cctctctctcacacacaatgaatcttcctccgtctccaatagttgacc948
<210>8
<211>910
<212>dna
<213>artificialsequence
<220>
<221>misc_feature
<222>(352)
<223>mistatttataataaatattattataagtcttttttatctttordeletion
<400>8
aaaatgaggtggatgataaagaaaagagaggtaaaaaacagaaataaaaaaattacacta60
atctttcctaagtctttttgatgacataattttaaacaagcacttacctcctgcatattt120
attgtaacacacaccttttttttttatttaatacagtgacacacaccctttctttaatgt180
tatttagaagagatgagtgttttctaaaaaaagccaaagggtgtcaatgtgatttagaaa240
gacctcaattgaattagggtcattctcctaataagaaaaagagagaggtaaaataataga300
tataactaatattcctttttcataaaaaaaatattaaaaaaagttaacgaamctcttcac360
atcacatcattatcaaatttatcattttttttctttgttgttcactatttttctcttcta420
tttatccacacatcccaaatatgtaaaaataataatattcaattcattcatatattttat480
tttacatctatttttatctctttttttttctctttttcaccttcaacgtcacatcatata540
tcgcatttattattatttctgattttttttttaataagaaataagttatagtatgatatt600
ttatattgtaacttatttcttataatttgattattataaataaaaaggaagataatatta660
tttttttttctctatctattcatttccttcatcccattccgcctaaaaataaggtgaacg720
ttcacaatttccgaatttataatgtatgaatatatgactgccctgcccttatatttgtat780
tgttagctcattttatgaaattcaaattgtcaagtgacccacatctagtgatcttatcct840
ttttcctaatttcccattgaaacctctctctcacacacaatgaatcttcctccgtctcca900
atagttgacc910
<210>9
<211>735
<212>dna
<213>artificialsequence
<220>
<221>misc_feature
<222>(388)
<223>nistordeletion
<400>9
ccaaagggtgtcaatgtgatttagaaagacctcaattgaattagggtcattctcctaata60
agaaaaagagagaggtaaaataatagatataactaatattcctttttcataaaaaaaata120
ttaaaaaaagttaacgaatatttataataaatattattataagtcttttttatctttctc180
ttcacatcacatcattatcaaatttatcattttttttctttgttgttcactatttttctc240
ttctatttatccacacatcccaaatatgtaaaaataataatattcaattcattcatatat300
tttattttacatctatttttatctctttttttttctctttttcaccttcaacgtcacatc360
atatatcgcatttattattatttctgantttttttttaataagaaataagttatagtatg420
atattttatattgtaacttatttcttataatttgattattataaataaaaaggaagataa480
tattatttttttttctctatctattcatttccttcatcccattccgcctaaaaataaggt540
gaacgttcacaatttccgaatttataatgtatgaatatatgactgccctgcccttatatt600
tgtattgttagctcattttatgaaattcaaattgtcaagtgacccacatctagtgatctt660
atcctttttcctaatttcccattgaaacctctctctcacacacaatgaatcttcctccgt720
ctccaatagttgacc735
<210>10
<211>735
<212>dna
<213>artificialsequence
<220>
<221>misc_feature
<222>(486)
<223>ristordeletion
<400>10
ccaaagggtgtcaatgtgatttagaaagacctcaattgaattagggtcattctcctaata60
agaaaaagagagaggtaaaataatagatataactaatattcctttttcataaaaaaaata120
ttaaaaaaagttaacgaatatttataataaatattattataagtcttttttatctttctc180
ttcacatcacatcattatcaaatttatcattttttttctttgttgttcactatttttctc240
ttctatttatccacacatcccaaatatgtaaaaataataatattcaattcattcatatat300
tttattttacatctatttttatctctttttttttctctttttcaccttcaacgtcacatc360
atatatcgcatttattattatttctgattttttttttaataagaaataagttatagtatg420
atattttatattgtaacttatttcttataatttgattattataaataaaaaggaagataa480
tattarttttttttctctatctattcatttccttcatcccattccgcctaaaaataaggt540
gaacgttcacaatttccgaatttataatgtatgaatatatgactgccctgcccttatatt600
tgtattgttagctcattttatgaaattcaaattgtcaagtgacccacatctagtgatctt660
atcctttttcctaatttcccattgaaacctctctctcacacacaatgaatcttcctccgt720
ctccaatagttgacc735
<210>11
<211>901
<212>dna
<213>artificialsequence
<220>
<221>misc_feature
<222>(747)
<223>wiscora
<400>11
aagggtgtcaatgtgatttagaaagacctcaattgaattagggtcattctcctaataaga60
aaaagagagaggtaaaataatagatataactaatattcctttttcataaaaaaaatatta120
aaaaaagttaacgaatatttataataaatattattataagtcttttttatctttctcttc180
acatcacatcattatcaaatttatcattttttttctttgttgttcactatttttctcttc240
tatttatccacacatcccaaatatgtaaaaataataatattcaattcattcatatatttt300
attttacatctatttttatctctttttttttctctttttcaccttcaacgtcacatcata360
tatcgcatttattattatttctgattttttttttaataagaaataagttatagtatgata420
ttttatattgtaacttatttcttataatttgattattataaataaaaaggaagataatat480
tatttttttttctctatctattcatttccttcatcccattccgcctaaaaataaggtgaa540
cgttcacaatttccgaatttataatgtatgaatatatgactgccctgcccttatatttgt600
attgttagctcattttatgaaattcaaattgtcaagtgacccacatctagtgatcttatc660
ctttttcctaatttcccattgaaacctctctctcacacacaatgaatcttcctccgtctc720
caatagttgacccacccatcctccatwcacgctcacatttaatgaaataatattatataa780
gtaatcttccatgatgcctaaatcataaaaacctcattacattagctcaggttcctccta840
tatatatatatatatacatacataatatccttctccctcaaaccaaaccatacctattct900
c901
<210>12
<211>900
<212>dna
<213>artificialsequence
<220>
<221>misc_feature
<222>(839)
<223>yistaordeletion
<400>12
aagggtgtcaatgtgatttagaaagacctcaattgaattagggtcattctcctaataaga60
aaaagagagaggtaaaataatagatataactaatattcctttttcataaaaaaaatatta120
aaaaaagttaacgaatatttataataaatattattataagtcttttttatctttctcttc180
acatcacatcattatcaaatttatcattttttttctttgttgttcactatttttctcttc240
tatttatccacacatcccaaatatgtaaaaataataatattcaattcattcatatatttt300
attttacatctatttttatctctttttttttctctttttcaccttcaacgtcacatcata360
tatcgcatttattattatttctgattttttttttaataagaaataagttatagtatgata420
ttttatattgtaacttatttcttataatttgattattataaataaaaaggaagataatat480
tatttttttttctctatctattcatttccttcatcccattccgcctaaaaataaggtgaa540
cgttcacaatttccgaatttataatgtatgaatatatgactgccctgcccttatatttgt600
attgttagctcattttatgaaattcaaattgtcaagtgacccacatctagtgatcttatc660
ctttttcctaatttcccattgaaacctctctctcacacacaatgaatcttcctccgtctc720
caatagttgacccacccatcctccatccacgctcacatttaatgaaataatattatataa780
gtaatcttccatgatgcctaaatcataaaaacctcattacattagctcaggttcctccyt840
atatatatatatatacatacataatatccttctccctcaaaccaaaccatacctattctc900
- 还没有人留言评论。精彩留言会获得点赞!