一种适用于Hi-C的小片段DNA文库构建方法与流程
本发明涉及基因测序领域,更具体地,涉及一种适用于hi-c的小片段dna文库构建方法。
背景技术:
高通量测序技术又称为下一代测序技术、以能一次并行对几十万到几百万条dna分子进行序列测定和读长较短等为标志。第二代高通量illumina测序平台具有测序通量高、准确度高和成本低等优点,并广泛应用于多个领域。
染色体构象捕获(chromosomeconformationcapture,3c)技术是一种研究染色体和蛋白互作和染色体构象的技术,可以提供遥远的基因位点之间的关联性的详细信息,此关联性可以从甲醛固定的细胞核中捕获,并且可以从染色体的三维折叠方式被推断。近年来,在第二代测序技术的迅速发展下,由3c技术衍生出来的hi-c则是以整个细胞核为研究对象,研究整个基因组范围内基因位点之间的关联性。在hi-c技术中,以整个细胞系为研究对象,利用高通量测序技术,结合生物信息学方法,研究全基因组范围内整个染色质dna在空间位置上的关系;通过对染色质内全部dna相互作用模式进行捕获,获得高分辨率的染色质三维结构信息。将通过第二代测序得到的dna序列对映射到参考基因组后,如果一对序列对应于不同的n段酶切片段,那么就认为这两个片段之间有n次相互作用,从而能够构建一个全基因组中所有酶切片段之间接合频率的矩阵。利用这个频率矩阵,将基因组组装过程中形成的contig、scaffold进行染色体定位、方向没定位,延长拼接结果,辅助基因组组装,尤其对于获得群体困难无法构建遗传图谱的物种意义重大。
基于高通量进行染色体构象捕获的hi-c技术,是染色体相互作用研究领域的一个巨大的飞跃。但是距离研究普遍揭示生物学功能的目标还很遥远,hi-c交互数据包含相当高的随机噪声。同时,实验发现,现有hi-c高通量测序文库的构建也存在着信息丢失、捕获方法存在一定偏好性造成的获得的有效数据率不高的问题。
技术实现要素:
针对现有技术中存在的技术问题,本发明提出了一种适用于hi-c的小片段dna文库构建方法,该方法曾加生物素捕获步骤,可用于hi-c的小片段文库构建;同时该方法可显著减少非特异性扩增,明显改善dna纯度,对杂质较多的材料明显可提升其建库成功率。
本发明的第一个目的在于提出一种适用于hi-c的小片段dna文库构建方法,所述方法包括以下步骤:首先超声打断目标dna片段至主带集中在400bp,凝胶回收其中300-500bp的dna片段,对回收片段进行末端修复与加a获得末端修复后dna,对所述末端修复后的dna进行index连接获得连接物料,再对所述连接物料进行纯化获得dna纯化物料,最后将dna纯化物料进行pcr扩增获得dna测序文库。
进一步地,所述超声打断目标dna片段的操作为:
(1)取2μgdna样本纯化,溶于50μl去离子水中,使用qubit定量;
(2)取1μg纯化后的dna,使用去离子水补足体系至50μl;
(3)将50μl纯化后dna加入0.2mlpcr管中,并固定在浮漂上,超声波清洗仪水位加至最高并固定浮漂,打断功率为80w,时间为5min。
进一步地,所述末端修复与加a反应体系包括以下用量的各组分:片段dna50μl、nebnextultraiiendprepenzymemix3μl、nebnextultraiiendprepreactionbuffer3.0μl;反应体系20℃孵育30min,65℃灭活30min,4℃保温。
进一步地,所述index连接反应体系包括以下用量的各组分:末端修复后的dna60μl、nebnextultraiiligationmastermix30μl、nebnextligationenhancer1μl、ebnextadaptorforillumina2.5μl;反应体系混匀,20℃孵育15min后,加入3μluser酶,37℃孵育15min。
进一步地,所述pcr扩增体系包括以下用量的各组分:dna纯化物料20μl、nebnextultraiiq5mastermix25μl、indexprimer/i7primer2.5μl、universalpcrprimer/i5primer2.5μl;所述pcr程序为:98℃,30s;(98℃,10s;65℃,75s)6-12cycle;65℃,5min;降至4℃保存。
进一步地,所述方法还包括在pcr扩增后凝胶回收400-600bp文库片段来上机测序获得dna测序文库。
本发明的第二个目的是提出一种小片段dna文库,该文库由上述任一项所述的方法构建。
与现有技术相比,本发明的有益效果在于:
(1)本发明提出的一种适用于hi-c的小片段dna文库构建方法,对现有dna小片段文库构建实验技术进行了多项优化,使得文库构建成功率大大提升,成本显著降低,本发明的技术适用于绝大数物种;该方法操作流程简单,可以复制到其他具备分子生物学基础的其它实验室。
(2)本发明在现有dna小片段文库构建的基础上,曾加生物素捕获步骤,使该方法可用于hi-c小片段文库构建。
(3)本发明的方法利用超声波破碎的方式将基因组大片段打断至400bp左右,覆盖全基因组范围信息;在dna片段化后增加凝胶回收步骤,不仅进行了片段筛选,而且纯化了dna,对杂质较多的材料可显著提升其建库存成功率,对于物种的适应性大大增加。
(4)本发明的方法在末端修复之前进行凝胶回收,缩小目标片段范围至300-500bp,可显著减少非特异性扩增,有效提高有效数据率。
附图说明
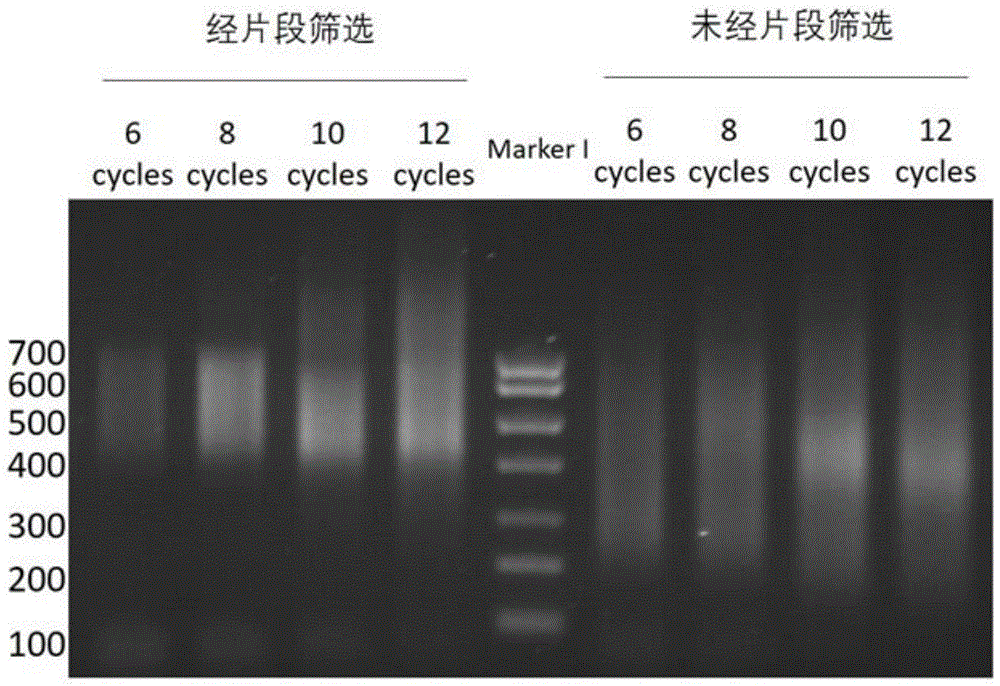
图1为实施例1中末端修复之前进行片段筛选和不进行片段筛选两种情况,后续进行pcr扩增的结果示意图。
图2为本实施例1构建的钉螺hi-c的小片段dna文库的agilent2100大小检测图。
具体实施方式
展示一下实例来具体说明本发明的某些实施例,且不应解释为限制本发明的范围。对本发明公开的内容可以同时从材料、方法和反应条件进行改进,所有这些改进,均应落入本发明的的精神和范围之内。若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
实施例1:
本实施例提供钉螺hi-c的小片段dna文库构建方法,所述方法包括以下步骤。
1.超声打断目标dna片段
1)取2μg左右dna样本进行纯化,溶于50μl去离子水中,使用qubit定量;
2)取1μg纯化后的dna,使用去离子水补足体系至50μl;
3)将50μl纯化后dna加入0.2mlpcr管中,并固定在浮漂上,超声波清洗仪水位加至最高并固定浮漂,打断功率选择最大,时间选择1-10min梯度摸索合适的打断条件;
4)打断后取2.5μl样品电泳检测打断效果,打断至主带集中在400bp的时间,即最佳打断时间;
5)打断条件摸索一次后即可适用于大多数纯化dna样本,无需多次摸索适合条件;最终确定的打断功率为80w,时间为5min。
2.目的片段回收
1)使用1.5×琼脂糖凝胶电泳与qiaquickgelextractionkit回收300-500bp大小的片段;
2)在紫外灯下小心地把大小约300-500bp的dna的片段切下来,并尽量去除多余的凝胶;
3)空离心管重称,切下的凝胶装在15ml离心管中并称重,算出凝胶重,1:1确定其体积,并加入3倍体积的bufferqg,常温摇晃10min;
4)待凝胶块彻底溶解后,观察混合溶液的颜色是不是黄色,若颜色为黄色,则继续下步操作,若为橙色,则加入100μl醋酸钠,使溶液恢复黄色再继续下步操作;
5)向溶液中加入一个胶体积的异丙醇并充分混匀;
6)将离心柱(qiaquickspincolumn)装在一个干净的2ml收集管中;
7)把5)中的溶液加入吸附柱中,12000rpm离心1min,弃掉废液,并把qiaquick离心柱重新套回2ml收集管中,若溶液体积大于700μl,则分数次加入,直至全部液体过柱;
8)在离心柱中加入0.5mlbufferqg12,000rpm离心1min,弃滤液;
9)在离心柱中加0.75mlbufferpe12,000rpm离心1min,弃滤液,在离心柱中再次加0.75mlbufferpe12,000rpm离心1min,弃滤液;
10)再次12,000rpm离心1min;
11)取出离心柱,把离心柱装在一个干净的1.5ml离心管上;
12)在吸附柱的中间部位加25μlbuffereb(10mmtris-hcl,ph,8.0),温室放置3min,再次12,000rpm离心1min,取上清液,用于后续文库构建或-20℃保存备用。
3.目的片段的末端修复与接头连接
1)dna末端修复与加a
参照如下体系依次加入试剂
反应体系20℃孵育30min,65℃灭活30min,4℃保温。
2)接头连接
参照如下体系依次加入试剂:
混匀,20℃孵育15min后,加入3μluser酶,37℃孵育15min。
4.ampurexpbeads纯化
1)加dna溶液的1.5倍体积磁珠溶液到dna连接产物中;振荡数秒混匀,室温孵育5min;
2)瞬时离心,将离心管放在磁力架上静置2min,移去上清;
3)离心管保持在磁力架上,加入1ml的75%乙醇清洗磁珠,弃乙醇,重复此步骤一次;
4)打开管盖,室温风干30s;加入52μl去离子水,振荡将磁珠重新混悬,室温孵育5min;
5)瞬时离心,将离心管放到磁力架上静置1min,吸取50μl上清转移至新离心管中,若感觉吸到磁珠,可再用磁力架吸附一次,保证磁珠完全去除,文库可进行下步操作或保存于-20℃冰箱。
5.目标片段富集
1)配置streptaridinbeads结合液及洗涤液
2)涡旋磁珠,取10μl加入1.5mllobind离心管中;使用100μl1×twb(tweenwashingbuffer)清洗,室温震荡3min;磁力架吸附磁珠,弃上清;
3)再次使用100μl1×twb清洗磁珠,室温震荡3min;磁力架吸附磁珠,弃上清;
4)50μl2×bb(bindingbuffer)和50μlhi-cdna重悬磁珠;室温震荡15min;磁力架吸附磁珠2-3min,弃上清;
5)使用100μl1×twb清洗磁珠并转移至一新lobind离心管中;磁力架吸附磁珠,弃上清;
6)重复2次1×twb清洗磁珠;
7)加入25μl水,70℃温浴5min洗脱dna,磁力架吸附,回收上清;
8)加入20μl水,70℃温浴5min洗脱dna,磁力架吸附,回收上清;
9)总体积45μl,4μl用于跑圈,20μl用于pcr扩增切胶,剩余21μl文库可于-20℃长期保存。
6.illumina试剂盒扩增嵌合片段
1)将pcr仪设置如下参数,并预热pcr仪
2)参照如下体系依次加入试剂:
3)pcr扩增放大目的片段
7.片段筛选
1)1.5%agarose,100v约60min凝胶回收400-600bp片段,使用qiagen凝胶回收试剂盒进行操作,溶于20-30μleb,浓度≥3ng/μl;
图1为末端修复之前进行凝胶回收和不进行凝胶回收两种情况下的结果,后续进行pcr扩增的结果示意图。从图上可以看到,pcr在第6-8个循环即可产生足够的产物,且经凝胶回收片段筛选的文库,可显著减少非特异性扩增,文库大小集中在400-600bp(pcr扩增的文库会加上引物接头,接头长度约为120bp,所以pcr电泳图会比实际凝胶回收获得的片段大120bp左右),而未经片段筛选的文库,非特异性扩增十分明显,片段范围增大至200-800bp。
2)回收产物使用agilent2100对文库大小分布进行检测,文库大小合适分布均匀,可以进行高通量测序。
图2为本实施例构建的钉螺hi-c的小片段dna文库的agilent2100大小检测图,结果表明主峰在483bp,且峰宽不超过400bp,是一个良好的文库大小分布峰图。
实施例2:
本实施例以钉螺hi-c连接产物dna为研究对象,对连接产物进行超声打断,凝胶回收300-500bp片段,最后进行文库构建,构建文库的中间步骤增加一步生物素捕获步骤,即可获得含有生物素标记的片段,再进行文库的pcr扩增,第二次凝胶回收400-600bp的片段,即可获得可上机测序的文库,在上机测序前进行agilent2100对文库大小分布进行检测,文库大小合适分布均匀,可以进行高通量测序。
测序完成后,对质控得到的cleandata,使用ice3软件对数据进行迭代比对,并进行噪音reads过滤。
表5钉螺dna小片段文库数据分析结果说明
以上所述,仅为本发明较佳的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,根据本发明的技术方案及其发明构思加以等同替换或改变,都应涵盖在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!