核酸序列测序接头及其构建测序文库的方法与流程
本发明涉及分子生物学
技术领域:
,特别涉及核酸序列测序接头及其构建测序文库的方法。
背景技术:
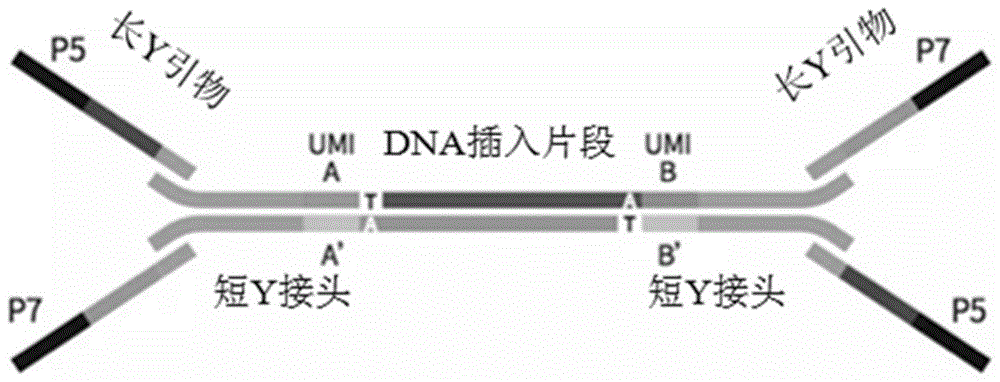
:液体活检弥补组织检测的不足,同一癌种的不同患者,同一个肿瘤患者的不同治疗阶段、同一肿瘤组织的不同区域,肿瘤的生物学特征均存在一定的差异。液体活检既可以克服组织检测的异质性,又具有简便、安全、无创、实时等特点,尤其是对于无法进行手术或穿刺,又或者肿瘤位置导致取样困难的患者而言,液体活检是一项可以弥补组织检测局限性的方法,近年来在肿瘤靶向治疗、耐药监测的实时评估等方面发挥着重要的作用。目前液体活检的靶标包括:游离的循环肿瘤细胞(CTCs)、循环肿瘤DNA(ctDNA)、循环肿瘤RNA(ctRNA)和外泌体(携带有细胞来源相关的多种蛋白质,脂类,DNA,RNA等)。ctDNA是肿瘤细胞在坏死、凋亡后释放的一种游离DNA(cfDNA),在血液中的半衰期短,可以实时反映肿瘤的动态变化。肿瘤患者体内的肿瘤细胞数量远远低于正常细胞,cfDNA在血浆中的含量很低,而ctDNA仅占cfDNA的0.1%~5%,且不同癌种,不同病程的肿瘤患者ctDNA在血浆中含量差异较大,因此相比于组织检测,ctDNA的检测需要更高的灵敏度和特异性。目前用于ctDNA液体活检的技术主要有ARMS-PCR、数字PCR(ddPCR)和第二代测序(NGS)。NGS能同时检测多个基因的多种不同变异形式,是应用最广泛的的基因检测技术。但由于NGS的实验流程技术较为复杂,在文库构建、目标区域捕获及测序过程中不可避免的会引入一些扩增和测序的错误,这些错误我们把它们叫做背景噪音,而ctDNA检测往往突变频率较低,受到背景噪音干扰较大,来自ctDNA样本中的低频突变往往淹没在背景噪音之中,造成假阴性或假阳性结果,这就限制了ctDNA检测的灵敏度和特异性。分子条形码又称分子标签(UniqueMolecularindentifier,UMI),它的原理就是给每一条原始DNA片段加上一段特有的标签序列,经文库构建及PCR扩增后一起进行测序。这样,根据不同的标签序列我们就可以区分不同来源的DNA模板,分辨哪些是PCR扩增及测序过程中的随机错误造成的假阳性突变,哪些是患者真正的携带的突变,从而提高检测灵敏度和特异性。技术实现要素:在一种实施方式中,本发明提供提供一种核酸序列测序接头,所述测序接头包括长Y引物和短Y接头,在测序的插入片段二端都连接有所述短Y接头和长Y引物;所述短Y接头具有部分互补的上游引物F和下游引物R,上游引物F和下游引物R分别含有标签序列UMI和标签序列UMI’,标签序列UMI和标签序列UMI’互补,且标签序列位于上游引物F和下游引物R形成的互补区域内;所述长Y引物具有上游引物P5和下游引物P7,所述上游引物P5含有标签序列indexD;所述下游引物P7含有标签序列indexN;和所述短接头是用来对同一个文库内部的不同分子进行标签识别,而所述长Y引物是用来对不同文库进行标签识别。在一种实施方式中,所述短Y接头的上游引物F和下游引物R通过退火形成部分双链结构。在一种实施方式中,所述上游引物F自5’端依次为通用序列、标签序列和游离碱基T;所述下游引物R自5’端依次为标签序列和通用序列,下游引物R的5’末端进行磷酸化修饰;所述标签序列由A、G、C、T任意排列组成。在一种实施方式中,所述长Y引物具有上游引物P5和下游引物P7,所述上游引物P5自5’端依次为通用序列、标签序列indexD和第一连接序列;所述下游引物P7自5’端依次为通用序列、标签序列indexN和第二连接序列;所述上游引物P5的第一连接序列与所述上游引物F的通用序列的一部分或全部通过互补结合,所述下游引物P7的第二连接序列和所述下游引物R的通用序列的一部分或全部通过互补结合。在一种实施方式中,所述标签序列UMI和标签序列UMI’中碱基数量为4-6个,和标签序列indexD和标签序列indexN中碱基的数量为6-8个。在一种实施方式中,当所述标签序列为4个碱基自由组合时,所述短Y接头的上游引物F为序列SEQNo.1:CCTACACGACGCTCTTCCGATCNNNNT,所述短Y接头的下游引物R为序列SEQNo.2:NNNNGATCGGAAGAGCACACGTCTGAA;所述上游引物F中的标签序列位于自5’端第23至第26位,所述下游引物R中的标签序列位于自5’端第1至第4位;所述N可以为A、G、C、T四种碱基的任意一种,但是不出现连续四个相同碱基。在一种实施方式中,当所述indexD标签序列和标签序列indexN由8个碱基组成时,所述长Y引物P5序列为:;长Y引物P5序列中加粗的碱基为indexD标签序列所在的位置;所述长Y引物P7序列为:长Y引物P7序列中加粗的碱基为标签序列indexN所在的位置。在一种实施方式中,本发明提供一种利用上述测序接头构建核酸测序文库的方法,所述方法包括:步骤1,利用酶反应将cfDNA样本进行末端修复及加A;步骤2,将上述经过末端修复、加A的cfDNA和短Y接头进行连接反应;步骤3,将连接产物利用长Y引物进行PCR扩增;和步骤4,纯化PCR扩增产物得到游离DNA样本文库。本发明在以上技术方案的基础上的优势表现为:(1)使用本发明的核酸测序接头,能够有效的区分和原始分子一样的其他ctDNA分子,一般ctDNA的提取量都比较低,通常只有十几纳克,本发明的核酸测序接头标签的优势在于有效区分不同的ctDNA分子,降低背景噪音,提高检测的灵敏度。(2)本发明的核酸测序接头由短Y接头和长Y引物两部分构成,短Y接头用来标记特定文库内部的不同分子,所以当进行多个文库构建时,连接过程可以采用同一种短Y接头混合物,在后期PCR扩增的过程中只要加入不同的长Y扩增引物就可以进行区分,这样设计的优势在于极大的节省了引物反复合成的成本,而且操作方便。本发明的核酸测序接头和建库方法能够更加准确的识别突变位点信息,大大提高检测体系的阳性预测能力,克服液体活检应用中存在的问题。附图说明为了更清楚地说明本申请实施例中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本申请中记载的一些实施例,对于本领域普通技术人员来说,在不付出创造性劳动的前提下,还可以根据这些附图获得其它的附图。图1为本发明的核酸序列测序接头的结构示意图;图2为ctDNA标准品的PPV结果对比分析图。具体实施方式为了使本领域
技术领域:
人员更好地理解本申请中的技术方案,下面将结合实施例对本发明作进一步说明,显然,所描述的实施例仅仅是本申请一部分实施例,而不是全部的实施例。基于本申请中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其它实施例,都应当属于本申请保护的范围。下述实施例中,如无特殊说明,均为本领域常规方法。以下实施例的定量实验中,均是设置三次重复实验,结果取平均值。实施例一本发明的核酸序列测序接头1.本发明的核酸序列测序接头如图1所示,在测序的插入片段一端连接短Y接头,短Y接头包括上游引物F和下游引物R,上游引物F和下游引物R具有部分互补的序列,它们分别含有互补的标签序列UMI和UMI’,标签序列UMI和标签序列UMI’位于上游引物F和下游引物R形成的互补区域内;在测序的插入片段另一端同样连接短Y接头,只是上游引物F和下游引物R的上下顺序相反。标签序列UMI由A、G、C、T任意排列组成,标签序列UMI中碱基数量优选地为4-6个。在一种实施方式中,上游引物F自5’端依次为通用序列、标签序列UMI和游离的碱基T。下游引物Y自5’端依次为标签序列UMI和通用序列。在一种实施方式中,上游引物F的3’末端第一个核苷酸是一个带有修饰的游离端T;下游引物R的5’末端进行磷酸化修饰。短接头是用来对同一个文库内部的不同分子进行标签识别,短Y接头可以通过上游引物F和下游引物R进行退火获得。如图1所示,长Y引物具有上游引物P5和下游引物P7,所述上游引物P5含有标签序列indexD;所述下游引物P7含有标签序列indexN;长Y引物是用来对不同文库进行标签识别的。长Y引物的上游引物P5和下游引物P7的序列之间不形成互补连接。长Y引物的上游引物P5自5’端依次为通用序列、标签序列indexD和第一连接序列;下游引物P7自5’端依次为通用序列、标签序列indexN和第二连接序列。标签序列indexD和标签序列indexN由多个碱基组成,如A、G、C、T任意排列组成的一种,indexD和标签序列indexN中碱基的数量优选地为6-8个。在PCR过程中所形成的文库长Y引物位于短Y接头的外侧。所述长Y引物上游引物P5的第一连接序列和短Y接头的上游引物F形成序列互补结合,互补的区域介于上游引物P5的标签序列indexD之后和标签序列UMI之前的序列。所述的长Y引物下游引物P7的第二连接序列和短Y接头的下游引物R形成序列互补结合,互补的区域介于标签序列indexN之后和标签序列UMI’之前的序列。当标签序列UMI中碱基数量为4时且测序平台为illumina平台时,上游引物F和下游引物R序列如表1所示,上游引物F中的标签序列UMI位于自5’端第23至第26位,下游引物R中的标签序列UMI’位于自5’端第1至第4位,其中的N表示可任意为A、G、C、T碱基中的一种。短Y接头如下:短Y接头由表1上游引物F所示的单链DNA分子序列和下游引物R所示的单链DNA分子序列形成部分双链得到。具体表现为上游引物F的5’端第26个碱基和下游引物R的5’端第1个碱基互补,上游引物F的5’端第25个碱基和下游引物R的5’端第2个碱基互补,上游引物F的5’端第24个碱基和下游引物R的5’端第3个碱基互补,依次类推,直到上游引物F的3’端第16个碱基和下游引物R的5’端第12个碱基互补。其余碱基都是处于非互补状态。且由于标签序列UMI的随机性,每个分子每一侧的接头的种类有44=256种,那么每个分子两侧标签的种类可以有256×256=65536种,足以满足特定ctDNA文库中每个分子都拥有一个独一无二的标签序列UMI。当标签序列indexD和标签序列indexN中碱基数量为8且测序平台为illumina平台时,扩增引物P5和P7序列如表2和表3所示,引物P5中的indexD标签序列位于自5’端第30至第37位,引物P7中的标签序列indexN位于自5’端第26至第33位。长Y引物和短Y接头的互补关系如下:(1)长Y引物的P5引物所示的单链DNA分子序列的任意一种和短Y接头的上游引物F所示的单链DNA分子序列的任意一种形成部分双链得到。具体表现为引物P5的5’端第48个碱基到第57个碱基的区域和上游引物F的5’端第1个碱基到第10个碱基区域存在10bp的碱基互补。(2)长Y引物的P7引物所示的单链DNA分子序列的任意一种和短Y接头的Y引物所示的单链DNA分子序列的任意一种形成部分双链得到。具体表现为引物P7的5’端第44个碱基到第53个碱基的区域和下游引物R的5’端第18个碱基到第27个碱基区域存在10bp的碱基重合。如表2和表3所示,由于P5序列有8种,P7序列有12种,可以满足同时构建8×12=96个文库的需要。双标签UMI的短Y接头上游引物F、下游引物R和长Y引物均合成自IDT公司,具体序列如表1、表2和表3所示。表1短Y接头的序列短Y接头序列(5’-3’)序列名称上游引物FCCTACACGACGCTCTTCCGATCNNNNT(27)SEQNo.1下游引物RNNNNGATCGGAAGAGCACACGTCTGAA(27)SEQNo.2表2长Y引物P5序列表3长Y引物P7序列表1中短Y接头的上游引物F和下游引物R可以通过退火形成部分双链结构,由于N可以为A、G、C、T四种碱基的任意一种,但是不能出现连续四个相同碱基的情况,因此实际的短Y接头的种类为44×44-4×4=65520种;另外上游引物F末端T可以与原始分子末端的A形成互补,从而发生连接反应。表2中长Y引物P5序列中加粗的碱基为indexD所在的位置,indexD有8种组合方式,在PCR过程中与上游引物F的第一连接序列互补,促进PCR的进行。表3中长Y引物P7序列中加粗的碱基为indexN所在的位置,indexN有12种组合方式,在PCR过程中与下游引物R的第二连接序列互补,促进PCR的进行。表2和表3引物在PCR过程中的不同组合用于区分不同样本,且在本发明中适用于illuminia的测序平台,如用其他测序平台需要更换相应的引物序列。实施例二含有本发明的核酸序列测序接头的文库构建和捕获(一)、文库构建ctDNA标准品购买自HorizonDiscovery公司,名称为MultiplexIcfDNAReferenceStandardSet,货号为HD780,ctDNA标准品的突变位点包括EGFR,KRAS,NRAS,PI3KCA基因的8个突变位点,本次实验使用的标准品突变频率为0.5%。根据不同建库起始量将实验分位组,分别取10ng、20ng或者30ng的标准品ctDNA进行样本文库构建,其中包括ctDNA末端修复、末端加A并且和短Y接头混合物的连接反应、磁珠纯化、利用长Y引物进行PCR扩增等过程。1.利用酶反应将提取好的质量较高的cfDNA样本进行末端修复及加A(相关生物酶来自商品化建库试剂盒KAPAHyperPrepKitIlluminaplatforms)。反应条件:20℃30min,65℃30min,4℃ever。2.将上述经过末端修复、加A的cfDNA和短Y接头进行连接反应,其中短Y接头储存液为15μM,根据不同建库起始量加入短Y接头使用量不同,10ng、20ng、30ng建库起始量分别加入1μL、2μL、3μL短Y接头。反应条件:20℃15min。连接后产物经过0.8×磁珠纯化后得到连接产物。3.将上一步的连接产物利用长Y引物进行PCR扩增反应条件:98℃45s,98℃15s;60℃30s;72℃30s,10-13cycles,72℃1min,4℃ever。PCR后产物经过0.5-0.9×磁珠筛选得到游离DNA样本文库。(二)目标区域捕获使用实验室自配试剂肺癌15基因的捕获体系进行目标区域捕获,具体捕获流程如下:1.游离DNA文库混合、封闭及干燥:将构建好的游离DNA文库按照数据量分配比例混合,总量1μg,随后将COTDNA、基因组DNA文库与封闭引物按照如下比例混合。其中COTDNA作为基因组中重复率较高的一部分DNA片段,在杂交时有助于提高杂交效率,封闭引物用来封闭文库中的测序接头。将上述混合好的样本在真空浓缩仪中60℃蒸干,用于后续杂交。2.重新溶解及变性:向上述干燥后的混合物中加入10.5μL的杂交缓冲液,充分涡旋溶解后于95℃变性10min。3.杂交:将变性好的10.5uL混合物加入到4.5μL杂交捕获探针文库中,涡旋30s充分溶解混匀后全速离心30s。于55℃杂交16h。4.生物亲和素磁珠捕获目标区域DNA文库片段:杂交反应后,将15uL样品转移至事先经过生物亲和素磁珠洗涤溶液洗涤重悬的100uL链霉亲和素标记磁珠中,混匀后于55℃孵育45min,每隔15分钟吹打混匀一次,让捕获的片段与磁珠结合,特异吸附目标片段,将杂交到的片段抓取出来。5.洗涤非目标区域DNA片段:①加入100μL55℃预热的洗涤溶液I,混匀后磁力悬浮弃上清,重复一次。②加入200μL55℃预热的Stringent洗涤溶液,混匀后55℃孵育5min,磁力悬浮弃上清,重复三次,共四次。③加入200μL洗涤溶液I,涡旋2min,磁力悬浮弃上清。④加入200μL洗涤溶液II,涡旋1min,磁力悬浮弃上清。⑤加入200μL洗涤溶液III,涡旋30S,磁力悬浮弃上清。⑥加入50μLPCR级别ddH2O重悬磁珠。6.捕获后富集:洗脱后磁珠带着捕获的目标DNA片段,进入样本的LM-PCR富集。LM-PCR反应体系如下(一个捕获样本做两管PCR反应):PCR反应条件为:98℃预变性45s;98℃变性15s,60℃退火30s,72℃延伸30s,共14个循环;72℃延伸1min;4℃保温。反应后,利用磁珠纯化PCR扩增产物,得到捕获后的样本文库,质检后用于测序分析。实施例三、测序结果分析与方法验证实施例1中制备的文库在illuminaNextSeq500测序仪上进行双端的151bp的测序,测序深度均在10000×以上。如表4所示,ctDNA标准品选择突变频率为0.5%的标准品。对于每一种突变频率的ctDNA标准品,建库投入的起始量分别为30ng、20ng和10ng三个不同的投入量。对于每一种突变频率的标准品的每种投入起始量在连接过程中分别加入短Y接头和普通接头,两者的差别在于前者的文库含有本发明接头标签序列,后者不含。表4建库标准品的条件设置标准品突变频率(%)建库方法投入起始量(ng)0.5%本发明接头100.5%普通接头100.5%本发明接头200.5%普通接头200.5%本发明接头300.5%普通接头30如表5和图2所示,是对以上设置条件的实验分析结果,我们发现在同一ctDNA建库起始量的条件下,本发明接头接头的阳性预测值(PPV)较普通接头有了大幅度的提高,尤其是随着ctDNA投入起始量的减少,本发明接头的优势更加明显,从30ng起始量的100%vs87.5%变化为10ng起始量的75%vs25%,从1.14倍增加到3倍之高。虽然在去duplex之前每一种ctDNA起始量的平均测序深度上,本发明接头和普通接头之间基本没有太大差别。但是在去duplication之后每一种ctDNA起始量的平均测序深度上,本发明接头都是普通接头的2倍,这也是本发明接头能够提高测序灵敏度的重要原因,并且能够减少假阴性的产生和干扰。表5ctDNA标准品的测序结果分析应该理解到披露的本发明不仅仅限于描述的特定的方法、方案和物质,因为这些均可变化。还应理解这里所用的术语仅仅是为了描述特定的实施方式方案的目的,而不是意欲限制本发明的范围,本发明的范围仅受限于所附的权利要求。本领域的技术人员还将认识到,或者能够确认使用不超过常规实验,在本文中所述的本发明的具体的实施方案的许多等价物。这些等价物也包含在所附的权利要求中。序列表<110>北京京诺玛特科技有限公司<120>核酸序列测序接头及其构建测序文库的方法<160>23<170>SIPOSequenceListing1.0<210>1<211>27<212>DNA<213>人工序列(ArtificialSequence)<400>1cctacacgacgctcttccgatcnnnnt27<210>2<211>27<212>DNA<213>人工序列(ArtificialSequence)<400>2nnnngatcggaagagcacacgtctgaa27<210>3<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>3aatgatacggcgaccaccgagatctacacatatgcgcacactctttccctacacgac57<210>4<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>4aatgatacggcgaccaccgagatctacactggtacagacactctttccctacacgac57<210>5<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>5aatgatacggcgaccaccgagatctacacaaccgttcacactctttccctacacgac57<210>6<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>6aatgatacggcgaccaccgagatctacactaaccggtacactctttccctacacgac57<210>7<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>7aatgatacggcgaccaccgagatctacacccttgtagacactctttccctacacgac57<210>8<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>8aatgatacggcgaccaccgagatctacacccttgtagacactctttccctacacgac57<210>9<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>9aatgatacggcgaccaccgagatctacactcaggcttacactctttccctacacgac57<210>10<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>10aatgatacggcgaccaccgagatctacacgttctcgtacactctttccctacacgac57<210>11<211>57<212>DNA<213>人工序列(ArtificialSequence)<400>11aatgatacggcgaccaccgagatctacacagaacgagacactctttccctacacgac57<210>12<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>12caagcagaagacggcatacgagatacgatcaggtgactggagttcagacgtgt53<210>13<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>13caagcagaagacggcatacgagattcgagagtgtgactggagttcagacgtgt53<210>14<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>14caagcagaagacggcatacgagatctagctcagtgactggagttcagacgtgt53<210>15<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>15caagcagaagacggcatacgagatatcgtctcgtgactggagttcagacgtgt53<210>16<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>16caagcagaagacggcatacgagattcgacaaggtgactggagttcagacgtgt53<210>17<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>17caagcagaagacggcatacgagatccttggaagtgactggagttcagacgtgt53<210>18<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>18caagcagaagacggcatacgagatatcatgcggtgactggagttcagacgtgt53<210>19<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>19caagcagaagacggcatacgagattgttccgtgtgactggagttcagacgtgt53<210>20<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>20caagcagaagacggcatacgagatattagccggtgactggagttcagacgtgt53<210>21<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>21caagcagaagacggcatacgagatcgatcgatgtgactggagttcagacgtgt53<210>22<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>22caagcagaagacggcatacgagatgatcttgcgtgactggagttcagacgtgt53<210>23<211>53<212>DNA<213>人工序列(ArtificialSequence)<400>23caagcagaagacggcatacgagataggatagcgtgactggagttcagacgtgt53当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1