异柠檬酸脱氢酶激酶突变体及其在制备芳香族氨基酸中的应用的制作方法
技术领域:
本发明属于生物技术与分子生物学领域,具体地涉及一种大肠杆菌编码的异柠檬酸脱氢酶激酶突变基因acek及该基因编码的氨基酸序列及其应用。
背景技术:
l-苯丙氨酸(l-phenylalanine)又名l-苯基-α-氨基丙酸,为白色结晶粉末,有苦味,熔点为283℃,在自然界中广泛存在于卵、乳和动物蛋白中,其含量5为%~6%,在植物性蛋白质中约含1%。苯丙氨酸可溶于水,在水中的溶解度为3%,难溶于乙醇、乙醚等有机溶剂,在100耐水中的溶解度为51℃:4.4g,等电点为5.48。苯丙氨酸有外消旋dl-型,l-型和d-型,其中最重要的是l-苯丙氨酸。
苯丙氨酸是8种必须氨基酸之一,其作为营养物质的同时也是体内的生糖兼生酮氨基酸,在代谢中有着不可代替的作用。在工业上,l-苯丙氨酸主要用于与天冬氨酸反应形成人工甜味剂阿斯巴甜。这种甜味剂在功能食品添加剂中有着广泛的需求。在医药领域上,l-苯丙氨酸是静脉输液的必需物质,同时可以用作许多抗肿瘤药物靶向供给的载体。目前全球市场l-苯丙氨酸年需求量超过50000吨,并且逐年以15%的速率稳定增长,其中阿斯巴甜的年需求量约为20000吨;另一方面,医药行业每年l-苯丙氨酸需求量约为8000吨。因此,l-苯丙氨酸具有巨大的市场开发空间(liux,niuh,liq,etal.metabolicengineeringfortheproductionofl-phenylalanineinescherichiacoli[j].3biotech,2019,9(3).)。
苯丙氨酸的制备方法有发酵法、酶法与化学合成法。用化学合成法经拆分可制得苯丙氨酸。化学合成法用苯甲醛和n-乙酞甘氨酸为原料,合成n-乙酞苯丙氨酸,再经氨基酞化酶拆分得l-苯丙氨酸。用氯节和丙二酸二乙醋或乙酞氨基丙二酸二乙醋或乙酞氨基氮基乙酸乙醋为原料,经氨基酞化酶水解制成l-苯丙氨酸和d-苯丙氨酸。国内生产厂家主要采用发酵法。苯丙氨酸的合成途径目前比较清晰,大肠杆菌利用自身的pts系统将外源的葡萄糖转运至细胞内,同时将其磷酸化为六磷酸葡萄糖。磷酸化后的葡萄糖一部分通过糖酵解途径(emp)进入三羧酸循环产生atp为机体的生命活动提供能量和一些中间物质(作为前体物为参与机体其他物质的合成),其中就包括磷酸烯醇式丙酮酸(pep),另一部分则进入磷酸戊糖非氧化还原途径(pp)产生赤藓糖4-磷酸(e4p),pep和e4p经过莽草酸途径生成分支酸并最终生成苯丙氨酸。苯丙氨酸的生物合成受其自身的反馈抑制和反馈阻遏。苯丙氨酸的生产菌株的代谢工程改造策略主要包括以下几部分内容:1.增加前体pep和e4p的供给;2.增强莽草酸途径;3.降低其他芳香族氨基酸竞争途径通量;4.解除反馈阻遏和反馈抑制;5.增强苯丙氨酸外排;6.敲除苯丙氨酸去路途径。(rodrigueza,martínez,juana,flores,noemí,etal.engineeringescherichiacolitooverproducearomaticaminoacidsandderivedcompounds[j].microbialcellfactories,2014,13(1):126.)
目前很少有通过改造中央碳代谢tca循环相关酶的活性进而有效提高苯丙氨酸产量的报道。异柠檬酸脱氢酶激酶/磷酸酶acek(由acek编码)处在两个中央代谢途径:tca循环和乙醛酸循环之间的分支点的位置,它通过控制tca循环中异柠檬酸脱氢酶(idh)的活性来控制微生物体内代谢流在这两个代谢途径中的分布,具体表现为acek通过磷酸化idh并使其失活使其底物异柠檬酸转移到乙醛酸循环中。当生物需要tca提供atp时acek通过使idh去磷酸化重新激活该酶,回复其对异柠檬酸的强亲和力并重新提高tca循环的通量。上述调控是由胞内的amp诱导acek的构象转换并决定acek是激酶还是磷酸化酶。acek作用于idh活性位点的丝氨酸残基。目前尚未有大肠杆菌编码异柠檬酸脱氢酶激酶突变基因acek用于选育芳香族氨基酸高产菌株的报道。
技术实现要素:
本发明人在前期的工作中构建了一种大肠杆菌高产苯丙氨酸的高通量筛选方法,通过对一株苯丙氨酸菌株进行诱变后筛选获得一株苯丙氨酸产量提升的突变菌,再挖掘分析其中对苯丙氨酸高产相关的突变基因,并做了进一步研究,最终完成本发明。
本发明提供一种异柠檬酸脱氢酶激酶的突变体,其氨基酸序列在如seqidno:1所示氨基酸序列存在下述突变:第26位氨基酸突变为k。
本发明进一步提供编码如上述异柠檬酸脱氢酶激酶的突变体的编码基因。更具体地,其核苷酸序列如seqidno:3所示。
本发明尤其提供异柠檬酸脱氢酶激酶编码基因在制备芳香族氨基酸中的应用。
在一个具体实施方式中,其是通过含有所述异柠檬酸脱氢酶激酶导入大肠杆菌生产芳香族氨基酸的工程菌的基因组中。
其中,所述异柠檬酸脱氢酶激酶编码基因优选编码具有seqidno:1或seqidno:2所示氨基酸序列的多肽。更优选地,所述异柠檬酸脱氢酶激酶编码基因具有seqidno:3所示核苷酸序列。
本发明构建的包含有大肠杆菌编码异柠檬酸脱氢酶激酶突变基因acek的工程菌生物安全,遗传背景清晰,可以有效的提高大肠杆菌生产苯丙氨酸等芳香族氨基酸的能力,在摇瓶水平可提高苯丙氨酸积累水平达12.1%以上,可提高色氨酸积累水平11.1%以上。
附图说明
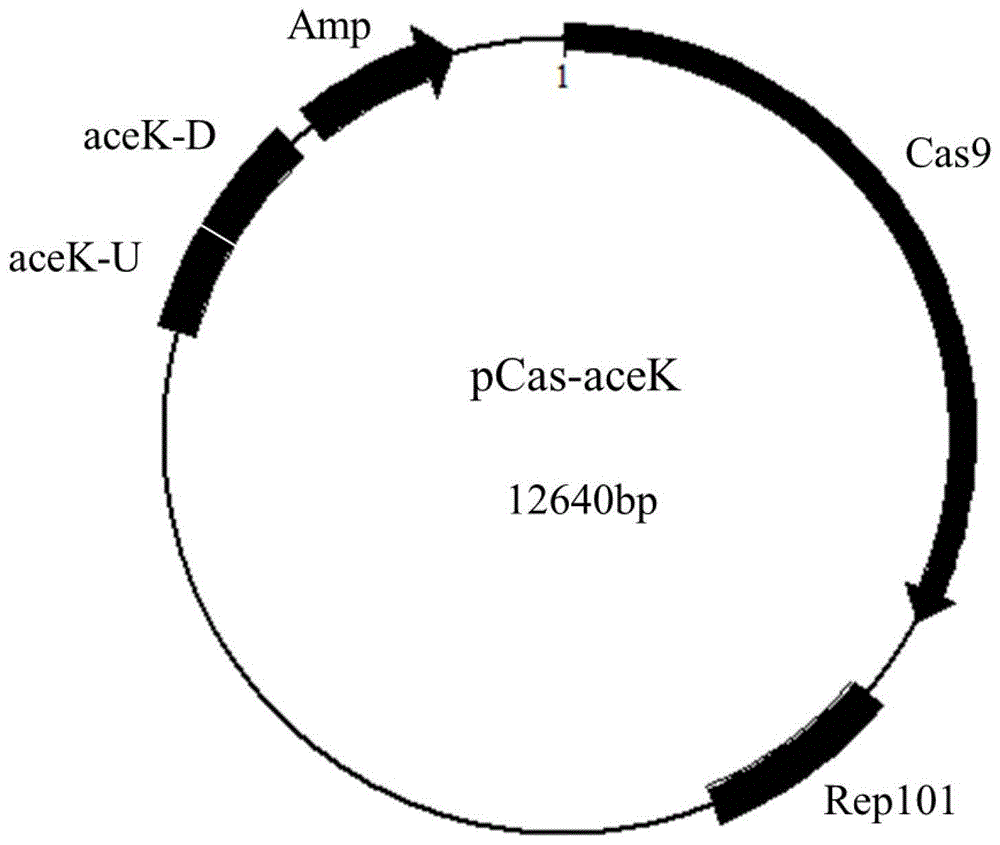
图1为用于基因组编辑的质粒pcas-acek的图谱。
图2为大肠杆菌苯丙氨酸工程菌株hdh5和hdh5-k和为色氨酸工程菌株kw002和kw002-k摇瓶水平发酵验证结果。
具体实施方式
本发明人在前期的工作中发明了一种基于苯丙氨酸的高通量的筛选方法,并利用该方法筛选出多株比出发菌的苯丙氨酸产量不同程度上提高的突变株(liu,y.,zhuang,y.,ding,d.,xu,y.,sun,j.,andzhang,d.(2017)biosensor-basedevolutionandelucidationofabiosyntheticpathwayinescherichiacoli,acssyntheticbiology,2017may19;6(5):837-848.doi:10.1021/acssynbio.6b00328.),该苯丙氨酸高产菌株的基因组测序结果已经公开发表。测序后发现acek基因的突变型e26k对苯丙氨酸产量提高具有重要作用。将该突变引入本实验保存的一株大肠杆菌高产色氨酸工程菌株中发现在摇瓶水平也可显著提升色氨酸发酵产量。本专利中提及的苯丙氨酸菌株hdh5菌株和kw002菌株的背景及相关操作已在发表的论文中详细说明(liu,y.,zhuang,y.,ding,d.,xu,y.,sun,j.,andzhang,d.biosensor-basedevolutionandelucidationofabiosyntheticpathwayinescherichiacoli,acssyntheticbiology,2017may19;6(5):837-848.doi:10.1021/acssynbio.6b00328;chen,y.,liu,y.,ding,d.,cong,l.,andzhang,d.(2018)rationaldesignandanalysisofanescherichiacolistrainforhigh-efficiencytryptophanproduction,jindmicrobiolbiotechnol,2018may;45(5):357-367.doi:10.1007/s10295-018-2020-x.)。
本发明中的“芳香族氨基酸工程菌株”是指以大肠杆菌或者谷氨酸棒状杆菌等工业菌株,经过适当的培养条件,在适当的培养基中发酵后可以生产苯丙氨酸或者色氨酸的菌株,优先特指大肠杆菌。目前通过本领域的科研人员掌握的各种常规的改造手段获取的大肠杆菌或者谷氨酸棒状杆菌菌株均为芳香族氨基酸工程菌株。
下面结合实例对本发明做进一步说明,下述实施例是为了使本领域的技术人员能够更好的理解本发明,单对本发明不作任何限制。
实施例1.构建pcas-acek
本实施例中构建的pcas-acek质粒用于将大肠杆菌色氨酸和苯丙氨酸工程菌株基因组上acek野生型基因替换为acek突变型(e26k),该质粒的具体工作原理已公开发表(zhaod,yuans,xiongb,sunh,yel,lij,zhangx,bic.developmentofafastandeasymethodforescherichiacoligenomeeditingwithcrispr/cas9..2016dec1;15(1):205.)’。pcas-red的具体构建过程描述如下:将cas9蛋白(以含有cas9蛋白的质粒(addgene编号为:42876)),合成不含有n20序列的grna蛋白(序列为:5’-gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgctt-3’)以及它的启动子序列(5’-ctaggtttatacataggcgagtactctgttatggagtcagatct-3’)和来自pkd46质粒(kirilla.datsenkoandbarryl.wanner,one-stepinactivationofchromosomalgenesinescherichiacolik-12usingpcrproducts,pnasjune6,200097(12)6640-6645;https://doi.org/10.1073/pnas.120163297)的大肠杆菌同源重组系统这三部分利用吉布森组装整合为一个质粒,并命名为pcas-red。
pcas-acek的质粒是由pcas-red衍生而来,在同源臂及n20序列等进行针对acek基因进行特异性改造,构建过程如下:以pcas-red质粒为模板,以pcas-acek-cas-lf、pcas-acek-cas-lr为引物,pcr扩增出质粒片段pcas1,pcr体系见表1。以pcas-red质粒为模板,以pcas-acek-n20-lf、pcas-acek-n20-lr为引物,pcr扩增出质粒片段pcas2。以大肠杆菌w3110基因组为模板,以pcas-acek-lf为上游引物,以pcas-acek-mut-lr为下游引物,pcr扩增片段分别为acek-u;以大肠杆菌w3110基因组为模板,以pcas-acek-mut-lf和pcas-acek-lr为引物,pcr扩增片段分别为acek-d。按照表2的体系进行吉布森组装,反应条件为50℃,1h。将10μl连接体系转入dh5α感受态细胞中于30℃利用lb培养基过夜培养,挑选阳性克隆后送金唯智公司测序进行验证,构建成功的质粒命名为pcas-acek。本实例中所用引物见表3.
表1.pcr反应体系
表2.吉布森连接反应体系
表3.构建pcas-acek质粒所需引物
实施例2.构建hdh5-k
将大肠杆菌苯丙氨酸工程菌hdh5制备成感受态,将pcas-acek质粒转入感受态细胞后涂布于含有氨苄抗性的平板上30°过夜培养。挑取阳性单菌落接种到含有氨苄lb液体培养基(胰蛋白胨10g/l,酵母提取物5g/l,氯化钠10g/l)试管中,在30℃摇床下培养6h后加入2g/l的阿拉伯糖(诱导cas9蛋白和pkd46上的重组蛋白的表达),促进cas9蛋白的筛选。然后继续在30℃摇床下培养6h,将菌液在含有氨苄和2g/l阿拉伯糖的lb平板上进行三区划线,30℃下恒温过夜培养。利用菌落pcr进行验证并进行测序,测序正确的菌株分别命名hdh5-k。
实施例3.构建kw002-k
将大肠杆菌苯丙氨酸工程菌kw002制备成感受态,将pcas-acek质粒转入感受态细胞后涂布于含有氨苄抗性的平板上30°过夜培养。挑取阳性单菌落接种到含有氨苄lb试管中,在30℃摇床下培养6h后加入2g/l的阿拉伯糖(诱导cas9蛋白和pkd46重组蛋白的表达),促进cas9蛋白的筛选。然后继续在30℃摇床下培养6h,将菌液在含有氨苄和2g/l阿拉伯糖的lb平板上进行三区划线,30℃下恒温过夜培养。利用菌落pcr进行验证并进行测序,测序正确的菌株分别命名kw002-k。
实施例4.苯丙氨酸菌株hdh5和hdh5-k摇瓶发酵
大肠杆菌高产苯丙氨酸菌株hdh5和hdh5-k的摇瓶发酵工艺如下:
(1)斜面活化培养:从-80℃冰箱取出保藏菌种划线于含有卡纳抗性的lb固体培养基(胰蛋白胨10g/l,酵母提取物5g/l,氯化钠10g/l,琼脂15g/l),37℃培养12-18h。
(2)种子培养:用接种环从新鲜活化斜面上挑取2环大肠杆菌于lb培养基中(500ml三角瓶中装50mllb培养基,封口膜封口),37℃220r/min振荡培养6-8h至od600约为2-3。
(3)摇瓶分批发酵培养:将种子培养液按10%的接种量接入含有卡纳抗性发酵基本培养基中(500ml三角瓶,装液量为50ml,封口膜封口),37℃220r/min振荡培养进行苯丙氨酸分批发酵。发酵培养基如下:
表4.苯丙氨酸发酵培养基配方
发酵结束后,10000rpm离心10min取出上清,利用液相进行检测,检测方法如下:
hplc水相:去离子水,0.22μm孔径的水系微孔过滤膜过滤,超声30min。
hplc盐相:色谱级nah2po36.218g溶解于1l去离子水中,用0.22μm孔径的水系微孔过滤膜过滤,超声30min。
hplc有机相:色谱级甲醇、色谱级乙腈、去离子水体积比为4.5:4.5:1,用0.22μm有机系溶剂微孔过滤膜过滤,超声30min。
hplc程序采用梯度洗脱,洗脱程序为:
表5.hplc梯度洗脱程序
发酵结束后对所有的工程菌株进行苯丙氨酸产量的检测。发酵结果见图2。从结果可以看出,与出发菌株比较,含有大肠杆菌编码异柠檬酸脱氢酶激酶突变基因acek突变基因的大肠杆菌工程菌株后,苯丙氨酸的产量提高了12.1%,说明该修饰对于大肠杆菌生产苯丙氨酸具有明显的提升作用。
实施例5.色氨酸菌株kw002和kw002-k摇瓶发酵
大肠杆菌高产色氨酸菌株kw002和kw002-k的摇瓶发酵工艺如下:
(1)斜面活化培养:从-80℃冰箱取出保藏菌种划线于含有四环素抗性的固体lb培养基,37℃培养12-18h。
(2)种子培养:用接种环从新鲜活化斜面上挑取2环大肠杆菌于lb培养基中(500ml三角瓶中装50mllb培养基,封口膜封口),37℃220r/min振荡培养6-8h,od600约为2-3。
(3)摇瓶分批发酵培养:将种子培养液按10%的接种量接入含有四环素抗性发酵培养基中(500ml三角瓶,装液量为50ml,封口膜封口),37℃220r/min振荡培养进行色氨酸分批发酵。发酵培养基如下。
表6色氨酸发酵培养基配方
发酵结束后,10000rpm离心10min取出上清,利用液相进行检测,检测方法如下:
hplc水相:去离子水,0.22μm孔径的水系微孔过滤膜过滤,超声30min。
hplc盐相:色谱级nah2po36.218g溶解于1l去离子水中,用0.22μm孔径的水系微孔过滤膜过滤,超声30min。
hplc有机相:色谱级甲醇、色谱级乙腈、去离子水体积比为4.5:4.5:1,用0.22μm有机系溶剂微孔过滤膜过滤,超声30min。
hplc程序采用梯度洗脱,洗脱程序为如表5所示。
发酵结束后对所有的工程菌株进行色氨酸产量的检测。发酵结果见图2。从结果可以看出,与出发菌株比较,含有大肠杆菌编码异柠檬酸脱氢酶激酶突变基因acek突变基因的大肠杆菌工程菌株后,色氨酸的产量提高了11.1%,说明该修饰对于大肠杆菌生产色氨酸具有明显的提升作用。
本发明的菌株的构建,其步骤的前后顺序不限定,本领域的技术人员按本发明公开的内容达到本发明的目的均属于本发明的保护范围。
本发明中的菌株代号如hdh5,hdh5-k,kw002和kw002-k等是为了方便描述,但不应理解为对本发明的限定。
上述方法构建的包含有大肠杆菌编码异柠檬酸脱氢酶激酶突变基因acek突变基因的工程菌的用途,包含但不局限于苯丙氨酸和色氨酸。
序列表
<110>中国科学院天津工业生物技术研究所
<120>异柠檬酸脱氢酶激酶突变体及其在制备芳香族氨基酸中的应用
<130>2019.11.5
<160>11
<170>siposequencelisting1.0
<210>1
<211>578
<212>prt
<213>escherichiacoli
<400>1
metproargglyleugluleuleuilealaglnthrileleuglngly
151015
pheaspalaglntyrglyargpheleugluvalthrserglyalagln
202530
glnargphegluglnalaasptrphisalavalglnglnalametlys
354045
asnargilehisleutyrasphishisvalglyleuvalvalglugln
505560
leuargcysilethrasnglyglnserthraspalaalapheleuleu
65707580
argvallysgluhistyrthrargleuleuproasptyrproargphe
859095
gluilealagluserphepheasnservaltyrcysargleupheasp
100105110
hisargserleuthrprogluargleupheilepheserserglnpro
115120125
gluargargpheargthrileproargproleualalysaspphehis
130135140
proasphisglytrpgluserleuleumetargvalileseraspleu
145150155160
proleuargleuargtrpglnasnlysserargaspilehistyrile
165170175
ilearghisleuthrgluthrleuglythraspasnleualagluser
180185190
hisleuglnvalalaasngluleuphetyrargasnlysalaalatrp
195200205
leuvalglylysleuilethrproserglythrleupropheleuleu
210215220
proilehisglnthraspaspglygluleupheileaspthrcysleu
225230235240
thrthrthralaglualaserilevalpheglyphealaargsertyr
245250255
phemetvaltyralaproleuproalaalaleuvalglutrpleuarg
260265270
gluileleuproglylysthrthralagluleutyrmetalailegly
275280285
cysglnlyshisalalysthrglusertyrargglutyrleuvaltyr
290295300
leuglnglycysasngluglnpheileglualaproglyilearggly
305310315320
metvalmetleuvalphethrleuproglypheaspargvalphelys
325330335
valilelysaspargphealaproglnlysglumetseralaalahis
340345350
valargalacystyrglnleuvallysgluhisaspargvalglyarg
355360365
metalaaspthrglngluphegluasnphevalleuglulysarghis
370375380
ileserproalaleumetgluleuleuleuglnglualaalaglulys
385390395400
ilethraspleuglygluglnilevalilearghisleutyrileglu
405410415
argargmetvalproleuasniletrpleugluglnvalgluglygln
420425430
glnleuargaspalailegluglutyrglyasnalaileargglnleu
435440445
alaalaalaasnilepheproglyaspmetleuphelysasnphegly
450455460
valthrarghisglyargvalvalphetyrasptyraspgluilecys
465470475480
tyrmetthrgluvalasnpheargaspileproproproargtyrpro
485490495
gluaspgluleualasergluprotrptyrservalserproglyasp
500505510
valpheproglugluphearghistrpleucysalaaspproargile
515520525
glyproleuphegluglumethisalaaspleupheargalaasptyr
530535540
trpargalaleuglnasnargilearggluglyhisvalgluaspval
545550555560
tyralatyrargargargglnargpheservalargtyrglyglumet
565570575
leuphe
<210>2
<211>578
<212>prt
<213>escherichiacoli
<400>2
metproargglyleugluleuleuilealaglnthrileleuglngly
151015
pheaspalaglntyrglyargpheleulysvalthrserglyalagln
202530
glnargphegluglnalaasptrphisalavalglnglnalametlys
354045
asnargilehisleutyrasphishisvalglyleuvalvalglugln
505560
leuargcysilethrasnglyglnserthraspalaalapheleuleu
65707580
argvallysgluhistyrthrargleuleuproasptyrproargphe
859095
gluilealagluserphepheasnservaltyrcysargleupheasp
100105110
hisargserleuthrprogluargleupheilepheserserglnpro
115120125
gluargargpheargthrileproargproleualalysaspphehis
130135140
proasphisglytrpgluserleuleumetargvalileseraspleu
145150155160
proleuargleuargtrpglnasnlysserargaspilehistyrile
165170175
ilearghisleuthrgluthrleuglythraspasnleualagluser
180185190
hisleuglnvalalaasngluleuphetyrargasnlysalaalatrp
195200205
leuvalglylysleuilethrproserglythrleupropheleuleu
210215220
proilehisglnthraspaspglygluleupheileaspthrcysleu
225230235240
thrthrthralaglualaserilevalpheglyphealaargsertyr
245250255
phemetvaltyralaproleuproalaalaleuvalglutrpleuarg
260265270
gluileleuproglylysthrthralagluleutyrmetalailegly
275280285
cysglnlyshisalalysthrglusertyrargglutyrleuvaltyr
290295300
leuglnglycysasngluglnpheileglualaproglyilearggly
305310315320
metvalmetleuvalphethrleuproglypheaspargvalphelys
325330335
valilelysaspargphealaproglnlysglumetseralaalahis
340345350
valargalacystyrglnleuvallysgluhisaspargvalglyarg
355360365
metalaaspthrglngluphegluasnphevalleuglulysarghis
370375380
ileserproalaleumetgluleuleuleuglnglualaalaglulys
385390395400
ilethraspleuglygluglnilevalilearghisleutyrileglu
405410415
argargmetvalproleuasniletrpleugluglnvalgluglygln
420425430
glnleuargaspalailegluglutyrglyasnalaileargglnleu
435440445
alaalaalaasnilepheproglyaspmetleuphelysasnphegly
450455460
valthrarghisglyargvalvalphetyrasptyraspgluilecys
465470475480
tyrmetthrgluvalasnpheargaspileproproproargtyrpro
485490495
gluaspgluleualasergluprotrptyrservalserproglyasp
500505510
valpheproglugluphearghistrpleucysalaaspproargile
515520525
glyproleuphegluglumethisalaaspleupheargalaasptyr
530535540
trpargalaleuglnasnargilearggluglyhisvalgluaspval
545550555560
tyralatyrargargargglnargpheservalargtyrglyglumet
565570575
leuphe
<210>3
<211>1737
<212>dna
<213>escherichiacoli
<400>3
atgccgcgtggcctggaattattgattgctcaaaccattttgcaaggcttcgatgctcag60
tatggtcgattcctcaaagtgacctccggtgcgcagcagcgtttcgaacaggccgactgg120
catgctgtccagcaggcgatgaaaaaccgtatccatctttacgatcatcacgttggtctg180
gtcgtggagcaactgcgctgcattactaacggccaaagtacggacgcggcatttttacta240
cgtgttaaagagcattacacccggctgttgccggattacccgcgcttcgagattgcggag300
agcttttttaactccgtgtactgtcggttatttgaccaccgctcgcttactcccgagcgg360
ctttttatctttagctctcagccagagcgccgctttcgtaccattccccgcccgctggcg420
aaagactttcaccccgatcacggctgggaatctctactgatgcgcgttatcagcgaccta480
ccgctgcgcctgcgctggcagaataaaagccgtgacatccattacattattcgccatctg540
acggaaacgctggggacagacaacctcgcggaaagtcatttacaggtggcgaacgaactg600
ttttaccgcaataaagccgcctggctggtaggcaaactgatcacaccttccggcacattg660
ccatttttgctgccgatccaccagacggacgacggcgagttatttattgatacctgcctg720
acgacgaccgccgaagcgagcattgtttttggctttgcgcgttcttattttatggtttat780
gcgccgctgcccgcagcgctggtcgagtggctacgggaaattctgccaggtaaaaccacc840
gctgaattgtatatggctatcggctgccagaagcacgccaaaaccgaaagctaccgcgaa900
tatctcgtttatctacagggctgtaatgagcagttcattgaagcgccgggtattcgtgga960
atggtgatgttggtgtttacgctgccgggctttgatcgggtattcaaagtcatcaaagac1020
aggttcgcgccgcagaaagagatgtctgccgctcacgttcgtgcctgctatcaactggtg1080
aaagagcacgatcgcgtgggccgaatggcggacacccaggagtttgaaaactttgtgctg1140
gagaagcggcatatttccccggcattaatggaattactgcttcaggaagcagcggaaaaa1200
atcaccgatctcggcgaacaaattgtgattcgccatctttatattgagcggcggatggtg1260
ccgctcaatatctggctggaacaagtggaaggtcagcagttgcgcgacgccattgaagaa1320
tacggtaacgctattcgccagcttgccgctgctaacattttccctggcgacatgctgttt1380
aaaaacttcggtgtcacccgtcacgggcgtgtggttttttatgattacgatgaaatttgc1440
tacatgacggaagtgaatttccgcgacatcccgccgccgcgctatccggaagacgaactt1500
gccagcgaaccgtggtacagcgtctcgccgggcgatgttttcccggaagagtttcgccac1560
tggctatgcgccgacccgcgtattggtccgctgtttgaagagatgcacgccgacctgttc1620
cgcgctgattactggcgcgcactacaaaaccgcatacgtgaagggcatgtggaagatgtt1680
tatgcgtatcggcgcaggcaaagatttagcgtacggtatggggagatgcttttttga1737
<210>4
<211>36
<212>dna
<213>artificialsequence
<400>4
aatccatgggcctgtactggcgcgtcgctttgcaca36
<210>5
<211>29
<212>dna
<213>artificialsequence
<400>5
ccggaggtcactttgaggaatcgaccata29
<210>6
<211>28
<212>dna
<213>artificialsequence
<400>6
tatggtcgattcctcaaagtgacctccg28
<210>7
<211>29
<212>dna
<213>artificialsequence
<400>7
ccattcacaggcaggtatcaataaataac29
<210>8
<211>32
<212>dna
<213>artificialsequence
<400>8
gatacctgcctgtgaatggaagcttggattct32
<210>9
<211>39
<212>dna
<213>artificialsequence
<400>9
tactgagcatcgaagccttggctaagatctgactccata39
<210>10
<211>46
<212>dna
<213>artificialsequence
<400>10
caaggcttcgatgctcagtagttttagagctagaaatagcaagtta46
<210>11
<211>35
<212>dna
<213>artificialsequence
<400>11
cgccagtacaggcccatggattcttcgtctgtttc35
- 还没有人留言评论。精彩留言会获得点赞!