检测菌群的试剂在制备食管鳞癌患者预后预测标志物的试剂或试剂盒中的应用的制作方法
本发明涉及食管鳞癌预后检测技术领域,尤其涉及检测菌群的试剂在制备食管鳞癌患者预后预测标志物的试剂或试剂盒中的应用。
背景技术:
食管癌是中国最常见的严重危害人民健康的恶性肿瘤之一,95%为食管鳞癌,具有恶性程度高、转移快、术后生存质量差及明显的区域发病率等特点。
现有技术中,gao等通过qpcr测序技术检测了食管鳞癌患者癌组织菌群分布情况,结果发现牙龈卟啉单胞菌在癌组织当中丰度较高,且牙龈卟啉单胞菌是食管鳞癌患者预后的影响因素,相比于牙龈卟啉单胞菌阴性患者,阳性患者的生存率较低。yamamura等通过qpcr测序检测了食管鳞癌患者癌组织具核梭菌的分布情况,结果显示具核梭菌在癌组织当中丰度较高,相比于具核梭菌检测阴性患者,阳性患者的生存率较低,具核梭菌是食管鳞状细胞癌患者预后的危险因素。但是仅对某一种特定的菌进行预后分析,并未考虑组织中其他菌群的影响,存在鉴定指标单一,代表性不足的问题。
技术实现要素:
本发明的目的在于提供检测菌群的试剂在制备食管鳞癌预后预测标志物的试剂或试剂盒中的应用,利用本发明的检测菌群的试剂对食管鳞癌预后进行检测,代表性好,结果可靠。
为了实现上述发明目的,本发明提供以下技术方案:
本发明提供了检测菌群的试剂在制备食管鳞癌患者预后预测标志物的试剂或试剂盒中的应用,所述菌群包括食管鳞癌患者癌组织中克罗诺杆菌属(cronobacter)、食管鳞癌患者癌旁组织中盐单胞菌科(familyhalomonadaceae)和食管鳞癌患者癌旁组织中肉杆菌菌属(carnobacterium)中的一种或几种。
本发明提供了检测肉杆菌菌属(carnobacterium)的试剂在制备食管鳞癌患者术后化疗预后预测标志物的试剂或试剂盒中的应用。
本发明提供了一种食管鳞癌患者预后预测标志物,所述食管鳞癌患者预后预测标志物包括食管鳞癌患者癌组织中克罗诺杆菌属(cronobacter)、食管鳞癌患者癌旁组织中盐单胞菌科(familyhalomonadaceae)和食管鳞癌患者癌旁组织中肉杆菌菌属(carnobacterium)中的一种或几种。
本发明提供了一种食管鳞癌患者术后化疗预后预测标志物,所述食管鳞癌患者术后化疗预后预测标志物包括肉杆菌菌属(carnobacterium)。
本发明的有益效果:本发明提供了检测菌群的试剂在制备食管鳞癌患者预后预测标志物的试剂或试剂盒中的应用,所述菌群包括食管鳞癌患者癌组织中克罗诺杆菌属(cronobacter)、食管鳞癌患者癌旁组织中盐单胞菌科(familyhalomonadaceae)和食管鳞癌患者癌旁组织中肉杆菌菌属(carnobacterium)中的一种或几种;所述克罗诺杆菌属(cronobacter)为食管鳞癌患者的癌组织中菌群,与食管鳞癌患者的预后有关,克罗诺杆菌属(cronobacter)阳性患者的预后较好(hr=0.436,95%ci:0.272-0.698);所述盐单胞菌科(familyhalomonadaceae)为食管鳞癌患者的癌旁组织中菌群,与食管鳞癌患者的预后有关,盐单胞菌科阳性患者的预后较好(hr=0.161,95%ci:0.058-0.447);所述肉杆菌菌属(carnobacterium)为食管鳞癌术后化疗患者的癌旁组织中菌群,与食管鳞癌术后化疗患者预后有关,未检出肉杆菌菌属(carnobacterium)的患者,术后化疗者预后劣于单纯手术者(hr=2.650,95%ci=1.199-5.856,p=0.016);而对于体内存在肉杆菌菌属(carnobacterium)且接受手术+化疗的患者的预后较好(hr=0.150,95%ci=0.033-0.686,p=0.014),因此临床使用化疗治疗食管鳞癌术后患者,可以参考其体内是否存在肉杆菌菌属(carnobacterium)进行选择是否选择化疗方案,为实现食管鳞癌患者术后的精准化治疗提供参考依据。本发明为食管鳞癌预后预测提供了微生物相关证据,为食管鳞癌患者预后预测提供了参考依据。
附图说明
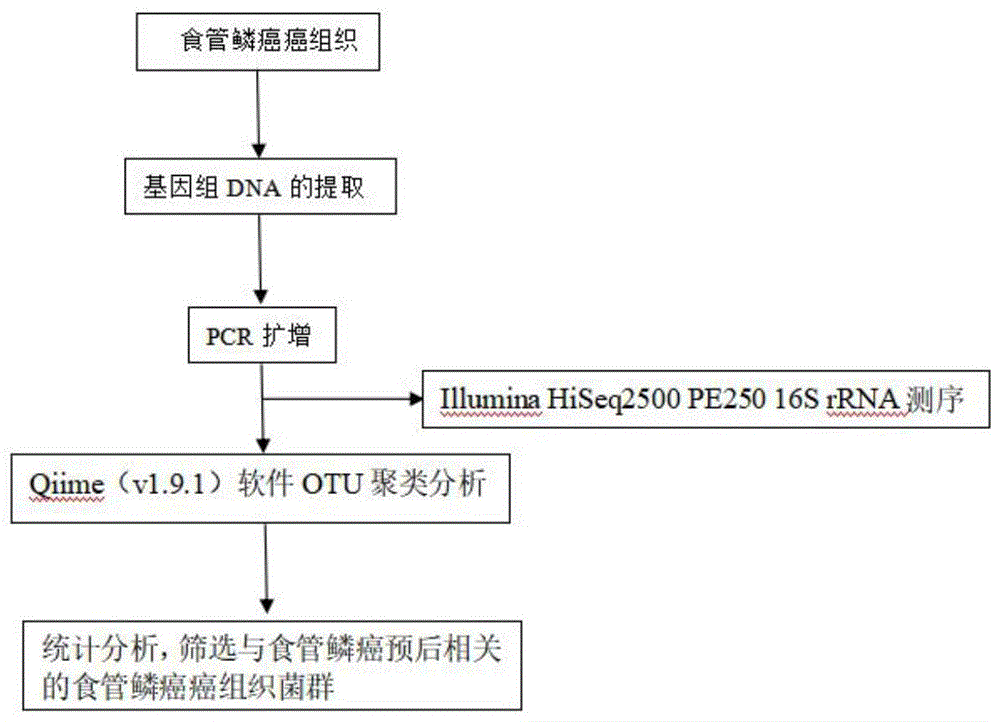
图1为实施例1流程图;
图2为实施例1中部分食管鳞癌患者的癌组织基因组dna检测图,其中编号为a25~a30癌组织样本对应的泳道为3~8泳道;
图3为实施例1中部分食管鳞癌患者的癌旁组织基因组dna检测图,其中编号为b1~b5癌旁组织样本对应的泳道为3~7泳道;
图4为实施例1中部分食管鳞癌患者的癌组织16srrnav3-v4区检测图,其中编号为a37,a59,a58,a99癌组织样本对应的泳道为2~5泳道;
图5为实施例1中部分食管鳞癌患者的癌旁组织16srrnav3-v4区检测图,其中编号为b31、b59、b32和b43癌旁组织样本对应的泳道为2~4、6泳道;
图6-a为实施例1中癌组织中产碱菌科(alcaligenaceae)分布情况不同患者生存曲线;
图6-b为实施例1中癌组织中克罗诺杆菌属(cronobacter)分布情况不同患者生存曲线;
图6-c为实施例1中癌组织中瘤胃球菌属(ruminococcus)分布情况不同患者生存曲线;
图6-d为实施例1中癌组织中气球菌属(aerococcus)分布情况不同患者生存曲线;
图6-e为实施例1中癌组织中都柏林克洛诺斯氏菌(cronobacterdublinensis)分布情况不同患者生存曲线;
图6-f为实施例1中癌组织中水草盐孢菌(salinisporatropica)分布情况不同患者生存曲线;
图6-g为实施例1中癌组织中卡托氏菌属(catonella)分布情况不同患者生存曲线;
图6-h为实施例1中癌组织中坦纳氏普拉沃氏菌(prevotellatannerae)分布情况不同患者生存曲线;
图7为实施例2流程图;
图8为实施例2中部分食管鳞癌患者癌组织的基因组dna检测图,其中编号为a25~29癌组织样本对应的泳道为3~7泳道;
图9为实施例2中部分食管鳞癌患者癌旁组织的基因组dna检测图,其中编号为b16~20癌旁组织样本对应的泳道为3~7泳道;
图10为实施例2中部分食管鳞癌患者16srrnav3-v4区检测图,其中编号为b18,b29,b19,b20癌旁组织样本对应的泳道为2,3,4,6泳道;
图11-a为实施例2中癌旁组织中间孢囊菌科(intrasporangiaceae)分布情况不同患者生存曲线;
图11-b为实施例2中癌旁组织中间生丝微菌科(hyphomicrobiaceae)分布情况不同患者生存曲线;
图11-c为实施例2中癌旁组织中盐单胞菌科(halomonadaceae)分布情况不同患者生存曲线;
图11-d为实施例2中癌旁组织中硝化螺旋菌属(nitrospira)分布情况不同患者生存曲线;
图11-e为实施例2中癌旁组织中甲基杆菌属(methylobacterium)分布情况不同患者生存曲线;
图11-f为实施例2中癌旁组织中克罗诺杆菌属(cronobacter)分布情况不同患者生存曲线;
图11-g为实施例2中癌旁组织中普氏菌(prevotellacopri)分布情况不同患者生存曲线;
图11-h为实施例2中癌旁组织中嗜热嗜盐厌氧菌(anaerophagathermohalophila)分布情况不同患者生存曲线;
图11-i为实施例2中癌旁组织中高丰度线粒体菌科(mitochondria)分布情况不同患者生存曲线;
图11-j为实施例2中癌旁组织中高丰度月形单胞菌属(selenomonas)分布情况不同患者生存曲线;
图12为实施例3流程图;
图13为实施例3中部分食管鳞癌患者癌组织的基因组dna检测图,其中编号为a49~53癌组织样本对应的泳道为3~7泳道;
图14为实施例4中部分食管鳞癌患者癌旁组织的基因组dna检测图,其中编号为b31~35癌旁组织样本对应的泳道为3~7泳道;
图15为实施例3中部分食管鳞癌患者癌旁组织的16srrnav3-v4区检测图,其中编号为b1,b29,b2,b13癌旁组织样本对应的泳道为2,3,4,6泳道。
具体实施方式
本发明提供了检测菌群的试剂在制备食管鳞癌患者预后预测标志物的试剂或试剂盒中的应用,所述菌群包括食管鳞癌患者癌组织中克罗诺杆菌属(cronobacter)、食管鳞癌患者癌旁组织中盐单胞菌科(familyhalomonadaceae)和食管鳞癌患者癌旁组织中肉杆菌菌属(carnobacterium)中的一种或几种。
本发明提供了检测肉杆菌菌属(carnobacterium)的试剂在制备食管鳞癌患者术后化疗预后预测标志物的试剂或试剂盒中的应用。
本发明提供了一种食管鳞癌患者预后预测标志物,所述食管鳞癌患者预后预测标志物包括食管鳞癌患者癌组织中克罗诺杆菌属(cronobacter)、食管鳞癌患者癌旁组织中盐单胞菌科(familyhalomonadaceae)和食管鳞癌患者癌旁组织中肉杆菌菌属(carnobacterium)中的一种或几种。
本发明提供了一种食管鳞癌患者术后化疗预后预测标志物,所述食管鳞癌患者术后化疗预后预测标志物包括肉杆菌菌属(carnobacterium)。
本发明中,所述克罗诺杆菌属(cronobacter)为食管鳞癌患者的癌组织中菌群,与食管鳞癌患者的预后有关,克罗诺杆菌属(cronobacter)阳性患者的预后较好;所述盐单胞菌科(familyhalomonadaceae)为食管鳞癌患者的癌旁组织中菌群,与食管鳞癌患者的预后有关,盐单胞菌科阳性患者的预后较好;所述肉杆菌菌属(carnobacterium)为食管鳞癌术后化疗患者的癌旁组织中菌群,与食管鳞癌术后化疗患者预后有关,未检出肉杆菌菌属(carnobacterium)的患者,术后化疗者预后劣于单纯手术者。
下面将结合本发明中的实施例,对本发明中的技术方案进行清楚、完整地描述。显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1
一、流程图参见图1。
二、操作步骤
1、食管鳞癌患者筛选
选取2013年2月~2017年10月就诊于福建省肿瘤医院和福建省漳州市医院的食管鳞癌患者120名。纳入标准:接受食管癌根治性手术治疗;所有研究对象均经过手术病理确诊患有食管鳞状细胞癌;术前均未接受放疗或者化疗等治疗;术前2个月无抗生素使用记录;无其他感染性疾病。排除标准:临床病理资料、组织标本不完整者;转移性肿瘤和食管癌复发病例;存在严重心脑器质性病变、精神病史等患者。
2、方法
2.1组织样本采集
使用手术室无菌剪刀分别剪取每位食管鳞癌患者的癌组织,剪取不同组织时更换剪刀,防止交叉污染。将组织剪成小块后分别放入高压灭菌的冻存管中,于1h之内置于液氮中储存,之后转入-80℃冰箱储存备用。所有耗材均经高压消毒灭菌。癌组织标本均经过病理苏木素-伊红染色(hematoxylin-eosinstaining,he)确诊为食管鳞状细胞癌,同时并未在癌旁组织标本中检测到有肿瘤细胞存在。
2.2基因组dna的提取与检测
采用sds法提取癌组织中菌群的dna。
(1)将样本从-80℃超低温冰箱中取出,使用无菌剪刀剪取50~100mg组织,放入1.5毫升ep管中,并加入1ml的dnazol,使用匀浆机处理5-10次。将经匀浆机处理后的ep管放入离心机当中,10000g离心10min,将得到的上清液转入新管当中。
(2)在离心好的ep管中加入edta(0.5m,ph8.0)和50μl溶菌酶,混匀后加入proteinasek,颠倒混匀,55℃孵育2h。
(3)12000rpm离心5min,吸取上清,加入蛋白沉淀液,轻柔涡旋10s,-20℃放置数分钟,12000rpm离心10min。
(4)吸取上清至新的ep管中,12000rpm离心5min,管底可能还有微量蛋白沉淀。吸取上清至新的ep管中,加入异丙醇,混匀-20℃放置20min后,12000rpm离心10min。
(5)倒出液体,用1ml75%乙醇洗涤两次,剩余的少量液体可再次离心收集,然后用枪头吸出。超净工作台吹干或者室温晾干。
(6)加入50μl的ddh2o溶解dna样品,振荡器助溶。加入1μl的rnasea,颠倒混匀后37℃温育15min。
(7)使用qubit仪对提取的dna进行定量检测,检测结果均合格。使用1%琼脂糖凝胶电泳检测:电压100v,40min。uvi凝胶成像系统拍照记录:dna电泳未出现杂带、拖尾,说明dna片段纯度良好,无明显降解,见图2和图3,其中图2为部分食管鳞癌患者的癌组织基因组dna检测图,其中编号为a25~a30癌组织样本对应的泳道为3~8泳道;图3为部分食管鳞癌患者的癌旁组织基因组dna检测图,其中编号为b1~b5癌旁组织样本对应的泳道为3~7泳道。取适量样品于离心管当中,使用无菌水稀释样品至1ng/μl。dna于-20℃冰箱内储存备用。
2.3pcr扩增
以稀释后的基因组dna为模板,根据测序区域的选择,使用带barcode的特异引物,采用细菌通用引物341f(cctaygggrbgcascag,如seqidno.1所示)和806r(ggactacnngggtatctaat,如seqidno.2所示)扩增细菌16srrna基因的v3-v4区域。使用
2.4pcr扩增产物混样与纯化
根据pcr产物浓度进行等质量混样,充分混匀后使用1×tae浓度2%的琼脂糖胶电泳纯化pcr产物,选择主带大小在400-450bp之间的序列,割胶回收目标条带。产物纯化试剂盒使用的是thermoscientific公司genejet胶回收试剂盒。
2.5文库构建与上机测序
使用illumina公司truseqdnapcr-freelibrarypreparationkit的建库试剂盒进行文库的构建,构建好的文库经过qubit定量和文库检测,合格后,使用illuminahiseq2500pe250测序平台进行菌群测序。
2.6测序数据的预处理
对食管鳞癌患者的食管菌群测序的原始数据原始数据质量控制具体步骤:1.数据拼接:去除3’端引物接头,根据pereads之间的重复关系将成对的序列拼接成一条序列。2.割库:通过标签序列(barcode)区分样品序列,并去除barcode和低质量、未检出的碱基。barcode位于reads的开头,用于区分这一条序列属于哪一个样本。分配完毕之后barcode会被删除。3.去除嵌合体:在pcr的反应中,会产生一些杂合的dna片段,通过将这些嵌合体去除,可以提高数据分析的准确性。
基于otu聚类分析:利用qiime(v1.9.1)软件对质控后的数据进行分析,将多条序列按序列间的距离进行聚类,再根据序列之间97%的相似性做阈值形成不同的操作分类单元(otus),排除总序列数和总样本量小于2的otu,挑选每个otu中的代表性序列并与greengenes(v_13.5)数据库中的基因序列进行比对,对每一个otu进行物种注释。
3.质量控制
两个研究中心均采用相同的方式进行组织取样,每个研究对象至少获取3个组织样本。组织标本取材过程中,剪取不同组织时需更换剪刀、镊子,防止交叉污染。观察组织的大小、形状、深度、范围、色泽和质地,剔除表面有腐败、糜烂的组织,通过病理学检查确诊有无鳞癌细胞存在。实验所需剪刀、镊子等器具均经过高压灭菌消毒。
4.统计方法
采用stata12.0软件对不同分组的人群的基本信息进行比较,通过kaplan-meier法绘制生存曲线,采用log-rank法进行生存率的比较,运用cox比例风险模型分析食管癌预后影响因素,并计算一致性指数(c-index)。p≤0.05有统计学意义。
采用qiime(v1.9.1)软件对癌组织的菌群测序数据进行分析,根据组织的性质将癌组织和癌旁组织各分为一组,将在某一组当中中位相对丰度大于0.01%的菌定为常见菌,常见菌的截断点为中位数,将菌群丰度大于中位数的研究对象定义为高相对丰度人群,小于中位数的定义为低相对丰度人群;中位相对丰度小于0.01%的菌定义为罕见菌,且罕见菌的检出率要超过检测对象的10%,罕见菌的研究中,将研究对象分为阳性和阴性患者。为了控制菌群之间相互的影响,将pcoa研究当中特征值大于1的坐标轴放入回归方程中一起分析,特征值大于1,说明对菌群总体分布解释的百分比较高,对菌群分布的说明能力更强。最终,在回归方程的研究中,癌组织中加入4个主坐标轴变量,4个变量对癌组织菌群分布的解释水平为60%。采用picrust软件对菌群的功能进行初步的分析,通过16s测序获得的物种构成来推测样本中的功能基因的构成,从而分析不同样本或分组之间在功能上的差异。
5.结果
5.1基本情况
截至2018年12月,共对120名食管鳞状细胞癌患者完成随访,研究对象当中,生存46人,死亡74人,最短随访时间为3个月,中位随访和中位生存时间均为28个月。总体研究对象的1年、3年和5年生存率分别为80%、42%和35%。
5.2癌组织中菌群分布与食管鳞癌患者预后的关系
在生存分析的研究中,首先绘制k-m生存曲线,并进行log-rank检验,结果发现罕见菌中,产碱菌科(alcaligenaceae)、都柏林克洛诺斯氏菌(cronobacterdublinensis)和水草盐孢菌(salinisporatropica)阳性患者的总体生存率低于阴性患者,克罗诺杆菌属(cronobacter)、瘤胃球菌属(ruminococcus)和气球菌属(aerococcus)阳性患者的总体生存率高于阴性患者。常见菌中,卡托氏菌属(catonella)丰度较高患者的总体生存率低于丰度低患者,而坦纳氏普拉沃氏菌(prevotellatannerae)丰度较高患者的总体生存率高于低丰度患者。见图6-a~图6-h,其中图6-a为癌组织中产碱菌科(alcaligenaceae)分布情况不同患者生存曲线;图6-b为癌组织中克罗诺杆菌属(cronobacter)分布情况不同患者生存曲线;图6-c为癌组织中瘤胃球菌属(ruminococcus)分布情况不同患者生存曲线;图6-d为癌组织中气球菌属(aerococcus)分布情况不同患者生存曲线;图6-e为癌组织中都柏林克洛诺斯氏菌(cronobacterdublinensis)分布情况不同患者生存曲线;图6-f为癌组织中水草盐孢菌(salinisporatropica)分布情况不同患者生存曲线;图6-g为癌组织中卡托氏菌属(catonella)分布情况不同患者生存曲线;图6-h为癌组织中坦纳氏普拉沃氏菌(prevotellatannerae)分布情况不同患者生存曲线。
在调整了年龄、性别、肿瘤分期、术后是否治疗等因素后,结果显示有5个菌科、7个菌属、3个菌种与患者的预后有关,在对初步研究结果进行fdr校正后,结果显示癌组织中坦纳氏普拉沃氏菌(prevotellatannerae)和克罗诺杆菌属(cronobacter)是食管鳞癌患者预后的影响因素,相比于低丰度坦纳氏普拉沃氏菌(prevotellatannerae)患者(中位生存时间22个月),高丰度患者的预后较好(hr=0.426,95%ci:0.260-0.700)。克罗诺杆菌属(cronobacter)阴性患者的中位生存时间为22个月,而阳性患者的中位生存时间为45个月,克罗诺杆菌属(cronobacter)阳性患者的预后较好(hr=0.436,95%ci:0.272-0.698)。见表1和表2。
表1癌组织中常见菌多因素cox回归分析
a模型调整的变量为年龄、性别、肿瘤分期、术后是否治疗。
b模型调整的变量为年龄、性别、肿瘤分期、术后是否治疗和特征值大于1的主坐标轴。
c对b模型得到的p值进行fdr校正。
表2癌组织中罕见菌多因素cox回归分析
a模型调整的变量为年龄、性别、肿瘤分期、术后是否治疗。
b模型调整的变量为年龄、性别、肿瘤分期、术后是否治疗和特征值大于1的主坐标轴。
c对b模型得到的p值进行fdr校正。
5.3预后影响菌群与患者一般情况的关系及其所具有的功能
克罗诺杆菌属(cronobacter)的功能进行分析,发现克罗诺杆菌属(cronobacter)阳性患者则是在非同源末端连接和细胞分裂这两个功能上数值较低,以通过这两个功能影响患者预后。见表3。
表3克罗诺杆菌属(cronobacter)功能水平分析
5.4菌群预后预测能力分析
通过多因素cox回归分析发现克罗诺杆菌属(cronobacter)与食管鳞癌患者的预后有关,通过一致性指数来评价每一类菌的模型对患者预后预测的准确性。结果显示,在模型中只含有年龄、性别、肿瘤分期以及术后是否化疗这4个变量时,一致性指数为0.6375,癌组织中的克罗诺杆菌属(cronobacter)一致性指数为0.7025,见表4。
表4预后有关菌群一致性指数分析
由实施例1可以看出,使用16srrna测序的方法检测食管鳞癌患者的癌组织和癌旁组织中菌群,使用cox回归分析发现cronobacter(克罗诺杆菌属)食管鳞癌患者的预后有关,为食管鳞癌预后预测提供微生物相关证据,为食管鳞癌患者预后预测提供参考依据。
实施例2
一、流程图参见图7。
二、操作步骤
1.食管鳞癌患者筛选
选取2013年2月~2017年10月就诊于福建省肿瘤医院和福建省漳州市医院的食管鳞癌患者120名。纳入标准:接受食管癌根治性手术治疗;所有研究对象均经过手术病理确诊患有食管鳞状细胞癌;术前均未接受放疗或者化疗等治疗;术前2个月无抗生素使用记录;无其他感染性疾病。排除标准:临床病理资料、组织标本不完整者;转移性肿瘤和食管癌复发病例;存在严重心脑器质性病变、精神病史等患者。
2.方法
2.1组织样本采集
使用手术室无菌剪刀分别剪取每位食管鳞癌患者的癌旁组织,剪取不同组织时更换剪刀,防止交叉污染。将组织剪成小块后分别放入高压灭菌的冻存管中,于1h之内置于液氮中储存,之后转入-80℃冰箱储存备用。所有耗材均经高压消毒灭菌。癌组织标本均经过病理苏木素-伊红染色(hematoxylin-eosinstaining,he)确诊为食管鳞状细胞癌,同时并未在癌旁组织标本中检测到有肿瘤细胞存在。
2.2基因组dna的提取与检测
采用sds法提取癌组织中菌群的dna。
(1)将样本从-80℃超低温冰箱中取出,使用无菌剪刀剪取50~100mg组织,放入1.5毫升ep管中,并加入1ml的dnazol,使用匀浆机处理5~10次。将经匀浆机处理后的ep管放入离心机当中,10000g离心10min,将得到的上清液转入新管当中。
(2)在离心好的ep管中加入edta(0.5m,ph8.0)和50μl溶菌酶,混匀后加入proteinasek,颠倒混匀,55℃孵育2h。
(3)12000rpm离心5min,吸取上清,加入蛋白沉淀液,轻柔涡旋10s,-20℃放置数分钟,12000rpm离心10min。
(4)吸取上清至新的ep管中,12000rpm离心5min,管底可能还有微量蛋白沉淀。吸取上清至新的ep管中,加入异丙醇,混匀-20℃放置20min后,12000rpm离心10min。
(5)倒出液体,用1ml75%乙醇洗涤两次,剩余的少量液体可再次离心收集,然后用枪头吸出。超净工作台吹干或者室温晾干。
(6)加入50μl的ddh2o溶解dna样品,振荡器助溶。加入1μl的rnasea,颠倒混匀后37℃温育15min。
(7)使用qubit仪对提取的dna进行定量检测,检测结果均合格。使用1%琼脂糖凝胶电泳检测:电压100v,40min。uvi凝胶成像系统拍照记录:dna电泳未出现杂带、拖尾,说明dna片段纯度良好,无明显降解,见图8和图9,其中图8为部分食管鳞癌患者癌组织的基因组dna检测图,其中编号为a25~29癌组织样本对应的泳道为3~7泳道;图9为部分食管鳞癌患者癌旁组织的基因组dna检测图,其中编号为b16~20癌旁组织样本对应的泳道为3~7泳道。取适量样品于离心管当中,使用无菌水稀释样品至1ng/μl。dna于-20℃冰箱内储存备用。
2.3pcr扩增
以稀释后的基因组dna为模板,根据测序区域的选择,使用带barcode的特异引物,采用细菌通用引物341f(cctaygggrbgcascag,如seqidno.1所示)和806r(ggactacnngggtatctaat,如seqidno.2所示)扩增细菌16srrna基因的v3-v4区域。使用
2.4pcr扩增产物混样与纯化
根据pcr产物浓度进行等质量混样,充分混匀后使用1×tae浓度2%的琼脂糖胶电泳纯化pcr产物,选择主带大小在400-450bp之间的序列,割胶回收目标条带。产物纯化试剂盒使用的是thermoscientific公司genejet胶回收试剂盒。
2.5文库构建与上机测序
使用illumina公司truseqdnapcr-freelibrarypreparationkit的建库试剂盒进行文库的构建,构建好的文库经过qubit定量和文库检测,合格后,使用illuminahiseq2500pe250测序平台进行菌群测序。
2.6测序数据的预处理
对食管鳞癌患者的食管菌群测序的原始数据原始数据质量控制具体步骤:1.数据拼接:去除3’端引物接头,根据pereads之间的重复关系将成对的序列拼接成一条序列。2.割库:通过标签序列(barcode)区分样品序列,并去除barcode和低质量、未检出的碱基。barcode位于reads的开头,用于区分这一条序列属于哪一个样本。分配完毕之后barcode会被删除。3.去除嵌合体:在pcr的反应中,会产生一些杂合的dna片段,通过将这些嵌合体去除,可以提高数据分析的准确性。
基于otu聚类分析:利用qiime(v1.9.1)软件对质控后的数据进行分析,将多条序列按序列间的距离进行聚类,再根据序列之间97%的相似性做阈值形成不同的操作分类单元(otus),排除总序列数和总样本量小于2的otu,挑选每个otu中的代表性序列并与greengenes(v_13.5)数据库中的基因序列进行比对,对每一个otu进行物种注释。
3.质量控制
两个研究中心均采用相同的方式进行组织取样,每个研究对象至少获取3个组织样本。组织标本取材过程中,剪取不同组织时需更换剪刀、镊子,防止交叉污染。观察组织的大小、形状、深度、范围、色泽和质地,剔除表面有腐败、糜烂的组织,通过病理学检查确诊有无鳞癌细胞存在。实验所需剪刀、镊子等器具均经过高压灭菌消毒。
4.统计方法
采用stata12.0软件对不同分组的人群的基本信息进行比较,通过kaplan-meier法绘制生存曲线,采用log-rank法进行生存率的比较,运用cox比例风险模型分析食管癌预后影响因素,并计算一致性指数(c-index)。p≤0.05有统计学意义。
采用qiime(v1.9.1)软件对癌旁组织的菌群测序数据进行分析,将在某一组当中中位相对丰度大于0.01%的菌定为常见菌,常见菌的截断点为中位数,将菌群丰度大于中位数的研究对象定义为高相对丰度人群,小于中位数的定义为低相对丰度人群;中位相对丰度小于0.01%的菌定义为罕见菌,且罕见菌的检出率要超过检测对象的10%,罕见菌的研究中,将研究对象分为阳性和阴性患者。为了控制菌群之间相互的影响,将pcoa研究当中特征值大于1的坐标轴放入回归方程中一起分析,特征值大于1,说明对菌群总体分布解释的百分比较高,对菌群分布的说明能力更强。最终,在回归方程的研究中,癌组织中加入4个主坐标轴变量,4个变量对癌组织菌群分布的解释水平为60%。采用picrust软件对菌群的功能进行初步的分析,通过16s测序获得的物种构成来推测样本中的功能基因的构成,从而分析不同样本或分组之间在功能上的差异。
5.结果
5.1基本情况
截至2018年12月,共对120名食管鳞状细胞癌患者完成随访,研究对象当中,生存46人,死亡74人,最短随访时间为3个月,中位随访和中位生存时间均为28个月。总体研究对象的1年、3年和5年生存率分别为80%、42%和35%。
5.2癌旁组织中菌群分布与食管鳞癌患者预后的关系
对癌旁组织中的菌群绘制k-m生存曲线,并通过log-rank检验进行生存率的比较,结果显示:间孢囊菌科(intrasporangiaceae)、生丝微菌科(hyphomicrobiaceae)、盐单胞菌科(halomonadaceae)、普氏菌(prevotellacopri)和嗜热嗜盐厌氧菌(anaerophagathermohalophila)阳性患者的总体生存率高于阴性患者,硝化螺旋菌属(nitrospira)、甲基杆菌属(methylobacterium)、克罗诺杆菌属阳性患者的总体生存率低于阴性患者。高丰度线粒体菌科(mitochondria)患者的总体生存率高于低丰度患者,而高丰度月形单胞菌属(selenomonas)患者的总体生存率较差。见图11-a~图11-j,其中图11-a为癌旁组织中间孢囊菌科(intrasporangiaceae)分布情况不同患者生存曲线;图11-b为癌旁组织中间生丝微菌科(hyphomicrobiaceae)分布情况不同患者生存曲线;图11-c为癌旁组织中盐单胞菌科(halomonadaceae)分布情况不同患者生存曲线;图11-d为癌旁组织中硝化螺旋菌属(nitrospira)分布情况不同患者生存曲线;图11-e为癌旁组织中甲基杆菌属(methylobacterium)分布情况不同患者生存曲线;图11-f为癌旁组织中克罗诺杆菌属(cronobacter)分布情况不同患者生存曲线;图11-g为癌旁组织中普氏菌(prevotellacopri)分布情况不同患者生存曲线;图11-h为癌旁组织中嗜热嗜盐厌氧菌(anaerophagathermohalophila)分布情况不同患者生存曲线;图11-i为癌旁组织中高丰度线粒体菌科(mitochondria)分布情况不同患者生存曲线;图11-j为癌旁组织中高丰度月形单胞菌属(selenomonas)分布情况不同患者生存曲线;在调整了年龄、性别、肿瘤分期、术后化疗情况和主坐标轴变量后,结果显示有6个菌科、7个菌属、2个菌种与患者的预后有关,在对初步研究结果进行fdr校正后,结果显示癌旁组织中盐单胞菌科(halomonadaceae)是食管鳞癌患者预后的影响因素,相比于盐单胞菌科阴性患者(中位生存时间24个月),盐单胞菌科阳性患者的预后较好(hr=0.161,95%ci:0.058-0.447)。见表5和表6。
表5癌旁组织中常见菌多因素cox回归分析
a模型调整的变量为年龄、性别、肿瘤分期、术后是否治疗。
b模型调整的变量为年龄、性别、肿瘤分期、术后是否治疗和特征值大于1的主坐标轴。
c对b模型得到的p值进行fdr校正。
表6癌旁组织中罕见菌多因素cox回归分析
a模型调整的变量为年龄、性别、肿瘤分期、术后是否治疗。
b模型调整的变量为年龄、性别、肿瘤分期、术后是否治疗和特征值大于1的主坐标轴。
c对b模型得到的p值进行fdr校正。
5.3预后影响菌群与患者一般情况的关系及其所具有的功能
对癌旁组织的盐单胞菌科(halomonadaceae)功能进行分析,发现盐单胞菌科(halomonadaceae)阳性患者在有关癌症通路功能上数值较低,而在黄酮和黄酮醇的生物合成的功能上数值较高,说明盐单胞菌科(halomonadaceae)可能通过这两种功能来影响食管鳞癌患者的预后,见表7。
表7盐单胞菌科(halomonadaceae)功能水平分析
5.4菌群预后预测能力分析
通过多因素cox回归分析发现癌旁组织盐单胞菌科(halomonadaceae)与食管鳞癌患者的预后有关,且这该菌均可通过不同的功能作用影响食管鳞癌患者的预后,通过一致性指数来评价每一类菌的模型对患者预后预测的准确性。结果显示,在模型中只含有年龄、性别、肿瘤分期以及术后是否化疗这4个变量时,一致性指数为0.6375,在加入菌群丰度的变量后,癌旁组织中的盐单胞菌科(halomonadaceae)的一致性指数为0.7151,见表8。
表8预后有关菌群一致性指数分析
由实施例2可知,使用16srrna测序的方法检测食管鳞癌患者的癌旁组织中菌群,使用cox回归分析发现盐单胞菌科(halomonadaceae)与食管鳞癌患者的预后有关,为食管鳞癌预后预测提供微生物相关证据,为食管鳞癌患者预后预测提供参考依据。
实施例3
一、流程图参见图12。
二、治疗分组
将研究对象分为2组,手术组、手术+术后化疗(铂类+紫杉醇类)组。
三、操作步骤
1.食管鳞癌患者筛选
选取2013年2月~2017年10月就诊于福建省肿瘤医院和福建省漳州市医院的食管鳞癌患者120名。纳入标准:接受食管癌根治性手术治疗;所有研究对象均经过手术病理确诊患有食管鳞状细胞癌;术前均未接受放疗或者化疗等治疗;术前2个月无抗生素使用记录;无其他感染性疾病。排除标准:临床病理资料、组织标本不完整者;转移性肿瘤和食管癌复发病例;存在严重心脑器质性病变、精神病史等患者。
2.方法
2.1组织样本采集
使用手术室无菌剪刀分别剪取每位食管鳞癌患者的癌旁组织,剪取不同组织时更换剪刀,防止交叉污染。将组织剪成小块后分别放入高压灭菌的冻存管中,于1h之内置于液氮中储存,之后转入-80℃冰箱储存备用。所有耗材均经高压消毒灭菌。癌组织标本均经过病理苏木素-伊红染色(hematoxylin-eosinstaining,he)确诊为食管鳞状细胞癌,同时并未在癌旁组织标本中检测到有肿瘤细胞存在。
2.2基因组dna的提取与检测
采用sds法提取癌组织中菌群的dna。
(1)将样本从-80℃超低温冰箱中取出,使用无菌剪刀剪取50~100mg组织,放入1.5毫升ep管中,并加入1ml的dnazol,使用匀浆机处理5-10次。将经匀浆机处理后的ep管放入离心机当中,10000g离心10min,将得到的上清液转入新管当中。
(2)在离心好的ep管中加入edta(0.5m,ph8.0)和50μl溶菌酶,混匀后加入proteinasek,颠倒混匀,55℃孵育2h。
(3)12000rpm离心5min,吸取上清,加入蛋白沉淀液,轻柔涡旋10s,-20℃放置数分钟,12000rpm离心10min。
(4)吸取上清至新的ep管中,12000rpm离心5min,管底可能还有微量蛋白沉淀。吸取上清至新的ep管中,加入异丙醇,混匀-20℃放置20min后,12000rpm离心10min。
(5)倒出液体,用1ml75%乙醇洗涤两次,剩余的少量液体可再次离心收集,然后用枪头吸出。超净工作台吹干或者室温晾干。
(6)加入50μl的ddh2o溶解dna样品,振荡器助溶。加入1μl的rnasea,颠倒混匀后37℃温育15min。
(7)使用qubit仪对提取的dna进行定量检测,检测结果均合格。使用1%琼脂糖凝胶电泳检测:电压100v,40min。uvi凝胶成像系统拍照记录:dna电泳未出现杂带、拖尾,说明dna片段纯度良好,无明显降解,见图13和图14,其中图13为部分食管鳞癌患者癌组织的基因组dna检测图,其中编号为a49~53癌组织样本对应的泳道为3~7泳道;图14为部分食管鳞癌患者癌旁组织的基因组dna检测图,其中编号为b31~35癌旁组织样本对应的泳道为3~7泳道。取适量样品于离心管当中,使用无菌水稀释样品至1ng/μl。dna于-20℃冰箱内储存备用。
2.3pcr扩增
以稀释后的基因组dna为模板,根据测序区域的选择,使用带barcode的特异引物,采用细菌通用引物341f(cctaygggrbgcascag,如seqidno.1所示)和806r(ggactacnngggtatctaat,如seqidno.2所示)扩增细菌16srrna基因的v3-v4区域。使用
2.4pcr扩增产物混样与纯化
根据pcr产物浓度进行等质量混样,充分混匀后使用1×tae浓度2%的琼脂糖胶电泳纯化pcr产物,选择主带大小在400~450bp之间的序列,割胶回收目标条带。产物纯化试剂盒使用的是thermoscientific公司genejet胶回收试剂盒。
2.5文库构建与上机测序
使用illumina公司truseqdnapcr-freelibrarypreparationkit的建库试剂盒进行文库的构建,构建好的文库经过qubit定量和文库检测,合格后,使用illuminahiseq2500pe250测序平台进行菌群测序。
2.6测序数据的预处理
对食管鳞癌患者的食管菌群测序的原始数据原始数据质量控制具体步骤:1.数据拼接:去除3’端引物接头,根据pereads之间的重复关系将成对的序列拼接成一条序列。2.割库:通过标签序列(barcode)区分样品序列,并去除barcode和低质量、未检出的碱基。barcode位于reads的开头,用于区分这一条序列属于哪一个样本。分配完毕之后barcode会被删除。3.去除嵌合体:在pcr的反应中,会产生一些杂合的dna片段,通过将这些嵌合体去除,可以提高数据分析的准确性。
基于otu聚类分析:利用qiime(v1.9.1)软件对质控后的数据进行分析,将多条序列按序列间的距离进行聚类,再根据序列之间97%的相似性做阈值形成不同的操作分类单元(otus),排除总序列数和总样本量小于2的otu,挑选每个otu中的代表性序列并与greengenes(v_13.5)数据库中的基因序列进行比对,对每一个otu进行物种注释。
3.随访
采用门诊、科室复查和电话随访相结合的方式对患者进行定期随访,术后两年内每3个月随访1次,两年后每6个月随访1次。末次随访时间为2018年12月。结局为患者发生死亡,失访者按删失值处理。中位随访时间为28个月。
4.质量控制
两个研究中心均采用相同的方式进行组织取样,每个研究对象至少获取3个组织样本。组织标本取材过程中,剪取不同组织时需更换剪刀、镊子,防止交叉污染。观察组织的大小、形状、深度、范围、色泽和质地,剔除表面有腐败、糜烂的组织,通过病理学检查确诊有无鳞癌细胞存在。实验所需剪刀、镊子等器具均经过高压灭菌消毒。
5.统计方法
采用stata12.0软件对不同分组的人群的基本信息进行比较,通过kaplan-meier法绘制生存曲线,采用log-rank法进行生存率的比较,运用cox比例风险模型分析食管癌预后影响因素,p≤0.05有统计学意义。
采用qiime(v1.9.1)软件对癌旁组织的菌群测序数据进行分析,将在某一组当中中位相对丰度大于0.01%的菌定为常见菌,常见菌的截断点为中位数,将菌群丰度大于中位数的研究对象定义为高相对丰度人群,小于中位数的定义为低相对丰度人群;中位相对丰度小于0.01%的菌定义为罕见菌,且罕见菌的检出率要超过检测对象的10%,罕见菌的研究中,将研究对象分为阳性和阴性患者。
5.研究结果
5.1基本情况
截至2018年12月,共对120名食管鳞状细胞癌患者完成随访,研究对象当中,生存46人,死亡74人,最短随访时间为3个月,中位随访和中位生存时间均为28个月。总体研究对象的1年、3年和5年生存率分别为80%、42%和35%。
5.2一般情况分析
首先对研究对象进行筛选,120例食管鳞癌手术患者中在排除了不符合纳入标准及的患者后剩余96名研究对象。该96名食管鳞癌患者,名就诊于福建省肿瘤医院名就诊于漳州市医院,研究结果显示,两家医院的患者在年龄、性别、肿瘤部位、t分期、n分期和肿瘤分化程度的分布上不存在统计学差异,因此,将两家医院的患者合并,共同分析,见表9。
表9食管鳞癌患者一般情况分析
5.3食管鳞癌患者基线特征的比较
对该96名患者根据治疗方式的不同分为单纯手术组和术后化疗组。并对一般情况的分布进行卡方检验,其中性别、肿瘤部位和分化程度的分布上在两组间的分布相近,而年龄和n分期的分布存在差异(p<0.05),且t分期在两组间的差异接近临界值(p=0.066)。对两组患者进行个案匹配,匹配因素为年龄和分期。再次进行卡方检验,两组患者的一般情况分布均无差异(p>0.05),见表10。
表10手术组和手术+化疗组匹配前后一般情况分析
5.4食管鳞癌癌旁组织菌群与食管鳞癌患者预后的关系。
在调整了年龄、性别、肿瘤分期、治疗方案后,cox多因素分析结果显示在常见菌中有3个菌科、5个菌属、3个菌种与患者的预后有关(见表11),在罕见菌中有8个菌科、14个菌属与患者的预后有关(见表12)。
表11匹配后患者中常见菌单因素多因素cox回归分析
*调整变量:性别、年龄、肿瘤分期、治疗方式
表12匹配后患者中罕见菌单因素多因素cox回归分析
*调整变量:性别、年龄、肿瘤分期、治疗方式
5.5不同治疗方案食管鳞癌癌旁组织菌群与食管鳞癌患者预后的关系(匹配人群)
对有关菌群与治疗方案进行交互作用分析,初步研究结果进行fdr方法多重检验校正后,结果显示,食管鳞癌患者癌旁组织中肉杆菌菌属(carnobacterium菌属)与治疗方案存在交互作用(p=0.008),因此根据患者体内肉杆菌菌属(carnobacterium菌属)存在情况和治疗方式进行分层分析,结果表明对于未检出肉杆菌菌属(carnobacterium菌属)的患者,术后化疗者预后劣于单纯手术者(hr=2.650,95%ci=1.199-5.856,p=0.016);而对于体内存在肉杆菌菌属(carnobacterium菌属)且接受手术+化疗的患者的预后较好(hr=0.150,95%ci=0.033-0.686,p=0.014),见表13。
表13治疗方案、carnobacterium菌属分布与食管鳞癌患者总生存之间的关系
注:调整了年龄、性别、分期。
由实施例3可知,使用16srrna测序的方法检测食管鳞癌患者的癌旁组织中菌群,使用cox回归分析发现肉杆菌菌属(carnobacterium菌属)与食管鳞癌术后化疗患者的预后有关,对于未检出肉杆菌菌属(carnobacterium菌属)的患者,术后化疗者预后劣于单纯手术者(hr=2.650,95%ci=1.199-5.856,p=0.016);而对于体内存在肉杆菌菌属(carnobacterium菌属)且接受手术+化疗的患者的预后较好(hr=0.150,95%ci=0.033-0.686,p=0.014),因此临床使用化疗治疗食管鳞癌术后患者,可以参考其体内是否存在肉杆菌菌属(carnobacterium菌属)进行选择是否选择化疗方案,能够为实现食管鳞癌患者术后的精准化治疗提供参考依据。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110>福建医科大学
<120>检测菌群的试剂在制备食管鳞癌患者预后预测标志物的试剂或试剂盒中的应用
<160>2
<170>siposequencelisting1.0
<210>1
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>1
cctaygggrbgcascag17
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
ggactacnngggtatctaat20
- 还没有人留言评论。精彩留言会获得点赞!