一种与猪背膘厚相关的SNP标记及其应用
一种与猪背膘厚相关的snp标记及其应用
技术领域
1.本发明属于分子生物学技术领域,涉及一种分子标记,具体涉及与猪背膘厚相关的snp标记及其应用。
背景技术:
2.在猪现代化养殖生产过程中,背膘厚是衡量瘦肉率和繁殖母猪体康等性状的重要指标,因而选择符合生产需求的种猪是猪育种工作的重要目标。大量研究表明,背膘厚度与瘦肉率呈显著负相关,背膘越薄,瘦肉率越高,薄背膘高瘦肉率是生猪生产过程中的重要育种方向。此外,猪繁殖性能的高低和背膘厚度也有着直接的关系,选择背膘厚度13.5-18.5mm的母猪可以从一定程度上提高母猪的繁殖性能。而猪的背膘厚度性状是由多基因位点决定的数量性状,通过对影响猪背膘厚度性状相关的位点进行鉴定并筛选,可以提高猪群的整体繁殖性能和满足人们对肉食产品以及育种工作的需求。
3.目前,猪场的遗传育种多以表型选择为主,表型选择具有育种周期长、假阳性率高、易受饲养环境影响等缺点,会造成基因的误选、误淘等问题。
4.因此,亟需发掘功能明确、效应显著的snp标记,实现背膘厚度性状的早期选择,加快遗传进展,改善猪的背膘厚性状。
技术实现要素:
5.为了克服上述问题,本发明人进行了锐意研究,提出了一种与猪背膘厚相关的分子标记及其获得方法与应用、预测猪背膘厚的方法和系统。所述分子标记可以用于猪的早期选育,降低育种成本,缩短育种周期,加快遗传进展,可以通过直接鉴定该标记来筛选更低背膘厚的猪品系,能够准确、高效地选育出低背膘厚的优良猪品种,从而完成了本发明。
6.具体来说,本发明的目的在于提供以下方面:
7.本发明提供了一种与猪背膘厚相关的分子标记,其中,所述分子标记为snp标记,其位于国际猪基因组10.2版本参考序列2号染色体上第161,686,082bp处,存在t/g多态性。
8.其中,所述位于国际猪基因组10.2版本参考序列2号染色体上第161,686,082bp处的snp标记的等位基因为g和t,其对应的基因型有三种,分别为gg基因型、tg基因型和tt基因型,
9.优选地,所述位于国际猪基因组10.2版本参考序列2号染色体上第161,686,082bp处的snp标记的基因型为tt的猪的背膘厚度显著低于基因型为tg和gg的猪。
10.本发明还提供了一种上述分子标记的获得方法,其中,所述方法包括以下步骤:
11.步骤1,选择猪群体,提取基因组dna;
12.步骤2,测定表型性状;
13.步骤3,进行基因分型;
14.步骤4,进行表型性状的全基因组关联分析。
15.其中,步骤1中,所述猪群体中的猪种选自杜洛克猪、长白猪和大白猪中的一种或
多种。
16.其中,步骤3中,所述基因分型采用直接测序法、限制性片段长度多态性聚合酶链式反应、飞行时间质谱技术和芯片技术中的一种或多种。
17.本发明还提供了一种预测猪背膘厚的方法,其中,所述方法包括通过对待测猪进行所述snp标记的检测,预测待测猪的背膘厚的步骤;
18.优选地,所述方法包括以下步骤:
19.步骤i,提取待测猪的基因组dna;
20.步骤ii,对基因组dna进行扩增,获得pcr产物;
21.步骤iii,对pcr产物进行测序,确定猪的背膘厚。
22.本发明还提供了一种预测猪背膘厚的系统,其中,所述系统包括:
23.扩增单元,用于对待测猪的基因组dna进行扩增;
24.测序单元,其与扩增单元相连,用于对扩增得到的产物进行测序;
25.预测单元,其与测序单元相连,用于根据测序结果预测猪背膘厚度。
26.本发明还提供了一种上述分子标记或上述方法获得的snp标记或上述的预测猪背膘厚的系统在鉴定猪背膘厚或筛选低背膘厚猪品种方面的应用。
27.本发明还提供了一种上述分子标记或上述方法获得的snp标记或上述的预测猪背膘厚的系统在猪育种中的应用。
28.其中,所述应用包括以下步骤:
29.步骤i,获取猪的基因组dna;
30.步骤ii,检测猪位于国际猪基因组10.2版本参考序列2号染色体上第161,686,082bp处的snp标记的基因型;
31.步骤iii,根据基因型选择低背膘厚的猪作为种猪,以进行育种。
32.本发明所具有的有益效果包括:
33.(1)本发明提供的与猪背膘厚相关的分子标记,可以用于猪的早期选育,降低育种成本,缩短育种周期,加快遗传进展,丰富了分子标记辅助育种的snp标记;
34.(2)本发明提供的与猪背膘厚相关的分子标记,可以通过直接鉴定该标记来筛选更低背膘厚的猪品系,提高了养殖企业以及整个生猪养殖业的经济效益与社会价值;
35.(3)本发明提供的预测猪背膘厚的方法,操作简单,预测准确性高,可以为猪生产性状的分子标记辅助选择提供科学依据。
附图说明
36.图1示出本发明实施例1中的全基因组关联分析曼哈顿图;
37.图2示出本发明实施例2中基因效应的箱线图。
具体实施方式
38.下面通过优选实施方式和实施例对本发明进一步详细说明。通过这些说明,本发明的特点和优点将变得更为清楚明确。
39.在这里专用的词“示例性”意为“用作例子、实施例或说明性”。这里作为“示例性”所说明的任何实施例不必解释为优于或好于其它实施例。
13/t 2065-2014)文件《种猪场场内生产性能测定技术规程》的遗传评估性状测定规程对采集的数据进行表型数据校正。
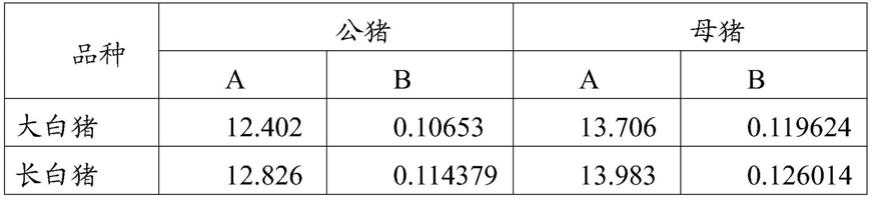
57.采用b超扫描测定倒数第3-4肋间处的背膘厚,以毫米为单位。最后按如下校正公式转换成达100kg体重的活体背膘厚(计算方法如下,校正背膘厚度的校正系数参考表1):
58.校正背膘厚(mm)=实测背膘厚(mm)
×
cf
59.其中:
60.cf=a
÷
{a+[b
×
(实测体重(kg)-100)]}
[0061]
表1、公母猪背膘厚度校正表
[0062][0063][0064]
注:a、b为不同猪种背膘厚度校正系数
[0065]
对测定后的表型性状数据进行质量控制:清除表型值缺失的个体,清除与平均值的偏差大于3倍标准差的个体。
[0066]
步骤3,进行基因分型。
[0067]
在本发明中,所述基因分型方法选自直接测序法、限制性片段长度多态性聚合酶链式反应、飞行时间质谱和芯片技术中的一种或多种,优选采用高密度snp芯片技术进行基因分型,其可以在很短的时间周期内对大量的snp进行分型,效率高,成本低。
[0068]
优选地,采用纽勤公司的neogen_pop80k芯片进行基因分型,并对基因型数据进行质量控制。
[0069]
所述质量控制为:清除基因型检出率小于95%的snp位点;清除检出率小于95%的个体;清除最小等位基因频率小于(minimum allele frequency,maf)小于1%的个体;清除哈代-温伯格平衡(hardy
–
weinberg equilibrium,hwe)卡方检验p值小于1.0e-4的snp位点;清除性染色体上的snp位点。
[0070]
步骤4,进行表型性状的全基因组关联分析。
[0071]
根据本发明一种优选的实施方式,所述全基因关联分析采用的统计分析模型如下所示:
[0072]
y=xβ+zμ+e
[0073]
其中,y是观察到的表型值;β是含有固定效应的未知值,包括遗传标记,种群结构(q矩阵)和截距;μ是来自个体或者系的多个背景qtl的随机加性遗传效应的未知值;x和z是已知的设计矩阵;e是未观察到的残差向量。
[0074]
在本发明中,进行全基因关联分析采用的分析软件包为r语言包gapit version 3,此软件包的统计模型为压缩混合线性模型。gapit的设计目的是在大数据集上准确地执行gwas和基因组预测。混合线性模型(mixed liner model,mlm)包括固定和随机效应,模型
将种群结构作为固定效应,同时将个体纳入随机效应进行个体亲缘关系矩阵的构建。
[0075]
基于上述的混合线性模型,对所有分型snp位点进行校正背膘厚度关联分析,获得snp位点(g/t;chr2:161,686,082)与猪背膘厚度性状显著相关。
[0076]
进一步地,对基因型数据和表型数据进行差异显著性检验,能够获得与低背膘厚显著相关的基因型类型。
[0077]
本发明的第三方面,提供了一种预测猪背膘厚的方法,所述方法包括:通过对待测猪进行第一方面所述snp标记的检测,预测待测猪的背膘厚。
[0078]
优选地,所述方法包括以下步骤:
[0079]
步骤i,提取待测猪的基因组dna;
[0080]
步骤ii,对基因组dna进行扩增,获得pcr产物;
[0081]
步骤iii,对pcr产物进行测序,确定猪的背膘厚。
[0082]
其中,步骤ii中,所述pcr扩增的引物为p1和p2,其核苷酸序列分别如seq id no:1和seq id no:2所示。
[0083]
步骤iii中,优选地,snp标记处的核苷酸为碱基t的猪背膘厚低于核苷酸为碱基g的猪背膘厚。
[0084]
采用上述方法,能够准确、高效地预测出猪背膘厚。
[0085]
本发明的第四方面,提供了一种预测猪背膘厚的系统,所述系统包括:
[0086]
扩增单元,用于对待测猪的基因组dna进行扩增;
[0087]
测序单元,其与扩增单元相连,用于对扩增得到的产物进行测序;
[0088]
预测单元,其与测序单元相连,用于根据测序结果预测猪背膘厚度。
[0089]
本发明的第五方面,提供了一种第一方面所述snp标记或第二方面所述方法获得的snp标记或第四方面所述的预测猪背膘厚的系统在鉴定猪背膘厚或筛选低背膘厚猪品种方面的应用。
[0090]
本发明的第六方面,提供了一种第一方面所述snp标记或第二方面所述方法获得的snp标记或第四方面所述的预测猪背膘厚的系统在猪育种中的应用。
[0091]
其中,所述猪优选为杜洛克猪、长白猪或大白猪。
[0092]
优选地,所述应用包括以下步骤:
[0093]
步骤i,获取猪的基因组dna;
[0094]
步骤ii,检测猪位于国际猪基因组10.2版本参考序列2号染色体上第161,686,082bp处的snp标记的基因型;
[0095]
步骤iii,根据基因型选择低背膘厚的猪作为种猪,以进行育种。
[0096]
其中,选择国际猪基因组10.2版本参考序列2号染色体上第161,686,082bp处的snp标记的基因型为tt的个体作为种猪,淘汰该snp位点基因型为tg和gg的个体,以在继代的过程中逐代提高该位点的基因型tt的频率,从而降低背膘厚度。
[0097]
实施例
[0098]
以下通过具体实例进一步描述本发明,不过这些实例仅仅是范例性的,并不对本发明的保护范围构成任何限制。
[0099]
实施例1 snp标记的鉴定
[0100]
1、试验群体
[0101]
本实施例所采用的试验猪群体为1,173头来自河北美神原种猪场的猪,其中,杜洛克公猪23头,杜洛克母猪177头,长白公猪15头,长白母猪363头,大白公猪2头,大白母猪593头。
[0102]
2、背膘厚度测定与校正
[0103]
在猪个体体重处于85~105kg范围时测定活体背膘厚(backfat thickness,bft),采用b超扫描测定群体中猪个体的倒数第3~4肋间处背膘厚,以毫米为单位。然后,采用河北省地方标准(db13/t 2065-2014)文件《种猪场场内生产性能测定技术规程》的遗传评估性状测定规程对采集的数据进行表型数据校正,具体按如下校正公式计算猪达100kg体重的活体背膘厚:
[0104]
校正背膘厚(mm)=实测背膘厚(mm)
×
cf
[0105]
其中:cf=a
÷
{a+[b
×
(实测体重(kg)-100)]};
[0106]
校正系数(a、b)如前述表1所示。
[0107]
3、dna提取与snp分型
[0108]
3.1、基因组dna提取
[0109]
本实施例采用北京百泰克生物技术有限公司的dp1902型号细胞/组织基因组dna提取试剂盒,从猪的耳组织中提取dna,用紫外分光光度计和凝胶电泳进行dna质量检测,将检测合格的dna放置于-20℃保存,以用于后续分型测定。
[0110]
3.2、基因分型
[0111]
取检测合格的dna,利用纽勤公司的neogen_por80k芯片进行基因型分型。
[0112]
4、表型数据和基因型数据的质量控制
[0113]
4.1、表型数据的质量控制标准:
[0114]
清除表型值缺失的个体;清除与平均值的偏差大于3倍标准差的个体。
[0115]
4.2、snp芯片分型的过滤标准:清除基因型检出率小于95%的snp位点;清除检出率小于95%的个体;清除最小等位基因频率小于(minimum allele frequency,maf)小于1%的个体;清除哈代-温伯格平衡(hardy
–
weinberg equilibrium,hwe)卡方检验p值小于1.0e-4的snp位点;清除性染色体上的snp位点。
[0116]
5、猪背膘厚的全基因组关联分析
[0117]
采用r语言包gapit version 3(华盛顿大学研发)进行全基因组关联分析,此软件包的统计模型为压缩混合线性模型。混合线性模型(mixed liner model,mlm)包括固定和随机效应,模型将种群结构作为固定效应,同时将个体纳入随机效应进行个体亲缘关系矩阵的构建。其统计分析模型为:
[0118]
y=xβ+zμ+e
[0119]
其中,y是观察到的表型值;β是含有固定效应的未知值,包括遗传标记,种群结构(q矩阵)和截距;μ是来自个体或者系的多个背景qtl的随机加性遗传效应的未知值;x和z是已知的设计矩阵;e是未观察到的残差向量。
[0120]
基于混合线性模型,采用gapit统计分析软件对所有分型snp位点进行校正背膘厚度关联分析,统计分析模型同上所述。
[0121]
结果如图1所示,由图1可知:snp位点2_161686082(g/t;chr2:161,686,082)与猪背膘厚度性状显著相关(其校正p值为3.14e-03)。
[0122]
实施例2 snp位点2_161686082(g/t)不同基因型与背膘厚度的关联分析
[0123]
1、试验群体
[0124]
本实施例采用的试验猪群体与实施例1相同。
[0125]
2、背膘厚度的测定与校正
[0126]
本实施例采用的背膘厚度的测定和校正方法与实施例1相同。
[0127]
3、snp的检测
[0128]
3.1、基因组dna提取
[0129]
本实施例中采用的基因组dna提取方法与实施例1相同。
[0130]
3.2、基因型确定
[0131]
取检测合格的dna,利用纽勤公司的neogen_por80k芯片检测每个个体的基因型,通过r语言提取多态性位点(2_161686082)的序列,并统计其基因型频率和基因频率分布,结果如表2所示:
[0132]
表2
[0133][0134]
由表2可知,gg基因型为实验群体优势基因型,g为优势等位基因。
[0135]
4、基因型与背膘厚度关联分析
[0136]
采用rstudio软件对基因型数据和表型数据利用kruskal-wallis方法进行差异显著性检验,p-value<0.01表明差异极显著,结果如表3所示;利用r语言的ggplot2、ggpubr和magrittr函数包绘制箱线图,结果如图2所示。
[0137]
表3
[0138][0139]
由表3和图2可以看出,三种基因型的猪校正背膘厚度有显著差异。在100kg体重时,tt基因型猪的背膘厚度显著低于tg和gg基因型的猪。位点2_161686082的tt基因型的个体背膘厚度比gg型的背膘厚度少2.12mm。该snp位点可以作为猪背膘厚度筛选的分子标记,应用于猪分子育种过程中
[0140]
以上结合具体实施方式和范例性实例对本发明进行了详细说明,不过这些说明并不能理解为对本发明的限制。本领域技术人员理解,在不偏离本发明精神和范围的情况下,可以对本发明技术方案及其实施方式进行多种等价替换、修饰或改进,这些均落入本发明
的范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1