蛋白FoUPE1在调控香蕉枯萎病菌致病力中的应用
本发明属于植物基因工程领域,特别涉及一种香蕉枯萎病菌未知蛋白(uncharacterizedprotein)foupe1在调控香蕉枯萎病菌致病力中的应用。
背景技术:
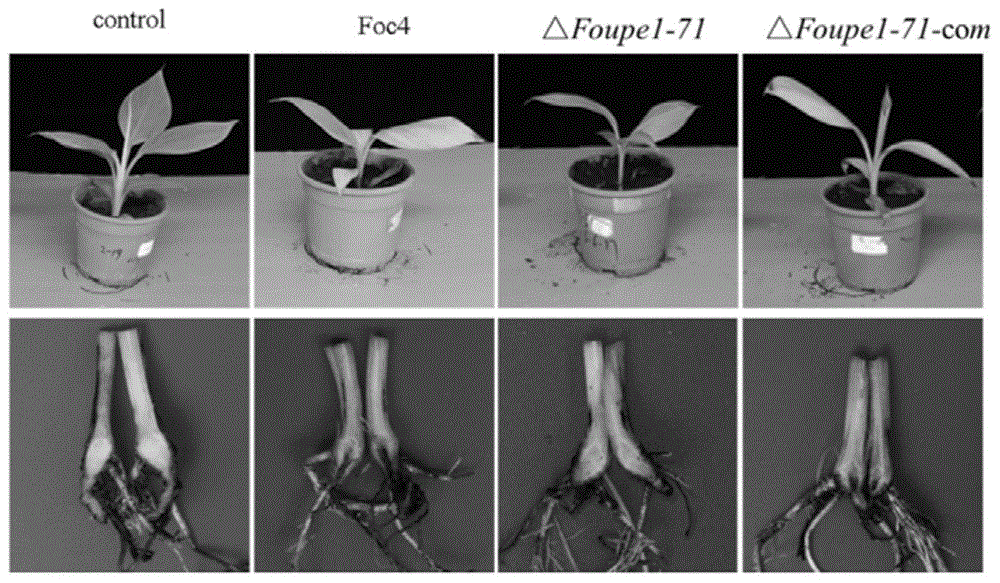
:香蕉枯萎病,是由尖孢镰刀菌古巴专化型(fusariumoxysporumf.sp.cubense,foc)引起的一种土传性真菌流行病害,该病在我国几乎所有的香蕉主产区均有发生,成为制约我国香蕉产业发展的最重要因素之一。foc可分为3个小种,其中以4号小种(foc4)危害最为严重,几乎能危害目前所有的栽培品种。充分挖掘香蕉枯萎病菌致病相关基因并开展其功能研究,有助于全面了解香蕉枯萎病菌的致病分子机理,并为香蕉枯萎病的防控提供理论基础。foupe1(uncharacterizedprotein)是一个未知蛋白,不含已知结构域,在镰刀菌(fusarium)中具有高度保守性。关于foupe1在香蕉枯萎病菌中的具体功能尚不清楚。技术实现要素:为了克服现有技术的缺点与不足,本发明的目的在于提供一种蛋白foupe1在调控香蕉枯萎病菌致病力中的应用。本发明的目的是公开一种香蕉枯萎病菌基因foupe1及其编码蛋白foupe1的新功能。基因foupe1为seqidno:1中第1位至第2733位所示的核苷酸序列,其所编码的蛋白foupe1为seqidno:2所示的蛋白质。本发明通过构建基因敲除载体,将其导入香蕉枯萎病菌原生质体;利用同源重组方法将该基因从香蕉枯萎病菌中敲除,获得敲除突变体△foupe1;通过构建基因回补载体,将其导入△foupe1原生质体;利用随机插入的方法将该基因回补到敲除突变体中,获得回补突变体△foupe1-com。该基因的敲除突变体在分生孢子产生和应对氧化胁迫存在缺陷,并且菌落直径减小。致病性测定表明,敲除突变体△foupe1致病性显著降低;回补突变体△foupe1-com的致病性则恢复到foc4水平。上述试验证明,香蕉枯萎病菌foupe1为香蕉枯萎病菌的致病相关基因。本发明的目的通过下述技术方案实现:本发明提供一种蛋白foupe1在调控香蕉枯萎病菌致病力中的应用。进一步的,所述的蛋白foupe1在调控香蕉枯萎病菌生长发育中的应用。进一步的,所述的蛋白foupe1在调控香蕉枯萎病菌产孢量中的应用。进一步的,所述的蛋白foupe1在调控香蕉枯萎病菌抗胁迫中的应用。优选的,所述的胁迫为氧化胁迫。本发明提供一种蛋白foupe1在防治由香蕉枯萎病菌导致的香蕉枯萎病中的应用,所述的防治是通过阻断或抑制编码蛋白foupe1的基因的表达来实现的。本发明提供一种蛋白foupe1作为用于植物病害防治的药物的靶标的应用,所述的植物病害是由香蕉枯萎病菌导致的香蕉枯萎病。本发明再提供一种治疗由香蕉枯萎病菌导致的香蕉枯萎病的方法,包含阻断或抑制香蕉枯萎病菌中编码蛋白foupe1的基因的表达(例如利用该基因的反义rna或sirna等)。阻断或抑制香蕉枯萎病菌中编码蛋白foupe1的基因的表达的药剂(例如利用该基因的反义rna或sirna等)在制备药物中的应用,所述药物用于控制由香蕉枯萎病菌导致的香蕉枯萎病。其中,所述的蛋白foupe1,其氨基酸序列如seqidno:2所示,或者是如seqidno:2所示的氨基酸序列通过一个或多个氨基酸替换、插入、缺失而获得的仍具有控制香蕉枯萎病菌致病力功能的类似物;编码蛋白foupe1的基因,其核苷酸序列为下列a、b、c之一:a、编码seqidno:2所示氨基酸序列的dna序列;b、如seqidno:1所示的dna序列;c、以上a和b通过碱基插入、缺失、或替换而获得的仍具有控制香蕉枯萎病菌致病力功能的类似物;进一步的,所述的香蕉枯萎病菌为香蕉枯萎病菌4号生理小种(foc4)。含有上述编码蛋白foupe1的基因的敲除载体、重组菌在上述方面的应用也属于本发明的保护范围。本发明相对于现有技术具有如下的优点及效果:本发明提供了有一个香蕉枯萎病菌4号小种(foc4)未表征蛋白foupe1(uncharacterizedprotein)及其编码基因foupe1的新功能。所述基因foupe1为seqidno:1中第1位至第2733位所示的核苷酸序列,其所编码的蛋白foupe1为seqidno:2所示的蛋白质;foupe1蛋白不含有已知结构域,uniprot分析其亚细胞定位未知,其在香蕉枯萎病菌的生物学功能并不清楚。将潮霉素磷酸转移酶基因(hph)和荧光蛋白基因(sgfp)置换foupe1编码基因foupe1,得到foc4敲除突变体△foupe1;试验证明,与foc4相比,△foupe1菌落生长速率下降且产孢量显著降低,对氧化胁迫耐受性下降;致病性试验表明,foupe1的缺失使foc4的致病力显著降低;将该基因回补后,其致病力得到恢复。本发明证实foupe1是香蕉枯萎病菌分生孢子产生、应对氧化胁迫和致病性所必需的。我们的研究有助于深入阐明香蕉枯萎病菌的致病分子机制,为开发有效杀菌剂提供了靶标基因。附图说明图1是香蕉枯萎病菌基因foupe1敲除载体的构建示意图。图2是部分候选敲除转化子hph基因的pcr扩增产物的琼脂糖凝胶电泳图;其中,m:2000dnamarker;泳道1:pct74质粒;泳道2:无菌水;泳道3-6:转化子71、76、113、117。图3是部分候选敲除转化子目的基因foupe1的pcr扩增产物的琼脂糖凝胶电泳图;其中,m:5000dnamarker;泳道1:foc4;泳道2:无菌水;泳道3-6:转化子71、76、113、117。图4是以foupe1片段为探针的foc4敲除转化子的southernblot分析;其中,泳道1:foc4;泳道2-4:转化子71、76、113。图5是以hph片段为探针的foc4敲除转化子的southernblot分析;其中,泳道1:foc4;泳道2-4:转化子71、76、113。图6是香蕉枯萎病菌基因foupe1回补载体示意图。图7是部分候选回补转化子foupe1基因扩增产物的琼脂糖凝胶电泳图;m:5000dnamarker;泳道1:foc4;泳道2:无菌水;3-6:回补转化子2、7、10、21。图8是敲除突变体△foupe1和回补突变体△foupe1-com对不同胁迫条件的分析;其中,a:菌落直径;b:菌落生长抑制率。图9是敲除突变体△foupe1和回补突变体△foupe1-com的致病性分析。具体实施方式下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。下列实施例中未注明具体实验条件的试验方法,通常按照常规实验条件或按照制造厂所建议的实验条件。所使用的材料、试剂等,如无特殊说明,为从商业途径得到的试剂和材料。实施例11、实验材料1.1供试菌株及植物供试菌株为香蕉枯萎病菌4号生理小种(foc4),供试植物为具有4~5片叶的巴西蕉(cavendish,aaa)。1.2宿主菌及质粒载体宿主菌为大肠杆菌(escherichiacoli)dh5α菌株。克隆载体为pmd18-tvector,基因敲除载体为丝状真菌表达载体pct74,基因回补载体为pctzn(由本实验室在pct74质粒基础上改造得来,即将pct74上的sgfp和hph基因替换成博来霉素(zeocin)基因)。2、实验方法2.1香蕉枯萎病菌foupe1基因上下游同源片段的扩增香蕉枯萎病菌foupe1基因敲除载体的构建如图1所示。分别选取foupe1基因的上游和下游长度大小约为1000bp左右的序列(分别命名为同源臂a片段和同源臂b片段),并设计引物(表1)。表1foupe1基因同源臂a片段和b片段的扩增引物引物名称引物序列5’-3’酶切位点foupe1-afggggtaccggtgcgacgggtccattkpnifoupe1-arccgctcgagatgtctccgtctgttagcaagtxhoifoupe1-bftccccccgggctgatgacgattctcgggctxmaifoupe1-brgctctagaacctgatcgccaactattctttacxbai按照omega公司的柱式真菌dna提取试剂盒(omegafungaldnakit)的说明书提取foc4基因组dna;以foc4基因组dna为模板,用引物foupe1-af和foupe1-ar进行pcr扩增,获得foupe1基因的同源臂a片段(foupe1-a);用引物foupe1-bf和foupe1-br进行pcr扩增,获得foupe1基因的同源臂b片段(foupe1-b)。pcr反应体系为:2×tsingkemastermix12.5μl模板dna0.5μlfoupe1-af/bf(10μmol/l)0.5μlfoupe1-ar/br(10μmol/l)0.5μlddh2o11.0μltotal25.0μlpcr反应条件为:94℃反应5min;94℃反应1min,55℃反应1min,72℃反应1min,共30个循环;72℃反应10min。使用omegacyclepurekit试剂盒,对pcr扩增产物进行清洁回收。2.2foupe1基因敲除载体的构建参考pmd18-tvectorcloningkit(takara公司)试剂盒说明书,将foupe1-a和foupe1-b分别与t载体连接,获得重组质粒pmd18t-foupe1-a和pmd18t-foupe1-b。具体为:取1μlpmd18-t载体,分别加入4μl上述pcr回收产物(同源臂a片段或同源臂b片段)和5μlsolutioni,于16℃下连接3~4h。取10μl连接产物加入到100μl大肠杆菌dh5α感受态细胞中,冰上放置30min;于42℃下水浴热击90s,冰上冷却5min;加入800μllb液体培养基,于37℃下150rpm培养1h;再于4000rpm离心5min,弃上清,留100μl菌液与沉淀混匀,涂布于lb固体培养基(含50μg/mlamp);于37℃下培养8~12h。挑取具有amp抗性的阳性转化子,提取重组质粒dna,并进行测序鉴定。用kpni和xhoi分别对pmd18t-foupe1-a和pct74载体进行双酶切,回收a片段和pct74载体。用t4dna连接酶将a片段与pct74连接,转化大肠杆菌dh5α感受态细胞;获得重组质粒pct74-foupe1-a。按同样程序,用xmai和xbai分别对pmd18t-foupe1-b和重组质粒pct74-foupe1-a进行双酶切,回收b片段和重组质粒。用t4dna连接酶将b片段与pct74-foupe1-a连接,转化大肠杆菌dh5α感受态细胞;经酶切鉴定,获得基因敲除载体pct74-foupe1-ko。2.3foupe1回补片段的扩增香蕉枯萎病菌foupe1基因回补载体的构建如图6所示。选取foupe1基因的上游长度为1500bp的启动子序列,下游长度为500bp的终止子序列,并设计引物(表2)。表2foupe1基因回补片段的扩增引物引物名称引物序列5’-3’酶切位点foupe1-com-fgactagttgcatagtgatgagaaagtccaaspeifoupe1-com-rataagaatgcggccgcaagtcaaagagtggtgagcacatnoti用真菌dna提取试剂盒(omegafungaldnakit),提取foc4基因组dna;以该基因组dna为模板,用引物foupe1-com-f和foupe1-com-r进行pcr扩增,获得foupe1基因的回补片段(foupe1-com)。具体的pcr反应体系为:模板dna1.0μlfoupe1-com-f(10μmol/l)1.0μlfoupe1-com-r(10μmol/l)1.0μl10×taqbuffer(mg2+plus)5.0μldntps(2.5mmol/l)4.0μlextaq(5u/μl)0.5μlddh2o37.5μltotal50.0μlpcr反应条件为:94℃反应5min;94℃反应1min,60℃反应1min,72℃反应4min,共30个循环;72℃反应10min。用omegacyclepurekit试剂盒,对pcr扩增产物进行清洁回收。2.4foupe1基因回补载体的构建用spei和noti分别对foupe1-com和pctzn载体进行双酶切,回收foupe1-com片段和pctzn载体。用t4dna连接酶将foupe1-com片段与pctzn连接,转化大肠杆菌dh5α感受态细胞;获得重组质粒pctzn-foupe1-com。经酶切鉴定,获得基因回补载体pctzn-foupe1-com。2.5foc4原生质体的制备将foc4接种到查氏培养基(feso4·7h2o0.018g,kcl0.5g,k2hpo4·3h2o1g,mgso4·7h2o0.5g,nano33g,蔗糖30g,蒸馏水定容至1l)中,于28℃下150rpm培养3d,经200目细胞筛过滤后,获得分生孢子液,于4℃下10000×g离心10min,弃上清,获得浓缩的分生孢子液,加入cm培养基(葡萄糖10.0g,蛋白胨2.0g,水解酪蛋白1.0g,酵母浸粉1.0g,20×硝酸盐50ml,1000×维生素1ml,1000×微量元素1ml,定容至1l,调节ph至6.5,其中,20×硝酸盐、1000×维生素、1000×微量元素的成分在“201710903818.8、一种香蕉枯萎病菌培养基及其应用”中公开),使分生孢子液终浓度为1×106个/ml;于28℃下120rpm培养11~12h,用100目细胞筛过滤,并用0.8mol/lnacl溶液(渗透压稳定剂)冲洗3~5次,获得新鲜菌丝体。按酶液与菌丝比例(体积质量比10:1),加入适量15g/l崩溃酶酶液,于30℃下120rpm条件下酶解3h,得到原生质体酶解液。于4℃下4000×g离心10min,弃上清。加入1ml预冷的stc溶液(含10mmol/ltris-hcl(ph7.5),1.2mol/l山梨醇,50mmol/lcacl2)重悬沉淀;离心,弃上清。再加入10~20ml预冷的stc将沉淀重悬,得到foc4原生质体悬液,使原生质体终浓度约为1×107个/ml。香蕉枯萎病菌敲除突变体原生质体,参照上述香蕉枯萎病菌原生质体的制备步骤制备得到。2.6foc4敲除突变体原生质体的转化用noti对敲除载体pct74-foupe1-ko进行单酶切,获得敲除载体线性化片段。将200μlfoc4原生质体置于冰上解冻后加入约5μg的敲除载体线性化片段,轻弹混合均匀,冰上静置20min;或者,将pctzn-foupe1-com质粒与200μl香蕉枯萎病菌敲除突变体原生质体混匀;逐滴加入1mlptc(40%peg-4000,1.2mol/l山梨醇,50mmol/lcacl2,10mmol/ltris-hcl,ph7.5),混匀后冰上放置15min;加入15ml预冷的stc,混匀;于4℃下4000rpm离心15min;去上清,留下5ml混合液,加入3mlpsb再生培养基(马铃薯200.0g,蔗糖273.6g,蒸馏水定容至1l)重悬沉淀,于28℃下100rpm震荡培养16h。于4℃下4000rpm离心15min,去掉5ml上清,加入12mlpsa再生培养基(psb再生培养基中加入1.5%琼脂粉、150μg/ml潮霉素或200μg/ml博来霉素),混匀倒板,于28℃黑暗培养2~3d;挑取潮霉素(或博来霉素)抗性转化子,转移到含有150μg/ml潮霉素(或200μg/ml博来霉素)的pda培养基(含马铃薯200.0g,无水葡萄糖20.0g,琼脂15.0g,蒸馏水定容至1l),于28℃黑暗培养2~3d,挑取单菌落用于鉴定。2.7foc4敲除突变体的pcr验证分析按照真菌dna提取试剂盒(omegafungaldnakit)说明书,提取上述潮霉素阳性转化子的基因组dna,进行pcr验证分析。分别用引物hph-f/hph-r进行hph基因片段的pcr扩增;用引物foupe1-f/foupe1-r进行foupe1基因片段的pcr扩增分析。hph-f:5′-tgctgctccatacaagccaa-3′,hph-r:5′-gacattggggagttcagcga-3′;foupe1-f:5′-agaccaacaactaggcaatccttt-3′,foupe1-r:5′-gtcttgggccggtactggtt-3′,pcr反应体系如下:模板dna0.5μlfoupe1-f/hph-f(10μmol/l)0.5μlfoupe1-r/hph-r(10μmol/l)0.5μl10×taqbuffer(mg2+plus)2.5μldntps(2.5mmol/l)2.0μltaq(5u/μl)0.25μlddh2o18.75μltotal25.0μlpcr反应条件为:94℃反应5min;94℃反应1min,55℃反应1min,72℃反应1min,共30个循环;72℃反应10min,得到扩增产物。2.8foupe1回补突变体的pcr验证分析按照真菌dna提取试剂盒法(omegafungaldnakit)说明书,提取上述博来霉素阳性转化子的基因组dna,进行pcr验证分析。用引物foupe1-f/foupe1-r进行基因片段foupe1的pcr扩增。pcr反应体系如下:模板dna0.5μlfoupe1-f(10μmol/l)0.5μlfoupe1-r(10μmol/l)0.5μl10×taqbuffer(mg2+plus)2.5μldntps(2.5mmol/l)2.0μltaq(5u/μl)0.25μlddh2o18.75μltotal25.0μlpcr反应条件为:94℃反应5min;94℃反应1min,55℃反应1min,72℃反应1min,共30个循环;72℃反应10min,得到扩增产物。2.9foc4敲除突变体的southernblot分析按照dighighprimednalabelinganddetectionstarterkiti(roche公司)说明书,进行southernblot杂交。用引物foupe1probe-f/foupe1probe-r扩增目的基因探针,用hph-f/hph-r扩增hph基因探针。foupe1probe-f:5′-ccaccctagaaatcgctcgt-3′,foupe1probe-r:5′-gatcttagcggtctcagcgt-3′,hph-f:5′-tgctgctccatacaagccaa-3′,hph-r:5′-gacattggggagttcagcga-3′;dna探针的pcr扩增体系如下:模板dna1.0μlfoupe1probe-f/hph-f(20μmol/l)1.0μlfoupe1probe-r/hph-r(20μmol/l)1.0μl10×extaqbuffer(mg2+plus)5.0μldntps(2.5mmol/l)4.0μlextaq(5u/μl)0.5μlddh2o37.5μltotal50.0μlpcr反应条件为:94℃反应5min;94℃反应1min,55℃反应1min,72℃反应1min,共30个循环;72℃反应10min,得到扩增产物。2.10foc4敲除突变体△foupe1和回补突变体△foupe1-com的表型观察(1)菌落形态观察及生长速度测定。将foc4、△foupe1和△foupe1-com分别接种于pda培养基上,于28℃黑暗条件下培养。在5d时采用十字交叉法测量菌落直径,并观察其菌落形态。每个处理设置3个重复。(2)分生孢子的获得。将香蕉枯萎病菌接种至查氏培养基,于28℃下120rpm培养,3d后统计产孢量。2.11敲除突变体△foupe1和回补突变体△foupe1-com抗胁迫分析将foc4、△foupe1和△foupe1-com分别接种至不同胁迫培养基(分别含1mol/lnacl、1mol/l山梨醇、50mmol/lh2o2、0.1%sds、100μg/ml荧光增白剂(cfw,钙荧光白)和100μg/ml刚果红)中,于28℃下培养5d,以pda培养基作为空白对照,采用十字交叉法测量菌落直径,计算不同胁迫条件下的菌落生长抑制率,每个处理设置3个重复。2.12敲除突变体△foupe1和回补突变体△foupe1-com的致病性分析取4叶期的巴西蕉,分别用foc4、△foupe1和△foupe1-com的分生孢子(2×105个/ml)悬浮液进行浸根30min,再移栽于营养土中;置于25±1℃的植物培养室内培养,于光/暗12h/12h交替培养,28d后观察香蕉苗叶片和球茎的发病情况。分别以foc4野生菌株和无菌水作为阳性和阴性对照。3结果与分析3.1香蕉枯萎病菌foupe1基因敲除载体的构建采用pcr扩增方法,以foc4基因组dna为模板分别克隆获得foupe1基因同源臂a片段和同源臂b片段;分别将其与t载体连接,经转化大肠杆菌、amp抗性筛选、质粒提取与测序鉴定,获得重组质粒pmd18t-foupe1-a和pmd18t-foupe1-b。将pmd18t-foupe1-a与pct74质粒连接,获得重组质粒pct74-foupe1-a;将其与pmd18t-foupe1-b进行双酶切,经dna连接、大肠杆菌转化、酶切鉴定,获得基因敲除载体pct74-foupe1-ko(图1)。3.2敲除突变体△foupe1的筛选3.2.1基因片段hph的pcr验证利用同源重组方法,将基因敲除载体pct74-foupe1-ko转化香蕉枯萎病菌原生质体,获得了117个潮霉素抗性转化子。经dna的提取,利用hph基因特异性引物,对117个潮霉素阳性转化子进行了pcr验证分析。结果表明,上述117个转化子均扩增到了hph基因,转化子71、76、113、117的验证结果如图2所示。3.2.2基因片段foupe1的pcr验证进一步利用foupe1基因特异性引物,对上述pcr扩增到hph基因的117个阳性转化子进行foupe1的pcr验证分析。结果表明,在117个转化子中,有4个转化子(转化子71、76、113、117)没有扩增到foupe1基因,进一步说明这4个转化子为阳性转化子(图3)。3.2.3敲除突变体△foupe1的southernblot验证对扩增到hph基因、同时没有扩增到foupe1基因的3个阳性转化子(转化子71、76、113)进行了southernblot分析。结果表明,以目的基因作为探针进行杂交,3个转化子均未有杂交条带(图4)。以hph为探针进行杂交,3个转化子均有单拷贝条带出现(图5)。上述试验进一步证明这3个转化子为阳性转化子。3.3回补突变体的筛选采用pcr扩增方法,克隆获得foupe1基因回补片段;将其与pctzn载体连接,经大肠杆菌转化、amp抗性筛选、质粒提取与测序鉴定,获得重组质粒pctzn-foupe1-com(图6)。利用随机插入的方法,将基因回补载体pctzn-foupe1-com转化敲除突变体△foupe1(△foupe1-71)原生质体,获得了21个博来霉素抗性转化子。经香蕉枯萎病菌基因组dna的提取,利用foupe1基因特异性引物,对上述转化子进行了pcr验证(图7)。结果表明,4个转化子可以扩增出目的基因,进一步说明这4个转化子为阳性转化子。3.4敲除突变体△foupe1和回补突变体△foupe1-com的菌落形态和生长速率测定将foc4、敲除突变体△foupe1(△foupe1-71)和回补突变体△foupe1-com(△foupe1-71-com-2)分别接种于pda培养基中,在接种5d后进行了菌落形态观察及菌落直径测定。结果表明,与foc4相比,△foupe1的生长速率显著下降,而回补突变体的生长速率恢复至野生型foc4水平。3.5敲除突变体△foupe1和回补突变体△foupe1-com的产孢量分析将foc4、敲除突变体△foupe1(△foupe1-71)、回补突变体△foupe1-com(△foupe1-71-com-2)分别接种于查氏培养基,培养3d后进行产孢量分析。结果表明,敲除突变体△foupe1的产孢量显著低于其foc4,而回补突变体△foupe1-com的产孢量恢复至野生型foc4水平。3.6敲除突变体△foupe1和回补突变体△foupe1-com对不同胁迫条件的分析将foc4、△foupe1(△foupe1-71、△foupe1-113)和△foupe1-com(△foupe1-71-com-2)分别接种在分别含1mol/lnacl、1mol/l山梨醇、0.1%sds、50mmol/lh2o2、100μg/ml荧光增白剂(cfw)和100μg/ml刚果红的pda培养基中,于28℃下培养5d后对其菌落直径进行测定。结果表明,与foc4相比,△foupe1对50mmol/lh2o2更敏感,对1mol/lnacl、1mol/l山梨醇、0.1%sds、100μg/ml荧光增白剂(cfw)和100μg/ml刚果红的敏感性无明显差异,说明foupe1在foc4应对氧化胁迫反应中具有重要作用(图8)。3.7敲除突变体△foupe1和回补突变体△foupe1-com的致病性分析利用伤根接种法,将foc4、△foupe1(△foupe1-71)和△foupe1-com(△foupe1-71-com-2)分生孢子液分别接种巴西蕉,于28d后进行观察。结果表明,foc4和△foupe1-com孢子液接种28d后,巴西蕉苗下部叶片出现黄化,叶片黄化面积约占叶面积的50~60%。纵剖发病巴西蕉苗球茎,可观察到黑褐色病变,褐变面积接近球茎面积的50%;而△foupe1-71孢子液接种28d后,巴西蕉下部叶片黄化面积约占叶面积的30%。纵剖发病巴西蕉苗,可观察到黑褐色病变,褐变面积约占球茎面积的25%。说明敲除foupe1基因后,香蕉枯萎病菌致病力明显下降(图9)。因此,本发明提供的基因可以用于植物病害防治,特别是由香蕉枯萎病菌所导致的香蕉枯萎病。另,本发明提供的基因可以作为用于植物病害防治的药物的靶标。本领域技术人员可以跟进本说明书的教导和启示,开发用于防治植物病害、特别是香蕉枯萎病的药物。上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。序列表<110>华南农业大学<120>蛋白foupe1在调控香蕉枯萎病菌致病力中的应用<160>14<170>siposequencelisting1.0<210>1<211>2733<212>dna<213>人工序列(artificialsequence)<220><223>foupe1基因的碱基序列<220><222>(1)..(1041)<223>非编码区1<220><222>(1042)..(1099)<223>外显子1<220><222>(1100)..(1155)<223>内含子1<220><222>(1156)..(1996)<223>外显子2<220><222>(1997)..(2051)<223>内含子2<220><222>(2052)..(2349)<223>外显子3<220><222>(2350)..(2733)<223>非编码区2<400>1cgtccagtccacactggagtctggagtccagctcgccctggcatcgtcttgggccggtac60tggttcctttttggtcttcagtgtctgagtgcccctgggataatgggccccgaaactggt120gactgacctgtcggtgccctgcttttgtccatgaacttaggttttgccatccaactcgaa180cccaccctggtagctccccccatgccccatgcaggcggcgccgaattcccaccgtgcact240catctcggtcgaccatctttcatctcctccctcccatctcctcctctctttttctctcgc300tccttcgaacttcttctttcctctccttgtcggtaatattctcgtccagtttttctcttc360acgccactcgttttcgtcaggctctactctgccagactttatctagttctaggaatattc420ttaactgttccgtgatatatctcagtcgttcgccgagaaactctcaaggatttgaagaat480caacaccatccaacaaaatcacaggatcctttccccttacaaattaggtacgtacgaacg540ttcatcgctcggtcgactggctcgatccattgtgaggtgcttgccgagacgcgcatttac600ttggcttgtgctccttgattggtttcgaccgttccgtcccgtctttcttgccgtcttgtt660ttctttcttccttcccctgcctcaaataatctttttgtgtacactcttcctcctccattc720actttgcgatttggtctattcttgcctacgattggaattgacgcatctcctcatcccccc780ttcacgcattccttccaggttggttgccattgaacattccctttcctgccaaagggggcg840ttgctatcgagcatcgacatctccacgacattgtactgcagttgcttgctcgacaccact900ctacatcagcatcacttctttcgaggcggttctactttggaccgacattcatagggacca960ccgcgacagaccagcccacagttttcttcacgcactcgtgtataatcaacgttatctaat1020tgtttgtttatccaggccataatgcattccgttaaggtcctctcggccattgcggccctc1080agtgtctctgccgtttctggtaggtgaaatcagatacaccgtctcggatatcctgctgac1140aacgaccttgtctagctgccacttgtaccaaggacgtcaagatcaccgaacctacacaag1200tcatcagctgcgatgttgtcgacgccgatatcatcattgacaagtctgttgctggtgccg1260ttgtcatcaacggccccaagcagatcaagggcaacttccaggccaagaacgccggtgacc1320ttatctctatctccagtacctctattaattccatcactggaaagttcgagctcaacaacc1380tcgaggctctcaacaacctcgacttctcttctctcgagagccttggcgagcttagcttca1440tcaagcttccccgactcggcgagctgaacttcggtaccgaaggcgttactaagatcaaga1500gcattcgaattaccgacaccttcatcagtgatcttagcggtctcagcgtcgccagtgtcg1560agagcttccagattgacaacaaccgaaagatgaacgtcttcaactccgaccttgttaatg1620ttactacccagctcctgatcttcgacaatggcaacgacgttatggaaattaccatgaaca1680agctcgagactgctgccgagatccagatttctagcgccaagtccttcaaggtccctgctc1740tccaggaggtgaccaagagcttgaagttgagcgccaaccccgagctcaagtctttctccg1800cccccaacctgaccacgatcacggagaccctgtcccttatcgacatgaacaagctcacca1860acgtttccttccccgccctcgaggagattggcggcggtttcaccatccagaacaacacca1920agcttgaggctattgatgatttccccaagctcgagaaggtcaccggtggtattgccctcc1980gaggtagcttcgagaagtgagtaaaacccctctagcatatagcatttccatatctaacga2040gcgatttctagggtggaacttcccaagctcgaccaagttagtggcagtgtcgttgtttcc2100tcgaccaccgatattaaggagttctgtgattacttcgacaacctcaagaaggacaagaag2160atcgatggtgaggagaagtgcacatccaacaacaaggcggccaatgagggcaaggacggt2220ggtgaggagagcgacggctcttccgagagcagcaacagtgacgatcagaaggatgctgct2280ggcattgtcagcgtcaacatggctgttcttgctcttgcaggtattgctgccgttgcccag2340ctcttttaagcatggagatgaagattgctggcttttcttttaaaggcgtcattatcgtcg2400tcatcatgtttcctgacttttctctacgacatattgtgcttggtcaacatggggcacaaa2460atgcttttcatgggtgggggtaactgtgtgtaaaggataacctgttagagcttaataatc2520gattgtatttggactttattcttgagatcatgttcgccgagctctgaaaattgacatctc2580tcttaactcatcatataaagatgtttagtttgtgataagcgcttcttatttcttctgaac2640atcgagagtcatcaggaaaccttttcggtctgcaactgatataaaggattgcctagttgt2700tggtctattatgaatataagtatatctccatcc2733<210>2<211>398<212>prt<213>人工序列(artificialsequence)<220><223>foupe1蛋白质的氨基酸序列<400>2methisservallysvalleuseralailealaalaleuservalser151015alavalseralaalathrcysthrlysaspvallysilethrglupro202530thrglnvalilesercysaspvalvalaspalaaspileileileasp354045lysservalalaglyalavalvalileasnglyprolysglnilelys505560glyasnpheglnalalysasnalaglyaspleuileserileserser65707580thrserileasnserilethrglylysphegluleuasnasnleuglu859095alaleuasnasnleuasppheserserleugluserleuglygluleu100105110serpheilelysleuproargleuglygluleuasnpheglythrglu115120125glyvalthrlysilelysserileargilethraspthrpheileser130135140aspleuserglyleuservalalaservalgluserpheglnileasp145150155160asnasnarglysmetasnvalpheasnseraspleuvalasnvalthr165170175thrglnleuleuilepheaspasnglyasnaspvalmetgluilethr180185190metasnlysleugluthralaalagluileglnileserseralalys195200205serphelysvalproalaleuglngluvalthrlysserleulysleu210215220seralaasnprogluleulysserpheseralaproasnleuthrthr225230235240ilethrgluthrleuserleuileaspmetasnlysleuthrasnval245250255serpheproalaleuglugluileglyglyglyphethrileglnasn260265270asnthrlysleuglualaileaspasppheprolysleuglulysval275280285thrglyglyilealaleuargglyserpheglulysvalgluleupro290295300lysleuaspglnvalserglyservalvalvalserserthrthrasp305310315320ilelysgluphecysasptyrpheaspasnleulyslysasplyslys325330335ileaspglygluglulyscysthrserasnasnlysalaalaasnglu340345350glylysaspglyglyglugluseraspglysersergluserserasn355360365seraspaspglnlysaspalaalaglyilevalservalasnmetala370375380valleualaleualaglyilealaalavalalaglnleuphe385390395<210>3<211>25<212>dna<213>人工序列(artificialsequence)<220><223>foupe1-af<400>3ggggtaccggtgcgacgggtccatt25<210>4<211>31<212>dna<213>人工序列(artificialsequence)<220><223>foupe1-ar<400>4ccgctcgagatgtctccgtctgttagcaagt31<210>5<211>30<212>dna<213>人工序列(artificialsequence)<220><223>foupe1-bf<400>5tccccccgggctgatgacgattctcgggct30<210>6<211>32<212>dna<213>人工序列(artificialsequence)<220><223>foupe1-br<400>6gctctagaacctgatcgccaactattctttac32<210>7<211>30<212>dna<213>人工序列(artificialsequence)<220><223>foupe1-com-f<400>7gactagttgcatagtgatgagaaagtccaa30<210>8<211>39<212>dna<213>人工序列(artificialsequence)<220><223>foupe1-com-r<400>8ataagaatgcggccgcaagtcaaagagtggtgagcacat39<210>9<211>20<212>dna<213>人工序列(artificialsequence)<220><223>hph-f<400>9tgctgctccatacaagccaa20<210>10<211>20<212>dna<213>人工序列(artificialsequence)<220><223>hph-r<400>10gacattggggagttcagcga20<210>11<211>24<212>dna<213>人工序列(artificialsequence)<220><223>foupe1-f<400>11agaccaacaactaggcaatccttt24<210>12<211>20<212>dna<213>人工序列(artificialsequence)<220><223>foupe1-r<400>12gtcttgggccggtactggtt20<210>13<211>20<212>dna<213>人工序列(artificialsequence)<220><223>foupe1probe-f<400>13ccaccctagaaatcgctcgt20<210>14<211>20<212>dna<213>人工序列(artificialsequence)<220><223>foupe1probe-r<400>14gatcttagcggtctcagcgt20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1