一种基于快速扫描和无动态排除的质谱采集模式的制作方法
本发明涉及一种基于快速扫描和无动态排除的质谱采集模式及其在蛋白质组学相对定量方面的应用。该方法在不影响蛋白质组鉴定数目的情况下能有效提高无标记相对定量及基于等重标记的相对定量的定量准确度。
背景技术:
定量分析蛋白质组的动态变化,是当前研究蛋白质功能、揭示细胞生物机理、寻找疾病蛋白标记物和药物靶标的迫切需要。在过去的数十年中,基于质谱的蛋白质组定量方法已逐渐取代传统的基于二维凝胶电泳的定量方法。到目前为止,基于质谱的蛋白质组学定量方法主要是在基于数据依赖模式的质谱采集模式下完成的。
基于数据依赖的质谱采集模式一个重要的功能就是动态排除。它是指对于已经碎裂过的母离子,在一定的时间窗口内不再碎裂该母离子,这种功能的好处在于能够将有限的质谱扫描次数充分利用,以更好的鉴定低丰度的肽段。但对于蛋白质组定量来说,这种功能存在如下缺点:1、动态排除功能的存在会使得每一个肽段只被碎裂一次或很有限的几次,而对于基于二级谱的定量来说,对应于同一肽段的谱图越多则定量越准(即碎裂次数越多越好);2、在基于等重标记的相对定量方法里,某些标记方法会引入H/D同位素差异,从而引起两份等重标记的肽段之间发生色谱峰偏移,如果只碎裂一次或有限的次数,则会进一步影响这种方法的定量准确度(Proteomics.doi:10.1002/pmic.201400262)。
为了解决上述问题,基于选择性扫描的目标蛋白质组学是一个很好的途径,但它的通量有限,一次实验只能分析几十到几百条肽段。近两年出现的基于数据非依赖模式的质谱扫描方法理论上是解决上述问题的最佳途径,但目前由于对该方法获得的数据存在数据解析的困难,因此还未能得到广泛的使用(Mol.Cell.Proteomics 2012,11(6),1-17)。
在本发明中,我们发展了一种介于数据非依赖扫描和基于动态排除的数据依赖扫描之间的质谱扫描模式,即基于质谱快速扫描和无动态排除模式相结合的质谱扫描模式,有效的提高了蛋白质组无标记相对定量和基于等重标记的相对定量的定量准确度。
技术实现要素:
本发明发展了一种基于快速扫描和无动态排除的质谱采集模式并将其应用于蛋白质组学相对定量分析,该方法在不影响蛋白质组鉴定数目的情况下能有效提高无标记相对定量及基于等重标记的相对定量的定量准确度,本发明的技术方案是:
在质谱数据采集方面,使用的是无动态排除模式下一个一级质谱扫描后接二十一次以上二级质谱扫描的质谱扫描模式;在无标记定量数据分析方面,使用两次质谱分析中同一色谱保留时间窗口下对应于同一肽段的谱图中匹配到的一个以上碎片离子的强度比值平均值作为最终的肽段强度比值;在基于等重标记的相对定量方面,使用的是同一肽段对应的一张以上二级质谱谱图所反映的比例的平均值作为最终的肽段强度比值。
在质谱数据采集方面,使用的是无动态排除模式下一个一级质谱扫描后接二十一次以上二级质谱扫描的质谱扫描模式,其中二级质谱扫描的次数为21-100中的任意整数。
在无标记定量数据分析方面,使用的是两次质谱分析中同一色谱保留时间窗口下对应于同一肽段的谱图中共同鉴定到的一个以上碎片离子的强度比值平均值作为最终的肽段强度比值,所采用的碎片离子个数为1个到共同鉴定到的全部碎片离子个数之间的任意整数。
在基于等重标记的相对定量方面,使用的是同一肽段对应的一张以上二级质谱谱图所反映的比例的平均值作为最终的肽段强度比值,计算比例所用的谱图数目为1张谱图到该肽段对应的所有谱图个数之间的任意整数,且计算每张谱图比例的算法为PISA算法、force search或force find算法中的任意一种。
质谱数据采集所采用的质谱仪按其质量分析器类型为四极杆质谱仪、离子阱质谱仪、飞行时间质谱仪、傅里叶变换离子回旋共振质谱仪、傅里叶变换静电场轨道阱质谱仪或磁质谱仪中的任意一种或二种以上。
上述模式可以应用于正常样本与疾病样本间蛋白质组的差异定量分析、疾病发展各时期间的蛋白质组定量分析、生物体受外界刺激所产生应激反应时蛋白质组变化的定量分析。
本发明的有益效果为:
1、在不影响蛋白质鉴定数目的前提下,提高了分析的重现性;
2、提高了无标记相对定量的定量准确度、精密度;
3、提高了基于等重标记的相对定量的定量准确度、精密度,改善了由于同位素效应导致的色谱峰偏移带来的定量不准问题;
4、无需复杂的数据处理软件,仅通过常规的蛋白质组学数据处理软件就能完成数据的分析。
5、适用面广,可以适用于所有基于质谱的蛋白质组学研究。
附图说明
图1方法的操作流程图,A:分别在传统的基于动态排除的数据依赖模式下和我们提出的基于质谱快速扫描和无动态排除的模式下对酵母酶解产物进行蛋白质组鉴定;B:分别在传统的基于动态排除的数据依赖模式下和我们提出的基于质谱快速扫描和无动态排除的模式下对等重标记的等比 例混合HeLa蛋白进行蛋白质组定量分析;C:分别在传统的基于动态排除的数据依赖模式下和我们提出的基于质谱快速扫描和无动态排除的模式下对等比例的酵母酶解产物进行无标记相对定量分析。
图2两种质谱采集模式下酵母蛋白质鉴定的结果及三次进样蛋白质鉴定结果间的overlap,A:采用基于质谱快速扫描和无动态排除的模式下对酵母酶解产物进行蛋白质组鉴定的结果及三次进样蛋白质鉴定结果间的overlap;B:采用传统的基于动态排除的数据依赖模式下对酵母酶解产物进行蛋白质组鉴定的结果及三次进样蛋白质鉴定结果间的overlap。
图3两种质谱采集模式下对1:1混合的等重标记的HeLa细胞进行蛋白质组定量分析的结果,A:采用基于质谱快速扫描和无动态排除的模式下对1:1混合的等重标记的HeLa细胞进行蛋白质组定量分析的结果;B:采用传统的基于动态排除的数据依赖模式下对1:1混合的等重标记的HeLa细胞进行蛋白质组定量分析的结果。
图4两种质谱采集模式下对等比例的酵母细胞进行酶解产物进行无标记相对定量分析的结果,A:采用基于质谱快速扫描和无动态排除的模式下对等比例的酵母细胞进行酶解产物进行无标记相对定量分析的结果;B:采用传统的基于动态排除的数据依赖模式下对等比例的酵母细胞进行酶解产物进行无标记相对定量分析的结果。
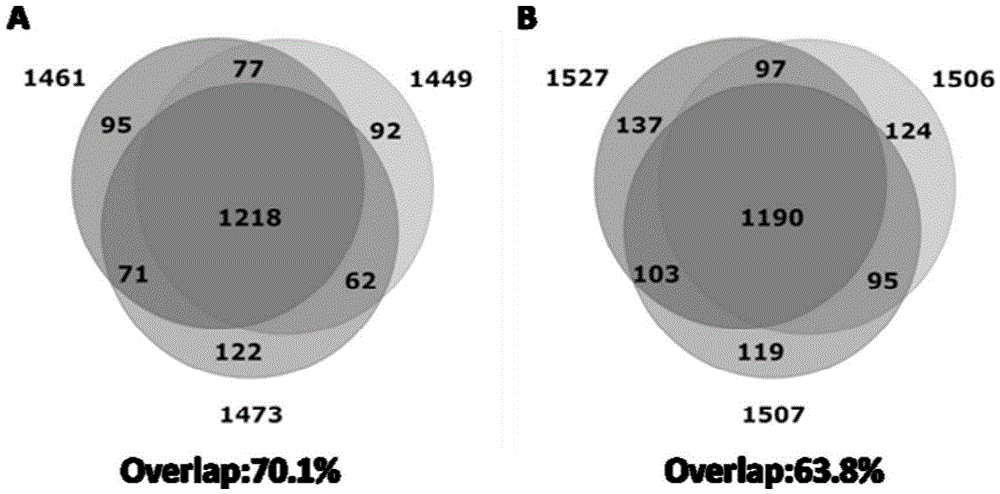
图5两种质谱采集模式下HeLa蛋白质鉴定的结果及三次进样蛋白质鉴定结果间的overlap,A:采用传统的基于动态排除的数据依赖模式下对酵母酶解产物进行蛋白质组鉴定的结果及三次进样蛋白质鉴定结果间的overlap;B:采用基于质谱快速扫描和无动态排除的模式下对酵母酶解产物进行蛋白质组鉴定的结果及三次进样蛋白质鉴定结果间的overlap。
具体实施方式
实施例1
如图1A所示,酵母蛋白首先由胰蛋白酶酶解,其中酶用量为1/25(w/w),温度为37℃,pH=7.5,酶解16h。将酶解产物使用Eksigent液相系统进行上样和液相分离(上样量为2μg,分离时间为120min)、AB Triple-TOF 5600进行质谱分析,质谱分析分别在传统的基于动态排除的数据依赖模式下(一个一级质谱后跟40个二级质谱,二级质谱累积时间为50ms,动态排除设定为21s排除1次)和我们提出的基于质谱快速扫描和无动态排除的模式下(一个一级质谱后跟100个二级质谱,二级质谱累积时间为30ms,无动态排除模式)进行蛋白质组鉴定,具体鉴定结果如果2所示,图2A为基于质谱快速扫描和无动态排除的模式下的三次质谱进样的鉴定结果及其overlap,图2B为传统的基于动态排除的数据依赖模式下的三次质谱进样的鉴定结果及其overlap。说明采用本发明与传统的基于动态排除的数据依赖模式采集相比,蛋白质组鉴定能力基本相当,且采用本发明的鉴定结果重 现性更好。
实施例2
如图1B所示,SILAC标记的两份HeLa蛋白(K0,K6)首先由Lys-C酶解,其中酶用量为1/50(w/w),温度为37℃,pH=7.5,酶解16h。向两份酶解产物中加入40μL 2M邻甲基异脲硫酸盐,于pH 11下进行胍基化2h,然后使用1%TFA调节pH至7.5,然后向K0标记的肽段中加入32μL 4%(v/v)13CD2O以及32μL 0.6M氰基硼氰化钠,向K6标记的肽段中加入32μL4%(v/v)CH2O以及32μL 0.6M氰基硼氰化钠,均反应1h后除盐蒸干,混合后使用Eksigent液相系统进行上样和液相分离(上样量为2μg,分离时间为120min)、AB Triple-TOF 5600进行质谱分析,分别在传统的基于动态排除的数据依赖模式下(一个一级质谱后跟40个二级质谱,二级质谱累积时间为50ms,动态排除设定为21s排除1次)和我们提出的基于质谱快速扫描和无动态排除的模式下(一个一级质谱后跟100个二级质谱,二级质谱累积时间为30ms,无动态排除模式)进行蛋白质定量分析,具体定量结果如图3所示,图3A为基于质谱快速扫描和无动态排除的模式下的肽段和蛋白质定量结果,图3B为传统的基于动态排除的数据依赖模式下的肽段和蛋白质定量结果。说明采用本发明能提高基于等重标记的相对定量的定量准确度、精密度,改善了由于同位素效应导致的色谱峰偏移带来的定量不准问题。
实施例3
如图1C所示,两份酵母蛋白首先由Trypsin酶解,其中酶用量为1/25(w/w),温度为37℃,pH=7.5,酶解16h。将两份酶解产物使用Eksigent液相系统进行上样和液相分离(上样量为2μg,分离时间为120min)、AB Triple-TOF 5600进行质谱分析,分别在传统的基于动态排除的数据依赖模式下(一个一级质谱后跟40个二级质谱,二级质谱累积时间为50ms,动态排除设定为21s排除1次)和我们提出的基于质谱快速扫描和无动态排除的模式下(一个一级质谱后跟100个二级质谱,二级质谱累积时间为30ms,无动态排除模式)进行蛋白质组定量分析,具体定量结果如图4所示,图4A为基于质谱快速扫描和无动态排除的模式下的肽段和蛋白质定量结果,图4B为传统的基于动态排除的数据依赖模式下的肽段和蛋白质定量结果。说明采用本发明能提高了无标记相对定量的定量准确度、精密度。
实施例4
如图5所示,将Hela细胞溶于2mL 8M的尿素溶液(溶于50mM碳酸氢铵溶液,pH 8)对蛋白质进行提取并测浓度后,取200μg的蛋白,加入200μL 10mM二硫苏糖醇溶液,56℃下,反应2h进行蛋白质的变性和还原处理。随后,通入200μL 25mM碘乙酰胺溶液,室温下避光反应40min进行蛋白质的烷基化处理。最后,加入25μg的胰蛋白酶(溶于50mM碳酸氢铵溶液,pH 8)进行蛋白质的酶解,酶解时间为16h。之后进行蛋白质 酶解产物的液相分级:将200μg的上述蛋白质酶解产物溶液经高pH反相色谱分级,流动相A(98%(v/v)水和2%(v/v)乙腈,使用氨水将pH调至10)和流动相B(2%(v/v)水和98%(v/v)乙腈,使用氨水将pH调制10)用于进行梯度分离。梯度为0-20%B(0-40min),20%-30%B(40-50min),30-80%B(50-60min)。自动馏分收集器以3min的时间间隔收集馏分,将相隔30min的间隔组分进行混合(馏分1和11,馏分2和12等),最终得到10个混合馏分进行冻干,保存待用。最后分别在传统的基于动态排除的数据依赖模式下(一个一级质谱后跟40个二级质谱,二级质谱累积时间为50ms,动态排除设定为21s排除1次)和我们提出的基于质谱快速扫描和无动态排除的模式下(一个一级质谱后跟100个二级质谱,二级质谱累积时间为30ms,无动态排除模式)进行蛋白质鉴定。图5A为传统的基于动态排除的数据依赖模式下的三次质谱进样的鉴定结果及其overlap,图5B为基于质谱快速扫描和无动态排除的模式下的三次质谱进样的鉴定结果及其overlap。说明在常规的蛋白质组大规模分级鉴定时,本发明相比于传统的基于动态排除的数据依赖模式在蛋白质组鉴定数目上更具优势。
实施例5
将经SILAC试剂盒培养且赖氨酸轻、重标记的人肝癌高低转移细胞株细胞按1:1(细胞计数比)混合,首先由Lys-C酶解,其中酶用量为1/50(w/w),温度为37℃,pH=7.5,酶解16h。将两份酶解产物中加入40μL 2M邻甲基异脲硫酸盐,于pH 11下进行胍基化2h,然后使用1%TFA调节pH至7.5,然后向K0标记的肽段中加入32μL 4%(v/v)13CD2O以及32μL 0.6M氰基硼氰化钠,向K6标记的肽段中加入32μL 4%(v/v)CH2O以及32μL 0.6M氰基硼氰化钠,均反应1h后除盐蒸干至干粉状。将两份肽段以1:1的质量比重溶混合后进行蛋白质酶解产物的液相分级:将200μg的上述蛋白质酶解产物溶液经高pH反相色谱分级,流动相A为98%(v/v)水和2%(v/v)乙腈,并使用氨水将pH调至10,流动相B为2%(v/v)水和98%(v/v)乙腈,并使用氨水将pH调制10。梯度为0-20%B(0-40min),20%-30%B(40-50min),30-80%B(50-60min)。自动馏分收集器以3min的时间间隔收集馏分,将相隔30min的间隔组分进行混合(馏分1和11,馏分2和12等),最终得到10个混合馏分进行冻干,保存待用。最后在我们提出的基于质谱快速扫描和无动态排除的模式下(一个一级质谱后跟100个二级质谱,二级质谱累积时间为30ms,无动态排除模式)进行蛋白质鉴定和定量分析。对结果进行分析发现大部分差异蛋白都已经被文献报道,说明本发明具有较好的定量准确度。
- 还没有人留言评论。精彩留言会获得点赞!