一种Y单倍群检测方法与流程
本发明涉及基因测绘技术,更具体地说,涉及一种y单倍群检测方法。
背景技术:
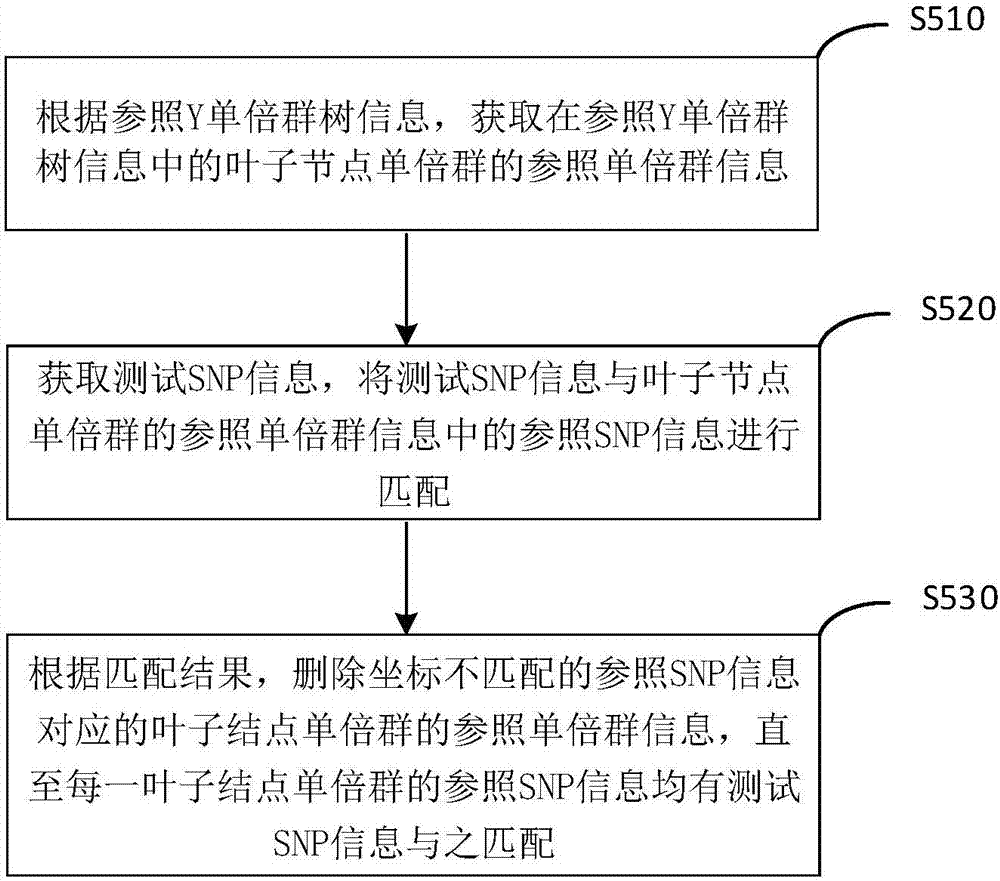
:人的基因组由22对常染色体和xy两条性染色体组成,其中22对常染色体在男性和女性中都有两条;而性染色体,女性有两个x染色体,男性则有一条x染色体和一条y染色体。所以,y染色体在人类遗传中,仅能由男性个体从其父亲遗传得到,而且该染色体不会发生同源重组的现象,因此可以用其上的snp(单核苷酸多态性singlenucleotidepolymorphism)标记,利用分子生物学的方法,来反演推算父系祖源,并形成一个有根的y染色体单倍群树。这个y染色体单倍群树,将现代人类分为18个大类型,用a到r的十八个字母作为索引。树上的父节点对应的snp是所有子节点共有的。这个树表征了人类从最早的根结点,在迁徙繁衍过程中的不断在y染色体上累积突变的过程。目前,有isogg、yfull等机构收录并升级各个研究的y单倍群树结构的结果,包括树上的分支结构,以及各个节点对应的单倍群名字和所包含的snp位点。现有的y单倍体检测算法大多是科学研究工具,如amy-tree算法,使用的方法适用于二代测序结果,在其算法中会挑选y单倍群树中,对研究深入的单倍群会有倾向性输出,而且少量在单倍群树的叶子节点的假阳性测序结果,会很容被输出为计算结果,导致计算错误。yhap则是利用群体的低深度测序结果进行预测,不适用直接得到基因型的snp分型数据。因此,上述检测算法存在不能同时适用二代测序结果和高通量microarray(芯片数据)的snp分型结果,使得检测算法适用性不够高,且容易出错的问题。技术实现要素:本发明要解决的技术问题在于,针对现有技术的上述检测算法存在不能同时适用二代测序结果和高通量microarray的snp分型结果,使得检测算法适用性不够高,且容易出错的的缺陷,提供一种y单倍群检测方法。本发明解决其技术问题所采用的技术方案是:构造一种y单倍群检测方法,所述方法包括以下步骤:获取用于参照的y单倍群树的参照y单倍群树信息,其中,所述参照y单倍群树信息包括所述y单倍群树中每个参照单倍群的参照单倍群信息,所述参照单倍群信息包括参照单倍群坐标信息;将所述每个参照单倍群的参照单倍群信息与每个测试snp的测试snp信息进行匹配,得到至少一个满足预设条件的目标参照单倍群;根据所述目标参照单倍群的参照单倍群坐标信息,获取与所述目标参照单倍群相关的第一数值与第二数值,其中,所述第一数值为从所述目标参照单倍群到与所述目标参照单倍群对应的根节点单倍群之间路径上的其他目标参照单倍群数量,所述第二数值为所述其他目标参照单倍群数量与在所述路径上总的有效单倍群数量的比值;根据所述第一数值与第二数值对每个所述目标参照单倍群进行评分,输出评分最高的目标参照单倍群所对应的结果信息。实施本发明的y单倍群检测方法,具有以下有益效果:1、通过将参照单倍群与测试单倍群进行比对,利用从根节点出发到目标单倍群的路径所获得的数据对该目标单倍群的评分,同时适用二代测序结果和高通量microarray的snp分型结果,使得检测适用性更高,提高检测手段的灵活度及应用广泛度;2、利用高通量的snp位点分型结果,通过遍历y单倍群树的所有可能结果,基于从根节点出发的全路径评分系统,得到准确的y单倍群分型结果,具有使y单倍群的检测过程更加高效、稳定的特点;3、该检测方法具有很强的容错性,可以根据实际检测的y染色体dna数量和质量,灵活调整参数,即使是质量较低的检测数据集也能得到较好的结果。附图说明下面将结合附图及实施例对本发明作进一步说明,附图中:图1是本发明实施例一种y单倍群检测方法的实现流程示意图;图2是本发明实施例中删除不相关叶子节点单倍群的实现流程示意图;图3是本发明实施例中测试snp信息与叶子节点单倍群中参照snp信息的匹配实现流程示意图;图4是本发明实施例中获得目标参照单倍群的实现流程示意图;图5是本发明实施例中参照单倍群的参照snp信息与测试snp信息匹配的实现流程示意图;图6是本发明实施例中对目标参照单倍群进行标记的实现流程示意图;图7是本发明实施例中一种输出评分最高的目标参照单倍群所对应的结果信息的实现流程示意图。具体实施方式为了使本发明的目的、技术方案及优点更加清楚明白,以下结合附图及实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。人的基因组由22对常染色体和xy两条性染色体组成,其中22对常染色体在男性和女性中都有两条;而性染色体,女性有两个x染色体,男性则有一条x染色体和一条y染色体。所以,y染色体在人类遗传中,仅能由男性个体从其父亲遗传得到,而且该染色体不会发生同源重组的现象,因此可以用其上的snp标记,利用分子生物学的方法,来反演推算父系祖源,并形成一个有根的y染色体单倍群树。这个y染色体单倍群树,将现代人类分为18个大类型,用a到r的十八个字母作为索引。树上的父节点对应的snp是所有子节点共有的。这个树表征了人类从最早的根结点,在迁徙繁衍过程中的不断在y染色体上累积突变的过程。snp是单核苷酸多态性(singlenucleotidepolymorphism)的简写,是指基因组上单个核苷酸变异,即a、t、c、g四种碱基的互相改变,形成基因组上同一位置会有多种碱基存在的多态性。snp在人群中广泛存在,多态性丰富,是很好的遗传标记物。尤其高通量的snp检测方法出现之后,被广泛用于生物信息学的分析。本发明是基于y染色体上的单核苷酸多态性(snp)建立的应用。本发明实施例适用于运行在python环境上,并且,python可以运行于debian系统,也可将其移植并运行于linux和windows系统,当然除了上述环境或平台,也可采用其他的环境或平台,本发明实施例对此不作限定。图1示出了本发明实施例中一种y单倍群检测方法的流程,为了便于说明,仅示出了与本发明实施例相关的部分。如图1所示,在本发明的实施例中,包括以下步骤s:步骤s100,获取用于参照的y单倍群树的参照y单倍群树信息,其中,参照y单倍群树信息包括y单倍群树中每个参照单倍群的参照单倍群信息,参照单倍群信息包括参照单倍群坐标信息;步骤s200,将每个参照单倍群的参照单倍群信息与测试y单倍群树中每个测试单倍群的测试单倍群信息进行匹配,得到至少一个满足预设条件的目标参照单倍群;步骤s300,根据目标参照单倍群的参照单倍群坐标信息,获取与目标参照单倍群相关的第一数值与第二数值,其中,第一数值为从目标参照单倍群到与目标参照单倍群对应的根节点单倍群之间路径上的其他目标参照单倍群数量,第二数值为其他目标参照单倍群数量与在路径上总的有效单倍群数量的比值;步骤s400,根据第一数值与第二数值对每个目标参照单倍群进行评分,输出评分最高的目标参照单倍群所对应的结果信息。本发明实施例中,通过将参照单倍群与测试单倍群进行比对,利用从根节点出发到目标单倍群的路径所获得的数据对该目标单倍群的评分,可同时适用二代测序结果和高通量microarray的snp分型结果,使得检测适用性更好,提高检测手段的灵活度及应用广泛度;且具有使y单倍群的检测过程更加高效、稳定的有益效果。在本发明实施例中,用于参照的y单倍群树的参照y单倍群树信息,可以是采用isogg公司或者yfull公司的y单倍群树信息作为参照y单倍群树信息。以isogg的y单倍群树举例子,其中共包含2531个参照单倍群。具体的,在参照y单倍群树信息中,还包含该参照y单倍群信息中的每个y单倍群对应的snp文件每个snp对应的坐标,突变位点的状态以及对应的参照单倍群名称。本发明实施例所采用的isogg发布的y单倍群树信息如表1所示:表1a00rootl1122l1104l1106l1107l1284l1102af05a0-trootl1155l1105l1124l1129l1095l1098l1116根据表1,可以很清楚的看到,图中每一行代表一个参照y单倍群节点,其中第一列是该参照单倍群名称,第二列是该参照单倍群的父节点的名称。第三列开始,是该参照单倍群所包含的snp的名字。本发明实施例所采用的y单倍群树信息中的每个参照单倍群对应的snp文件如表2所示:表2a945702a2b1a1a5b8550434c→aa945802a2b1a1a5b19335462t→c根据表2,图中每一行代表一个参照snp,其中第一列是该snp的名字,第二列是其所属的单倍群,第三列是该snp在y染色体上的坐标,第四列是该snp的碱基突变类型,如第一行的c->a,代表该snp正常情况下是碱基c,不属于o2a2b1a1a5b单倍群对应的突变,如果突变成了a,则代表这个snp属于o2a2b1a1a5b单倍群对应的突变。本发明实施例所采用的用户的测试snp数据,可以是微整列芯片的snp的结果,也可以是二代测序的snp结果。需要给出测试snp的检测基因型,包括突变和未突变的测试snp位点。数据如表3所示:表3chry21152971cchry23541348gchry14263051c根据表3,图中每一行代表y染色体上一个snp,每一列分别代表染色体名字,测试snp的坐标信息,测试snp的检测结果。如图2所示,图2示出了本发明实施例的为了提高相关检测的效率及降低检测出错率,删除不相关叶子节点单倍群的流程,为了便于说明,仅示出了与本发明实施例相关的部分。本发明实施例提供了一种删除不相关叶子节点单倍群的流程,如图2所示,该流程包括:步骤s510,根据参照y单倍群树信息,获取在参照y单倍群树信息中的叶子节点单倍群的参照单倍群信息;叶子节点单倍群为没有子节点单倍群的参照单倍群,位于y单倍群树的末端。步骤s520,获取测试snp信息,将测试snp信息与叶子节点单倍群的参照单倍群信息中的参照snp信息进行坐标匹配;步骤s530,根据匹配结果,删除坐标不匹配的参照snp信息对应的所述叶子节点单倍群的参照单倍群信息,直至每一叶子节点单倍群的参照snp信息均有所述测试snp信息与之匹配。如图3所示,图3示出了一种测试snp信息与叶子节点单倍群中参照snp信息的匹配实现流程,为了便于说明,仅示出了与本发明实施例相关的部分。在本发明实施例中,该匹配实现流程包括:步骤s521,获取测试snp信息,以及参照单倍群信息的参照snp信息;作为本发明的一种实施例,测试snp信息从检测人员提供的对用户检测所获得的snp检测结果上获取,具体的,所获取的测试snp信息具有如表2中所列举的数据格式。步骤s522,将测试snp信息中的测试snp位点信息与参照snp信息中的参照snp位点信息进行匹配。例如,若叶子节点单倍群为o2a2b1a1a5,其包含cts1017,cts7316,cts10738这三个参照snp,将上述cts1017,cts7316,cts10738的参照snp位点信息即其坐标,与用户的测试snp位点信息进行比对,看该叶子节点单倍群o2a2b1a1a5中的参照snp位点信息是否在测试snp位点信息中。本发明实施例对所有经获取的叶子节点单倍群的参照snp进行与测试snp信息的匹配遍历,查找每个是否有测试snp的坐标位点与其参照snp匹配。根据匹配结果,删除坐标匹配的参照snp信息的叶子节点单倍群,直至每一叶子节点单倍群的参照snp信息均有测试snp信息与之匹配。例如,若叶子节点单倍群为o2a2b1a1a5,其包含cts1017,cts7316,cts10738这三个参照snp,将上述cts1017,cts7316,cts10738的参照snp位点信息即其坐标,与用户的测试snp位点信息进行比对,若该叶子节点单倍群o2a2b1a1a5中的参照snp位点信息均未在该测试snp位点信息中,则可以认定该叶子节点单倍群o2a2b1a1a5为不相关的叶子节点单倍群,执行将其名称及参照单倍群信息删除的指令,以此一一对所有叶子节点单倍群进行匹配测试,以修剪不相关的叶子节点单倍群,最后使得参照y单倍群树信息中的叶子节点单倍群均包含有与测试snp位点相对的参照单倍群。若该参照y单倍群树信息中,包含有过多与测试snp不相关的叶子节点单倍群,不仅会影响测试snp的检测效率,而且可能会提高出错率。本发明实施例先获取参照y单倍群树信息中的叶子节点单倍群的参照单倍群信息,将不含有测试snp位点的叶子节点单倍群的信息删除,当对参照snp与测试snp进行遍历匹配的过程中,可以有效降低检测出错率,提高检测效率。如图4所示,图4示出了本发明实施例中获得目标参照单倍群的具体流程,为了便于说明,仅示出了与本发明实施例相关的部分。在本发明实施例中,该流程包括:步骤s210,遍历所述参照y单倍群树信息;步骤s220,若所述参照y单倍群树信息中有参照单倍群的参照snp信息与所述测试snp信息匹配,则将所述参照snp信息对应的参照snp定义为目标snp;在本发明实施例中,匹配时,可将上述表2中的参照snp信息与表3中的测试snp信息进行对比匹配。优选的,如图5所示,图5示出了本发明实施例的参照单倍群的参照snp信息与测试snp信息匹配的具体流程,为了便于说明,仅示出了与本发明实施例相关的部分。该流程包括:步骤s221,获取每个测试snp信息的测试snp位点信息,以及每个所述参照snp信息的参照snp位点信息;步骤s222,若一所述测试snp位点信息与一参照snp位点信息匹配,则将配对后的所述测试snp的测试snp碱基突变类型信息,和与之配对的参照snp的参照snp分型结果信息进行再次匹配;在本发明实施例中,首先,将测试snp遍历所有的树上的节点,若有一个测试snp位点信息与一个参照y单倍群树节点上的单倍群a0a1a所包含的snp名称为v169的参照snp位点信息一样,则对该测试snp的分型结果与该单倍群a0a1a的碱基突变类型进行再次匹配。步骤s223,若两次匹配均成功,则将匹配成功的所述测试snp定义为目标snp。可以理解的,若两次匹配均成功,该测试snp为大概率与所匹配的参照单倍群一致,两次匹配可以保证其检测结果的准确性。步骤s230,若有所述参照单倍群满足至少包含有预设比例值的目标snp,则将满足条件的所述参照单倍群定义为目标参照单倍群。例如,将含有目标snp的数量与该参照单倍群的总参照snp数量的比值为0.1设置为该预设比例值的阈值,对于参照y单倍群树某一节点的单倍群名称为o2a2b1a1a5,包含cts1017,cts7316,cts10738,cts1017,m1543,m1694,cts10738,m1726共8个参照snp,如果检测结果中检测了其中7个,并且7个中有cts1017,cts7316两个测试snp信息符合o2a2b1a1a5中的突变情况,则该节点满足上述匹配条件的测试snp比例达到了0.286(2/7),超过了预设的0.1的阈值,将o2a2b1a1a5标记为目标参照单倍群。优选的,图6示出了本发明实施例中对目标参照单倍群进行标记的流程,为了便于说明,仅示出了与本发明实施例相关的部分。在本发明实施例中,包括以下判断方式:若所述参照单倍群为目标参照单倍群,则将所述参照单倍群的状态信息定义为true;若所述参照单倍群包含有目标snp但比例低于预设比例值,则将所述参照单倍群的状态信息定义为false;若所述参照单倍群不包含与测试snp坐标匹配的参照snp,则将所述参照单倍群的状态信息定义为none。进一步的,有效单倍群是状态信息为true或false的参照单倍群。如此标记,可以清楚地区分用户不同测试snp与参照y单倍群树中的参照snp之间的匹配程度,将用户所有的测试snp区分为true、false、none三种匹配度,在评分过程中,只将定义为true和false两种状态的参照单倍群作为有效单倍群,进而将有效单倍群作为评分基础,不仅有利于提高检测的效率,还可以提高该评分的可靠度与准确度。在评分的时候为了能更加方便、直观,目标参照单倍群的评分为所述第一数值与第二数值的乘积,其中,第一数值为从目标参照单倍群到与目标参照单倍群对应的根节点单倍群之间路径上的其他目标参照单倍群数量,第二数值为其他目标参照单倍群数量与在路径上总的有效单倍群数量的比值。在本发明实施例中,计算的公式可以为score=p(true)*n(true)。其中p(true)是从y单倍群树的根节点单倍群出发,到该目标参照单倍群的整个路径上通过的所有的节点,其状态为true占所有不是none的节点的比例。举例来说,如某一目标参照单倍群o2a2b1a1a5为true,则找到从root开始到它,经过所有的节点为“a0-t”,“a1”,“a1b”,“bt”,“ct”,“cf”,“f”,“ghijk”,“hijk”,“ijk”,“k”,“k2”,“no”,“o”,“o2”,“o2a”,“o2a2”,“o2a2b”,“o2a2b1”,“o2a2b1a”,“o2a2b1a1”,“o2a2b1a1a”,“o2a2b1a1a5”,共23个,如果其中20个节点状态为true,2个为false,1个为none,则p(true)等于0.91(20/22)。n(true)为路径上所有状态为true的点,在举例中n(true)等于20。整体的评分所得分数(score)等于p(true)*n(true),即18.2(0.91*20)。对所有的点,都计算出对应的分数值。可以理解的,除了上述将第一数值与第二数值相乘得出数值的方式,也可以将第一数值与第二数值作为参考值,进行任何方式的权重计算,以得出对检测结果的比对有利的评分方式。结合上述标记的方法,对所有状态为true的节点,计算评价分数,可以给出一个较为具有参考价值的结果。图7示出了本发明实施例中一种输出评分最高的目标参照单倍群所对应的结果信息的流程,为了便于说明,仅示出了与本发明实施例相关的部分。作为本发明的一种实施例,该具体流程包括:步骤s410,若评分最高的目标参照单倍群为唯一,则将评分最高的目标参照单倍群所对应的结果进行输出;例如,当有“o2a2a”“a0a2”“a0b”3个目标参照单倍群时,若其评分分别是“16.8”“21.3”“19.1”,则将获得最高分“21.3”的“a0a2”结果进行输出。步骤s420,若评分最高的目标参照单倍群不唯一,则比较各个评分最高的目标参照单倍群的目标snp所占比例,将其中目标snp所占比例最高的目标参照单倍群所对应的结果信息进行输出。例如,当有“o2a2a”,“a0a2”,“a0b”3个目标参照单倍群时,若其评分分别是“16.8”“19.1”“19.1”,则“a0a2”,“a0b”同时获得相同的最高分,然后需对比“a0a2”,“a0b”中目标snp所占比例,若“a0a2”,“a0b”的目标snp所占比例分别是“0.4”,“0.3”,则将“a0a2”的结果进行输出。步骤s430,若评分最高的目标参照单倍群不唯一,且其中目标snp所占比例均相同,则随机输出其中一个评分最高的目标参照单倍群所对应的结果信息。例如,当有“o2a2a”,“a0a2”,“a0b”3个目标参照单倍群时,若其评分分别是“19.1”,“19.1”,“19.1”,则“o2a2a”,“a0a2”,“a0b”同时获得相同的最高分,且若其目标snp所占比例均相同,则直接随机输出一个目标参照单倍群的结果。可以理解的,上述结果输出方法只是其中一种优选方案,也可以根据实际需要进行设计,例如同时输出最高的两个或两个以上数据作为参考,本发明实施例对此不作限定。在本发明实施例中,结果信息包括评分最高的目标参照单倍群所对应的名称信息、坐标信息、与目标参照单倍群对应的根节点单倍群之间的路径信息的一个或多个。上述信息可以清楚的表示检测结果的信息,方便检测者根据该信息作出分析及处理。下面将列举23andme、amytree和本方法(记为“本发明实施例”)在不同数据集中的表现情况,来展示本方法的有益效果。1、使用千人基因组phase3的数据比较,得到的y染色体单倍型结果如表4所示:表4可以看到,三种发放在大部分人的结果分析上有一致的结论(如果不同的单倍群结果,单倍群名字前面的字母和数字一样的话,可以认为是大致一致的,只是精度有差别)。在不一致的部分,本发明实施例与23andme的结果一致性较好,amytree部分结果与这两个有较大差异(三角形标记)。2.使用本发明实施例检测的部分microarray数据比较三种方法,得到的y染色体单倍型结果如表5所示:表5id23andmeyhaplo本发明实施例amytree22271602272658ao-tδn1c2b2n1c2b227311602271750ao-tδr1a1a1b2a2ar1a1a1b2a2a33241602272739ao-tδc1a1a1c1a1a237661602274626ao-tδo1b1a1a1a1a2o1b1a1a1a1a237726031800398ao-tδo2b1ao2b1a3839548230069ao-tδr1a1a1b2a2b1br1a1a1b2a2b1b43385432200146ao-tδo2a2b1a2a1a1a1δc2e1b2δ47061602272612ao-tδc2e1a1ac2e1b1a48085262300489ao-tδo2a1a2o2a1a253071602274893ao-tδo1a2o1b1a1a1b57635432200796ao-tδe1a2b1a2δd1b2a2δ61295432200700ao-tδq1a2a1c1δt1a1a3δ67541602272634ao-tδo1a1a1a1a1o1b1a1a1b79941602272898ao-tδo1b2ao1b1a1a1b92551602274266ao-tδq1a1a1q1a1a1可以看到,23andme的方法出现了明显的问题,所有人的结果都被认为是相同的,a0-t是非常原始的单倍群,几乎不可能在中国人群总检测到。而本发明实施例的结果更具合理性,得到的结果都是中国人群可能出现的单倍群类型。3.考察三个方法在缺失数据情况下的表现。a.随机取千人基因组50%的数据,得到的y染色体单倍型结果如表6所示:表6b.随机取千人基因组10%的数据,得到的y染色体单倍型结果,如表7所示:表7c.随机取千人基因组5%的数据,得到的y染色体单倍型结果如表8所示:表8通过随机挑选一定比例的千人基因组可以看到,23andme和本方法的一致性很好而amytree的算法则很不稳定。对比挑选位点之前的结果,23andme和本发明实施例的方法也有很稳定的输出,而amytree的结果则很容易受异常值得影响,导致不同数据集之间的结果波动很大。在本发明实施例中,结合上述测试实验结果可知本发明实施例具有以下有益效果:1、通过将参照单倍群与测试单倍群进行比对,利用从根节点出发到目标单倍群的路径所获得的数据对该目标单倍群的评分,可以同时适用二代测序结果和高通量microarray的snp分型结果,使得检测适用性更高,提高检测手段的灵活度及应用广泛度;2、利用高通量的snp位点分型结果,通过遍历y单倍群树的所有可能结果,基于从根节点出发的全路径评分系统,得到准确的y单倍群分型结果,具有使y单倍群的检测过程更加高效、稳定的特点;3、该检测方法具有很强的容错性,可以根据实际检测的y染色体dna数量和质量,灵活调整参数,即使是质量较低的检测数据集也能得到较好的结果。本领域普通技术人员可以理解实现上述实施例方法中的全部或部分流程,是可以通过计算机程序来指令相关的硬件来完成,所述的程序可存储于计算机、手机等终端设备的可读取存储介质中,该程序在执行时,可包括如上述各方法的实施例的流程。其中,所述的存储介质可为磁碟、光盘、只读存储记忆体(read-onlymemory,rom)或随机存储记忆体(randomaccessmemory,ram)等。可以理解的,以上实施例仅表达了本发明的优选实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制;应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,可以对上述技术特点进行自由组合,还可以做出若干变形和改进,这些都属于本发明的保护范围;因此,凡跟本发明权利要求范围所做的等同变换与修饰,均应属于本发明权利要求的涵盖范围。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1