一种针对胚胎染色体的序列数据处理装置的制作方法
本发明涉及数据处理技术,尤其涉及一种针对胚胎染色体的序列数据处理装置,适用于胚胎染色体非整倍体检测技术中。
背景技术:
染色体异常是导致自然流产、出生缺陷、胎儿多发畸形等重要临床因素。所述染色体异常包括有染色体数目异常和染色体微缺失微重复。其中,孕早期原因未明的自然流产中大部分是染色体非整倍体所致,b超显示存在多发畸形的胎儿中10%异常存在染色体非整倍体,出生缺陷的新生儿中约20%也为染色体异常所致。因此,对染色体异常进行检测,这一方面对于早期自然流产,有利于排查流产是否为胎儿染色体异常所致,特别是对多次未明原因的孕早期反复流产的孕妇,可以对夫妻双方进行染色体异常检测,以减少再次妊娠时异常患儿出生的可能性;另一方面,有利于早期发现胎儿异常是否为染色体异常所致,为医生提供诊断的辅助信息,从而实现胎儿异常的早期治疗,进而降低出生缺陷。
此外,近年来,人类辅助生殖技术的快速发展使得“试管婴儿”技术逐渐应用于临床,帮助更多不孕不育或年龄较大或携带遗传疾病的夫妻获得下一代。然而大量临床研究发现,在体外受精形成的胚胎中,大约一半左右的胚胎存在染色体异常的现象,这往往是许多孕妇会出现反复种植失败或自然流产或死产的主要原因[1]。而且随着孕妇年龄增加,胚胎发生染色体异常的风险也越高,极大地限制了辅助生殖技术的成功率。因此,胚胎植入前能对胚胎染色体异常的准确筛查,进而选择健康的胚胎植入,是能显著提高试管婴儿的妊娠率和活产率。
目前,针对染色体异常检测的方法主要包括有fish、微阵列-比较基因组杂交(array-cgh)技术和高通量测序技术。荧光原位杂交技术(fluorescenceinsituhybridization,fish)是早期染色体异常检测的黄金标准。虽然fish具有快速、特异性高等优势,但是由于受到探针种类和标记荧光素种类限制,使得该技术仅能一次对部分染色体数目异常进行检测,而不能在全基因组的水平上进行检测。目前更为普遍应用于染色体异常检测的方法是微阵列-比较基因组杂交(array-cgh)技术[2]。相比fish技术,array-cgh技术可以仅通过一次杂交实验就能检测全部23对染色体数目变化,但是其检测的分辨率取决于探针的密度(探针未覆盖的区域是无法检测到的),若要从全基因组水平上检测23对染色体异常的情况,就必须增加探针的数量,大大地增加了成本费用。而随着高通量测序成本的降低,近几年来,基于高通量测序技术进行胚胎染色体非整倍体检测的方法逐渐成为主流。
基于高通量测序技术来检测胚胎染色体非整倍体的主要过程如下:1)、获取合理数量的dna模板(流产物组织或胚胎组织则可以直接酶切或超声将dna片段化;而囊胚细胞或者卵裂细胞由于起始的dna模板为微克级别所以需要提前进行单细胞扩增);2)、选择一定片段大小的dna分子(如150-250bp);3)、构建文库,在上述dna分子两端加上测序用接头;4)、上机测序获得一定长度的序列(reads);5)、利用比对软件将序列(reads)比对到人类参考基因组,过滤重复序列和低质量的序列,得到各染色体不同位置的序列数目(readsnumber)和序列比例(readsratio);6)、利用统计模型判断胚胎是否存在染色体异常。当胚胎出现染色体非整倍体时,相应染色体总数会有一定比例的升高或降低,因此可以与一定量样本构成的参考集合相比较或者自身样本内比较来判断染色体是否存在异常。染色体异常检测的统计学方法主要可以分为参考样本集合比较和自身样本内比较两种方法。
参考样本集合比较的代表性方法是z检验[3]:z检验模型利用大量正常样本构建参考数据库,得到参考数据集中各染色体的读长比例(readsratio)的均值和标准差,然后计算待测样本在每条染色体中的z-score,根据z-score来判断样本是否为非整倍体。但是,z检验模型所存有的主要问题是待测样本的z-score大小对参考数据集的模型依赖性很强,如果待测样本和参考样本集合的数据一致性低的时候会导致灵敏性和特异性严重降低。对于胚胎植入前非整倍体筛查(pgs),胚胎的起始dna含量约为6.6pg~30pg,dna起始的模板含量非常低,所以需要进行全基因组扩增(wholegenomeamplification,wga)然后测序,而全基因组扩增会引入严重的gc偏好,这往往导致待测样本和参考数据集样本的一致性很差,可见,z-score模型不适用于胚胎植入前染色体非整倍体检测方法。
因此,胚胎植入前筛查主要采用自身样本内比较的方法:将基因组分成不同窗口大小的bins(数据箱),统计所有bins的序列比例(copyratio),然后通过读长比例的变化趋势来推断是否存在染色体异常[4]。而基于自身样本内比较的检验方法的主要问题则在于检验的结果只基于单一样本的单一的统计指标“copyratio”,当单细胞扩增均一性较差时,“copyratio”的波动性很大,会出现大量的异常值以及假阳性的结果。因此为了解决传统自身样本内比较方法所产生的结果准确度和可靠性低下的问题,本发明针对自身样本内比较方法的数据处理过程提出了改进。
参考文献
1.bielanska,m.,s.l.tan,anda.ao,chromosomalmosaicismthroughouthumanpreimplantationdevelopmentinvitro:incidence,type,andrelevancetoembryooutcome.humreprod,2002.17(2):p.413-9.
2.gutierrez-mateo,c.,etal.,validationofmicroarraycomparativegenomichybridizationforcomprehensivechromosomeanalysisofembryos.fertilsteril,2011.95(3):p.953-8.
3.chiu,r.w.,etal.,noninvasiveprenataldiagnosisoffetalchromosomalaneuploidybymassivelyparalelgenomicsequencingofdnainmaternalplasma.procnatlacadsciusa,2008.105(51):p.20458-63.
4.fu,y.,etal.,uniformandaccuratesingle-celsequencingbasedonemulsionwhole-genomeamplification.procnatlacadsciusa,2015.112(38):p.11923-8.
技术实现要素:
为了解决上述技术问题,本发明的目的是提供一种针对胚胎染色体的序列数据处理装置。
本发明所采用的技术方案是:一种针对胚胎染色体的序列数据处理装置,该装置包括:
测序数据获取单元,用于获取经高通量测序后得到的dna读长片段;
测序数据处理单元,用于将获得的dna读长片段与人类基因组标准序列进行比对,将各dna读长片段比对到染色体相应位置,从而得到各dna读长片段所对应的染色体、起始位点及序列长度,以及唯一完全匹配序列;
数据结果分析单元,用于根据唯一完全匹配序列的读长片段分布情况,划分不同的读长区间,计算每条染色体上每个长度区间的dna片段比例,根据待测染色体不同长度区间下的dna片段比例与已知常染色体在不同长度区间下的dna片段比例两者之间的差异,判断待测染色体是否为非整倍体;
其中,所述dna片段比例是根据长度区间下的dna片段数目、样本在长度区间下的所有常染色体的dna片段数总和以及染色体的长度计算得出。
进一步,所述染色体上长度区间的dna片段比例,其所采用的计算公式如下所示:
其中,i表示为染色体编号;j表示为长度区间编号;ratioij表示为第i号染色体上第j个长度区间下的dna片段比例;reads_nij表示为第i号染色体上第j个长度区间下的dna片段数目;reads_nj表示为样本在第j个长度区间下的所有常染色体的dna片段数总和;chr_leni表示为第i号染色体的长度。
进一步,所述根据待测染色体不同长度区间下的dna片段比例与已知常染色体在不同长度区间下的dna片段比例两者之间的差异,判断待测染色体是否为非整倍体这一步骤,其具体包括:
判断待测染色体不同长度区间下的dna片段比例与已知常染色体在不同长度区间下的dna片段比例两者之间的差异是否符合统计学意义上显著差异的标准,若是,则判断待测染色体为非整倍体,反之,则判断待测染色体不为非整倍体。
进一步,所述染色体的长度指的是染色体过滤掉着丝粒、端粒和随体区后的长度。
进一步,所述读长区间的划分采用滑窗法来实现。
本发明所采用的另一技术方案是:一种针对胚胎染色体的序列数据处理装置,包括处理器,适于实现各种指令,所述指令适于由处理器加载并执行以下步骤:
获取经高通量测序后得到的dna读长片段;
将获得的dna读长片段与人类基因组标准序列进行比对,将各dna读长片段比对到染色体相应位置,从而得到各dna读长片段所对应的染色体、起始位点及序列长度,以及唯一完全匹配序列;
根据唯一完全匹配序列的读长片段分布情况,划分不同的读长区间,计算每条染色体上每个长度区间的dna片段比例,根据待测染色体不同长度区间下的dna片段比例与已知常染色体在不同长度区间下的dna片段比例两者之间的差异,判断待测染色体是否为非整倍体;
其中,所述dna片段比例是根据长度区间下的dna片段数目、样本在长度区间下的所有常染色体的dna片段数总和以及染色体的长度计算得出。
进一步,所述染色体上长度区间的dna片段比例,其所采用的计算公式如下所示:
其中,i表示为染色体编号;j表示为长度区间编号;ratioij表示为第i号染色体上第j个长度区间下的dna片段比例;reads_nij表示为第i号染色体上第j个长度区间下的dna片段数目;reads_nj表示为样本在第j个长度区间下的所有常染色体的dna片段数总和;chr_leni表示为第i号染色体的长度。
进一步,所述根据待测染色体不同长度区间下的dna片段比例与已知常染色体在不同长度区间下的dna片段比例两者之间的差异,判断待测染色体是否为非整倍体这一步骤,其具体包括:
判断待测染色体不同长度区间下的dna片段比例与已知常染色体在不同长度区间下的dna片段比例两者之间的差异是否符合统计学意义上显著差异的标准,若是,则判断待测染色体为非整倍体,反之,则判断待测染色体不为非整倍体。
进一步,所述染色体的长度指的是染色体过滤掉着丝粒、端粒和随体区后的长度。
进一步,所述读长区间的划分采用滑窗法来实现。
本发明的有益效果是:通过将本发明装置应用于传统自身样本内比较方法,来实现胚胎染色体数目异常时,不仅准确率高,而且本装置不需要利用正常阴性样本构建的参考集作为参照,避免了参考样本集合比较方法在参考样本集和待测样本存在严重偏差导致的假阳性和假阴性。同时,本发明装置引入了各染色体的读长信息,令对染色体异常的判断不单单依赖于序列比例(copyratio)的数值变化,而且还需要考察copyratio在不同读长(readslength)比例下的特征变化是否合理,对染色体是否存在异常的判断更为准确,可以同时降低假阳性率和假阳性率。
附图说明
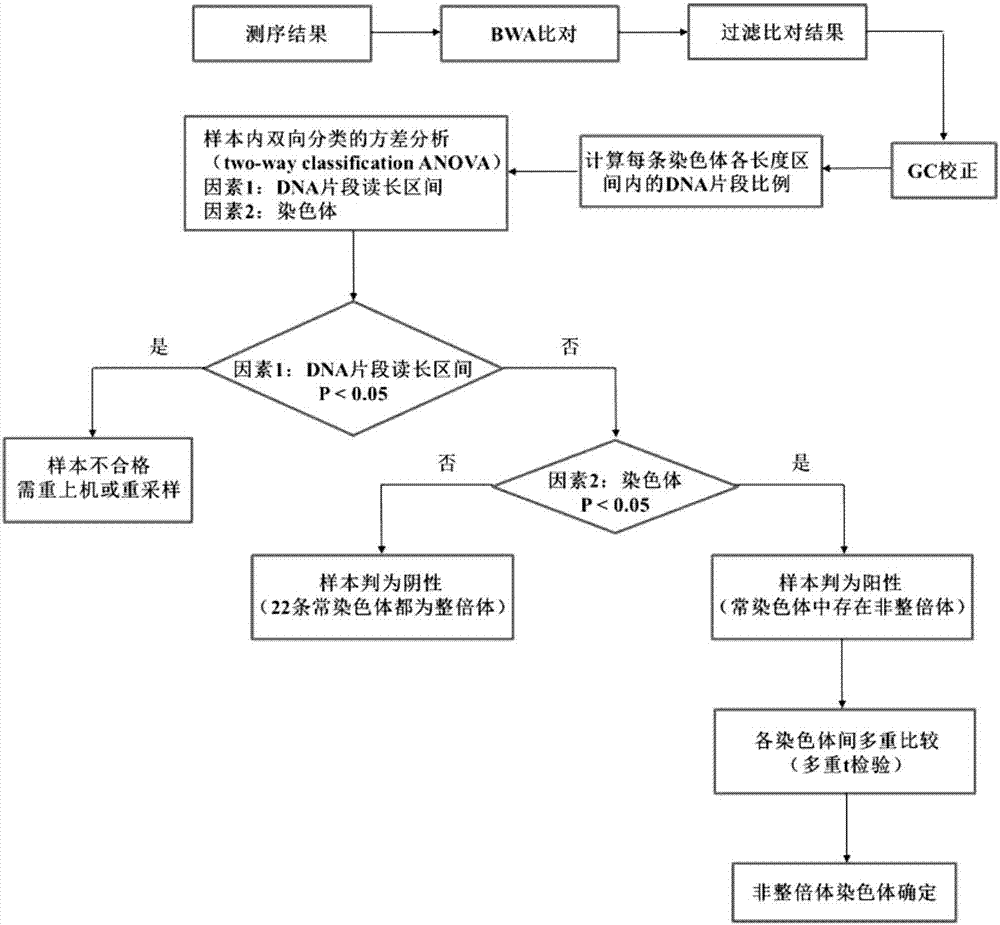
图1是基于高通量测序数据进行胚胎染色体非整倍体判定的分析流程图;
图2是羊水细胞t2样本各染色体多重比较后各染色体的p值指数分布图;
图3是羊水细胞t2样本各染色体多重比较的p值表;
图4是卵裂球单细胞扩增产物t4样本各染色体多重比较后各染色体的p值指数分布图;
图5是卵裂球单细胞扩增产物t4样本各染色体多重比较的p值表。
具体实施方式
本发明的思想为:在自身样本内比较方法的基础上,引入序列的长度信息,利用序列的长度对染色体的copyratio值进行分类,同时,本发明判断染色体是否存在异常时除了考虑序列比例(readsratio)的变化,还考虑了不同读长(readslength)的序列比例的数值是否合理,因此通过使用本发明装置而得出的检测结果更为准确可靠,且可同时减少假阳性率和假阴性率。可见,本发明不仅仅适用于流产物和胚胎组织的染色体异常检测,也适用于基于单细胞扩增的胚胎植入前筛查,是一项通用的检测装置。
以下结合具体实施例来对本发明装置进行详细阐述。
实施例1
一种针对胚胎染色体的序列数据处理装置,具体包括:
测序数据获取单元,用于获取经高通量测序后得到的dna读长片段;其中,所述dna读长片段指的是测序获得的dna信息,包括dna碱基序列和长度等信息;
其中,获取得到的dna读长片段,其是对卵裂球单细胞扩增产物或流产组织或羊水细胞中的dna进行高通量测序后而得到的dna读长片段;
测序数据处理单元,用于将获得的dna读长片段与人类基因组标准序列hg19进行比对,将各dna读长片段比对到染色体相应位置,从而得到各dna读长片段所对应的染色体、具体起始位点及序列长度;同时,在dna读长片段与人类基因组标准序列hg19进行比对过程中,通过剔除处于串联重复位置及转座子重复位置的核苷酸序列,以及低质量的、多匹配和非完全匹配到染色体上的核苷酸序列后,得到unique序列,即唯一完全匹配序列;
数据结果分析单元,用于根据unique序列的读长片段分布情况,划分不同的读长区间,不同的读长区间为不同长度区间;
采用滑窗法计算每条染色体上每个长度区间的dna片段比例,然后对计算出的dna片段比例进行gc校正,通过比较校正后的待测染色体不同长度区间下的dna片段比例与其他已知常染色体在不同长度区间下的dna片段比例的差异是否具有显著性,从而判断待测染色体是否为非整倍体;
优选地,所述采用滑窗法计算每条染色体上每个长度区间的dna片段比例这一步骤,其具体包括:
采用滑窗法,按照预设的长度梯度和step(步长),将dna读长片段分别划分到不同的长度区间,具体地,以10bp作为长度梯度(窗口),以10bp作为step(步长),得到不同长度片段区间为:[100,110),[110,120),[120,130),……,[210,220),[220,230);
然后,为了考虑到染色体之间长度不同,在dna片段比例计算公式中引入染色体长度变量,实现不同染色体之间readsratio的度量单位统一,即,所述染色体上长度区间的dna片段比例,其所采用的第一计算公式如下所示:
其中,i表示为染色体编号;j表示为长度区间编号;ratioij表示为第i号染色体上第j个长度区间下的dna片段比例;reads_nij表示为第i号染色体上第j个长度区间下的dna片段数目;reads_nj表示为样本在第j个长度区间下的所有常染色体的dna片段数总和;chr_leni表示为第i号染色体的长度;
其中,上述经统计得出的长度区间下的dna片段数目是基于gc校正后的读长片段分布情况进行统计得出的;
优选地,所述通过比较校正后的待测染色体不同长度区间下的dna片段比例与其他已知常染色体在不同长度区间下的dna片段比例的差异是否具有显著性,从而判断待测染色体是否为非整倍体这一步骤,其具体包括:
判断待测染色体不同长度区间下的dna片段比例与其他已知常染色体在不同长度区间下的dna片段比例,两者之间的差异是否符合统计学意义上显著差异的标准,具体地,即判断单位染色体长度下不同长度区间内dna读长片段比例是否有统计学意义上的显著差异,若是,则判断待测染色体为非整倍体,反之,则判断待测染色体不为非整倍体。
上述测序数据获取单元、测序数据处理单元及数据结果分析单元可为程序模块,也可为硬件设备模块。
实施例2
一种针对胚胎染色体的序列数据处理装置,包括处理器,适于实现各种指令,所述指令适于由处理器加载并执行以下步骤:
s101、获取经高通量测序后得到的dna读长片段,其中,所述dna读长片段指的是测序获得的dna信息,包括dna碱基序列和长度等信息;
其中,获取得到的dna读长片段,其是对卵裂球单细胞扩增产物或流产组织或羊水细胞中的dna进行高通量测序后而得到的dna读长片段;
s102、将获得的dna读长片段与人类基因组标准序列hg19进行比对,将各dna读长片段比对到染色体相应位置,从而得到各dna读长片段所对应的染色体、具体起始位点及序列长度;同时,在dna读长片段与人类基因组标准序列hg19进行比对过程中,通过剔除处于串联重复位置及转座子重复位置的核苷酸序列,以及低质量的、多匹配和非完全匹配到染色体上的核苷酸序列后,得到unique序列,即唯一完全匹配序列;
s103、根据unique序列的读长片段分布情况,划分不同的读长区间,不同的读长区间对应不同长度区间;统计出不同染色体不同长度区间下的dna片段数目,当待测染色体不同长度区间下的dna片段数目与对应长度区间下其他已知常染色体的dna片段数目,两者之间的数值差符合显著性条件时,即待测染色体不同长度区间下的dna片段数目显著多于或少于对应长度区间下其他常染色体的dna片段数目时,则判断该待测染色体为非整倍体;
优选地,在所述统计出不同染色体不同长度区间下的dna片段数目这一步骤之前设有校正步骤,所述校正步骤为:对unique序列的读长片段分布情况进行gc校正;也就是说,
不同染色体不同长度区间下的dna片段数目是基于gc校正后的dna片段分布情况进行统计的;
s104、采用滑窗法计算每条染色体上每个长度区间的dna片段比例,然后对计算出的dna片段比例进行gc校正,通过比较校正后的待测染色体不同长度区间下的dna片段比例与其他已知常染色体在不同长度区间下的dna片段比例的差异是否具有显著性,从而判断待测染色体是否为非整倍体;
优选地,所述采用滑窗法计算每条染色体上每个长度区间的dna片段比例这一步骤,其具体包括:
采用滑窗法,按照预设的长度梯度和step(步长),将dna读长片段分别划分到不同的长度区间,具体地,以10bp作为长度梯度(窗口),以10bp作为step(步长),得到不同长度片段区间为:[100,110),[110,120),[120,130),……,[210,220),[220,230);
然后,为了考虑到染色体之间长度不同,在dna片段比例计算公式中引入染色体长度变量,实现不同染色体之间readsratio的度量单位统一,即,所述染色体上长度区间的dna片段比例,其所采用的第一计算公式如下所示:
其中,i表示为染色体编号;j表示为长度区间编号;ratioij表示为第i号染色体上第j个长度区间下的dna片段比例;reads_nij表示为第i号染色体上第j个长度区间下的dna片段数目;reads_nj表示为样本在第j个长度区间下的所有常染色体的dna片段数总和;chr_leni表示为第i号染色体的长度;
优选地,所述通过比较校正后的待测染色体不同长度区间下的dna片段比例与其他已知常染色体在不同长度区间下的dna片段比例的差异是否具有显著性,从而判断待测染色体是否为非整倍体这一步骤,其具体包括:
判断待测染色体不同长度区间下的dna片段比例与其他已知常染色体在不同长度区间下的dna片段比例,两者之间的差异是否符合统计学意义上显著差异的标准,具体地,即判断单位染色体长度下不同长度区间内dna读长片段比例是否有统计学意义上的显著差异,若是,则判断待测染色体为非整倍体,反之,则判断待测染色体不为非整倍体。
实施例3
将上述一种针对胚胎染色体的序列数据处理装置应用在胚胎染色体非整倍体检测技术中,其具体检测实现部分包括以下六个部分,并且具体实现流程步骤如图1所示。
第一部分、样本来源:2例样本来自羊水细胞,其核型分析结果分别为46,xn和47,xn,+16;2例样本来自胚胎卵裂时期的卵裂球单细胞扩增产物,其array-cgh芯片分析结果分别为46,xn和47,xn,+9。
第二部分、测序数据比对与质控
将测序数据与人类基因组标准序列hg19进行比对,确定dna片段序列在染色体上的准确位置。为了保证测序结果的质量及避免一些重复序列的干扰,剔除低质量的序列,并对位于基因组串联重复及转座重复区域的碱基进行过滤,最终获得唯一匹配的dna片段,即unique序列。
第三部分、gc校正
为了消除gc含量对不同染色体不同长度区间内dna片段数目影响,统计不同gc含量组下dna片段数目,并利用中位数对其进行校正。
第四部分、计算待测样本内各染色体各长度区间的dna片段比例
a、实施例中以10bp作为长度梯度(窗口),以10bp作为step(步长),得到不同长度片段区间为:[100,110),[110,120),[120,130),……,[210,220),[220,230);
b、统计样本内各长度区间经gc校正后的dna片段总数;
c、统计样本内各染色体各长度区间经gc校正后的dna片段数;
d、根据上述第一计算公式,计算待测样本内各染色体各长度区间的dna片段比例。结果如表1-4所示,其中i为第i号染色体,j为第j组长度区间。
表1羊水细胞样本t1中各常染色体各长度区间对应的dna片段比例
表2羊水细胞样本t2中各常染色体各长度区间对应的dna片段比例
表3卵裂球单细胞扩增产物样本t3中各常染色体各长度区间对应的dna片段比例
表4卵裂球单细胞扩增产物样本t4中各常染色体各长度区间对应的dna片段比例
第五部分、对校正后的dna片段比例进行双向分类的方差分析(two-wayclassificationanova)
a、两个因素:因素1:dna片段读长区间,因素2:染色体,不考虑交互作用。根据p值和显著性水平,判断各染色体不同长度区间下dna片段比例有无差别;
b、考虑dna片段长度和染色体两个因素,对dna片段比例进行双向分类的方差分析(假设h0:22条常染色体dna片段比例总体均数都相等,即不考虑性染色体情况下,该样本为阴性样本;h1:22条常染色体dna片段比例总体均数不全相等,即该样本为阳性样本,存在非整倍体染色体);
c、方差分析结果判读:对于因素1—dna片段读长区间,如果p值(方差检验结果对应的概率值)小于显著水平0.05,说明不同染色体不同长度区间下的dna片段比例的差异受到该因素影响,因此该样本的结果是不可靠的(因为不同dna片段长度产生是通过酶切随机片段化产生,dna片段长度与dna片段比例是没有联系的);如果p值大于0.05,说明该样本结果是合理的,可以进一步对因素2结果进行分析;对于因素2—染色体,如果p值大于0.05,说明不同染色体之间的dna片段比例没有显著差异,22条常染色体都为整倍体,故可判断为正常样本(不考虑性染色体情况下);如果p值小于0.05,说明不同染色体之间dna片段存在显著差异,22条常染色体中存在非整倍体染色体,故接下来需要进行多条染色体间的多重比较,从而确定哪条染色体为非整倍体。
d、根据方差分析结果,计算p值。结果如表5所示(p1:不同dna片段读长区间因素;p2:染色体因素)。
表5方差分析的p值结果
注:t1和t2为羊水细胞;t3和t4为卵裂球单细胞扩增产物。
根据上述表5,判断如下:
1)对于t1,p1和p2都大于0.05,故可推断为正常样本;同理,推断出t3为正常样本。
2)对于t2,p1大于0.05,而p2小于0.05,则认为该样本存在非整倍体染色体,故判断
为阳性样本;同理,推断出t4也为阳性样本。
第六部分、对异常样本的各染色体间dna片段均值进行多重比较
由于方差分析只能判定该样本是否存在非整倍体染色体,而不能确定具体是哪条异常,因此,利用多重t检验对方差分析判定为异常的样本进行均值的多重比较。即对每条染色体的dna片段比例的总体均值而言,分别与其他21条染色体的dna片段比例的总体均值进行差异性比较,方法是采用两正态总体均值的t检验。由于多次重复使用t检验会增大犯ⅰ类错误(把本无差别的两个总体均数判为有差别)的概率,从而使得“有显著差异”的结论不一定可靠。因此,采用bonferroni方法对p值进行调整。
对上述两例异常样本(t2和t4)进行多重比较分析,p值结果如图3所示。
对于t2样本,从图2的方差分析的p值指数的分布图可以看出,16号染色体与其他染色体有明显差异,图3中多重比较的p值也可以看出,16号染色体与其他染色体之间都呈现显著性差异(p值小于0.05),但其他染色体相互之间没有显著性差异。且16号染色体在不同长度区间下的平均dna片段比例为5.627,其他染色体在不同长度区间下的平均dna片段比例在3.7~3.8之间,因此,认为多一条16号染色体,故判断t2样本核型为47,xn,+16(与核型分析结果一致)。
同理,对于t4样本,从图4的方差分析的p值指数的分布图可以看出,9号染色体与其他染色体有明显差异,图5中多重比较的p值也可以看出,9号染色体与其他染色体之间都呈现显著性差异(p值小于0.05),但其他染色体相互之间没有显著性差异。且9号染色体在不同长度区间下的平均dna片段比例为5.915,其他染色体在不同长度区间下的平均dna片段比例都在3.75左右,因此,认为多一条9号染色体,故判断t4样本核型为47,xn,+9(与array-cgh分析结果一致)。
以上是对本发明的较佳实施进行了具体说明,但本发明创造并不限于所述实施例,熟悉本领域的技术人员在不违背本发明精神的前提下还可做作出种种的等同变形或替换,这些等同的变形或替换均包含在本申请权利要求所限定的范围内。
- 还没有人留言评论。精彩留言会获得点赞!