用于基于不一致性度量根据生物数据的亚群检测的方法、系统和装置与流程
本文描述的各种实施例总体上涉及生物医学信息学技术。更具体地但非排他性地,本文公开的各种方法、系统和装置涉及生物信息学和基于生物数据对亚群的检测。
背景技术:
生物信息学技术提供了用于分析生物有机体的有效手段,并且是若干生物领域的重要方面。特别地,生物信息学技术过程已经在基因组学以及对包括癌症的疾病的研究和处置方面取得了重大进展。癌症以及其他基因组疾病的特征在于支持从正常细胞到肿瘤细胞的演变的基因组结构变异和基因表达的异种模式。出于临床研究的目的,并且特别是出于在肿瘤发展和增殖中识别驾驶员和乘客事件的目的,从可用基因组数据解读并表征独特模式的能力具有高度重要性。
技术实现要素:
目前可用的生物医学信息学和生物信息学技术的有效性相对有限,因为这些技术所采用的分析不能提供用于确定生物数据中的亚组或亚群的数量的确定且准确的手段。例如,基因图谱的复杂性和体积使得很难有效且准确地分析它们以便检测各种亚群,包括例如基于对整个肿瘤活组织检查的分析的癌症患者的同种亚组以及反映肿瘤细胞谱系和演变的克隆群体,以及异常、正常和疾病特异性细胞系的群体。
本公开涉及用于检测至少一种生物有机体的成分的亚群的方法、系统和装置。应用机器学习技术来发现这些类型的亚群是有问题的,因为数据内的类的数量通常是未知的。虽然非参数无监督式机器学习方法非常擅长检测个体样本的接近度并确定主要亚组(集群)的结构,但是它们无法提供正确数量的类的明确指示,而参数方法假设类的数量是事先知道的,这种情况很少发生。
为了在保持高准确度的同时提高亚群的检测的效率,能够对生物数据执行聚类过程以获得利用集群内不一致性度量(例如集群内的元素的成对统计方差)评估的集群分区。特别地,不是将单元素集群视为在集群内具有零不一致性,本申请的实施例而是将非零不一致性度量分配给单元素集群。本申请的发明人惊奇地发现,分析集群一致性并将一定程度的集群内不一致性分配给单元素集群使得能够出现具有根据分区水平来评估的不一致性得分的最小值的u形曲线。这里,已经发现对应于最小值的分区水平准确地表示生物数据中存在的集群和亚群的数量。因此,通过将非零不一致性度量分配给单元素集群,能够以高效且准确的方式检测亚群。
通常,在一个方面,示范性系统被配置为检测至少一种生物有机体的成分的亚群。这里,所述系统包括至少一个硬件处理器和非瞬态存储介质。所述处理器被配置为获得所述(一种或多种)生物有机体的所述成分的生物数据样本的多个分区,并且所述存储介质被配置为存储所述多个分区。另外,所述多个分区中的每个分区定义所述成分的所述生物数据样本的各自数量的集群。此外,所述处理器被配置为针对多个分区中的每个分区基于对集群内不一致性进行测量的不一致性度量来计算针对对应分区的不一致性得分,其中,针对所述多个分区中的至少一个,非零值被分配给仅具有一个生物数据样本的至少一个集群的所述不一致性度量。所述处理器还被配置为确定所述多个分区中的哪个分区具有最小不一致性得分,并且通过选择所述多个分区中具有所述最小不一致性得分的分区作为所述亚群来识别所述(一种或多种)生物有机体的所述成分的所述亚群。
类似地,在另一方面,示范性方法涉及检测至少一种生物有机体的成分的亚群。所述方法由至少一个硬件处理器实施。根据所述方法,获得所述(一种或多种)生物有机体的所述成分的生物数据样本的多个分区。另外,所述多个分区中的每个分区定义所述成分的所述生物数据样本的各自数量的集群。针对所述多个分区中的每个分区,基于对集群内不一致性进行测量的不一致性度量来计算针对对应分区的不一致性得分,其中,针对所述多个分区中的至少一个,非零值被分配给仅具有一个生物数据样本的至少一个集群的所述不一致性度量。此外,所述方法包括确定所述多个分区中的哪个分区具有最小不一致性得分,并且通过选择具有所述最小不一致性得分的分区作为所述亚群来识别所述亚群。
根据示范性实施例,所述生物数据包括基因组数据或蛋白质组数据中的至少一种。已经发现系统、方法和装置实施例由于在识别亚群中的显著准确性在被应用于基因组数据或蛋白质组数据时是特别有利的。
在一个示范性实施例中,所述计算还包括根据对应集群中的生物数据样本的总数和所述(一种或多种)生物有机体的所述成分的生物数据样本的总数来对所述对应分区中的集群的至少子集中的每个集群的所述不一致性度量进行加权。所述加权能够对具有低集群内不一致性以及相对较少数量的集群的分区提供有利的偏好。在该实施例的一个版本中,执行所述加权,使得所述对应集群的所述不一致性度量与所述对应集群中的生物数据样本的所述总数直接相关。
根据示范性实施例,所述非零值通过将所述(一种或多种)生物有机体的所述成分的所述生物数据样本的所述不一致性度量作为一个整体进行加权来确定。因此,例如,单样本集群能够被分配整个生物样本的分区不一致性度量的总体方差的一部分,从而使得能够形成根据分区水平来评估的不一致性得分中的u形曲线和最小值。如上所述,该最小值能够表示集群的总数,从而允许对亚群的准确且精确确定。在该实施例的一个版本中,所述成分的所述生物数据样本的所述不一致性度量利用所述成分的生物数据样本的总数进行加权。另外,在该实施例的相同或不同版本中,执行所述加权,使得所述非零值与所述成分的生物数据样本的所述总数反相关。
此外,根据示范性实施例,所述不一致性度量是所述对应分区的给定集群中的生物数据样本之间的成对距离的统计方差。已经发现使用统计方差作为不一致性度量对于基因组数据是显著准确的。
另外,在示范性实施例中,能够显示所选择的分区的至少一个集群的表示。此外,所述表示能够包括对所述(一个或多个)集群的临床注释或表型注释中的至少一种,以帮助临床医生评价数据。在该实施例的一个版本中,所述注释包括药物反应数据、疾病复发风险或疾病分型数据中的至少一种。
示范性实施例还能够包括提供诊断信息。例如,根据一种方法,所选择的分区的集群的至少子集与临床变量、临床结果或临床标签中的至少一个相关联。另外,所述方法包括接收至少一个生物数据样本并且通过将所述样本与所选择的分区的集群的表示进行比较来搜索与所述生物数据样本的至少一个匹配。此外,输出与匹配所述样本的集群中的至少一个集群的表示相关联的临床变量、临床结果或临床标签中的任何一个或多个作为诊断信息。这里,诊断信息能够用作健康护理提供者诊断或开具对患者的特定处置的指导。例如,诊断信息能够指示患者可能遭受的特定癌症分型。另外,诊断信息能够指示一种或多种特定药物在处置与生物数据样本匹配的集群的患者中的疾病或病痛中成功或不成功。由于本文描述的实施例所提供的灵活性和适应性,能够提供各种各样的诊断信息。
此外,在一个方面,一种计算机可读介质包括计算机可读程序,当在计算机上运行时,所述计算机可读程序使得所述计算机能够执行本文描述的方法中的任何一个或多个。例如,所述计算机可读程序能够被配置为检测至少一种生物有机体的成分的亚群,使得当在计算机上运行所述程序时,所述程序使所述计算机执行本文描述的方法实施例中的任何一个或多个的步骤。所述计算机可读介质能够是计算机可读存储介质或计算机可读信号介质。备选地或额外地,所述计算机可读介质能够包括所述计算机可读程序的更新部分或其他部分。
如本文中出于本公开的目的使用的,术语“至少一种生物有机体的成分”应当被理解为包括但不限于细胞、细胞系、细菌培养物、其他微生物或患者。
术语“生物数据”应当被理解为包括但不限于基因组数据,除了其他类型的生物数据之外,还包括例如以下中的一种或多种:突变、全基因组拷贝数改变、基因和/或非编码rna表达数据、dna甲基化数据、组蛋白修饰、dna结合数据(例如,chlpseq)和/或rna结合数据,和/或其他类型的基因组数据、蛋白质组数据(包括例如生物样本的蛋白质表达数据、磷酸化数据、泛素化数据和/或乙酰化数据)、生物医学数据(包括临床数据和个人健康数据,包括葡萄糖水平数据、血压数据、体重数据、体重指数(bmi)数据、饮食数据和/或每日卡路里摄入量)。
另外,“分区”应当被理解为包括一个或多个集群。
此外,在本文描述的实施例中,采用“不一致性度量”,“非零”值被分配给单元素或单样本集群,并且不一致性得分的“最小”值被确定并且用于识别亚群。然而,这些术语应当被理解为包括相反的等同术语。例如,如果采用一致性度量,例如采用统计方差的倒数,而不是采用不一致性度量,那么找出“一致性”得分的“最大”值来识别亚群应当被理解为等同于确定或找到“不一致”得分的“最小”值来识别亚群。类似地,在这些相反的等同情况下,将值(例如,一致性度量的非单位值)分配给单元素或单样本集群应当被理解为等同于将不一致度量的非零值分配给单元素或单样本集群。
本文使用的术语“控制器”通常用于描述与计算设备的操作有关的各种装置。控制器能够以多种方式(例如,使用专用硬件)来实施以执行本文所讨论的各种功能。“处理器”是采用可以使用软件(例如,微代码)编程以执行本文所讨论的各种功能的一个或多个硬件微处理器或者采用专用硬件的控制器的一个示例。控制器可以在采用或不采用处理器的情况下被实施,并且还可以被实施为执行一些功能的专用硬件和执行其他功能的微处理器(例如,一个或多个编程的微处理器和相关联的电路)的组合。可以在本公开的各种实施例中采用的控制器部件的示例包括但不限于传统的微处理器、专用集成电路(asic)和现场可编程门阵列(fpga)。
术语“模块”应当被理解为一个或多个专用硬件处理器和/或执行软件指令的一个或多个硬件处理器。
在各种实施方式中,处理器或控制器可以与一个或多个计算机可读存储介质(在本文中一般地称为“存储器”,例如,诸如ram、prom、eprom和eeprom、软盘、紧凑盘、光盘、磁带等的易失性和非易失性计算机存储器)相关联。如本文所使用的,术语“非瞬态机器可读存储介质”将被理解为包括易失性存储器和非易失性存储器两者,但是不包括瞬态信号。在一些实施方式中,存储介质可以被编码有一个或多个程序,当在一个或多个处理器和/或控制器上执行时,所述一个或多个程序执行本文所讨论的功能中的至少一些。各种存储介质可以固定在处理器或控制器内,或者可以是可传输的,使得存储在其上的一个或多个程序能够被加载到处理器或控制器中,以便实施本文所讨论的各个方面。术语“程序”或“计算机程序”在本文中在一般意义上用于指能够用于对一个或多个处理器或控制器进行编程的任何类型的计算机代码(例如,软件或微代码)。在一些实施方式中,计算机可读信号介质可以被编码有一个或多个程序,当在一个或多个处理器和/或控制器上执行时,所述一个或多个程序执行本文所讨论的功能中的至少一些。例如,信号介质能够是数据信号通过其传播的电磁介质,例如射频介质和/或光学介质。
术语“可寻址”在本文中用于指代被配置为接收意图用于多个设备(包括其自身)的信息(例如,数据)并且选择性地响应意图用于其的特定信息的设备(例如,控制器或处理器)。术语“可寻址”通常与网络环境(或“网络”,下面进一步讨论)结合使用,其中,多个设备经由一些通信介质或媒介耦合在一起。
在一个网络实施方式中,耦合到网络的一个或多个设备可以用作耦合到网络的一个或多个其他设备的控制器(例如,以主/从关系)。在另一实施方式中,联网环境可以包括被配置为控制耦合到网络的设备中的一个或多个的一个或多个专用控制器。通常,耦合到网络的多个设备中的每个都可以具有对存在于通信介质或媒介上的数据的访问;然而,给定设备可以是“可寻址的”,因为其被配置为例如基于分配给它的一个或多个特定标识符(例如,“地址”)来选择性地与网络交换数据(即,从网络接收数据和/或将数据发送到网络)。
如本文使用的术语“网络”是指两个或更多个设备(包括控制器或处理器)的任何互连,其有助于在耦合到网络的任何两个或更多设备之间和/或多个设备之间传输信息(例如,用于设备控制、数据存储、数据交换等)。如应当容易认识到的,适用于互连多个设备的网络的各种实施方式可以包括各种网络拓扑中的任何,并且采用各种通信协议中的任何。额外地,在根据本公开的各种网络中,两个设备之间的任何一个连接可以表示两个系统之间的专用连接,或者备选地非专用连接。除了承载意图用于两个设备的信息之外,这种非专用连接可以承载不一定意图用于两个设备中的任一个的信息(例如,开放网络连接)。此外,应当容易认识到,如本文所讨论的各种设备网络可以采用一个或多个无线、有线/线缆和/或光纤链接来促进整个网络中的信息传输。
如本文使用的术语“用户接口”指的是人类用户或操作者与一个或多个设备之间的接口,该接口使得能够在用户与(一个或多个)设备之间进行通信。可以在本公开的各种实施方式中采用的用户接口的示例包括但不限于开关、电位计、按钮、拨盘、滑块、鼠标、键盘、小键盘、各种类型的游戏控制器(例如,操纵杆)、跟踪球、显示屏、各种类型的图形用户接口(gui)、触摸屏、麦克风、以及可以接收某种形式的人类生成的刺激并响应于其而生成信号的其他类型的传感器。
应当认识到,下面更详细讨论的前述构思和额外构思的所有组合(假设这些构思不相互矛盾)被认为是本文公开的主题的一部分。特别地,要求保护的主题的所有组合都被预见为是本文公开的主题的一部分。还应当认识到,也可以出现在通过引用并入的任何公开内容中的本文明确采用的术语应当被赋予与本文公开的特定构思最一致的含义。
附图说明
在附图中,类似的附图标记在不同视图中通常指代相同的部分。而且,附图不一定按比例绘制,而是通常将重点放在说明各种原理上。
图1是根据示范性实施例的用于检测至少一种生物有机体的成分的亚群的系统的高级框图/流程图。
图2是根据示范性实施例的用于检测至少一种生物有机体的成分的亚群的方法的高级框图/流程图。
图3是说明根据示范性实施例的能够用于识别至少一种生物有机体的成分的亚群的不一致性得分的绘图的示图。
图4是根据示范性实施例的用于提供诊断信息的方法的高级框图/流程图。
图5是能够实施一个或多个示范性实施例的示范性计算机系统的高级框图/流程图。
具体实施方式
在生物医学信息学内,由于数据的复杂性和大小,基因组数据的生物信息学分析通常非常困难。特别地,目前可用的技术不能提供用于确定生物数据中的亚组或亚群的数量的适当手段。当将其应用于来自很大队列的患者、细胞系和/或细胞组群(其能够包括例如在群体研究水平的患者亚组以及疾病细胞的克隆群体或与疾病相关的不同细胞系)的患者临床数据、个人健康数据和基因组数据以用于检测亚群时,该分析尤其困难。为了提高检测亚群的准确性和效率,申请人已经意识到并且认识到,从单元素集群开始分配非零集群内不一致性度量将是有益的。以这种方式将非零集群内不一致性度量分配给单元素集群是违反直觉的,但是使得能够出现具有根据分区水平来评估的不一致性得分中的最小值的u形曲线。已经发现该最小值准确地表示生物数据中的亚群的正确数量。因此,通过将非零集群内不一致性度量分配给单元素集群,示范性实施例提供了用于识别亚群的有效且优雅的手段。
如本文所述的对亚群的识别能够被用作诊断工具。例如,对亚组/亚群的识别能够用于区分具有相似患者特性和相似结果的患者的亚群。此外,对亚群的识别能够在临床应用中用于在评价肿瘤样本的侵袭性中辨别克隆演变模式和肿瘤异质性。这种洞察为癌症以及其他疾病的处置提供了显著的优势。因此,实施例能够用于辅助患者行程的处置规划阶段。例如,基于在细胞群体水平的诊断,实施例能够用于治疗设计中。这里,对亚群的识别是特别有利的,因为医生能够为每个亚群定制药物和抑制剂,而不是在平均目标上使用一种抑制剂。因此,以这种方式,由实施例显示为特别具有侵袭性的某些亚群能够被特异性地靶向以处置患者。本文描述的实施例还能够用于发现细菌感染中的新群体长出,并且能够用于区分医院获得性(医院内)感染和社区获得性感染。
鉴于前述内容,本文描述的各种实施例和实施方式涉及用于检测至少一种生物有机体的成分的亚群的方法、系统和装置。这些实施例能够用于例如对基因组和/或转录组事件进行分类,表征克隆细胞群,并提取有价值的临床信息,例如肿瘤进展模式、处置计划功效的预后、以及患者风险。此外,实施例能够包括模式识别工具,其能够基于基因组数据来检测克隆群体,除了其他类型的基因组和蛋白质组和翻译后修饰数据之外,基因组数据还包括例如突变、全基因组拷贝数改变、基因和/或非编码rna表达数据、dna甲基化数据、组蛋白修饰、dna结合数据(例如chlpseq)和/或rna结合数据。根据示范性方面,能够从蛋白质组数据中检测克隆群体,所述蛋白质组数据是根据质谱法提取的并且能够并入整合分析中。mertins等人的“integratedproteomicanalysisofpost-translationalmodificationsbyserialenrichment”(naturemethods10,634-637(2013))描述了质谱法的示例,通过引用将其并入本文。蛋白质组数据能够包括生物样本的蛋白质表达数据、磷酸化数据、泛素化数据和乙酰化数据。此外,根据示范性实施例,为了基因组疾病研究和患者临床评价的目的,能够以自动方式表征细胞内和细胞间异质性。此外,实施例还能够检测细菌演变中的亚群,用于传染病管理以及抗生素抗性检测和预测。
示范性方法和系统实施例能够以组合或分开的方式识别各种类型的基因组/蛋白质组数据中的模式以表征患者数据用于临床结果预测和分型。如上所述,优选的实施例能够整合并提取来自基因组信息和/或蛋白质组信息的可用模态的有用信息。此外,示范性系统和方法实施例能够被实施为用于使用多级聚类架构的基因组模式识别以及用于临床环境中的数据解读的有效计算工具。
此外,示范性实施例能够用于确定一大群有机体(个体)内的具有从医学临床角度(包括来自电子病历的数据、生理学信号和/或健康数据)通过不同技术度量的总体特性的特定水平的异质性的亚群。例如,实施例能够用于基于疾病信息(例如肿瘤分级、结节损害、分期、转移状态、免疫组织化学状态、年龄、药物反应数据、总体存活和无进展存活数据等)、连续的健康数据(例如心率、每天的步数、深度和浅度睡眠模式、皮肤电反应测量结果)等对患者进行分类。
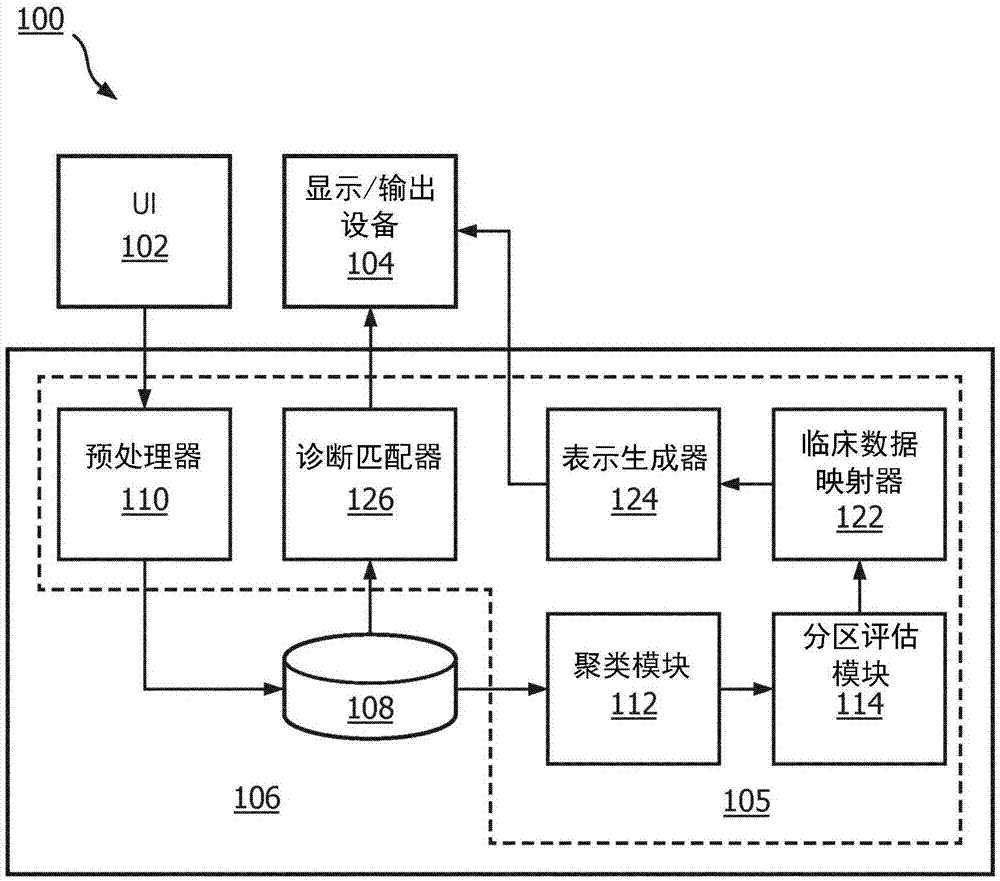
参考图1,说明性地描绘了根据示范性实施例的用于检测至少一种生物有机体的成分的模式和/或亚群的示范性系统100。系统100能够包括预处理器(pre-prcssr)110、聚类模块(clstr.mod.)112、分区评估模块(eval.mod.)114、临床数据映射器(clin.d.map.)122、表示生成器(rep.gen.)124和诊断匹配器(diag.mtchr)126。系统部件110、112、114、122、124和126中的每个能够由控制器(cntrlr)105实施,该控制器能够是作为硬件计算系统106的一部分的一个或多个硬件处理器。计算系统106还能够包括存储介质108,并且系统100能够包括用户接口(ui)102和显示/输出设备(dsply/out.dev.)104。在一些实施例中,用户接口102和显示/输出设备104能够被合并到单个设备(例如触摸屏设备)中。本文下面关于图2的方法200和图4的方法400描述了根据示范性实施例的各种系统部件的示范性功能。
参考图2,同时继续参考图1,说明性地描绘了用于检测至少一种生物有机体的成分的亚群的示范性方法200。这里,成分能够是细胞,例如克隆细胞,或一种或多种有机体的细胞系。备选地或额外地,成分能够是生物有机体本身,包括例如患者或甚至细菌培养物。方法200能够被应用于基于生物数据来检测这些成分中的任何一个或多个成分的亚群,所述生物数据包括例如根据这些成分汇编的基因组数据和/或蛋白质组数据。应当注意,方法200和400能够由系统100或106执行。例如,方法200和方法400的步骤能够是能够存储在存储介质108上并且由控制器实施的元件110、112、114、122、124和/或126执行的程序的指令,例如,如下文所讨论的。
方法200能够在步骤202处开始,在该步骤处,预处理器110能够根据一种或多种生物有机体的成分的生物数据样本来汇编特征数据集。例如,预处理器110能够在步骤204处接收生物数据样本,并且在一个实施例中,能够将数据直接汇编在一个或多个矩阵中。输入数据也能够以数据矩阵或数据矩阵的集合的形式来接收,数据矩阵的集合能够被合并或分开分析。例如,能够对集合中的数据矩阵中的每个执行方法200。
在步骤204处接收并汇编的生物数据能够包括基因组数据、蛋白质组数据或临床数据中的至少一种。对于该队列的每个成员,如上所述,基因组数据能够包括突变、小插入和缺失(indels)、重组、全基因组拷贝数改变、基因表达数据、甲基化数据和/或其他类型的基因组数据中的一种或多种。备选地或额外地,如上所述,蛋白质组数据能够包括生物样本的蛋白质表达数据、磷酸化数据、泛素化数据和/或乙酰化数据。蛋白质组数据是基因组结构和许多下游生物过程的功能读出。基因组数据和/或蛋白质组数据可以由上述类型之一的数据或不同类型的数据的任何组合组成。拷贝数改变能够表示针对该队列的每个成员的基因组的各个区域的缺失和扩增。基因表达数据和甲基化数据分别表示根据给定生物有机体中的基因的过表达/欠表达和基因沉默或活化程度的额外类型的基因组表征。这些数据被提供作为从测量过程导出的定量变量,并且能够为在步骤202处接收的输入的一部分。还应当注意,尽管这里将基因组数据和蛋白质组数据描述为示例,但是生物数据能够额外地或备选地包括其他类型的数据,如上所述。如本领域技术人员基于本说明书所理解的,数据能够以类似于下文关于基因组数据描述的示例的方式来形成并分析。
应当注意,除了生物数据之外,在步骤202处,用户可以任选地将注释/标签(其能够包括临床变量、临床结果和/或其他临床标签)输入到系统106。在下文中关于步骤222和224以及图4中描绘的方法400详细讨论注释/标签。
在如上所述的步骤204处,预处理器110能够将根据(一种或多种)生物有机体的成分的队列汇编的生物数据样本形成为存储介质108的至少一个数据结构内的矩阵。这里,矩阵的每列能够是(一种或多种)生物有机体的成分的生物数据样本。例如,根据该队列汇编的基因组数据能够被形成如下:
在该具体示例中,基因组数据由拷贝数改变(cna)、基因表达数据(ge)、甲基化数据(m)组成。然而,应当理解,矩阵能够由这些类型之一的数据或这些类型的数据或上面讨论的其他类型的数据的任何子组合组成。此外,如果矩阵表示有机体的整个群体,则矩阵能够包括表型表达的测量结果,例如肿瘤体积、分级、分期、年龄、对药物的反应、进展时间和/或死亡时间。备选地或额外地,矩阵能够包括在基因组和表观基因组以及蛋白质水平两者处对生物有机体的个体细胞的测量结果。此外,列的每个集合表示队列的特定成员,其能够是例如给定患者的特定细胞或特定患者。另外,如上所述,每列能够是生物数据样本。例如,如果队列成员是患者,则患者由矩阵(1)的元素中的第一个下标表示,其中,cna1,n、ge1,n和m1,n分别表示患者1的拷贝数改变数据、基因组表达数据和甲基化数据,cna2,n、ge2,n和m2,n分别表示患者2的拷贝数改变数据、基因组表达数据和甲基化数据,等等。这里,n表示基因组的任意染色体区域,其中,队列中的每个患者的基因组通过沿着基因组长度的1、2、3…m个区域划定。划定的区域由矩阵(1)中的行表示。例如,cna1,1、ge1,1和m1,1分别表示患者1的区域1的拷贝数改变数据、基因组表达数据和甲基化数据,cna1,2、ge1,2和m1,2分别表示患者1的区域2的拷贝数改变数据、基因组表达数据和甲基化数据,cna2,2、ge2,2和m2,2的分别表示患者2的区域2的拷贝数改变数据、基因组表达数据和甲基化数据,等等。因此,cnam,n能够表示患者n的基因组的区域m中的正常改变、缺失或扩增,而gem,n可以表示在患者n的基因组的区域m处表达的基因的值。也能够在步骤202处接收基因组的划定的区域,随后将其排列为列向量,该列向量能够被存储在存储介质108内的存储结构中,并且能够是由系统106的元件中的任何一个或多个用来将矩阵(1)的元素映射到特定基因组区域的参考。因此,在矩阵(1)中,每列能够表示队列中的不同患者,其中,任何矩阵元素中的第一个下标表示队列中的特定患者,并且每个区域1、2、3…m对应于针对该患者的基因组数据。
应当注意,优选地,方法200针对一种数据类型被实施。例如,能够针对由列cnan,1…cnan,m表示的拷贝数数据执行方法200。另外,方法200能够分别针对基因表达数据和甲基化数据并行执行。然而,应当理解,在该方法的一个实施方式中,能够对矩阵(1)的整个数据执行方法200。方法200还能够应用于有机体或有机体内细胞的生物活性的其他类型的数据测量。这里,基因组水平数据被分析,但是,应当理解该方法同样适用于疾病信息(例如肿瘤分级、结节损害、分期、转移状态、免疫组织化学状态、年龄等)、连续的健康数据(例如心率、每天的步数、深度和浅度睡眠模式、皮肤电反应测量结果)和治疗反应信息,包括药物反应/抗性数据、总体存活和无进展存活。
根据一个实施例,矩阵(1)能够是在步骤204中汇编的特征数据集。或者,能够进一步对矩阵(1)进行预处理以获得在步骤208/210及后续步骤中分析的特征数据集。例如,任选地,在步骤206处,预处理器110能够对在步骤204处接收并汇编的数据执行数据中心化、标准化和/或异常检测。这里,为了执行数据中心化,预处理器110能够计算并减去特征向量中的均值,如下所示:
x:=x-m(x)(2)
其中,x是特征向量,其能够是矩阵(1)、矩阵(1)的列或矩阵(1)中的列的集合,并且m(x)=求平均(x)。
此外,为了执行数据标准化,预处理器110能够采用最适合于特定数据类型的变换。例如,预处理器110能够通过执行以下过程之一来实施标准化。根据第一过程,预处理器110能够将每个特征向量除以最大元素,如下所示:
x:=x/max(x)(3)
其中,x是特征向量,其能够是矩阵(1)、矩阵(1)的列或矩阵(1)中的列的集合,并且max(x)是特征向量x中的最大元素。在第二过程中,预处理器110能够计算标准偏差并将每个特征向量除以各自的标准偏差,如下所示:
x:=x/std(x)(4)
其中,x是特征向量,其能够是矩阵(1)、矩阵(1)的列或矩阵(1)中的列的集合,并且是std(x)是特征向量x的标准偏差。根据第三过程,预处理器110能够计算每个特征范围并且能够将特征向量除以范围长度,如下所示:
x:=x/长度(范围(x))(5)
其中,x是特征向量,其能够是矩阵(1)、矩阵(1)的列或矩阵(1)中的列的集合,并且范围(x)是在特定样本队列中看到的特征向量中的值的范围,并且长度(范围(x))是范围的长度。
此外,为了执行异常值检测,在任选步骤206处,预处理器110能够识别在步骤204处接收的生物数据中的异常值,并且能够将异常值与生物数据分离。因此,在步骤202处由预处理器110汇编的特征数据集能够是没有任何识别到的异常值的中心化且标准化的数据集。例如,为了确定并分离异常值,预处理器110能够应用各种方法中的一种或多种,包括马哈拉诺比斯距离方法或主成分分析(pca)方法中的至少一种。这里,预处理器110能够应用这些方法之一、这两种方法或这些方法与识别并分离异常值的任何适当方法的任何子组合。对于这些方法中的每种方法,在步骤204处接收的生物数据能够被组成在数据矩阵中。
在马哈拉诺比斯距离方法中,预处理器110能够将通常具有高维度的数据矩阵拆分成区域。这里,每个数据类别能够在矩阵中被分组为例如相邻列。例如,全基因组拷贝数改变数据能够被分组在相邻列的集合中,基因表达数据能够被分组在相邻列的集合中,甲基化数据能够被分组在相邻列的集合中,等等。预处理器110拆分矩阵使得每个类别集合被拆分成多个区域,使得任何给定区域仅由来自一个类别的数据组成。对于每个区域和数据类别,预处理器110能够计算均值估计m(x)和协方差估计c(x),如下所示:
m(x)=求平均(x)(6)
其中,x表示数据类别,其能够是例如拷贝数改变类别、基因表达数据类别或甲基化数据类别,x表示区域中的值或元素,并且n在这里表示区域中的元素的数量(n≥2)。预处理器110能够以二次形式计算针对每个元素x的马哈拉诺比斯距离md(x,x),如下所示:
md(x,x)=(x-m(x))c-1(x)(x-m(x))(8)
此外,预处理器110能够将异常值检测为具有被确定为高于阈值的马哈拉诺比斯距离的大马哈拉诺比斯的点。预处理器110还能够使用从区域维度(n-1)识别的卡方(χ2)分布自由度来评估马哈拉诺比斯距离。
在pca分析方法中,预处理器110能够线性地变换(旋转)原始数据矩阵,使得相关矩阵在变换空间中被对角化。这里,预处理器110能够将相关矩阵拆分成区域,例如以上关于马哈拉诺比斯距离方法所讨论的,并且能够基于由这些分量捕获的方差的阈值来选择主分量的数量。例如,阈值能够被选择为90%。预处理器110能够如上面关于方程6-8所讨论的那样计算所获得的主分量上的马哈拉诺比斯距离,并且能够应用卡方检验来将异常高的值识别为异常值,如上所述。
根据示范性实施例,初步特征数据集能够由从步骤206或步骤204得到的数据组成。在步骤204和/或206处汇编特征数据集之后,预处理器110能够将特征数据集存储在存储介质108中的数据结构内用于随后由聚类模块112检索,或者能够将特征数据集直接提供给聚类模块112。
任选地,在步骤208处,聚类模块112能够选择集群完整性度量。例如,集群完整性度量能够是不一致性度量,例如对集群内不一致性进行测量的方差。集群完整性度量能够是由聚类过程确定的给定分区的每个集群/亚群中的样本之间的成对距离的方差,聚类过程可以在步骤210处执行。这里,方差应当被理解为统计方差度量。例如,方差可由下式表示
其中,var(cr)是集群cr的方差,di,i'是集群cr中的给定对的样本/成分i和i'之间的距离,dμ是在集群cr中的所有可能的样本对中取得的平均距离,k是集群cr中的样本/成分的总数。此外,距离度量di,i'、dμ能够是欧几里德距离度量、曼哈顿距离度量或其他适当的距离度量。或者,集群完整性度量能够是集群cr中的样本/成分的熵。出于说明目的,在下文中使用方差。然而,方法200能够采用其他类型的集群完整性度量,其中,var(x)可以总体上表示集群完整性度量或不一致性度量,并且可以取代下文的一个或多个其他完整性度量。例如,用户可以输入并定义将在下文描述的评估步骤212处采用的集群完整性度量。备选地或额外地,聚类模块112可以通过用户接口102向用户提供集群完整性度量的若干选项,并且聚类模块112可以选择由用户选择以用于在步骤212处使用的集群完整性度量。或者,集群完整性度量可以在所有情况下由系统106预先确定并应用。
任选地,在步骤209处,聚类模块112可以选择和/或增加特征集用于评估。例如,方法200可以迭代地评价来自例如矩阵(1)的行的不同基因集,以确定哪个基因集或一般地哪个特征集最佳地识别最佳数量的集群。根据一个示范性实施例,在步骤209处,聚类模块112可以确定特征的子集,其可以是例如具有最高方差的矩阵(1)的行的子集。例如,聚类模块112可以计算不同特征集的方差,并确定具有最高方差的特征或该示例中的基因的前1%。类似地,聚类模块112还可以确定具有最高方差的特征的前5%,10%,15%等。这里,结合任选步骤220,可以通过步骤210-219迭代地评估这些特征集中的每个,如下文所讨论的。因此,在由步骤209和220定义的循环的第一次迭代中,步骤210-219可以应用于对应于具有最高方差的前1%特征的特征集。然而,应当理解,步骤209和220是任选的,并且步骤210-219可以应用于由步骤202提供的特征数据集。
在步骤210处,聚类模块112可以获得集群分区的集合,其中,该集合或多个分区中的每个分区定义成分的生物数据样本的各自数量的集群。例如,聚类模块112可以执行聚类过程以生成分区的集合。备选地或额外地,在步骤202处,聚类模块112可以接收生物数据样本的集群分区的集合作为来自用户的输入。例如,对于样本的给定集合,例如矩阵(1)或者由给定的数据分型组成的矩阵,例如拷贝数变更(cna)、基因表达数据(ge)、甲基化数据(m),聚类模块112可以生成或接受输入样本的独特集群分区的集合作为给定输入。这里,聚类模块112可以执行无监督式聚类过程,例如,分层聚类、模糊聚类、k均值聚类或任何其他类型的聚类方案。另外,每个分区可以定义生物数据样本的不同数量的集群。例如,一个分区可以定义一个集群,第二分区可以定义两个集群等。
在步骤212处,分区评估模块114可以评估在步骤210获得的集群的分区完整性。例如,对于在步骤210处获得的分区的至少子集中的每个分区,分区评估模块114可以基于对集群内不一致性进行测量的不一致性度量来计算针对对应分区的不一致性得分。如上所述,不一致性度量可以是集群内的统计成对方差,或者可以是例如集群的熵度量。根据一个示范性实施方式,可以迭代地执行分区评估,其中,分区评估模块114在步骤214处评价下一个分区。例如,过程可以以设置为零的分区号开始并且在步骤214处这里增加到1。分区号可以用于识别分区,并且可以对应于由分区定义的集群的数量。备选地,评估模块114可以最初和/或随后将分区号增加大于1的值。例如,在整个迭代过程中可以增加和/或减少在步骤214处增加的分区号。然而,在下文描述的特定实施方式中,在步骤214的每次迭代中,分区号可以增加1。或者,与如上所述的增加分区号相反,评估模块114可以以与上述方式相同的方式减少分区号。实际上,步骤214的迭代可以以各种不同的方式来实施,只要足够数量的分区被评估以解密完整性/不一致性得分的最小值,如下文所讨论的。
在步骤216处,分区评估模块114可以将非零不一致性度量分配给仅具有一个生物数据样本的任何集群。例如,当采用统计方差作为不一致性度量时,可以通过将在步骤212的迭代中评价的分区的总体方差的一部分分配给单样本集群来确定单样本集群的方差(s_i)。样本集群。例如,当采用方程(9)的统计方差作为不一致性度量时,单样本集群var(s_i)的方差可以被确定如下:
其中,n是成分的生物数据样本的总数并且var(总分区)是总分区的方差。换句话说,var(总分区)是作为一个整体的(一种或多种)生物有机体的成分的所有生物数据样本的成对方差。另外,n在这里可以是例如针对(一种或多种)生物有机体的成分的拷贝数改变数据、基因表达数据或甲基化数据的矩阵(1)中的n。因此,分区评估模块114可以通过利用至少一种生物有机体的成分的生物数据样本n的总数对作为一个整体的(一种或多种)生物有机体的成分的生物数据样本的不一致性度量(例如var(总分区))进行加权来确定非零值var(s_i)。因此,根据示范性方面,可以执行加权,使得非零值var(s_i)与(一种或多种)生物有机体的成分的生物数据样本的总数(n)反相关。如上所述,针对单样本集群的非零不一致性度量的分配是违反直觉的,但是提供了实质性的优势,因为它使得能够形成不一致性得分的u形绘图,从而允许识别生物数据样本的集群的最佳分区。
在步骤218处,分区评估模块114可以基于集群完整性度量/不一致性度量来计算针对评估中的对应分区的不一致性得分。例如,如果不一致性度量是如上所述的成对统计方差,则在步骤218的当前迭代中针对评估中的分区的不一致性得分score_var(分区)可以被计算如下:
score_var(分区)=d1var(c1)+d2var(c2)+...+drvar(cr)(11)
其中,c1,c2,...cr分别表示分区中的集群1,2...r,r表示分区中的集群的总数。但是,应当理解,如果任何集群cr是单样本集群,则针对cr的“var(s_i)”应当取代方程(11)中的“drvar(cr)”。另外,根据优选实施例,系数dr可以被选择为集群cr中的元素的数量和生物样本的总数的函数,其中,r=1,...,r。换句话说,系数dr,r=1,...,r,可以是对应集群cr中的生物数据样本的总数和生物数据样本的总数(例如,针对(一种或多种)生物有机体的成分的拷贝数改变数据、基因表达数据或甲基化数据的矩阵(1)中的n)的函数。因此,通过例如根据方程(11)将系数dr应用于不一致性度量var(cr),分区评估模块114可以根据对应集群中的生物数据样本的总数和至少一种生物有机体的成分的生物数据样本的总数来对对应分区中的集群中的每个集群cr的不一致性度量进行加权。将系数配置为集群中的元素的总数和样本的总数的函数可以改善并更好地定义不一致性得分的绘图的u形,从而使得能够更好地确定最佳分区。根据一个示范性实施方式,系数dr可以被计算为
在步骤219处,分区评估模块114可以确定是否已找到不一致性得分的最小值。例如,分区评估模块114可以汇编通过步骤212的迭代评估的分区的所有不一致性得分,并且评价关于评估的分区的集群的总数的不一致性得分。例如,根据一个实施方式,分区评估模块114可以形成绘图,例如图3的绘图302,其中,垂直轴表示不一致性得分,并且水平轴表示给定分区中的集群的总数,在图3中称为分区水平。这里,绘图302上的每个点表示不同分区中的集群的总数,并且绘图可以被构建,因为针对分区的不一致性得分被确定。根据图3,每个分区具有从一个集群到约50个集群的范围中选择的独特数量的集群。例如,如果在步骤210处执行或单独执行,则集群过程可以确定大量分区(在该示例中,50个或更多个分区)的分区。另外,分区评估模块114可以迭代地确定不一致性得分并迭代地构建绘图,直到已经找到确定的最小值。例如,在步骤219处,分区评估模块114可以将最近确定的不一致性得分添加到绘图中并确定是否已经找到最小值。最小值可以通过评价绘图中的曲线以确定曲线中的一阶导数为零的点来找到。这里,曲线中的一阶导数为零的点对应于最小值。如果尚未找到最小值,则该方法可以前进到步骤212,在该步骤处可以确定针对下一个分区水平或数量的另一不一致性得分。绘图可以从分区水平一(一个总集群)到分区水平2(两个总集群)等连续地被构建,直到已经找到最小值。或者,如果尚未找到最小值,则该方法可以前进到步骤210。例如,分区评估模块114可以被配置为确定在最小值已经被添加到绘图之后的10或20个额外不一致性得分之后已经找到最小值。例如,该特征将阻止分区评估模块114检测到假阳性,如在点304处,其中,已经找到局部最小异常值。这里,分区评估模块114可以获得额外不一致性得分以找到真正的最小值,在这种情况下其在点306处。额外不一致性得分的阈值可以根据基于检查的特定数据的特征并且平衡处理效率和准确性的设计选择被设置。因此,如果在步骤210处尚未获得足够数量的分区,或者等价地,如果已经评价了所有可用分区并且尚未达到阈值,则该方法可以前进到步骤210以通过执行聚类过程来获得额外分区或者从外部源(例如外部或远程数据库)获得额外分区。
如果在步骤219处,分区评估模块114确定已经找到最小不一致性得分,则该方法可以前进到步骤221,或者可以前进到任选步骤220。例如,如以上所指示的,当在该最低值之后已经添加了阈值数量的不一致性得分并且未找到不一致性得分的更低值时,分区评估模块114可以确定最小不一致性得分是绘图上的最低值。在图3中的示例中,不一致性得分的最小值对应于点306,其表示定义总共六个集群的分区。
在任选步骤220处,聚类模块112可以确定是否已经找到最佳特征集。例如,在由步骤209和220定义的循环的第一次迭代中,聚类模块112可以评价与具有最高方差的特征的前1%相对应的特征集。这里,聚类模块112可以评价上面关于最小值讨论的绘图中的曲线的锐度或陡度。例如,聚类模块112可以确定方差得分曲线上的相邻点之间的标准化绝对差的序列:sn={100*|vs(n)-vs(n-1)|/(vsmax-vsmin)},n=2,3,…,nmin,其中,nmin是具有最小不一致性得分的分区,vs(n)是在分区水平n的不一致性得分,vsmax是分区n=2,3,…,nmin的最大不一致性得分,vsmin是分区nmin的最小不一致性得分,并且计算sn的p=第75个百分位数。例如,在图3的绘图300中,聚类模块112可以确定分区水平1与分区水平6之间的绘图的sn的第75个百分位数,其对应于点306。这里sn的第75个百分位数可以是关于最小值的曲线的锐度或陡度的度量。然而,应当理解,可以采用其他陡度或锐度度量。在确定对应于具有最高方差的特征的前1%的特征集的陡度或锐度度量之后,该方法可以前进到步骤209以选择另一特征集。例如,在步骤209处,聚类模块112可以选择与具有最高方差的特征的前5%相对应的特征集,并且步骤210-219可以应用于该特征集,如上所述。由步骤209-220定义的循环可以继续评估与具有最高方差的特征的前10%、前15%等相对应的特征集。根据一个示范性实施例,阈值可以被设置为15%,其中,在步骤220处,聚类模块112可以通过确定已经评估了与具有最高方差的特征的前1%、5%、10%和15%相对应的所有特征集来确定已经找到最佳特征集。这里,在步骤220处,聚类模块112可以确定特征集中的哪些具有最高大小的陡度或锐度度量。另外,聚类模块112可以选择具有最高大小陡度或锐度度量的特征集作为最佳特征集。应当注意的是,对特征集的最佳性的评估不需要通过4%或5%的增量来执行,而是可以根据取决于评价的生物数据的类型的设计选择使用其他百分比或区分参数来执行。另外,阈值不需要被设置为15%,而是也可以根据设计选择来选择。此外,根据一个示范性实施例,在步骤209处,在选择用于评估的任何特征集之前,聚类模块112可以从考虑中移除与具有最高方差的特征的前0.01%相对应的特征作为离群值。根据评价的生物数据的类型,也可以根据设计选择来选择离群值阈值(在该示例中为前.01%)。响应于聚类模块在步骤220处确定已经找到最佳特征集,该方法可以前进到步骤221。
在步骤221处,分区评估模块114可以通过选择具有最小不一致性得分的分区作为亚群来识别(一种或多种)生物有机体的成分的亚群。这里,最小不一致性得分可以是在步骤219处确定的最小值。如果执行任选步骤209和220,则用于识别亚群的最小不一致性得分是针对在步骤220选择的最佳特征集获得的最小不一致性得分。
任选地,在步骤222处,临床数据映射器122可以将临床数据与所选择的分区的集群的至少子集或全部进行映射和关联,并且/或者将标签和/或注释分配给所选择的分区的集群的子集或全部。例如,注释可以包括药物反应数据、疾病复发风险(例如,低风险、中等风险、高风险等)或疾病分型数据中的至少一种。注释/标签可以通过用户接口102从用户接收,存储在存储介质108中,并且与在以上讨论的步骤202处的队列的成分相关。基于该相关,临床数据映射器122可以将注释/标签映射到各自的集群。如果注释/标签不可用,则临床数据映射器可以生成注释/标签并将注释/标签分配给每个集群或子集中的每个集群。例如,可以从存储关于根据方法200聚类的生物数据的信息的外部数据库访问注释/标签。注释/标签可以指示哪些患者接受某种药物,哪些患者对药物反应良好以及哪些患者对药物反应不佳,以使得健康护理从业者确定药物是否有效。因此,如果集群表示表明患者对药物有反应,那么他们可以向健康护理从业者指示处置应当继续。注释/标签还可以包括临床数据或表型数据,包括分型数据,其又可以包括临床上相关的具体类型的癌症。
应当理解,注释可以包括临床变量、临床结果和/或其他临床标签。例如,根据示范性方面,在步骤222处,每个集群或子集的每个集群可以被分配有一个或多个临床变量、临床结果和/或其他临床标签或者与一个或多个临床变量、临床结果和/或其他临床标签相关联。例如,临床变量可以是施予给在步骤202处输入其生物数据的患者的一种或多种药物,患者遵循的规定饮食,和/或患者经历的物理治疗方案,以及其他变量。临床变量还可以包括药物、饮食和/或物理治疗旨在治愈的疾病或病痛。反过来,对应临床结果可以是药物、饮食或物理治疗是否导致治愈或改善患者所患的疾病或病痛的指示。在执行分区获得步骤210之前,可以先验地知道临床变量和临床结果。这里,在步骤222处,通过参考患者/生物数据与临床变量和临床结果之间的相关性,临床数据映射器122可以将对应临床变量/临床结果映射到在步骤221处识别的集群/亚群。例如,针对在步骤221处确定的集群/亚群的质心或其他数学表示的生物数据样本和值可以被映射到属于相应集群/亚群的患者的对应临床变量和临床结果。例如,表示生成器124可以将表示形成为蛋白质组和/或基因组的矩阵,并且对应的值表示例如针对形成在步骤221处确定的对应集群的成分成员的生物数据的质心的集合的拷贝数改变数据、基因表达数据和/或甲基化数据。这里,质心表示或其他表示以及临床变量/结果注释可以用作模型,该模型可以用作针对新患者的临床管理的指导,如下文所述。应当注意,任何类型的注释/标签都可以被映射到相应集群。例如,除了临床变量和结果之外,注释/标签可以是例如癌症分型数据。例如,与临床变量和结果一样,临床标签可以在执行分区获得步骤210之前先验地获知。在步骤222处,通过参考患者/生物数据和临床标签之间的相关性,临床数据映射器122可以将对应临床标签映射到在步骤221处被识别为所选择的分区的部分的集群。例如,类似于临床变量和结果,针对在步骤221处确定的集群/亚群的质心或其他数学表示的生物数据样本和值可以被映射到属于相应集群的患者的对应标签/分型。质心/数学表示与临床标签一起可以用作用于比较目的的模型,其可以帮助诊断患者。标记可以是任何临床上相关的数据,包括例如复发信息、存活率、针对特定基因或基因组的突变数据,和/或基因的表达水平或特定途径的基因的表达水平等。
在步骤224处,表示生成器124可以生成在步骤221处选择的分区中具有最小不一致性得分的集群的表示,和/或对应生物数据的表示,包括在步骤222处映射或分配的任何数据标签或注释,并且将所生成的表示存储在存储介质108内。例如,每个集群表示可以是针对所选择的分区的集群的质心或者另一适当表示,如上面关于步骤222所讨论的。或者,集群的表示可以是质心或其他适当的数学表示与在步骤222处映射到集群的临床变量/结果数据、临床标签和/或其他注释的组合。例如,一个集群表示可以包括集群的质心、施予给属于该集群的患者的药物的指示、药物处置旨在治愈的疾病或病痛以及药物成功的指示。类似地,另一集群表示可以包括不同集群的质心、施予给属于该集群的患者的相同药物的指示、对应的疾病或疾病以及药物不成功的指示。如下文所讨论的,集群表示可以用作可以帮助健康护理提供者评价新患者是否对药物反应良好的模型。或者,集群表示可以包括集群的质心和癌症分型标签。这里,集群表示可以用于比较目的,以帮助健康护理提供者诊断患者所患的疾病。计算出的模式/表示被可视化并提供有用于解读的临床注释。例如,表示可以是图形、热图或2d绘图,其中,点表示患者或其他类型的成分。此外,该表示可以包括表示在步骤221处选择的分区的集群的基因组或蛋白质组的表示。
在步骤226处,表示生成器124可以指引显示/输出设备104显示或输出所生成的表示。如上所述,该表示可以是在步骤221处选择的分区的集群中的至少一个或者表示这些集群的基因组或蛋白质组的表示。此外,还可以显示对集群的临床注释或表型注释中的至少一种。另外,所识别的亚群,或等价地,在步骤221处选择的分区的集群,可以是成分的所识别的亚群的简单列表。或者,所识别的亚群的输出可以进一步包括统计特性,例如,亚群间相似性和/或群体间差异性的描述性特性。
参考图4,同时继续参考图1和图2,说明性地描绘了根据示范性实施例的用于提供诊断信息的方法400。应当注意,方法400可以与方法200组合。此外,可以执行方法400以通知健康护理提供者应当被汇编以获得诊断信息的特定特征数据,并且额外地或备选地,可以执行该方法以向健康护理提供者提供诊断信息。例如,方法400可以在任选步骤402处开始,在该任选步骤处,系统预处理器110可以任选地通过用户接口102从用户接收搜索准则,并且可以将该准则存储在存储介质108中。例如,搜索准则可以表示特定疾病或分型和/或健康护理提供者正在考虑给患者开出的特定药物或其他处置。可以单独输入在步骤402处接收的搜索准则,或者,可以利用生物数据输入搜索准则。例如,在任选步骤404处,预处理器110可以通过用户接口102从用户接收至少一个其他生物数据样本,并将该样本存储在存储介质108中。生物数据样本可以是被形成为矩阵的生物数据,如上文关于步骤202所讨论的,并且可以由患者或成分的整个基因组组成,或者可以由基因的子集和/或上文讨论的蛋白质组数据的任何集或子集组成。步骤404也可以在具有或没有步骤402的情况下执行。例如,如果执行步骤402和步骤404两者,使得预处理器110接收搜索准则和(一个或多个)生物数据样本两者,则预处理器110可以将该准则与生物数据关联并且可以将该准则和生物数据存储在存储介质108中。提供具有数据样本的搜索准则可以将对诊断信息的搜索限制到特定类型的临床变量、结果或标签。例如,搜索可以限制于与特定分型和/或药物相关的诊断信息。或者,可以在没有步骤402的情况下执行步骤404,以使得健康护理提供者能够获得与提交给系统106的生物数据样本相关的所有信息。
在步骤406处,诊断匹配器126可以从存储介质108检索搜索准则和/或(一个或多个)生物数据样本,并且可以搜索存储介质108中存储的数据库内的与搜索准则和/或(一个或多个)生物数据样本的一个或多个匹配。例如,在步骤408处,诊断匹配器126可以将搜索准则与存储在存储介质108中的注释进行比较。例如,诊断匹配器126可以将搜索准则与在存储介质108中的数据库中存储且与集群表示相关联的临床变量、临床结果和/或临床标签进行比较。存储且与集群表示相关联的临床变量、临床结果和/或临床标签可以是在步骤222处映射的注释。类似地,诊断匹配器126可以将在步骤404处接收的生物数据与在方法200的步骤224处生成的并且存储在存储介质108的数据库中的质心或其他数学表示或模型进行比较。此外,如果在步骤402和404处接收到搜索准则和生物数据,则诊断匹配器126可以过滤集群表示并仅搜索与搜索准则相关联的集群表示。
在步骤410处,诊断匹配器126可以确定是否找到与搜索准则和/或(一个或多个)生物数据样本的任何一个匹配或多个匹配。这里,可以采用各种基于语义的搜索方法中的任何一种来实施在步骤406和410处关于搜索准则的搜索和匹配。类似地,通过选择落入相似性阈值距离内的集群表示,可以将(一个或多个)生物数据样本与集群表示匹配,该相似性阈值距离可以是预设的,或者可以通过在步骤402处接收的搜索准则来设置。例如,诊断匹配器126可以在比较步骤408处确定在步骤404处接收的生物数据样本与存储在存储介质108中的所有集群表示或集群表示的子集之间的欧几里德距离度量、曼哈顿距离度量和/或一些其他适当的度量,其中,如上所述,可以通过利用搜索准则对这些表示进行过滤来确定集群表示的子集。响应于确定在步骤404处接收的生物数据样本落入与任何一个或多个集群表示(其可以是如上所述的质心)的阈值距离内,诊断匹配器126确定这个表示或这些表示与生物数据匹配。否则,响应于确定在步骤404处接收的生物数据样本未落入与存储的表示中的任何表示的阈值距离内,诊断匹配器126确定未找到匹配。
如果未找到与搜索准则和/或生物数据的匹配,则该方法可以前进到步骤414,在该步骤处,诊断匹配器126可以通过显示/输出设备104指示没有找到匹配。因此,诊断匹配器126可以指示存储在存储介质108中的数据库缺少特定诊断信息,并且可以提示用户利用额外的生物数据来运行方法200以扩展数据库。
如果找到与搜索准则和/或生物数据的匹配,则该方法可以前进到步骤412,在该步骤处,诊断匹配器126可以输出与在步骤404处接收的(一个或多个)生物数据样本匹配的(一个或多个)表示/(一个或多个)模型、注释和/或与匹配的表示/(一个或多个)模型相关联的诊断信息,注释可以包括例如与搜索准则匹配的临床变量、临床结果和/或临床标签。例如,如果在步骤402处用户输入搜索准则,则诊断匹配器126可以通过显示/输出设备104输出与匹配的注释相关联的特征数据集和/或集群表示,例如质心,搜索准则可以表示例如特定疾病或分型和/或健康护理提供者正在考虑向患者开出的特定药物或其他处置,而没有生物数据样本。例如,如果用户输入特定癌症分型,则诊断匹配器126可以输出基因组和/或蛋白质组以及与匹配的表示中的基因组/蛋白质组相关的对应基因组/蛋白质组信息,例如,例如,拷贝数变异数据、基因表达数据和/或基因甲基化数据。输出可以通知健康护理提供者他或她应当获得以使用系统106确定患者是否具有搜索准则中的分型的特定生物数据。另外,如果健康护理提供者输入一个或多个生物数据样本并且找到一个或多个集群表示匹配,则诊断匹配器126可以输出与匹配的表示相关联的癌症分型以通知健康护理提供者患者可能患有这个特殊的分型。因此,以这种方式,例如,该系统可以帮助指导患者的临床管理。此外,如果健康护理提供者输入一个或多个生物数据样本并且找到一个或多个集群表示匹配,则诊断匹配器126可以备选地或额外地输出临床变量,例如药物处置或其他类型的处置,以及与匹配的(一个或多个)表示相关的临床结果。例如,诊断匹配器126可以以这种方式通知健康护理提供者,集群中与健康护理提供者的当前患者匹配的先前患者通过特定药物治疗治愈或对特定药物治疗有反应或无反应。这样,方法200、400和系统106可以在针对患者的治疗规划期间向健康护理提供者提供有效的临床指导。
现在参考图5,示出了可以通过其实施以上描述的本原理的方法实施例的示范性计算系统500。计算系统500包括硬件处理器或控制器510和存储介质508。处理器510可以通过中央处理单元(cpu)总线514访问随机存取存储器(ram)516和只读存储器(rom)520。此外,处理器510还可以通过输入/输出控制器512、输入/输出总线504和存储接口506访问计算机可读存储介质508,如图5所示。处理器510可以实施元件110、126、112、114、122或124中的任何一个或多个。系统500还可以包括输入/输出接口502,其可以耦合到显示/输出设备104、用户接口102、键盘、鼠标、触摸屏、外部驱动器或存储介质等,用于向系统500和从系统500输入和输出数据。根据一个示范性实施例,处理器510可以访问存储在存储介质508中的软件指令,并且可以访问存储器516和520以运行存储在存储介质508上的软件指令。具体地,软件指令可以实施或者可以是方法200和/或方法400的步骤。或者,实施方法200和/或400的软件指令可以被编码在计算机可读信号介质中,例如射频信号、电信号或光信号。
将显而易见的是,示例计算系统500的各种备选硬件可用于实施本文描述的方法和系统。例如,在一些实施例中,托管在云计算环境中的一个或多个虚拟机可以提供本文描述的功能中的一些或全部。因此,系统500的部件中的一些可以驻留在彼此分开的物理设备中,但是,也可以作为单个虚拟设备或其分组一起操作。对支持这种布置的系统的各种修改将是显而易见的。
如上所述,本文描述的生物信息学方法和系统通过将非零集群内不一致性度量分配给单样本集群来提供用于识别亚群的有效且准确的手段。本文描述的实施例可以在利用生物信息学技术的任何适当领域中使用。例如,如上所述,实施例可以在临床应用中用于检测克隆演变模式和肿瘤异质性以确定肿瘤的侵袭性。另外,如上所述,实施例可以用于发现细菌感染中的新群体长出以及其他应用中。此外,实施例可以用于治疗设计中。例如,如上所述,对亚群的识别可以使得健康护理专业人员能够为每个亚群定制药物,从而显著增强处置成功的几率。
虽然在本文中已经描述并且说明了若干实施例,但是本领域的普通技术人员将容易设想用于执行本文所描述的功能和/或获得本文所描述的结果和/或优点中的一个或多个的各种其他装置和/或结构,并且这样的变型和修改中的每个被认为是在本文所描述的实施例的范围内。更一般地,本领域的技术人员将容易认识到,在本文中所描述的所有参数、材料和配置旨在为示例性的,并且实际参数、材料和/或配置将取决于使用本教导的特定一个应用或多个应用。本领域的技术人员将认识到或者能够使用不超过常规试验确定本文所描述的特定实施例的许多等价方案。因此,应理解,前述实施例仅以示例的方式呈现,并且在随附权利要求书和其等价方案的范围内,可以实践除特别地描述和要求保护外的实施例。本公开的实施例涉及本文所描述的每个单独特征、系统、制品、材料、工具和/或方法。另外,如果这样的特征、系统、制品、材料、工具和/或方法不互相矛盾,则两个或两个以上这样的特征、系统、制品、材料、工具和/或方法的任何组合被包括在本公开的范围内。
如本文所定义并且所使用的所有定义应当被理解为控制在词典定义、通过引用并入的文档中的定义和/或定义的术语的普通含义上。
除非清楚地给出相反指示,否则如本文在说明书中并且在权利要求中所使用的词语“一”和“一个”应当被理解为意指“至少一个”。
如本文在说明书中并且在权利要求中所使用的短语“和/或”应当被理解为意指如此结合的元件“之一或两者”(即,结合地存在于一些情况中并且分离地存在于其他情况中的元件)。利用“和/或”列出的多个元件应当以相同的方式解释(即,如此结合的元件中的“一个或多个”)。除由“和/或”子句特别地识别的元件之外,可以任选地存在其他元件,无论与特别地识别的那些元件有关还是无关。因此,作为非限制性示例,在一个实施例中,当结合开放式语言(诸如“包括”)使用时,对“a和/或b”的参考可以指代仅a(任选地包括除b之外的元件);在另一实施例中,指代仅b(任选地包括除a之外的元件);在又一实施例中,指代a和b两者(任选地包括其他元件);等等。
如本文在说明书中并且在权利要求中所使用的,“或者”应当被理解为具有与如上文所定义的“和/或”相同的含义。例如,当分离列表中的项时,“或”或者“和/或”应当被解释为包括性的,即,包括至少一个,而且包括若干元件或者元件的列表中的超过一个元件,以及任选地额外的未列出项。只有在清楚地给出相反指示的术语(诸如“……中的仅一个”或者“……中的确切一个”或者当使用在权利要求中时“由……构成”)将指代包括若干元件或者元件的列表中的确切一个元件。一般而言,如本文所使用的术语“或者”应当仅当在排他性的术语(诸如“任一”、“之一”、“……中的仅一个”或者“……中的确切一个”)之前时才被解释为指示排他性备选方案(即,“一个或另一个但非两者”)。当使用在权利要求中时,“基本上由……构成”应当具有其如在专利法的领域中使用的普通含义。
如本文在说明书中并且在权利要求中所使用的,对一个或多个元件的列表的引用中的短语“至少一个”应当被理解为意指选自元件的列表中的元件中的任何一个或多个的至少一个元件,但是不必包括元件的列表内特别地列出的每一个元件中的至少一个并且不排除元件的列表中的元件的任何组合。该定义还允许可以任选地存在除短语“至少一个”指代的元件的列表内特别地识别的元件之外的元件,无论与特别地识别的那些元件有关还是无关。因此,作为非限制性示例,在一个实施例中,“a和b中的至少一个”(或者,等效地“a或b中的至少一个”或者等效地“a和/或b中的至少一个”)可以指代至少一个,任选地包括超过一个,a,并且没有b存在(并且任选地包括除b之外的元件);在另一实施例中,至少一个,任选地包括超过一个,b,并且没有a存在(并且任选地包括除a之外的元件);在又一实施例中,至少一个,任选地包括超过一个,a,以及至少一个,任选地包括超过一个,b(并且任选地包括其他元件);等等。
还应当理解,除非清楚地给出相反指示,否则在本文要求保护的包括超过一个步骤或者动作的任何方法中,方法的步骤或者动作的顺序不必限于方法的步骤或者动作被记载的顺序。
在权利要求中以及在以上说明书中,所有连接词(诸如“包括(comprising)”、“包括(including)”、“承载”、“具有”、“包含”、“涉及”、“保持”、“包括(composedof)”等)将被理解为开放式的(即,意指包括但不限于)。仅连接词“由……构成”和“基本上由……构成”应当分别是封闭式或者半封闭式连接词,如在美国专利局专利审查指南规程2111.03节中所阐述的。
- 还没有人留言评论。精彩留言会获得点赞!