一种化学分子式切分方法与流程
本发明属于化合物合成技术领域,尤其涉及一种化学分子式切分方法。
背景技术:
在化合物合成的流程中,要对带有保护基团的试剂分子进行脱保护基、互相反应得到新化合物。从化学角度来看,这两个步骤都是化学反应;从计算的角度来看,分子脱掉保护基的化学反应是分子的“位点切分”,得到可以与其他分子反应(连接)的位点,之后互相反应则是分子间的“位点拼接”,把可发生化学反应的位点相连,得到新化合物。
计算上目前的处理方式是把第一步得到的带位点的分子记录下来,将不同反应得到的位点区分标记,如反应类型1得到的位点标记为[r1],位点的位置即分子在反应中脱去部分的原子的位置,然后将带有相同位点标记的分子进行拼接,如两个分子都带有[r1]则可以拼接。
目前的计算化学工具如openbabel、rdkit等,是通过模拟真实化学反应过程进行切分的,即输入待反应的化合物,得到反应后的化合物。
技术实现要素:
本发明的目的在于针对现有技术的不足,在分子式的图结构基础实现了一种切分方法,即用特定类型的化学反应切掉分子式的某一基团,切分模拟了化学反应中的一个子过程,在真实化学反应中,发生化学反应的化合物会被切掉一部分,之后与其他化合物结合,形成新的化合物。在通过大量试剂分子进行化合物合成的应用场景下,分子式切分是一个固有的子环节,试剂分子脱去固定基团首先要脱掉某个基团,才能与其他化合物结合。
本发明的目的是通过以下技术方案来实现的:一种化学分子式切分方法,对于一个带位点或不带位点的分子式smi,将分子式中的位点记为[ri],i=0,1…n,[ri]对应于某一反应类型,n为反应类型的总数;
对一种反应类型reaction,定义一个反应类型串smi_sub和转换类型串smi_convert,表示在smi中如果存在smi_sub子串,则把该子串部分转换为smi_convert;smi_sub子串包含至少一个与smi其余部分连接的位点[ri],smi_convert在对应位置有相同的位点[ri],smi_convert还包含要切除的原子[*],切分过程如下:
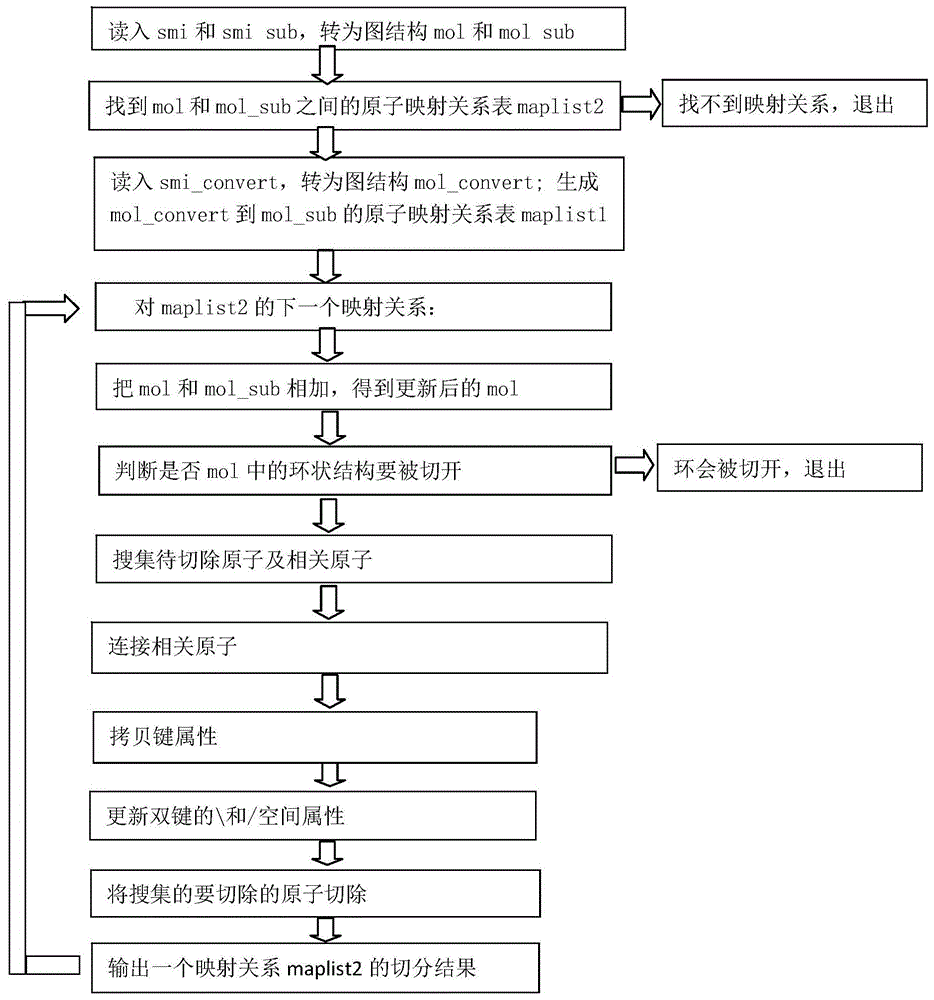
(1)读入smi和smi_sub,转为图结构mol和mol_sub,生成mol_sub到mol的原子映射关系表maplist2,如果mol中不存在mol_sub子结构,则不再进行切分。如果mol中存在多个mol_sub子结构,则maplist2中保存多个映射关系。映射时考虑连接位点对应的原子是否满足smi_sub中对[ri]类型的限定,如不满足则映射失败。在读入smi_sub时,保存连接位点[ri]的位置。
(2)读入smi_convert,转为图结构mol_convert,生成mol_convert到mol_sub的原子映射关系表maplist1,找到切除原子[*]的位置cut_idx,找到切除原子[*]连接的原子的位置cut_connect_idx。对于mol_convert中的每个原子,在mol_sub中找不到对应原子时,用-1表示该原子的映射原子位置。
(3)对maplist2中的每一个映射关系,得到一个切分结果:
(3.1)把mol和mol_sub相加,得到更新后的mol。
(3.2)判断该映射关系是否会导致mol中的环状结构被切开,如果是,则跳过这个映射关系,执行下一个映射关系,否则,执行步骤3.3。
(3.3)通过该映射关系,搜集映射到mol中不切除的原子的位置,即maplist1中>-1的元素对应的位置,存放到not_cut_idxs。
搜集映射到mol中切除原子[*]连接的原子的位置,即cut_connect_idx对应的原子,存放到cut_connect_idxs。
搜集映射到mol中的全部切除原子的位置,即maplist1中为-1的元素对应的位置,存放到cut_idxs,对于切分点位置的原子,将其邻接原子中不在not_cut_idxs中的原子存放到cut_idxs。每个切除位置维护一个cut_idxs列表,如果该位置找不到mol中对应的原子,则该位置列表为空。
对每个切除位置的cut_idxs,如果为空,则切除该位置上的氢原子,如果该位置没有氢原子,则跳过这个映射关系的切分,执行下一个映射关系。如果不为空,搜集该位置上要切除的原子,包括该位置的原子和与之相邻但不在cut_connect_idxs中的原子,递归搜集该位置相邻原子的相邻原子。
搜集mol中要切除的原子的位置,即cut_connect_idx中的那些原子,包括该位置的原子和与之相邻但不在cut_connect_idx和cut_idx中的原子,这些原子在新mol中有新的位置,递归搜集该位置相邻原子的相邻原子。
(3.4)连接原子:将cut_connect_idxs和cut_idx中的对应原子用化学键相连。
(3.5)拷贝键属性:从mol_sub中复制键属性到mol中新增的键。搜集新增原子,即新增键的两端的原子。
(3.6)更新双键的\和/空间属性:如果mol的空间属性结构中包含要切除的原子,则需要更新空间属性。双键空间属性结构中存储了双键两端的原子及与它们相连的原子,如果双键空间属性结构中的两端原子与上一步的新增原子之间有键,则把结构中的对应原子替换为新增原子。
(3.7)将搜集的要切除的原子切除。
(3.8)输出一个maplist2映射关系的切分后的分子。
进一步地,根据各反应类型的特点,对反应类型串smi_sub中的位点[ri]所代表的原子类型作一些限定,即对[ri]对应到待切分子式中的原子的类型作一些限定,包括位点[ri]不能为氢原子(h),或只能为c、n、o原子,或可以为任意原子,限定按照smarts规范。
进一步地,所述步骤(1)中,读入的分子式格式为canonicalsmiles,其他格式的输入转换为该格式。
进一步地,所述步骤(1)中,映射时考虑连接位点对应的原子是否满足smi_sub中对[ri]类型的限定,如不满足则映射失败。
进一步地,所述步骤(3.2)中,判断该映射关系是否会导致mol中的环状结构被切开即判断mol_sub是否映射到了mol的环状结构的一部分,具体为:通过maplist2中的映射关系找到mol中待切位点[ri]的对应原子a,如果a有一个相邻原子a_neighbor不在要切除的列表中,且a_neighbor在环上,则环会被切开。
进一步地,所述步骤(3.5)中,从mol_sub中复制键属性到mol中新增的键,即找到mol_sub中每个键两端的原子,找到在mol中对应的两个原子,将这两个原子之间的键属性更新为mol_sub中对应键的属性值。
本发明的有益效果是:本发明在原子、化学键的图结构上实现分子式的切分,操作灵活性强,可定制性强,适用于对于多种情形的切分。通过待切分化学式中与表示反应类型的反应类型串,及反应类型串表示转换类型的反应类型串之间的原子映射关系,找到待切分化学式中哪些原子上的哪些分枝要切除,哪些分枝要保留,实现正确的切分。
附图说明
图1为本发明方法流程图;
图2为本发明的一个切分示例。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,以下结合附图及实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
本发明提供的一种化学分子式切分方法,具体如下:
对于一个带位点或不带位点的分子式smi(假设输入格式为canonicalsmiles,其他格式的输入也可转换为该格式),将分子式中的位点记为[ri],i=0,1…n,[ri]对应于某一反应类型,是用试剂分子进行该类型的化学反应得到的,n为反应类型的总数;
对一种反应类型reaction,定义一个反应类型串smi_sub和转换类型串smi_convert,表示在smi中如果存在smi_sub子串,则把该子串部分转换为smi_convert。smi_sub子串包含至少一个与smi其余部分连接的位点[ri],smi_convert在对应位置有相同的位点[ri],smi_convert还包含要切除的原子[*]。特别地,根据各反应类型的特点,对反应类型串smi_sub中的位点[ri]所代表的原子类型作一些限定,即对[ri]对应到待切分子式中的原子的类型作一些限定,如位点[ri]不能为氢原子(h),或只能为c、n、o原子,或可以为任意原子,具体的限定处理主要按照smarts规范。切分过程如下:
1.读入smi和smi_sub,转为图结构mol和mol_sub,生成mol_sub到mol的原子映射关系表maplist2,如果mol中不存在mol_sub子结构,则不再进行切分,说明分子式smi不能发生反应类型reaction的化学反应。如果mol中存在多个mol_sub子结构,则maplist2中保存多个映射关系,此时说明分子式smi有多个基团可以发生该类型的化学反应。映射时考虑连接位点对应的原子是否满足smi_sub中对[ri]类型的限定,如不满足则映射失败。
在读入smi_sub时,保存连接位点[ri]的位置。
2.读入smi_convert,转为图结构mol_convert,生成mol_convert到mol_sub的原子映射关系表maplist1,找到切除原子[*]的位置cut_idx,找到切除原子[*]连接的原子的位置cut_connect_idx。对于mol_convert中的每个原子,maplist1中由于被切除或替换为更长的子串,导致在mol_sub中找不到对应原子时,用-1表示该原子的映射原子位置。
3.对maplist2中的每一个映射关系,得到一个切分结果:
3.1.把mol和mol_sub相加,即将两者的数据结构堆叠,得到更新后的mol。
3.2.判断该映射关系是否会导致mol中的环状结构被切开,如果是,则跳过这个映射关系,执行下一个映射关系,否则,执行步骤3.3;判断该映射关系是否会导致mol中的环状结构被切开即判断mol_sub是否映射到了mol的环状结构的一部分,具体为:通过maplist2中的映射关系找到mol中待切位点[ri]的对应原子a,如果a有一个相邻原子a_neighbor不在要切除的列表中,且a_neighbor在环上,则环会被切开。
3.3.通过该映射关系,搜集映射到mol中不切除的原子的位置,即maplist1中>-1的元素对应的位置,存放到not_cut_idxs。
搜集映射到mol中切除原子[*]连接的原子的位置,即cut_connect_idx对应的原子,存放到cut_connect_idxs。
搜集映射到mol中的全部切除原子的位置,即maplist1中为-1的元素对应的位置,存放到cut_idxs,对于切分点位置的原子,将其邻接原子中不在not_cut_idxs中的原子存放到cut_idxs,由于在转换串中已经不存在,所以这些原子也是要切除的。每个切除位置维护一个cut_idxs列表,如果该位置找不到mol中对应的原子,则该位置列表为空。
对每个切除位置的cut_idxs,如果为空,则切除该位置上的氢原子,如果该位置没有氢原子,则跳过这个映射关系的切分,执行下一个映射关系。如果不为空,搜集该位置上要切除的原子,包括该位置的原子和与之相邻但不在cut_connect_idxs中的原子,递归搜集该位置相邻原子的相邻原子。
搜集mol中要切除的原子的位置,即cut_connect_idx中的那些原子,包括该位置的原子和与之相邻但不在cut_connect_idx和cut_idx中的原子,这些原子在新mol中有新的位置,递归搜集该位置相邻原子的相邻原子。
3.4.连接原子:将cut_connect_idxs和cut_idx中的对应原子用化学键相连。
3.5.拷贝键属性:从mol_sub中复制键属性到mol中新增的键,即找到mol_sub中每个键两端的原子,找到在mol中对应的两个原子,将这两个原子之间的键属性更新为mol_sub中对应键的属性值。搜集新增原子,即新增键的两端的原子。
3.6.更新双键的\和/空间属性:由于空间属性是单独存储的,需要单独处理,如果mol的空间属性结构中包含要切除的原子,则需要更新空间属性。双键空间属性结构中存储了双键两端的原子及与它们相连的原子,如果双键空间属性结构中的两端原子与上一步的新增原子之间有键,则把结构中的对应原子替换为新增原子。
3.7.将搜集的要切除的原子切除。
3.8.输出一个maplist2映射关系的切分后的分子,即把mol转换为canonicalsmiles格式。
切分示例如下:如图2所示,(a)、(b)、(c)、(d)依次为:反应类型、转换类型、待切分子式、切分的最终结果,其中xx即为[*]原子,s为[r]原子;对应的化学式依次为:
[s]c=c
[s]c([*])c[*]
ccoc(=o)/c=c/c(o)=o
ccoc(=o)c(c(c(=o)o)[*])[*]
mol_convert(第2个化学式)到mol_sub(第1个化学式)的原子映射关系表maplist1为{0,1,-1,2,-1}。
需要声明的是,本发明内容及具体实施方式意在证明本发明所提供技术方案的实际应用,不应解释为对本发明保护范围的限定。在本发明的精神和权利要求的保护范围内,对本发明作出的任何修改和改变,都落入本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!