一种高效可并行运算且高准确性的全基因组选择方法与流程
本发明涉及动植物育种及人类疾病预测技术领域,特别是涉及一种高效可并行运算且高准确性的全基因组选择方法。
背景技术:
随着覆盖整个基因组高密度单核苷酸多态性(snp)基因分型技术的发展,全基因组选择(预测)作为基因组统计分析的强大工具,被广泛应用于植物和动物育种中复杂性状的遗传价值(种用价值)预测和评估,以及人类遗传学研究中。
现有的全基因组选择方法分为两类:一类是以全基因组最佳线性无偏预测gblup(genomicbestlinearunbiasedprediction)为代表的直接法,仅需构建个体间基因组关系矩阵,获取方差组分后可通过求解混合模型求得个体育种值;另一类以bayesb为代表的间接法,结合bayes理论和隐马尔可夫迭代过程求取标记效应值,然后依照个体基因型对标记效应进行累加获得个体育种值。其中,直接法计算效率高,但由于其对性状遗传构建的假设简单,估计的育种值准确性较差;间接法对性状遗传构建的假设相对复杂,更符合性状遗传机制,具有更好的预测准确性,但由于其假设引入众多的未知参数,导致其参数求解过程极其复杂,计算效率较差,限制了间接法在实际预测中的应用。
技术实现要素:
针对现有技术存在的问题,本发明提供一种高效可并行运算且高准确性的全基因组选择方法,能够快速准确且稳定地预测出个体基因组育种值,提升全基因组选择的准确性及计算效率。
本发明的技术方案为:
一种高效可并行运算且高准确性的全基因组选择方法,其特征在于,包括下述步骤:
步骤1:读取原始基因型文件和原始表型文件,提取原始基因型文件和原始表型文件中相同个体的基因型数据和表型数据,形成新的基因型文件和新的表型文件,并利用新的基因型文件计算所有个体间的亲缘关系矩阵g;
步骤2:提取新的表型文件中的所有个体作为参考群,提取原始基因型文件中无表型数据的所有个体作为预测群,得到参考群数据和预测群数据,将参考群随机分为m个规模相同的子参考群;其中,参考群数据包括参考群中每个个体的基因型数据和表型数据,预测群数据包括预测群中每个个体的基因型数据;
步骤3:利用参考群数据进行全基因组关联分析,提取全基因组关联分析的结果特征;构建性状特异的模型库,采用交叉验证策略,依次优化最佳固定效应、最佳随机效应,从模型库中选取最优预测模型;
步骤4:利用所述最优预测模型,计算所述预测群的基因组估计育种值。
所述步骤3包括下述步骤:
步骤3.1:使用l个线程并行执行相关系数计算;其中,使用第l∈{1,2,...,l}个线程执行相关系数计算包括:
步骤3.1.1:随机选取m-1个子参考群组合成测试群,将未被选取的子参考群作为验证群,利用测试群进行全基因组关联分析,采用选定的模型提取基因型中所有位点的显著p值为{p1l,p2l,...,pnl,...,pnl};其中,n为基因型中的位点总数,pnl为第l个线程计算的基因型中第n个位点的显著p值,n∈{1,2,...,n};
步骤3.1.2:用预设大小的窗口对所有位点按照基因组上的分布顺序进行划分,得到x个窗口内的位点;将每个窗口内的位点按照显著p值从小到大进行排序,选取每个窗口中显著p值最大的位点,形成位点集合x;
步骤3.1.3:利用测试群和gblup模型计算验证群的基因组估计育种值gebv1,gebv1只包含随机效应部分,计算gebv1与验证群真实表型间的相关系数c0l;利用所述位点集合x测试固定效应模型flm,从位点集合x中依次逐个不放回地取出位点并加入flm中作为协变量,计算验证群的基因组估计育种值gebv2,gebv2只包含固定效应部分,并计算gebv2与验证群真实表型间的相关系数集合为{cf1l,cf2l,...,cfil,...,cfxl};利用所述位点集合x测试混合效应模型mlm,从位点集合x中依次逐个不放回地取出位点并加入mlm中作为协变量,计算验证群的基因组估计育种值gebv3,gebv3包含固定效应及随机效应两部分,并计算gebv3与验证群真实表型间的相关系数集合为{cm1l,cm2l,...,cmil,...,cmxl};其中,i∈{1,2,...,x};
步骤3.1.4:若cfil>cf,i-1,l且cfil>c0l,则位点集合x中第i个位点为flm有效位点,第i个位点对应的窗口为flm有效窗口,得到flm有效窗口集合fl;若cmil>cm,i-1,l且cmil>c0l,则位点集合x中第i个位点为mlm有效位点,第i个位点对应的窗口为mlm有效窗口,得到mlm有效窗口集合ml;
步骤3.2:计算{c01,c02,...,c0l,...,c0l}的均值为
步骤3.3:若最优预测模型为flm,则对l个flm有效窗口集合{f1,f2,...,fl,...,fl}中的窗口进行计数,挑取出现次数大于或等于l×95%的flm有效窗口作为终选flm有效窗口;若最优预测模型为mlm,则对l个mlm有效窗口集合{m1,m2,...,ml,...,ml}中的窗口进行计数,挑取出现次数大于或等于l×95%的mlm有效窗口作为终选mlm有效窗口;
步骤3.4:计算{pn1,pn2,...,pnl,...,pnl}的指定值作为第n个位点的最终关联p值
步骤3.5:使用l个线程并行执行梯度下相关系数计算;其中,使用第l∈{1,2,...,l}个线程执行梯度下相关系数计算包括:
步骤3.5.1:若最优预测模型为gblup或mlm,则基于vanraden算法初始化n×n的对角权重矩阵w=diag(w1,w2,...,wn)=diag(1,1,...,1);对所述步骤3.1.1得到的所有位点的显著p值{p1l,p2l,...,pnl,...,pnl}按照从小到大的顺序进行排序,得到排序后的显著p值序列{p1l',p2l',...,pnl',...,pnl'},将排序后的显著p值序列中前α%的元素对应的权重设置为放大倍数、将后(1-α%)的元素对应的权重保持不变仍为1,得到新的对角权重矩阵w';其中,对α设置n1个梯度为{α1,α2,...,αp,...,αn1}、设置放大函数为logβp,对β设置n2个梯度{β1,β2,...,βq,...,βn2},梯度βq下前αp%的元素中第k个元素对应的放大倍数为
步骤3.5.2:利用矩阵t及所述步骤3.1.1中的测试群计算所述步骤3.1.1中的验证群的基因组估计育种值gebv4,并计算梯度{αp,βq}下gebv4与所述步骤3.1.1中的验证群真实表型间的相关系数cpql,继而得到所有梯度下的相关系数为{c11l,c12l,...,c1,n2,l,...,cpql,cp,q+1,l,...,cp,n2,l,...,cn1,1,l,cn1,2,l,...,cn1,n2,l};
步骤3.6:计算{cpq1,cpq2,...,cpql,...,cpql}的均值为
步骤3.7:若需要对亲缘关系矩阵g进行优化且最优前百分比为αp%、最优放大函数为
所述步骤3.1.1中,所述选定的模型为gblup模型或固定效应模型flm或混合效应模型mlm。
所述步骤3.4中,所述指定值为最大值或最小值或均值或中值。
所述步骤4包括下述步骤:
步骤4.1:若最优预测模型为flm,则利用所述flm最佳协变量位点及所述参考群计算所述预测群的基因组估计育种值;
步骤4.2:若最优预测模型为gblup且无需对亲缘关系矩阵g进行优化,则利用所述亲缘关系矩阵g及所述参考群计算所述预测群的基因组估计育种值;
步骤4.3:若最优预测模型为gblup且需要对亲缘关系矩阵g进行优化,则利用所述最佳性状特异的个体间亲缘关系矩阵g'及所述参考群计算所述预测群的基因组估计育种值;
步骤4.4:若最优预测模型为mlm且无需对亲缘关系矩阵g进行优化,则利用所述mlm最佳协变量位点、所述亲缘关系矩阵g及所述参考群计算所述预测群的基因组估计育种值;
步骤4.5:若最优预测模型为mlm且需要对亲缘关系矩阵g进行优化,则利用所述mlm最佳协变量位点、所述最佳性状特异的个体间亲缘关系矩阵g'及所述参考群计算所述预测群的基因组估计育种值。
本发明的有益效果为:
本发明在gblup模型的基础上融合bayes理论的先验假设,结合全基因组关联分析(gwas)信息,基于可并行的交叉验证,采用多元回归、网格搜索、二分法求极值等策略,针对不同性状自动选择最佳预测模型,准确筛选大效应标记以整合模型协变量,同时给予不同标记合适权重以构建个体关系矩阵,联合解析复杂性状遗传构建(geneticarchitecture),能够快速准确且稳定地预测出个体基因组育种值,提升全基因组选择的准确性及计算效率。
附图说明
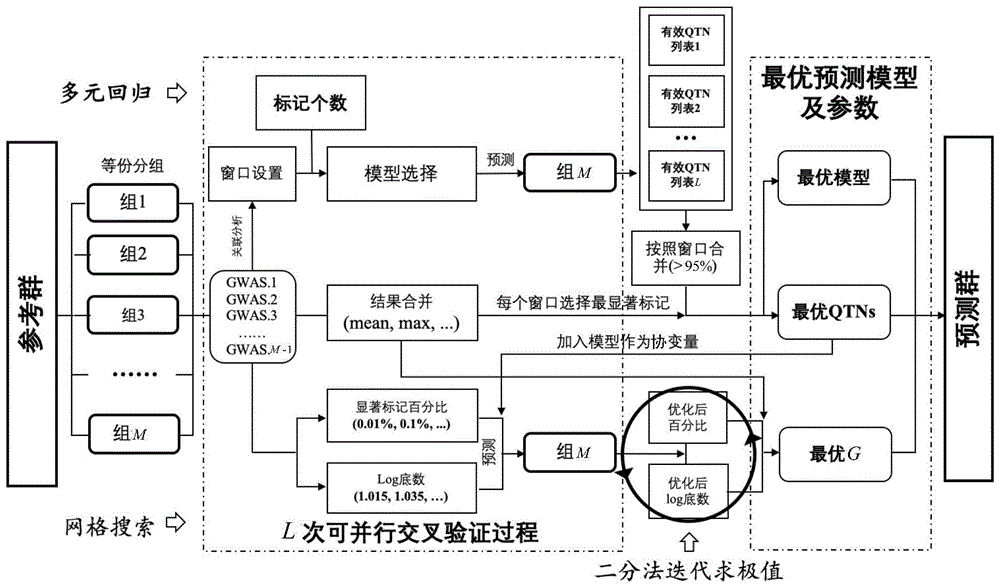
图1为本发明的高效可并行运算且高准确性的全基因组选择方法的原理图。
具体实施方式
下面将结合附图和具体实施方式,对本发明作进一步描述。
如图1所示,本发明的高效可并行运算且高准确性的全基因组选择方法,其特征在于,包括下述步骤:
步骤1:读取原始基因型文件和原始表型文件,提取原始基因型文件和原始表型文件中相同个体的基因型数据和表型数据,形成新的基因型文件和新的表型文件,并利用新的基因型文件计算所有个体间的亲缘关系矩阵g。
本实施例中,采用r语言中的s4数据格式来建立磁盘与内存之间的数据映射;基因型文件和表型文件的读取及储存采用rcran::bigmemory软件包中的big.matrix格式,并提供plink格式[.ped/.map,.bed/.bim/.fam]、hapmap格式、vcf格式和numeric格式[0/1/2]四种数据格式的转换。检查、调整原始基因型文件和原始表型文件中的个体顺序,使其一致,并选留两个文件中同时存在的个体,获得新的表型文件及基因型文件。
步骤2:提取新的表型文件中的所有个体作为参考群,提取原始基因型文件中无表型数据的所有个体作为预测群,得到参考群数据和预测群数据,将参考群随机分为m个规模相同的子参考群;其中,参考群数据包括参考群中每个个体的基因型数据和表型数据,预测群数据包括预测群中每个个体的基因型数据。
步骤3:利用参考群数据进行全基因组关联分析,提取全基因组关联分析的结果特征;构建性状特异的模型库,采用交叉验证策略,依次优化最佳固定效应、最佳随机效应,从模型库中选取最优预测模型。
所述步骤3包括下述步骤:
步骤3.1:使用l个线程并行执行相关系数计算;其中,使用第l∈{1,2,...,l}个线程执行相关系数计算包括:
步骤3.1.1:随机选取m-1个子参考群组合成测试群,将未被选取的子参考群作为验证群,利用测试群进行全基因组关联分析,采用选定的模型提取基因型中所有位点的显著p值为{p1l,p2l,...,pnl,...,pnl};其中,n为基因型中的位点总数,pnl为第l个线程计算的基因型中第n个位点的显著p值,n∈{1,2,...,n};本实施例中,所述选定的模型为gblup模型或固定效应模型flm或混合效应模型mlm;
步骤3.1.2:用预设大小的窗口对所有位点按照基因组上的分布顺序进行划分,得到x个窗口内的位点;将每个窗口内的位点按照显著p值从小到大进行排序,选取每个窗口中显著p值最大的位点,形成位点集合x;本实施例中,窗口大小为1mb;
步骤3.1.3:利用测试群和gblup模型计算验证群的基因组估计育种值gebv1,gebv1只包含随机效应部分,计算gebv1与验证群真实表型间的相关系数c0l;利用所述位点集合x测试固定效应模型flm(fixedeffectlinearmodel),从位点集合x中依次逐个不放回地取出位点并加入flm中作为协变量,计算验证群的基因组估计育种值gebv2,gebv2只包含固定效应部分,并计算gebv2与验证群真实表型间的相关系数集合为{cf1l,cf2l,...,cfil,...,cfxl};利用所述位点集合x测试混合效应模型mlm(mixedeffectlinearmodel),从位点集合x中依次逐个不放回地取出位点并加入mlm中作为协变量,计算验证群的基因组估计育种值gebv3,gebv3包含固定效应及随机效应两部分,并计算gebv3与验证群真实表型间的相关系数集合为{cm1l,cm2l,...,cmil,...,cmxl};其中,i∈{1,2,...,x};
步骤3.1.4:若cfil>cf,i-1,l且cfil>c0l,则位点集合x中第i个位点为flm有效位点,第i个位点对应的窗口为flm有效窗口,得到flm有效窗口集合fl;若cmil>cm,i-1,l且cmil>c0l,则位点集合x中第i个位点为mlm有效位点,第i个位点对应的窗口为mlm有效窗口,得到mlm有效窗口集合ml。
步骤3.2:计算{c01,c02,...,c0l,...,c0l}的均值为
步骤3.3:若最优预测模型为flm,则对l个flm有效窗口集合{f1,f2,...,fl,...,fl}中的窗口进行计数,挑取出现次数大于或等于l×95%的flm有效窗口作为终选flm有效窗口;若最优预测模型为mlm,则对l个mlm有效窗口集合{m1,m2,...,ml,...,ml}中的窗口进行计数,挑取出现次数大于或等于l×95%的mlm有效窗口作为终选mlm有效窗口。
步骤3.4:计算{pn1,pn2,…,pnl,…,pnl}的指定值作为第n个位点的最终关联p值
步骤3.5:使用l个线程并行执行梯度下相关系数计算;其中,使用第l∈{1,2,…,l}个线程执行梯度下相关系数计算包括:
步骤3.5.1:若最优预测模型为gblup或mlm,则基于vanraden算法初始化n×n的对角权重矩阵w=diag(w1,w2,…,wn)=diag(1,1,…,1);对所述步骤3.1.1得到的所有位点的显著p值{p1l,p2l,…,pnl,…,pnl}按照从小到大的顺序进行排序,得到排序后的显著p值序列{p1l',p2l',…,pnl',...,pnl'},将排序后的显著p值序列中前α%的元素对应的权重设置为放大倍数、将后(1-α%)的元素对应的权重保持不变仍为1,得到新的对角权重矩阵w';其中,对α设置n1个梯度为{α1,α2,...,αp,...,αn1}、设置放大函数为logβp,对β设置n2个梯度{β1,β2,...,βq,...,βn2},梯度βq下前αp%的元素中第k个元素对应的放大倍数为
步骤3.5.2:利用矩阵t及所述步骤3.1.1中的测试群计算所述步骤3.1.1中的验证群的基因组估计育种值gebv4,并计算梯度{αp,βq}下gebv4与所述步骤3.1.1中的验证群真实表型间的相关系数cpql,继而得到所有梯度下的相关系数为{c11l,c12l,...,c1,n2,l,...,cpql,cp,q+1,l,...,cp,n2,l,...,cn1,1,l,cn1,2,l,...,cn1,n2,l}。
本实施例中,对α设置n1个梯度为{0.01,0.1,...,1,5};对β设置n2个梯度{1.015,1.035,...,3,5,10}。
步骤3.6:计算{cpq1,cpq2,...,cpql,...,cpql}的均值为
步骤3.7:若需要对亲缘关系矩阵g进行优化且最优前百分比为αp%、最优放大函数为
步骤4:利用所述最优预测模型,计算所述预测群的基因组估计育种值,具体包括下述步骤:
步骤4.1:若最优预测模型为flm,则利用所述flm最佳协变量位点及所述参考群计算所述预测群的基因组估计育种值;
步骤4.2:若最优预测模型为gblup且无需对亲缘关系矩阵g进行优化,则利用所述亲缘关系矩阵g及所述参考群计算所述预测群的基因组估计育种值;
步骤4.3:若最优预测模型为gblup且需要对亲缘关系矩阵g进行优化,则利用所述最佳性状特异的个体间亲缘关系矩阵g'及所述参考群计算所述预测群的基因组估计育种值;
步骤4.4:若最优预测模型为mlm且无需对亲缘关系矩阵g进行优化,则利用所述mlm最佳协变量位点、所述亲缘关系矩阵g及所述参考群计算所述预测群的基因组估计育种值;
步骤4.5:若最优预测模型为mlm且需要对亲缘关系矩阵g进行优化,则利用所述mlm最佳协变量位点、所述最佳性状特异的个体间亲缘关系矩阵g'及所述参考群计算所述预测群的基因组估计育种值。
显然,上述实施例仅仅是本发明的一部分实施例,而不是全部的实施例。上述实施例仅用于解释本发明,并不构成对本发明保护范围的限定。基于上述实施例,本领域技术人员在没有做出创造性劳动的前提下所获得的所有其他实施例,也即凡在本申请的精神和原理之内所作的所有修改、等同替换和改进等,均落在本发明要求的保护范围内。
- 还没有人留言评论。精彩留言会获得点赞!