一种长链RNA特异性探针的设计方法与流程
本发明涉及生物
技术领域:
,特别是涉及一种长链rna特异性探针的设计方法。·
背景技术:
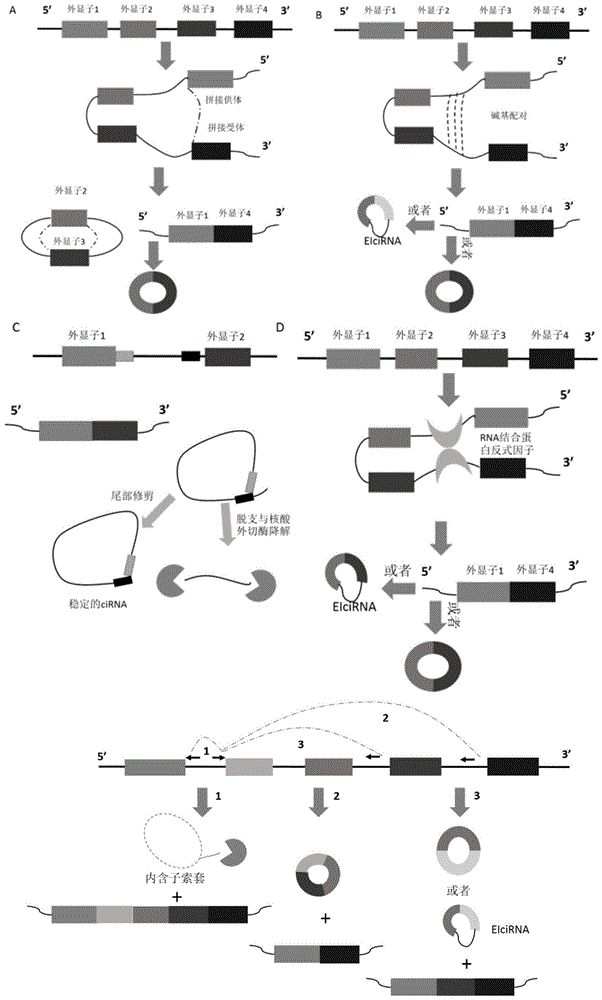
:环状rna(circrna)是一类以共价封闭环为特征,广泛存在于真核生物中的新型rna分子。circrna来源于基因的外显子或者内含子区域,在哺乳动物细胞中大量存在。circrnas的形成不同于线性rna的标准剪切模式,是通过供体外显子的5’端与受体外显子的3’端首尾相连,形成反向剪接位点(backsplicing)剪切而来。现有的circrna形成模型主要有以下几种(参见图1所示):(1)“套锁驱动成环(lariat-drivencircularization)”或者“外显子跳跃成环(exonskipping)”,如图1a所示;(2)“配对内含子驱动成环(intron-pairing-drivencircularization)”或者“直接反向拼接成环(directbacksplicing)”,如图1b所示;(3)环状内含子rna(cirnas)形成模式,如图1c所示;(4)依赖于rna结合蛋白(rnabindingproteins,rbps)环化模式,如图1d所示;(5)类似于可变剪切的可变环化模式,如图1e所示。现有研究表明大部分的circrna在不同物种间是保守的。同时,由于其环状结构能抵抗rnaser的降解而比较稳定。circrna由于其表达的特异性和调控的复杂性,以及在疾病发生中的重要作用,越来越受到大家的重视。就像mirna和长链非编码rna一样,circrna已经成为rna领域的一个新的研究热点。目前常用的环状rna表达丰度检测的技术手段是实时pcr,但该方法具有研究通量低的缺点,而circrna的研究刚刚起步,急需一个高通量的可靠的检测技术来满足研究需要。长链非编码rna(lncrna)泛指长度大于200个碱基的线性非编码rna,整体表达丰度低于mrna,保守性差,40%左右的lncrna具有polya尾巴,具有较强的组织表达特异。其在转录沉默、转录激活、染色体修饰、核内运输等均具有重要的功能。lncrna曾被比喻为宇宙的暗物质,最近几年的研究发现它参与多种生物学过程,是维持基因功能的重要基础,与多种复杂疾病相关。根据长链非编码rna与其最近的mrna的位置关系可以分为:反义长链非编码rna(antisenselncrna)、同义长链非编码rna(senselncrna)、内含子长链非编码rna(introniclncrna)、基因间长链非编码rna(intergeniclncrna)、双向长链非编码rna(bidirectionallncrna)、增强子长链非编码rna(enhancerlncrna)基因芯片技术是指通过微阵列技术将高密度dna片段通过高速机器人或原位合成方式以一定的顺序或排列方式使其附着在如玻片、硅片等固相表面,以荧光或生物素标记的目的片段,借助碱基互补杂交原理,进行大量的基因表达研究的革命性技术。基因芯片技术是伴随着人类基因组计划的实施而发展起来的生命科学领域里的前沿生物技术。目前,人们对疾病的分类和诊断的水平已经有了进一步的提高,基于基因芯片的特征选择技术在其中起到了关键性的作用。经过十几年的发展,基因芯片技术也在不断完善、成熟,并广泛运用于生命科学的各个领域,但现有技术中尚无能够同时检测mrna、环状rna、长链非编码rna等多种长链rna表达丰度的基因芯片及方法。技术实现要素:鉴于以上所述现有技术的缺点,本发明的目的在于提供一种长链rna特异性探针的设计方法,用于解决现有技术中的问题。为实现上述目的及其他相关目的,本发明第一方面提供一种用于同时检测两种以上长链rna的特异性探针的设计方法,包括下列步骤:s100根据预设探针种类设计目标基因的探针,作为候选探针,所述预设探针种类选自;mrna探针、环状rna探针或长链非编码rna探针中的至少两种探针;s200将候选探针序列与所有探针的全长靶序列进行比对;s300若一候选探针的比对结果符合预设值,则保留该候选探针保留作为特异性探针;若一候选探针的比对结果不符合预设值,则淘汰该候选探针。s400若一目标基因无保留的特异性探针,则根据预设的探针种类,重新设计该目标基因的探针作为候选探针,并继续执行步骤s200。若一目标基因保留的特异性探针中,仅包括预设探针种类中的部分种类,则重新设计该目标基因其余预设探针种类作为候选探针,并继续执行步骤s200,直至所有目标基因保留的特异性探针中均包括预设种类的探针;具体的,s300中符合预设值为:候选探针与所有探针的全长靶序列的相似性均不超过第一预设值,且与所有探针的全长靶序列之间连续相同的碱基长度均不超过第二预设值。不符合预设值为:候选探针与所有探针的全长靶序列中的至少一个靶序列的相似性超过第一预设值,或与所有探针的全长靶序列中的至少一个靶序列之间连续相同的碱基长度超过第二预设值。优选的,s100中用于设计所述mrna探针的序列选自各目标基因的mrna最长转录本序列。优选的,s100中用于设计所述环状rna探针的序列选自各目标基因的环状rna的反向剪接序列的片段。优选的,s100中所述环状rna探针选自与目标基因的反向剪接序列的结合位点位于反向剪接位点的环状rna探针。优选的,s100中用于设计反义长链非编码rna或同义长链非编码rna探针的特异性序列选自:反义长链非编码rna或同义长链非编码rna和mrna的非重叠区的片段;用于设计内含子长链非编码rna、基因间长链非编码rna、双向长链非编码rna或增强子长链非编码rna探针的特异性序列选自:各目标基因中最长的长链非编码rna的片段。本发明第二方面提供一种设计长链rna特异性探针的系统,所述系统可以用于设计同时检测mrna、环状rna或长链非编码rna中至少两种长链rna的特异性探针。所述系统包括:设计模块1,用于根据预设探针种类设计目标基因的探针,作为候选探针,所述预设探针种类选自;mrna探针、环状rna探针或长链非编码rna探针中的至少两种探针;比对模块2,用于将候选探针序列与所有探针的全长靶序列进行比对;筛选模块3,用于判断候选探针与所有探针的全长靶序列的比对结果是否符合预设值,若符合,则保留该候选探针保留作为特异性探针;若不符合,则淘汰该候选探针。迭代模块4:用于判断各目标基因有无保留的特异性探针,若无,则根据预设的探针种类,重新设计该目标基因的探针作为候选探针,并继续执行比对模块2。若各目标基因有保留的特异性探针,且保留的特异性探针中仅包括预设探针种类中的部分种类,则重新设计该目标基因其余预设探针种类作为候选探针,并继续执行比对模块2,直至所有目标基因保留的特异性探针中均包括预设种类的探针。进一步的,用于设计所述mrna探针的序列选自各目标基因的mrna最长转录本的序列。更进一步的,选择与目标基因的最长转录本的结合位点位于该最长转录本的3’端的片段,作为目标基因的mrna候选探针。进一步的,用于设计所述环状rna探针的序列选自:各目标基因的环状rna的反向剪接序列的。更进一步的,所述环状rna探针选自与目标基因的反向剪接序列的结合位点位于反向剪接位点的环状rna探针。进一步的,用于设计反义长链非编码rna或同义长链非编码rna探针的序列选自:目的基因的长链非编码rna与mrna非重叠区的序列;用于设计内含子长链非编码rna、基因间长链非编码rna、双向长链非编码rna或增强子长链非编码rna探针的序列选自:各目标基因中最长的长链非编码rna的片段。具体的,筛选模块3中,若一候选探针与所有探针的全长靶序列的相似性均不超过第一预设值,且与所有探针的全长靶序列之间连续相同的碱基长度均不超过第二预设值,则认定该候选探针与所述探针的全长靶序列的比对结果符合预设值。若一候选探针与所有探针的全长靶序列中的至少一个靶序列的相似性超过第一预设值,或与所有探针的全长靶序列中的至少一个靶序列之间连续相同的碱基长度超过第二预设值,则认定该候选探针与所述探针的全长靶序列的比对结果不符合预设值。本发明第三方面提供一种存储介质,所述存储介质上存储有计算机程序,所述计算机程序被计算机执行时实现设计同时检测两种以上长链rna的特异性探针的方法。本发明第四方面提供一种服务终端,所述服务终端包括处理器和存储器;所述存储器用于存储计算机程序,所述处理器用于执行所述存储器存储的计算机程序,以使所述服务终端执行时实现设计同时检测两种以上长链rna的特异性探针的方法。如上所述,本发明的一种长链rna特异性探针及其设计方法和基因芯片,具有以下有益效果:1)通过特异性设计的探针和基因芯片,利用基因芯片的杂交原理,无需线性rna去除,即能够特异性杂交捕获待检测长链rna;2)同时对近8万多个环状rna、7万多个长链非编码rna、2万个mrna同时进行检测,实现了对微量样本的环状rna、长链非编码rna等调控性rna分子与表达蛋白的mrna表达丰度的同时、快速、高通量检测。3)使基因芯片技术更好的应用于长链rna表达谱分析,弥补了现有转录组各种rna不能同时检测技术的不足,也为转录调控网络研究提供技术方法。附图说明图1显示为现有技术中5种circrna形成模型,其中,a图为套锁驱动成环或者外显子跳跃成环模型;b图为配对内含子驱动成环或者直接反向拼接成环模型;c图为cirnas形成模型;d图为依赖于rbps环化模型;e图为可变环化模型。图2显示为现有技术中根据长链非编码rna在基因组上的位置关系进行分类的六种长链非编码rna示意图,其中a为基因间长链非编码rna,b为内含子长链非编码rna,c为双向长链非编码rna,d为增强子长链非编码rna,e为同义长链非编码rna,f为反义长链非编码rna。图3显示为现有技术中长链非编码rna的六种调控机制示意图。图4显示为本发明长链rna特异性探针的整体设计流程图。图5显示为本发明长链rna探针的设计示意图。图6显示为本发明设计长链rna特异性探针的系统示意图。图7显示为本发明设计长链rna特异性探针的服务终端示意图。图8显示为本发明芯片探针表达值与探针灵敏度、特异性定量pcr验证相对表达值图。图9显示为本发明实施例的基因芯片检测的各种长链rna信号值统计图。图10显示为本发明实施例的检测长链rna表达丰度的基因芯片扫描图。图6和图7中元件标号说明1设计模块2比对模块3筛选模块4迭代模块5处理器6存储器具体实施方式本发明中,术语“长链rna”,包括mrna、长链非编码rna和circrna等。术语“探针”指是一段序列已知的、与目的基因互补的dna或rna核酸序列。术语“特异性探针”是指当同时检测多种长链rna时,各种长链rna的探针均无互相干扰、特异性强的探针。术语“全长靶序列”,是指探针能够识别的片段所在的rna的全长序列,序列数据库中给出的rna的序列一般为dna的有义链序列。术语“相似性”,dna序列是由a、t、c、g四种碱基排列组合而成,使用现有的各种打分方案(例如匹配打分矩阵)对两序列碱基的相似程度打分,所得分值即相似性,代表其相似的程度。术语“最长转录本”,转录本是基因通过转录形成的可供编码蛋白质的成熟的mrna,由于mrna形成时有不同的剪接方式,造成一个基因可能有多个转录本,其中序列最长的转录本即为该基因的最长转录本。本发明的一个实施例提供了一种用于同时检测两种以上长链rna的特异性探针的设计方法,包括下列步骤:s100根据预设探针种类设计目标基因的探针,作为候选探针,所述预设探针种类选自;mrna探针、环状rna探针或长链非编码rna探针中的至少两种探针;s200将候选探针序列与所有探针的全长靶序列进行比对;s300若一候选探针的比对结果符合预设值,则保留该候选探针保留作为特异性探针;若一候选探针的比对结果不符合预设值,则淘汰该候选探针。s400若一目标基因无保留的特异性探针,则根据预设的探针种类,重新设计该目标基因的探针作为候选探针,并继续执行步骤s200。若一目标基因保留的特异性探针中,仅包括预设探针种类中的部分种类,则重新设计该目标基因其余预设探针种类作为候选探针,并继续执行步骤s200,直至所有目标基因保留的特异性探针中均包括预设种类的探针;具体的,步骤s100中,预设探针种类可以是mrna探针和环状rna探针两种,也可以为mrna探针和长链非编码rna探针两种,还可以为环状rna探针和长链非编码rna探针两种,更可以为mrna探针、环状rna探针和长链非编码rna探针三种。在如图4所示的较佳实施方式中,预设探针种类为mrna探针、环状rna探针和长链非编码rna探针三种。具体的,步骤s300中:若一候选探针与所有探针的全长靶序列的相似性均不超过第一预设值,且与所有探针的全长靶序列之间连续相同的碱基长度均不超过第二预设值,则认定该候选探针与所述探针的全长靶序列的比对结果符合预设值。若一候选探针与所有探针的全长靶序列中的至少一个靶序列的相似性超过第一预设值,或与所有探针的全长靶序列中的至少一个靶序列之间连续相同的碱基长度超过第二预设值,则认定该候选探针与所述探针的全长靶序列的比对结果不符合预设值。对于不符合预设值的判断,可以是在将一候选探针与所有探针的全长靶序列都比对完后确定;也可以在发现第一个相似性超过第一预设值的全长靶序列或发现第一个与候选探针之间连续相同的碱基长度超过第二预设值的全长靶序列时即中止比对,确定为不符合预设值。进一步的,所述第一预设值和第二预设值会根据不同的比对程序发生变化,设计初期需要实验验证预设值的设置是否合理,从而保证该预设值下筛选出的探针的特异性,例如利用bedtools线下比对程序进行比对,所述第一预设值可以是不超过75%,第二预设值可以是不超过15。在如图4所示的较佳实施例中,s100中用于设计所述mrna探针的序列选自各目标基因的mrna最长转录本序列。可采用现有技术确认目标基因的序列,例如目标基因序列可以来源于genbank数据库。可采用现有技术确认目标基因的最长转录本,例如最长转录本可以来源于refseq数据库。进一步的,如图5所示,选择与目标基因的最长转录本的结合位点位于该最长转录本的3’端的mrna探针,作为目标基因的mrna候选探针。所述最长转录本的3’端通常指距离3’端第一个碱基300个碱基之内的片段。待测样本由3’端开始反转录,mrna探针在于3’端,能够提高检测的灵敏度。在鉴别反义长链非编码rna或同义长链非编码rna和mrna的非重叠区的片段时,使用候选基因最长转录本作为目的基因的全长靶序列,能够更准确地鉴定。进一步的,s100中用于设计所述环状rna探针的序列选自:该环状rna的反向剪接序列的片段。所述反向剪接序列是指将环状rna线性序列的剪接供体外显子的5’端与剪接受体外显子的3’端首尾相连成环形成的序列。可以利用现有技术获得所述环状rna线性序列,例如从来源于circbase、circpedia多数据库的序列,根据序列和染色体位置去除冗余后得到。去除冗余序列可以用现有的软件,例如可以用bedtools。进一步的,如图5所示,s100中,选择与目标基因的反向剪接序列的结合位点位于反向剪接位点的环状rna探针,作为目标基因的环状rna候选探针。所述反向剪接位点为环状rna与相应线性rna唯一不同的区域,它是由剪接供体外显子的5’端与剪接受体外显子的3’端首尾相连产生。根据环状rna特殊的剪接模式——反向剪接,因此其反向剪接序列具有特异的反向剪接位点(backsplicing),而线性rna不具有此位点,因此将环状rna探针设计在反向剪接位点,该探针能够特异性检测样本中的环状rna。进一步的,由于长链非编码rna具有不同的类型,不同类型的长链非编码rna在设计探针时的规则不同,因此需要根据长链非编码rna的类型设计探针。在如图4所示的较佳实施例中,s100中,用于设计反义长链非编码rna或同义长链非编码rna探针的序列选自:反义长链非编码rna或同义长链非编码rna和mrna的非重叠区的片段;用于设计内含子长链非编码rna、基因间长链非编码rna、双向长链非编码rna或增强子长链非编码rna探针的序列选自:各目标基因最长的长链非编码rna的片段。选择以上用于设计长链非编码rna探针的序列的原因是:避免长链非编码rna的探针检测到同基因的mrna。长链非编码rna的转录本序列可以从来源于ensembl、ncbi、ucsc、genecode、noncode等多数据库的序列,根据序列和染色体位置去除冗余后获得。s100中可以使用现有的探针设计软件进行探针设计,例如可以用agilent公司专业基因芯片探针设计软件earray。具体的,在如图4所示的较佳实施例中,所述特异性探针的设计方法可以设计用于同时检测mrna、环状rna和长链非编码rna的特异性探针。设计方法如下:将利用前述方法得到的mrna最长转录本、环状rna反向剪接序列以及用于设计长链非编码rna探针的序列合并成一个文件后导入earray软件,根据现有探针的设计原则例如gc比例、退火温度等条件设置相应的参数后进行探针设计,为保证设计出的所有探针拥有相同的杂交温度,探针的设计参数一旦设定后,直到同一实验中用到的所有探针设计完成前,设计参数不能随意更改。进一步的,由于基因组的复杂性,避免探针杂交多个靶序列,需要利用迭代检测进行特异性筛选:即将每个探针与所有探针的全长靶序列进行比对,根据是否符合预设值即是否符合每个候选探针与所有探针的全长靶序列相似性大小以及与所有探针的全长靶序列之间连续相同的碱基长度进行特异性筛选。根据设置的特异性筛选条件:相似性不超过75%,且与所述全长靶序列之间连续相同的碱基长度不超过15个,候选探针满足条件即视为特异性高,将候选探针保留作为特异性探针,候选探针不满足条件即视为特异性较差,放弃该候选探针。再判断是否有保留特异性探针,若无保留的特异性探针,则返回重新设计探针。若有保留的特异性探针,则继续判断保留的特异性探针中是否每个目标基因都具有mrna特异性探针、长链非编码rna特异性探针、环状rna特异性探针,如果是,则此目标基因的特异性探针设计完毕,如果否,则继续设计其余种类的探针。进一步的,利用软件设计探针时,针对各目标基因的每种探针种类,可以设计一个探针,也可以设计多个探针作为候选探针。设计一个探针时,如果探针满足特异性筛选的条件,则直接作为特异性探针。如果探针不满足特异性筛选的条件,则重新设计探针。重新设计探针时,可以将软件产生探针的个数设置为多个,其他参数不变。软件在满足设置参数的条件下随机产生多个探针后,将多个探针再与所有探针(包括新设计的探针及已筛选出的特异性探针)的全长靶序列进行比对,将满足特异性筛选的探针保存为特异性探针,不满足特异性筛选的探针不被保存。实验时可选择特异性探针中的任一。如果本次还未产生特异性探针,则再次重新设计探针,直到产生特异性探针为止。基于现有探针设计软件,在针对同一序列设计探针时,会根据要求输出的探针数,在诸多符合参数要求的探针中随机输出探针,因此重新设计探针时,即使采用同样的序列与设计参数,也能获得不同的探针序列。进一步的,mrna、环状rna或长链非编码rna的特异性探针的长度为50-70nt。例如可以为55nt、60nt、65nt、70nt。进一步的,所述特异性探针的设计方法设计出的mrna特异性探针、环状rna特异性探针或长链非编码rna特异性探针中的两种以上集成至基因芯片中,所述基因芯片即可以同时检测mrna、环状rna或长链非编码rna中的任意两种或三种长链rna。如图6所示的较佳实施例中,提供了一种设计长链rna特异性探针的系统,所述系统可以用于设计同时检测mrna、环状rna或长链非编码rna中至少两种长链rna的特异性探针。所述系统包括:设计模块1,用于根据预设探针种类设计目标基因的探针,作为候选探针,所述预设探针种类选自;mrna探针、环状rna探针或长链非编码rna探针中的至少两种探针;比对模块2,用于将候选探针序列与所有探针的全长靶序列进行比对;筛选模块3,用于判断候选探针与所有探针的全长靶序列的比对结果是否符合预设值,若符合,则保留该候选探针保留作为特异性探针;若不符合,则淘汰该候选探针。迭代模块4:用于判断各目标基因有无保留的特异性探针,若无,则根据预设的探针种类,重新设计该目标基因的探针作为候选探针,并继续执行比对模块2。若各目标基因有保留的特异性探针,且保留的特异性探针中仅包括预设探针种类中的部分种类,则重新设计该目标基因其余预设探针种类作为候选探针,并继续执行比对模块2,直至所有目标基因保留的特异性探针中均包括预设种类的探针。具体的,设计模块1中,预设探针种类可以是mrna探针和环状rna探针两种,也可以为mrna探针和长链非编码rna探针两种,还可以为环状rna探针和长链非编码rna探针两种,更可以为mrna探针、环状rna探针和长链非编码rna探针三种。进一步的,用于设计所述mrna探针的序列选自各目标基因的mrna最长转录本序列。更进一步的,选自与目标基因的最长转录本的结合位点位于该最长转录本的3’端的片段,作为目标基因的mrna候选探针。进一步的,用于设计所述环状rna探针的序列选自:选自该环状rna的反向剪接序列的片段。更进一步的,选择与目标基因的反向剪接序列的结合位点位于反向剪接位点的环状rna序列,作为目标基因的环状rna候选探针。进一步的,用于设计反义长链非编码rna或同义长链非编码rna探针的序列选自:目标基因中与反义长链非编码rna或同义长链非编码rna和mrna的非重叠区结合的片段;用于设计内含子长链非编码rna、基因间长链非编码rna、双向长链非编码rna或增强子长链非编码rna探针的序列选自:各目标基因中与长链非编码rna最长的序列结合的片段。具体的,筛选模块3中,若一候选探针与所有探针的全长靶序列的相似性均不超过第一预设值,且与所有探针的全长靶序列之间连续相同的碱基长度均不超过第二预设值,则认定该候选探针与所述探针的全长靶序列的比对结果符合预设值。若一候选探针与所有探针的全长靶序列中的至少一个靶序列的相似性超过第一预设值,或与所有探针的全长靶序列中的至少一个靶序列之间连续相同的碱基长度超过第二预设值,则认定该候选探针与所述探针的全长靶序列的比对结果不符合预设值。进一步的,所述第一预设值和第二预设值会根据不同的比对程序发生变化,设计初期需要实验验证预设值的设置是否合理,从而保证该预设值下筛选出的探针的特异性,例如所述第一预设值可以是不超过75%,第二预设值可以是不超过15。将候选探针执行筛选模块和迭代模块,是为了增加探针与探针之间、探针与目的rna之间的特异性。经过筛选和迭代以后得到的特异性探针,可使得环状rna检测探针与长链非编码rna、mrna的检测探针同时具有高度特异性和灵敏度。需要说明的是,应理解以上装置的各个模块的划分仅仅是一种逻辑功能的划分,实际实现时可以全部或部分集成到一个物理实体上,也可以物理上分开。且这些模块可以全部以软件通过处理元件调用的形式实现;也可以全部以硬件的形式实现;还可以部分模块通过处理元件调用软件的形式实现,部分模块通过硬件的形式实现。例如,x模块可以为单独设立的处理元件,也可以集成在上述装置的某一个芯片中实现,此外,也可以以程序代码的形式存储于上述装置的存储器中,由上述装置的某一个处理元件调用并执行以上x模块的功能。其它模块的实现与之类似。此外这些模块全部或部分可以集成在一起,也可以独立实现。这里所述的处理元件可以是一种集成电路,具有信号的处理能力。在实现过程中,上述方法的各步骤或以上各个模块可以通过处理器元件中的硬件的集成逻辑电路或者软件形式的指令完成。本发明的又一实施例提供了一种存储介质,所述存储介质上存储有计算机程序,所述计算机程序被计算机执行时实现设计同时检测两种以上长链rna的特异性探针的方法。进一步的,所述存储介质包括:rom、ram、磁碟、u盘、存储卡或者光盘等各种可以存储程序代码的介质。如图8所示,本发明的又一实施例提供了一种服务终端,所述服务终端包括处理器5和存储器6;所述存储器6用于存储计算机程序,所述处理器5用于执行所述存储器6存储的计算机程序,以使所述服务终端执行时实现设计同时检测两种以上长链rna的特异性探针的方法。所述存储器6用于存储计算机程序。优选地,所述存储器6包括:rom、ram、磁碟、u盘、存储卡或者光盘等各种可以存储程序代码的介质。所述处理器5与所述存储器6相连,用于执行所述存储器6存储的计算机程序,以使所述服务终端执行上述的设计方法。优选地,所述处理器5可以是通用处理器,包括中央处理器(centralprocessingunit,简称cpu)、网络处理器(networkprocessor,简称np)等;还可以是数字信号处理器(digitalsignalprocessor,简称dsp)、专用集成电路(applicationspecificintegratedcircuit,简称asic)、现场可编程门阵列(fieldprogrammablegatearray,简称fpga)或者其他可编程逻辑器件、分立门或者晶体管逻辑器件、分立硬件组件。以下以esr2基因为例,设计用于同时检测esr2基因的mrna、长链非编码rna和环状rna的特异性探针,即预设探针种类为mrna特异性探针、长链非编码rna特异性探针和环状rna特异性探针三种。s100:genbank中找到esr2基因序列(nm__001214902),refseq数据库中找到esr2基因的最长转录本(序列号为nm__001214902),根据检测esr2基因序列设计mrna的探针作为候选探针,序列如下:ataaaagagttttgggaatacactgagctttgagtgaaagaagctgcagtggcctccctg(seqidno:1)ensembl数据库中找到与esr2基因为同向转录也有部分外显子重叠的长链非编码rnaenst00000359491的探针,作为候选探针,序列如下:atacctgagcaagtgaaattaagaagggaattgaagcaaatattcctgacatccaagtgg(seqidno:2)circbase数据库中找到esr2基因来源的环状rnahsa_circ_0102409,它是由esr2基因的第7个外显子到第12个外显子首尾反向拼接而成的。依据该环状rna5’端序列(seqidno:3)和3’端序列(seqidno:4)的特点,设计出涵盖该拼接位点的探针,作为候选探针,序列如下(seqidno:5):ggatgaggggaaatgcgtagaaggaattctggaaatctttgacatgctcctggcaactacttcaaggtttcgagagttaaaactccaacacaaagaatatctctgtgtcaaggccatgatcctgctcaattcca(seqidno:3)ccattatacttgcccacgaatctttgagaacattataatgacctttgtgcctcttcttgcaaggtgttttctcagctgttatctcaagacatggatataaaaaactcaccatctagccttaattctccttcctcctacaactgcagtcaatccatcttacccctggagcacggctccatatacataccttcctcctatgtagacagccaccatgaatatccagccatgacattctatagccctgctgtgatgaattacagcattcccagcaatgtcactaacttggaaggtgggcctggtcggcagaccacaagcccaaatgtgttgtggccaacacctgggcacctttctcctttagtggtccatcgccagttatcacatctgtatgcggaacctcaaaagagtccctggtgtgaagcaagatcgctagaacacaccttacctgtaaacag(seqidno:4)gccatgatcctgctcaattccaccattatacttgcccacgaatctttgagaacattataa(seqidno:5)该探针可特异性检测环状rnahsa_circ_0102409。s200:将上述候选探针与所有探针的全长靶序列进行比对。s300:比对结果均符合预设值,保留为特异性探针。s400:特异性探针包括所有预设探针种类,完成该基因的特异性探针设计。基因芯片中检测其他目的基因的mrna、环状rna或长链非编码rna的特异性探针设计方法同esr2基因。验证特异性探针的特异性、灵敏度并合成芯片为验证经筛选、迭代检测后探针的灵敏度、特异性,选择thbs1基因,其内含子来源的长链非编码rnaenst00000478845,及其第2个外显子到第7个外显子环化形成的环状rnahsa_circ_0034426进行了定量pcr验证,结果如图7所示,thbs1基因的3个长链rna相对于对照组的变化倍数在芯片中和定量pcr表达变化趋势一致。定量pcr实验步骤如下:(一)cdna第一链合成1.从-80℃冰箱中取出rna,在4℃下解冻,然后在0.2mlpcr管中配制反应体系如下:2.将pcr管置于37℃孵育15min,98℃变性5min,4℃保温。(二)sybrgreenqpcr1.在1.5ml离心管中配置反应混合液(384孔板):2.将pcr管子置于pcr仪中进行反应,50℃孵育2min,然后95℃,10min;接着进行40个循环:95℃,15秒;60℃,1min,最后添加溶解曲线。(三)引物序列如下:thbs1上游引物:gaacgggacaactgccagta(seqidno:6)下游引物:acctacagcgagtccaggat(seqidno:7)enst00000478845上游引物:tcgcgcattcttggaagtct(seqidno:8)下游引物:tgccagagggtgaaaagcaa(seqidno:9)hsa_circ_0034426上游引物:ctgcaaaaaggtgtcctgcc(seqidno:10)下游引物:tcaggaacaggacgcctagt(seqidno:11)验证完成后,委托agilent公司利用喷墨打印化学原位合成技术,在严格质控条件下定制出长链rna基因表达丰度高通量检测芯片。检测长链rna表达丰度实验操作具体步骤如下:1.提取、纯化样本总rnatrizol抽提样本总rna,再用qiagenkit(货号74106)纯化总rna,详细操作方法如下(参见rneasyminiprotocol):1)取总rna≤100μg溶解于100μlrnasefree(无rna酶)的水中,加入350μl缓冲液rlt并充分混匀。2)加入250μl无水乙醇,用加样枪的枪头充分混匀。3)将共计700μl含总rna的溶液转入套在2ml离心管内的rneasy柱子内,13200rpm离心15秒,弃去滤过液。4)吸取350μl缓冲液rw1到rneasymini柱子内,13200rpm离心15秒,弃去滤过液。5)将10μl的dnaseⅰ加入到70μl的缓冲液rdd中,混匀,加入到柱子内,室温放置15min。6)吸取350μl缓冲液rw1到rneasymini柱子内,13200rpm离心15秒,弃去滤过液。7)吸取500μl缓冲液rpe到rneasymini柱子内,13200rpm离心15秒,弃去滤过液,重复本步骤一次。8)换新的套管,13200rpm,2min。并将柱子转入到洗脱管中。9)将rneasymini柱子转入收集管中。10)吸取30μlrnasefree水,静置1min,13200rpm离心1min。11)重新将洗脱管中的30ul样品转回柱子,静置1min,13200rpm离心1min。12)nanodrop(nanodropnd-1000uv-visspectrophotometer)测得rna浓度和a260/280。2.对rna进行线性扩增并标记荧光cy31)准备单标的spike-in(rnaspike-inkit,one-color,agilent5188-5282)。按照不同的rna起始量,用稀释缓冲液稀释spike-in,如表1所示:表1rnaspike-in2)反转录:配制组成如表2所示的反应溶液:表2反转录反应溶液组成总rna10-200ng1.5μl稀释好的one-dyespikein2.0μlt7promoterprimer0.8μl无核酸酶水(whitecap)1.0μl总体积5.3μlpcr仪(mjptc-100)上65℃保温10min,冰浴5min。同时将5xfirststrandbuffer在80℃预热3min,室温备用。配置反转录混合溶液,具体组成如表3所示:表3反转录混合溶液组成5xfirststrandbuffer2.0μl0.1mdtt1.0μl10mmdntpmix0.5μlaffinityscriptrnaseblockmix1.2μl总体积4.7μl将上述4.7μl反转录混合溶液加入变性后的冰浴的rna中,混匀,离心,进行pcr反应。pcr反应条件:40℃反应2小时;70℃灭活15分钟;4℃反应5分钟。3)荧光标记配制荧光标记混合溶液(lowinputquickamplabelingkit,one-color,agilent5190-2305),具体组成如表4所示:表4荧光标记混合溶液组成无核酸酶水0.75μl5*转录缓冲液3.2μl0.1mdtt0.6μlntpmix1.0μlt7rna聚合酶混合物0.21μlcy3-ctp0.24μl总体积6.0μl加入上述6.0μl荧光标记混合溶液,混匀,离心,进行pcr反应,得荧光标记产物。pcr反应条件:40℃反应2小时;4℃反应5分钟。4)荧光标记产物纯化a)加84μl的无核酸酶水,总体积至100μl。b)加350μl的rlt,混匀。c)加250μl的无水乙醇,混匀,不要离心。d)将700μl的mix,转到柱子上。13000rpm,4℃离心30sec。丢弃流过液。e)加500μl的rpe,13000rpm,4℃离心30秒。丢弃流过液。f)另加500μl的rpe,13000rpm,4℃离心60秒。丢弃流过液。g)换新的套管,13000rpm,4℃空转30秒,并将柱子转入到洗脱管中。h)加30μl的无核酸酶水,静置1min,13000rpm,4℃离心30秒。i)重新将洗脱管中的30μl样品转回柱子,静置1min,13000rpm,4℃离心30秒。j)用nanodrop测得rna浓度,cy3浓度,a260/280。荧光标记产物纯化所用探针量的要求如表5所示:表5荧光标记产物纯化所用的探针量1×芯片crna>5μgcy3>6pmol/μg2×芯片crna>3.75μgcy3>6pmol/μg4×芯片crna>1.65μgcy3>6pmol/μg5×芯片crna>0.825μgcy3>6pmol/μg3.基因芯片杂交纯化后的荧光标记产物与环状rna基因芯片上的探针利用碱基互补杂交原理进行杂交。杂交试剂盒采用geneexpressionhybridizationkit(agilent5188-5242)。具体步骤如下:1)按表6配片段化混合溶液:表6片段化混合溶液组成组成成分1x2x4x8xcy3-crna5μg3.75μg1.65μg600ng10x阻断剂50μl25μl11μl5μl无核酸酶水补至240μl补至120μl补至52.8μl补至24μl25x片段化缓冲液10μl5μl2.2μl1μl总体积250μl125μl55μl25μl2)60℃保温30min,然后冰浴1min,短暂离心。3)加等体积的2xgex杂交缓冲液hi-rpm,如表7所示,混匀。表7杂交混合溶液组成组成成分1x2x4x8x片段化混合溶液中的crna250μl125μl55μl25μl2xgex杂交缓冲液hi-rpm250μl125μl55μl25μl4)13000rpm,离心1min,然后置于冰上。5)将杂交仓(agilentg2534a)放在水平桌面上,放上带垫圈盖玻片,按表8所示体积加入样品:表8杂交加样体积组成成分1x2x4x8x制备体积500μl250μl110μl50μl杂交体积490μl240μl100μl40μl6)将基因芯片上带有“agilent”面朝下,盖到盖玻片上,迅速组装好杂交仓,在杂交炉(agilentg2545a)中,65℃,10rpm,杂交17h。4.基因芯片洗涤、扫描1)洗液1和洗液2加入2ml的10%tritonx-102,洗液2需要37℃预热过夜。2)将已经完成杂交的基因芯片从杂交炉中取出,拆开杂交仓,按表9中的步骤1-3洗涤基因芯片:表9基因芯片洗涤步骤操作步骤洗液成分温度洗涤时间拆片ge洗液1室温-洗液1洗涤ge洗液1室温1min洗液2洗涤ge洗液237℃1min表9中的ge洗液1、ge洗液2来自geneexpressionwashbufferkit(基因表达洗涤缓冲试剂盒,品牌:安捷伦,货号:5188-5327)3)将洗好的基因芯片装入片夹中,放入扫描仪(agilentmicroarrayscannerg2565ca)进行扫描。扫描参数如表10所示:表10基因芯片扫描参数5.数据分析原始数据用r软件中limma包归一化后,采用fold-change(表达差异倍数)以及t检验(student'st-test)统计学方法对长链rna的表达丰度、差异表达的rna进行分析。实验组和对照组中,检测到的mrna、长链非编码rna、环状rna的表达丰度如图9和图10所示。以前列腺癌肿瘤(实验组)及癌旁(对照组)为例,经过基因芯片筛选及数据分析后,筛选得到实验组相对于对照组差异2倍上调的mrnas有328个,差异2倍下调的mrna有892个,总共筛选到2倍差异表达mrna1220个。差异2倍上调的长链非编码rna有447个,差异2倍下调的长链非编码rna有840个,总共筛选到2倍差异表达长链非编码rna1287个。差异2倍上调的环状rna有508个,差异2倍下调的环状rna有1706个,总共筛选到2倍差异表达环状基因2214个。综上所述,本发明有效克服了现有技术中的种种缺点而具高度产业利用价值。上述实施例仅例示性说明本发明的原理及其功效,而非用于限制本发明。任何熟悉此技术的人士皆可在不违背本发明的精神及范畴下,对上述实施例进行修饰或改变。因此,举凡所属
技术领域:
中具有通常知识者在未脱离本发明所揭示的精神与技术思想下所完成的一切等效修饰或改变,仍应由本发明的权利要求所涵盖。序列表<110>上海生物芯片有限公司<120>一种长链rna特异性探针的设计方法<160>11<170>siposequencelisting1.0<210>1<211>60<212>dna<213>人工序列(artificialsequence)<400>1ataaaagagttttgggaatacactgagctttgagtgaaagaagctgcagtggcctccctg60<210>2<211>60<212>dna<213>人工序列(artificialsequence)<400>2atacctgagcaagtgaaattaagaagggaattgaagcaaatattcctgacatccaagtgg60<210>3<211>134<212>dna<213>人工序列(artificialsequence)<400>3ggatgaggggaaatgcgtagaaggaattctggaaatctttgacatgctcctggcaactac60ttcaaggtttcgagagttaaaactccaacacaaagaatatctctgtgtcaaggccatgat120cctgctcaattcca134<210>4<211>452<212>dna<213>人工序列(artificialsequence)<400>4ccattatacttgcccacgaatctttgagaacattataatgacctttgtgcctcttcttgc60aaggtgttttctcagctgttatctcaagacatggatataaaaaactcaccatctagcctt120aattctccttcctcctacaactgcagtcaatccatcttacccctggagcacggctccata180tacataccttcctcctatgtagacagccaccatgaatatccagccatgacattctatagc240cctgctgtgatgaattacagcattcccagcaatgtcactaacttggaaggtgggcctggt300cggcagaccacaagcccaaatgtgttgtggccaacacctgggcacctttctcctttagtg360gtccatcgccagttatcacatctgtatgcggaacctcaaaagagtccctggtgtgaagca420agatcgctagaacacaccttacctgtaaacag452<210>5<211>60<212>dna<213>人工序列(artificialsequence)<400>5gccatgatcctgctcaattccaccattatacttgcccacgaatctttgagaacattataa60<210>6<211>20<212>dna<213>人工序列(artificialsequence)<400>6gaacgggacaactgccagta20<210>7<211>20<212>dna<213>人工序列(artificialsequence)<400>7acctacagcgagtccaggat20<210>8<211>20<212>dna<213>人工序列(artificialsequence)<400>8tcgcgcattcttggaagtct20<210>9<211>20<212>dna<213>人工序列(artificialsequence)<400>9tgccagagggtgaaaagcaa20<210>10<211>20<212>dna<213>人工序列(artificialsequence)<400>10ctgcaaaaaggtgtcctgcc20<210>11<211>20<212>dna<213>人工序列(artificialsequence)<400>11tcaggaacaggacgcctagt20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1