基于宏基因组的抗性基因分析方法、装置、介质及终端与流程
1.本发明涉及计算机科学与生物信息学交叉的技术领域,特别是涉及基于宏基因组的抗性基因分析方法、装置、介质及终端。
背景技术:
2.近年来,由于抗生素滥用等因素造成的抗生素耐药性问题受到越来越多的政府及组织的关注。同时,抗生素抗性基因作为一种新兴的环境污染物,存在于土壤、水体、人体等各类环境中并且能够通过水平基因转移从宿主转移到其它生物体当中,在环境中进行传播,造成更严重的污染问题,所以研究环境中抗生素抗性基因的分布等情况成为很多学者的研究热点。
3.宏基因组学作为一种组学技术,范围可以覆盖到整个环境中的微生物,对于研究环境中的抗生素抗性基因是比较适合的技术。但是目前已有的一些面向宏基因组学数据的抗生素抗性基因分析工具,第一是功能不够完善,比如仅仅能够获取环境中的抗生素抗性基因信息,但是却无法知道其对应的物种情况或是可移动性;第二是数据适用性比较单一,只能支持二代测序平台产出的原始数据,或者是只支持质控好的优化数据;第三是无法进行更深入的分析,只能获取到基本的抗生素抗性基因的组成情况,但是其在不同环境处理下有无差异、与其它关注的功能有无关联等这些问题都无法得到进一步了解。这使得对抗生素抗性基因的研究不够方便,需要花费额外的时间去寻找不同的工具实现想要的一些分析。
技术实现要素:
4.鉴于以上所述现有技术的缺点,本发明的目的在于提供基于宏基因组的抗性基因分析方法、装置、介质及终端,面向宏基因组学数据,对其进行抗性基因注释、可移动元件识别、物种分类以及个性化功能标识,并根据研究者实际需求对数据进行进一步的挖掘分析,从而快速、全面地了解抗生素抗性基因在环境中的分布、传播等情况,同时也可获取环境中的物种组成、功能组成等其它情况,并且挖掘了抗生素抗性基因与物种、可移动元件及个性化功能之间的相关性。
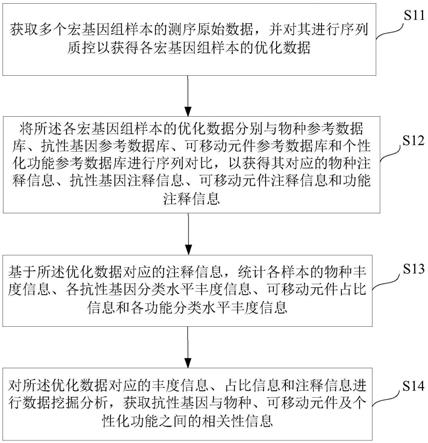
5.为实现上述目的及其它相关目的,本发明的第一方面提供一种基于宏基因组的抗性基因分析方法,包括:获取多个宏基因组样本的测序原始数据,并对其进行序列质控以获得各宏基因组样本的优化数据;将所述各宏基因组样本的优化数据分别与物种参考数据库、抗性基因参考数据库、可移动元件参考数据库和个性化功能参考数据库进行序列对比,以获得其对应的物种注释信息、抗性基因注释信息、可移动元件注释信息和功能注释信息;基于所述优化数据对应的注释信息,统计各样本的物种丰度信息、各抗性基因分类水平丰度信息、可移动元件占比信息和各功能分类水平丰度信息;对所述优化数据对应的丰度信息、占比信息和注释信息进行数据挖掘分析,获取抗性基因与物种、可移动元件及个性化功能之间的相关性信息。
6.于本发明的第一方面的一些实施例中,所述方法还包括:对所述优化数据进行序列组装,以获取其对应的重叠群;对所述重叠群进行基因预测,以获取其对应的开放阅读框;将所述开放阅读框分别与物种参考数据库、抗性基因参考数据库、可移动元件参考数据库和个性化功能参考数据库进行序列对比,以获得其对应的物种注释信息、抗性基因注释信息、可移动元件注释信息和功能注释信息;基于所述开放阅读框对应的注释信息,统计各样本的物种丰度信息、各抗性基因分类水平丰度信息、可移动元件占比信息和各功能分类水平丰度信息;对所述开放阅读框对应的丰度信息、占比信息和注释信息进行数据挖掘分析,获取抗性基因与物种、可移动元件及个性化功能之间的相关性信息。
7.于本发明的第一方面的一些实施例中,所述优化数据的获取方式包括:去除所述测序原始数据中的测序接头或序列尾部的质量值低于预设质量阈值的碱基片段,并去除所述测序原始数据中长度小于预设长度阈值的序列,以获得各宏基因组样本的优化数据。
8.于本发明的第一方面的一些实施例中,所述数据挖掘分析包括:利用r语言获取各个样本的组成分析结果。
9.于本发明的第一方面的一些实施例中,所述数据挖掘分析还包括:对不同样本之间的物种、抗性基因、可移动元件、个性化功能的整体组成进行比较,以获取不同样本之间的物种、抗性基因、可移动元件、个性化功能的差异性数据。
10.于本发明的第一方面的一些实施例中,所述数据挖掘分析包括:利用r语言分析获取抗性基因与物种、可移动元件及个性化功能之间的相关性信息。
11.于本发明的第一方面的一些实施例中,所述方法包括:将所述物种注释信息进行分类,其分类层级包括:界、门、纲、目、科、属、种,以供分析抗生素抗性基因与不同分类层级物种之间的相关性。
12.为实现上述目的及其它相关目的,本发明的第二方面提供一种基于宏基因组的抗性基因分析装置,包括:数据前处理模块,获取多个宏基因组样本的测序原始数据,并对其进行序列质控以获得各宏基因组样本的优化数据;信息注释模块,将所述各宏基因组样本的优化数据分别与物种参考数据库、抗性基因参考数据库、可移动元件参考数据库和个性化功能参考数据库进行序列对比,以获得其对应的物种注释信息、抗性基因注释信息、可移动元件注释信息和功能注释信息;统计模块,基于所述优化数据对应的注释信息,统计各样本的物种丰度信息、各抗性基因分类水平丰度信息、可移动元件占比信息和各功能分类水平丰度信息;数据挖掘分析模块,对所述优化数据对应的丰度信息、占比信息和注释信息进行数据挖掘分析,获取抗性基因与物种、可移动元件及个性化功能之间的相关性信息。
13.为实现上述目的及其它相关目的,本发明的第三方面提供一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现所述基于宏基因组的抗性基因分析方法。
14.为实现上述目的及其它相关目的,本发明的第四方面提供一种电子终端,包括:处理器及存储器;所述存储器用于存储计算机程序,所述处理器用于执行所述存储器存储的计算机程序,以使所述终端执行所述基于宏基因组的抗性基因分析方法。
15.如上所述,本发明的基于宏基因组的抗性基因分析方法、装置、介质及终端,具有以下有益效果:面向宏基因组数据,范围覆盖到整个环境的微生物,尤其适用于对环境中的抗生素抗性基因的研究;并且数据的适应性强,采用了二代或三代测序平台的测序原始数
据;通过对不同样本的物种、抗性基因、可移动元件和个性化功能的组成分析、比较分析和相关性分析,发现抗生素抗性基因在不同环境下的组成、整体组成的差异性,以及抗生素抗性基因与物种、可移动元件以及个性化功能之间的相关性;并且本发明实施例大大提高了对抗生素抗性基因研究的便利性,满足了研究者的多样性需求。
附图说明
16.图1显示为本发明一实施例中一种基于宏基因组的抗性基因分析方法流程示意图。
17.图2显示为本发明一实施例中另一种基于宏基因组的抗性基因分析方法流程示意图。
18.图3显示为本发明一实施例中一种基于宏基因组的抗性基因分析装置结构示意图。
19.图4显示为本发明一实施例中一种电子终端的结构示意图。
具体实施方式
20.以下通过特定的具体实例说明本发明的实施方式,本领域技术人员可由本说明书所揭露的内容轻易地了解本发明的其它优点与功效。本发明还可以通过另外不同的具体实施方式加以实施或应用,本说明书中的各项细节也可以基于不同观点与应用,在没有背离本发明的精神下进行各种修饰或改变。需说明的是,在不冲突的情况下,以下实施例及实施例中的特征可以相互组合。
21.需要说明的是,在下述描述中,参考附图,附图描述了本发明的若干实施例。应当理解,还可使用其它实施例,并且可以在不背离本发明的精神和范围的情况下进行机械组成、结构、电气以及操作上的改变。下面的详细描述不应该被认为是限制性的,并且本发明的实施例的范围仅由公布的专利的权利要求书所限定。这里使用的术语仅是为了描述特定实施例,而并非旨在限制本发明。
22.在本发明中,除非另有明确的规定和限定,对于本领域的普通技术人员而言,可以根据具体情况理解相关术语在本发明中的具体含义。
23.再者,如同在本文中所使用的,单数形式“一”、“一个”和“该”旨在也包括复数形式,除非上下文中有相反的指示。应当进一步理解,术语“包含”、“包括”表明存在所述的特征、操作、元件、组件、项目、种类、和/或组,但不排除一个或多个其它特征、操作、元件、组件、项目、种类、和/或组的存在、出现或添加。此处使用的术语“或”和“和/或”被解释为包括性的,或意味着任一个或任何组合。因此,“a、b或c”或者“a、b和/或c”意味着“以下任一个:a;b;c;a和b;a和c;b和c;a、b和c”。仅当元件、功能或操作的组合在某些方式下内在地互相排斥时,才会出现该定义的例外。
24.本发明的目的在于提供一种基于宏基因组的抗性基因分析方法、装置、介质及终端,面向宏基因组学数据,对其进行抗性基因注释、可移动元件识别、物种分类以及个性化功能标识等分析,从而快速、全面地了解抗生素抗性基因在环境中的分布、传播等情况,同时也可了解环境中的物种组成、功能组成等其它情况,并可根据研究者实际需求对数据进行进一步的挖掘分析,为环境中抗生素抗性基因的研究及风险评估提供了方便的分析手
段。
25.为了使本发明的目的、技术方案及优点更加清楚明白,通过下述实施例并结合附图,对本发明实施例中的技术方案的进一步详细说明。应当理解,此处所描述的具体实施例仅用以解释本发明,并不用于限定发明。
26.实施例一
27.图1显示为本发明实施例的一种基于宏基因组的抗性基因分析方法流程示意图,可具体表述如下:
28.步骤s11.获取多个宏基因组样本的测序原始数据,并对其进行序列质控以获得各宏基因组样本的优化数据。具体的,首先利用二代或三代测序平台获取各个宏基因组样本的测序原始数据,可以是数据格式满足fastq的任何二代或三代测序平台,本实施例对此不作限定,比如illumina、nanopore、ion torrent、pacbio等;然后通过质控软件对各样本的测序原始数据分别进行序列质控,从而获得各样本的优化数据(clean reads),其中,质控软件的选择取决于测序平台类型,可以是任何支持二代或者三代原始数据质控的软件,具体的,二代平台可以选择fastp、fastqc等,三代平台可以选择nanoplot等。
29.在本实施例较佳的实施方式中,所述优化数据的获取方式包括:去除所述测序原始数据中的测序接头或序列尾部的质量值低于预设质量阈值的碱基片段,并去除所述测序原始数据中长度小于预设长度阈值的序列,以获得各宏基因组样本的优化数据。其中,所述预设质量阈值根据不同测序平台设定,比如二代平台质量阈值可设置为20,三代测序平台质量阈值可设置为7;所述预设长度阈值也根据不同测序平台设定,比如二代平台长度阈值可设置为50,三代测序平台长度阈值可设置为2000bp等。
30.步骤s12.将所述各宏基因组样本的优化数据分别与物种参考数据库、抗性基因参考数据库、可移动元件参考数据库和个性化功能参考数据库进行序列对比,以获得其对应的物种注释信息、抗性基因注释信息、可移动元件注释信息和功能注释信息。
31.具体的,可通过软件(比如kraken2、centrifuge、metaphlan2等)将各样本的优化数据(clean reads)与物种参考数据库(比如nr数据库、silva数据库等)进行序列比对,获得每条优化数据对应的物种注释信息;通过序列对比软件(比如blast、diamond等)将各样本的优化数据与抗性基因参考数据库(比如card数据库、ardb数据库等)进行序列比对,获得每条优化数据对应的抗性基因注释信息(包括名称name、类型type、抗性机制resistance mechanism,等等);通过序列对比软件将各样本的优化数据与可移动元件参考数据库(比如isfinder、integrall、iceberg等)进行序列比对,获得每条优化数据对应的可移动元件注释信息(其中,isfinder数据库中的可移动元件注释信息包括:is名称、is编号、is家族等;integrall数据库中的可移动元件注释信息包括:整合子id、基因盒信息等);通过序列对比软件将各样本的优化数据与个性化功能参考数据库(比如bacmet、vfdb等)进行序列比对,获得每条优化数据对应的功能注释信息(以bacmet数据库为例,所述功能注释信息包括基因名称、抗性类型、抵抗的化合物等)。
32.在本实施例较佳的实施方式中,所述方法包括:将所述物种注释信息进行分类,其分类层级包括:界(kingdom)、门(phylum)、纲(class)、目(order)、科(family)、属(genus)、种(species),以供分析抗生素抗性基因与不同分类层级物种之间的相关性。
33.步骤s13.基于所述优化数据对应的注释信息,统计各样本的物种丰度信息、各抗
性基因分类水平丰度信息、可移动元件占比信息和各功能分类水平丰度信息。具体的,基于所述优化数据对应的物种注释信息,利用python等语言编写脚本,进行各样本的物种丰度、相对丰度统计,最终获得总的物种丰度表;基于所述优化数据对应的抗性基因注释信息,利用python等语言编写脚本,进行各样本的不同抗性基因分类水平上的丰度统计(例如:对不同名称、不同类型或不同抗性机制的抗性基因进行丰度统计),最终获得各抗性基因分类水平的丰度表;基于所述优化数据对应的可移动元件注释信息,利用python等语言编写脚本,进行各样本的可移动元件占比统计,最终获得可移动元件占比情况表;基于所述优化数据对应的个性化功能注释信息,利用python等语言编写脚本,进行各样本的不同功能分类水平上的丰度统计,最终获得各功能分类水平上的总丰度表。
34.步骤s14.对所述优化数据对应的丰度信息、占比信息和注释信息进行数据挖掘分析,获取抗性基因与物种、可移动元件及个性化功能之间的相关性信息。具体的,抗性基因与物种、可移动元件及个性化功能之间的相关性可以按照相关方向分为正相关和负相关,也可以分为完全相关、不完全相关和不相关,也可以分为线性相关和非线性相关。
35.在本实施例较佳的实施方式中,利用r语言对物种、抗性基因、可移动元件、个性化功能等因素之间进行相关性分析,从而获取抗性基因与物种、可移动元件及个性化功能之间的相关性信息。可选的,通过gephi等软件以网络图等形式进行可视化展示,还可以以分析结果表、pdf、png图片、气泡图、热图等格式进行相关性分析结果展示。这里通过物种与抗性基因之间的相关性分析进行举例说明:基于各样本中物种分布特征(即物种丰度信息)和各样本中抗性基因分布特征(即各抗性基因分类水平的丰度信息),利用r语言相关性计算模块计算物种与抗性基因之间的相关性(可选用spearman相关系数、pearson相关系数等),并对结果进行显著性检验(可选用false discovery rate,fdr校正法等),从而获得显著相关的物种与抗性基因。
36.在本实施例较佳的实施方式中,所述数据挖掘分析包括:利用r语言获取各个样本的组成分析结果。具体的,基于物种、抗性基因、可移动元件等因素的丰度信息或占比信息,利用r语言对各个样本的组成进行分析,并以特定图形类型如柱状图、条形图、扇形图、气泡图、热图等进行可视化展示,还可以以分析结果表、pdf、png图片等格式进行组成分析结果展示,直观展示出优势物种、优势抗性基因等信息,以及不同物种、抗性基因、可移动元件等在各样本中的整体分布情况。
37.在本实施例较佳的实施方式中,所述数据挖掘分析还包括:对不同样本之间的物种、抗性基因、可移动元件、个性化功能的整体组成进行比较,以获取不同样本之间的物种、抗性基因、可移动元件、个性化功能的差异性数据。比如比较分析不同地点、不同气候下的样本之间的物种、抗性基因、可移动元件等分布的差异性。具体的,可利用主成分分析法(pca)、主坐标分析法(pcoa)等方法来实现,比较结果可以以分析结果表、pdf、png图片、气泡图、热图等形式进行展示。
38.在本实施例较佳的实施方式中,所述数据挖掘分析还包括差异分析(lda effect size,lefse),通过对两个或多个分组的样本进行比较,发现样本之间具有统计学差异的生物标识,一般用lda值分布柱状图、进化分支图或特征展示差异分析结果,从而发现抗生素抗性基因更容易传播的环境或物种。
39.在一些实施方式中,所述方法可应用于控制器,所述电控单元例如为arm
(advanced risc machines)控制器、fpga(field programmable gate array)控制器、soc(system on chip)控制器、dsp(digital signal processing)控制器、或者mcu(micorcontroller unit)控制器等等。在一些实施方式中,所述方法也可应用于包括存储器、存储控制器、一个或多个处理单元(cpu)、外设接口、rf电路、音频电路、扬声器、麦克风、输入/输出(i/o)子系统、显示屏、其它输出或控制设备,以及外部端口等组件的计算机;所述计算机包括但不限于如台式电脑、笔记本电脑、平板电脑、智能手机、智能电视、个人数字助理(personal digital assistant,简称pda)等个人电脑。在另一些实施方式中,所述方法还可应用于服务器,所述服务器可以根据功能、负载等多种因素布置在一个或多个实体服务器上,也可以由分布的或集中的服务器集群构成。
40.综上所述,本实施例提出一种基于宏基因组的抗性基因分析方法,面向宏基因组数据,范围覆盖到整个环境的微生物,尤其适用于对环境中的抗生素抗性基因的研究;并且数据的适应性强,采用了二代或三代测序平台的测序原始数据;通过对不同样本的物种、抗性基因、可移动元件和个性化功能的组成分析、比较分析和相关性分析,发现抗生素抗性基因在不同环境下的组成、整体组成的差异性,以及抗生素抗性基因与物种、可移动元件以及个性化功能之间的相关性;并且本发明实施例大大提高了对抗生素抗性基因研究的便利性,满足了研究者的多样性需求。
41.实施例二
42.图2显示为本发明实施例的另一种基于宏基因组的抗性基因分析方法流程示意图,可具体表述如下:
43.步骤s21.获取多个宏基因组样本的测序原始数据,并对其进行序列质控以获得各宏基因组样本的优化数据。
44.步骤s22.对所述优化数据进行序列组装,以获取其对应的重叠群。可选的,通过软件进行序列比对和序列合并等演算,将短片段的dna建构成为较长的连续序列,获得重叠群(contigs),其格式为fasta。其中,序列组装软件的选择取决于测序平台类型,可以是任何支持二代或者三代序列组装的软件,例如二代平台可以选择megahit等,三代平台可以选择flye、canu等。
45.步骤s23.对所述重叠群进行基因预测,以获取其对应的开放阅读框。其中基因预测又包括同源预测和从头预测,优选从头预测以减少计算资源和时间的消耗。可选的,通过软件如prodigal、metagene等对重叠群contigs进行基因预测,获得开放阅读框(open reading frames,orfs),其格式为fasta。
46.步骤s24.将所述开放阅读框orfs分别与物种参考数据库、抗性基因参考数据库、可移动元件参考数据库和个性化功能参考数据库进行序列对比,以获得其对应的物种注释信息、抗性基因注释信息、可移动元件注释信息和功能注释信息。
47.步骤s25.基于所述开放阅读框orfs对应的注释信息,统计各样本的物种丰度信息、各抗性基因分类水平丰度信息、可移动元件占比信息和各功能分类水平丰度信息。
48.具体的,将各个宏基因组样本的优化数据(clean reads)与开放阅读框(orfs)进行比对(可选用软件soapaligner),获取各样本比对上各orfs的测序序列(reads)数目,即orfs的丰度信息;以orfs为桥梁,将其对应的物种注释信息、抗性基因注释信息和功能注释信息与orfs的丰度信息对应起来,分别统计获得各样本的物种丰度信息、各抗性基因分类
水平丰度信息、各功能分类水平丰度信息。
49.本实施例较佳的实施方式中,所述方法包括:对所述orfs的丰度信息进行标准化处理,将reads数目基于orfs长度及样本总reads数目进行转换,得到orfs的标准化丰度信息,比如tpm、ppm等;然后以orfs为桥梁,将orfs的物种注释信息、抗性基因注释信息和功能注释信息与orfs的标准化丰度信息对应起来,再统计获得各样本的物种丰度信息、各抗性基因分类水平丰度信息、各功能分类水平丰度信息。
50.在一些示例中,所述可移动元件占比信息的获取方式具体包括:将各个宏基因组样本的优化数据(clean reads)与orfs进行比对,获取各样本比对上各orfs的测序序列(reads)数目;以orfs为桥梁,将其对应的可移动元件注释信息与orfs在各样本中的测序序列数对应起来,得到各样本中注释为可移动元件的reads数目,再将其与各样本的总reads数目比较,最终获得可移动元件占比信息。
51.步骤s26.对所述开放阅读框orfs对应的丰度信息、占比信息和注释信息进行数据挖掘分析,获取抗性基因与物种、可移动元件及个性化功能之间的相关性信息。
52.其中,本实施例的步骤s21、步骤s24、s26分别与前述实施例一的步骤s12、s14类似,故此不再赘述。并且,本实施例同时支持对组装过的序列及未经组装的序列进行分析,为使用者提供了更多的选择。
53.实施例三
54.图3显示为本发明实施例的一种基于宏基因组的抗性基因分析装置,包括:数据前处理模块31,用于获取多个宏基因组样本的测序原始数据,并对其进行序列质控以获得各宏基因组样本的优化数据;信息注释模块32,用于将所述各宏基因组样本的优化数据分别与物种参考数据库、抗性基因参考数据库、可移动元件参考数据库和个性化功能参考数据库进行序列对比,以获得其对应的物种注释信息、抗性基因注释信息、可移动元件注释信息和功能注释信息;统计模块33,用于基于所述优化数据对应的注释信息,统计各样本的物种丰度信息、各抗性基因分类水平丰度信息、可移动元件占比信息和各功能分类水平丰度信息;数据挖掘分析模块34,用于对所述优化数据对应的丰度信息、占比信息和注释信息进行数据挖掘分析,获取抗性基因与物种、可移动元件及个性化功能之间的相关性信息。
55.需要说明的是,本实施例提供的各个模块与前文所提供的方法、实施方式类似,故不再赘述。另外需要说明的是,应理解以上装置的各个模块的划分仅仅是一种逻辑功能的划分,实际实现时可以全部或部分集成到一个物理实体上,也可以物理上分开。且这些模块可以全部以软件通过处理元件调用的形式实现;也可以全部以硬件的形式实现;还可以部分模块通过处理元件调用软件的形式实现,部分模块通过硬件的形式实现。例如,信息注释模块可以为单独设立的处理元件,也可以集成在上述装置的某一个芯片中实现,此外,也可以以程序代码的形式存储于上述装置的存储器中,由上述装置的某一个处理元件调用并执行以上信息注释模块的功能。其它模块的实现与之类似。此外这些模块全部或部分可以集成在一起,也可以独立实现。这里所述的处理元件可以是一种集成电路,具有信号的处理能力。在实现过程中,上述方法的各步骤或以上各个模块可以通过处理器元件中的硬件的集成逻辑电路或者软件形式的指令完成。
56.例如,以上这些模块可以是被配置成实施以上方法的一个或多个集成电路,例如:一个或多个特定集成电路(application specific integrated circuit,简称asic),或,
一个或多个微处理器(digital signal processor,简称dsp),或,一个或者多个现场可编程门阵列(field programmable gate array,简称fpga)等。再如,当以上某个模块通过处理元件调度程序代码的形式实现时,该处理元件可以是通用处理器,例如中央处理器(central processing unit,简称cpu)或其它可以调用程序代码的处理器。再如,这些模块可以集成在一起,以片上系统(system-on-a-chip,简称soc)的形式实现。
57.实施例四
58.图4显示为本发明实施例提供的一种电子终端的结构示意图。本实施例提供的电子终端,包括:处理器41、存储器42、通信器43;存储器42通过系统总线与处理器41和通信器43连接并完成相互间的通信,存储器42用于存储计算机程序,通信器43用于和其它设备进行通信,处理器41用于运行计算机程序,使电子终端执行如上一种基于宏基因组的抗性基因分析方法的各个步骤。
59.上述提到的系统总线可以是外设部件互连标准(peripheral component interconnect,简称pci)总线或扩展工业标准结构(extended industry standard architecture,简称eisa)总线等。该系统总线可以分为地址总线、数据总线、控制总线等。为便于表示,图中仅用一条粗线表示,但并不表示仅有一根总线或一种类型的总线。通信接口用于实现数据库访问装置与其它设备(例如客户端、读写库和只读库)之间的通信。存储器可能包含随机存取存储器(random access memory,简称ram),也可能还包括非易失性存储器(non-volatile memory),例如至少一个磁盘存储器。
60.上述的处理器可以是通用处理器,包括中央处理器(central processing unit,简称cpu)、网络处理器(network processor,简称np)等;还可以是数字信号处理器(digital signal processing,简称dsp)、专用集成电路(application specific integrated circuit,简称asic)、现场可编程门阵列(field-programmable gate array,简称fpga)或者其它可编程逻辑器件、分立门或者晶体管逻辑器件、分立硬件组件。
61.实施例五
62.本发明实施例提供一种计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行时实现前文所述的基于宏基因组的抗性基因分析方法。
63.本领域普通技术人员可以理解:实现上述各方法实施例的全部或部分步骤可以通过计算机程序相关的硬件来完成。前述的计算机程序可以存储于一计算机可读存储介质中。该程序在执行时,执行包括上述各方法实施例的步骤;而前述的存储介质包括:rom、ram、磁碟或者光盘等各种可以存储程序代码的介质。
64.综上所述,本发明提供提出基于宏基因组的抗性基因分析方法、装置、介质及终端,面向宏基因组数据,范围覆盖到整个环境的微生物,尤其适用于对环境中的抗生素抗性基因的研究;并且数据的适应性强,采用了二代或三代测序平台的测序原始数据;通过对不同样本的物种、抗性基因、可移动元件和个性化功能的组成分析、比较分析和相关性分析,发现抗生素抗性基因在不同环境下的组成、整体组成的差异性,以及抗生素抗性基因与物种、可移动元件以及个性化功能之间的相关性;并且本发明大大提高了对抗生素抗性基因研究的便利性,满足了研究者的多样性需求。因此,本发明有效克服了现有技术中的种种缺点而具高度产业利用价值。
65.上述实施例仅例示性说明本发明的原理及其功效,而非用于限制本发明。任何熟
悉此技术的人士皆可在不违背本发明的精神及范畴下,对上述实施例进行修饰或改变。因此,举凡所属技术领域中具有通常知识者在未脱离本发明所揭示的精神与技术思想下所完成的一切等效修饰或改变,仍应由本发明的权利要求所涵盖。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1