一种基于mRNA碱基的生物亲缘识别方法和系统
一种基于mrna碱基的生物亲缘识别方法和系统
技术领域
1.本发明涉及一种基于mrna碱基的生物亲缘关系识别方法与系统,属于亲缘智能 识别技术领域,特别属于基于碱基进行的生物亲缘智能识别技术领域。
背景技术:
2.mrna,又名信使rna,是由dna的一条链作为模板转录而来的、携带遗传信 息能指导蛋白质合成的一类单链核糖核酸,它将遗传信息从dna传递到核糖体,在 那里作为蛋白质合成模板并决定基因表达蛋白产物肽链的氨基酸序列。以细胞中基因 为模板,依据碱基互补配对原则转录生成mrna后,mrna就含有与dna分子中某 些功能片段相对应的碱基序列,作为蛋白质生物合成的直接模板。如在dna中一样, mrna遗传信息也保存在核苷酸序列中,其被排列成由每个三个碱基对组成的密码子。 每个密码子编码特定氨基酸,但终止密码子例外,因为其终止蛋白质合成。
3.mrna疫苗的研制表明mrna携带着病毒的某些信息,mrna中的遗传信息保 存在核苷酸序列中,不同的核苷酸序列代表着不同的病毒。近些年来,各类流行病毒 的爆发给生产生活造成了极大的不便,威胁着人们的生命健康,造成了很大的经济损 失。经过研究发现,很多病毒来自自然界或是自然界某种已经存在的病毒的变体,甚 至很多病毒之间有很强的相似性。因此病毒之间的亲缘识别成为了本领域中亟待解决 的问题。当前生物学界采用的亲缘识别方法没有考虑mrna的作用,而且步骤繁多, 所用时间久,对使用设备存在较强的依赖性,且人工参与多,人力成本很高。
技术实现要素:
4.针对上述问题,本发明的目的是提供一种基于mrna碱基的生物亲缘关系识别方 法与系统,其无需对数据进行人工标注,节省了人力成本,也避免了人工因素对分类 结果的影响,使用方法简单,程序运行效率高、速度快。
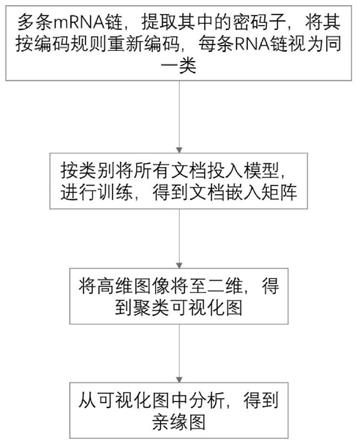
5.为实现上述目的,本发明采取以下技术方案:一种基于mrna碱基的生物亲缘识 别方法,包括以下步骤:s1提取mrna链中的碱基密码子,将其按编码规则重新编 码;s2将经过重新编码的碱基链转化成模型能够识别的文档;s3将文档输入模型进 行碱基文本向量化,并对向量化的碱基文本进行聚类;s4对聚类结果进行可视化显示, 从而获得生物亲缘识别结果。
6.进一步,步骤s1中编码通过两位二级制编码对四种碱基进行表征。
7.进一步,步骤s2中通过内容映射的方式将经过重新编码的碱基链转换为模型能够 识别的文档,文档包括mrna链代表的生物名称和对应的碱基链编码。
8.进一步,步骤s3中将文档输入模型进行碱基文本向量化的方法为:s3.1确定文 档嵌入中最优滑动窗口与模型构建维度两个参数;s3.2对每一个文档的归一化向量输 入模型进行流形学习,并对归一化向量进行降维,将高维矩阵转化为二维向量组,从 而将高维图像降至二维。
提取核苷酸序列,利用计算机方法对多种不同的序列进行分析,通过对不同mrna核 苷酸序列的再编码,将新的编码所形成的文档输入计算机模型,训练神经网络模型, 提取序列所代表的rna生物的特征,使得具有相似性或有着亲缘关系的序列链聚集 在一起,实现在计算机领域的生物亲缘性判断。下面结合附图,通过两个实施例对本 发明的方案进行详细说明。
23.实施例一
24.本实施例公开了一种基于mrna碱基的生物亲缘识别方法,如图1、2所示,包 括以下步骤:
25.s1提取mrna链中的碱基密码子,将其按编码规则重新编码。
26.其具体步骤为:通过生物手段获取多条mrna链,并提取一段mrna链上的碱 基密码子,生成碱基链,同时利用编写好的碱基转码程序将碱基转化为对应的由0和 1组成的可以被计算机识别的编码形式。本实施例中将编码由一位扩展为2位,由“00”、
ꢀ“
01”、“10”、“11”来表征构成rna的四种碱基编码。四类碱基的编码方式为:a(腺 嘌呤)重新编码为“00”,g(鸟嘌呤)重新编码为“01”,c(胞嘧啶)重新编码为“10”, u(尿嘧啶)重新编码为“11”。
27.s2将经过重新编码的碱基链转化成模型能够识别的文档,即将其转化为txt格式 的文档,采用内容映射的方式实现文本化转换。一种生物的mrna信息由多个文档组 成。其中,文本标题,即文本唯一标识码,是mrna链代表的生物名称;文本内容是 对应的碱基链编码,每个文本包含的碱基链长度为120。需要说明的是,此处txt格式 文档以及碱基链的具体长度均为本实施例的优选方案,但不排除其他格式的文档也可 以用于神经网络模型,且碱基链也可以是其他长度。其中,模型优选为神经网络模型, 更优选为无监督的深度学习模型doc2vec,但也不排除其他模型的适用。
28.s3将文档输入模型进行碱基文本向量化,并对向量化的碱基文本进行聚类,通过 引入先验知识和聚类方法来识别碱基所代表的mrna结构。
29.将文档输入模型进行碱基文本向量化的方法为:s3.1确定文档嵌入中最优滑动窗 口与模型构建维度两个参数。
30.步骤s3.1中确定最优滑动窗口与模型构建维度的方法为:构建不同维度下的文档 嵌入模型,得到不同维度下的文档嵌入矩阵,根据矩阵计算每个维度下的模型损失, 使模型损失最小化,先确定窗口,固定维度值为230,从而得到最优窗口;随后根据 损失函数计算模型的噪声绘制在最优窗口下不同维度的模型折线图,从而获得最优的 模型构建维度;通过最优的模型构建维度对最优窗口进行验证。
31.得到最优窗口的具体步骤为:固定窗口或维度;计算文档嵌入矩阵a,遍历窗口 或维度,获得矩阵集合{a};对于矩阵集合{a}中任一个矩阵m1,计算sumdvl= sum(dvl(m1,m
other
)),其中,m
other
为集合{x}内除m1以外的其他矩阵;取sumdvl 最小时的窗口为最优窗口。
32.s3.2对每一个文档的归一化向量输入模型进行流形学习,并对归一化向量进行降 维,将高维矩阵转化为二维向量组,从而将高维图像降至二维。
33.步骤s3.2中对归一化向量进行降维包括以下步骤:寻找高维空间中的数据集a
i
的 映射关系f,根据映射关系f构造低维数据集{y
i
=f(a
i
)},其中,{y
i
}在维度上满足给 定的条件。通过流形学习中非线性的t
‑
sne将高维向量降到二维,得到聚类可视化结 果。非线性
降维方式,在考虑距离的同时也考虑到了映射数据的拓扑结构,对于高维 数据的文档嵌入矩阵,采用非线性降维方式,能够保留向量数据的原始特征,同时对 于得到的维度低的数据可以进行可视化处理。
34.采用控制变量的方法依次确定这两个参数,构建不同维度下的文档嵌入模型,得 到不同维度下的文档嵌入矩阵,根据矩阵计算每个维度下的模型损失。确定最优窗口 后,根据损失函数计算模型的噪声,绘制在固定窗口下不同维度的模型折线图再确认 最优维度。在确定最优维度后,再次对窗口进行验证。
35.在最优维度和最优窗口两个参数确定之后,按类别将所有文档输入模型,进行训 练,对向量进行归一化变换,得到文档嵌入矩阵。
36.s4对聚类结果进行可视化显示,从而获得生物亲缘识别结果。
37.如图3所示,将每个文档的二维向量看做一个散点,绘制成图,得到聚类可视化 结果图,在可视化结果图中若两个散点之间的距离小于阈值,则二者具有亲缘关系, 否则二者不具有亲缘关系。当两类点混成一团时,代表这两类点所代表的mrna碱基 之间存在一定的相似度,进一步可以说明mrna所代表的rna之间存在某种联系, 甚至是亲缘关系;当两个散点聚成的团在坐标系上相差甚远时,说明这两类rna生 物之间没有联系。
38.实施例二
39.基于相同的发明构思,本实施例公开了一种基于mrna碱基的生物亲缘识别系统, 包括:
40.重编码模块,用于提取mrna链中的碱基密码子,将其按编码规则重新编码;
41.转化模块,用于将经过重新编码的碱基链转化成模型能够识别的文档;
42.聚类模块,用于将文档输入模型进行碱基文本向量化,并对向量化的碱基文本进 行聚类;
43.显示模块,用于对聚类结果进行可视化显示,从而获得生物亲缘识别结果。
44.最后应当说明的是:以上实施例仅用以说明本发明的技术方案而非对其限制,尽 管参照上述实施例对本发明进行了详细的说明,所属领域的普通技术人员应当理解: 依然可以对本发明的具体实施方式进行修改或者等同替换,而未脱离本发明精神和范 围的任何修改或者等同替换,其均应涵盖在本发明的权利要求保护范围之内。上述内 容仅为本技术的具体实施方式,但本技术的保护范围并不局限于此,任何熟悉本技术 领域的技术人员在本技术揭露的技术范围内,可轻易想到变化或替换,都应涵盖在本 申请的保护范围之内。因此,本技术的保护范围应以权利要求的保护范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1