参考蛋白质数据库的构建方法、存储介质、电子设备与流程
1.本发明涉及生物技术以及信息技术领域,具体涉及一种参考蛋白质数据库的构建方法,基于蛋白质组学测定snp位点的方法。
背景技术:
2.基于质谱的蛋白质组学已成为全面检测和表征蛋白质的主要方法,在整个生物学和医学领域起重要作用。为了鉴定肽或蛋白质,大多数蛋白质组学实验都依赖于公共通用的蛋白质序列参考数据库,以从公共通用的蛋白质序列参考数据库搜索得出的理论质谱为基准,对实验肽段质谱图进行比较、评分。但是,公共通用的蛋白质序列参考数据库中缺少的蛋白质就难以被检测到,比如尚未发现的未注释的蛋白质或包含单核苷酸多态性(single nucleotide polymorphism,snp)的蛋白质,然而,这些蛋白质却通常具有重要的生物学意义。
3.snp是指在基因组水平上由单个核苷酸变异引起的dna序列多态性,在人类基因组中广泛存在,据研究预计人类基因组中snp总数可达300万甚至更多,是新一代的可用于鉴定的遗传标记。个体特异性的snp可能是疾病的潜在突变,影响药物的个体敏感性或耐受性,也可作为个体身份识别鉴定的标志物。鉴于snp重要的应用价值,目前已经开发出多种方法用于snp位点的检测,主要包括dna分子杂交、引物延伸、等位基因特异的寡核苷酸连接反应、侧翼探针切割反应以及基于这些方法的变通技术。但是,上述对于snp的解析方法都是基于核酸层面。
4.蛋白质序列是基因组dna的外显子经过转录、翻译得到,因而部分dna中的snp会导致对应蛋白中氨基酸序列的改变,这让从单个氨基酸多态性(single amino acid polymorphism,sap)的位点反推snp位点有可靠的理论依据。sap通常是指单个氨基酸突变所造成的肽段的多态性,可遗传变异肽(genetically variant peptides,gvp)是指包括一个或多个氨基酸突变的特异性肽段,通常gvp包含了sap的情形。得益于质谱技术的重大突破和蛋白质分离、富集方法的进步,基于质谱的蛋白质组学研究的灵敏性和准确性都得到了很大的提升,通过蛋白质来鉴定gvp变得可行,从蛋白层面鉴定snp也可以更好地在蛋白质水平上表征这些突变的影响。特别是对一些样品,其核酸成分已经受到了外界因素,如酶等因素的破坏,然而蛋白质在某些场合远比dna更加稳定,甚至经历百年仍然保持其氨基酸的原始序列。对于此类样品,利用蛋白质组学测定snp是唯一的选择。
5.因此,基于蛋白质组学测定snp位点具有很高的现实应用意义。蛋白质基因组学得益于新一代测序(next
‑
generation sequencing,ngs)技术的发展,使用基因组或转录组核苷酸测序数据来创建每个样品定制的蛋白质组学数据库,用以基于质谱(mass spectrometry,ms)的蛋白质组学在定制的蛋白质组学数据库中搜索,以实现对样品中特异的蛋白质包含的变异进行检测。这种由ngs驱动的蛋白质基因组学的策略已越来越多地用于检测和研究人类蛋白质变异,其好处是多方面的,可以帮助描绘基因组中的蛋白编码基因和突变,可以提供蛋白质存在和蛋白质变异的直接证据,可以帮助发现个体特异性的治
疗靶标或生成用于诊断、定制治疗的生物标记物。
6.理论上可以针对每种样品类型利用测序数据自适应地构建最佳蛋白质数据库,尽管这非常有益于全面地检测样品的snp、提高肽段识别的准确性,但一方面会提高实验和分析成本,另一方面在有些场合难以获取到可以提取核酸的样品用来测序。建立通用的包含尽可能多的snp位点信息的蛋白质数据库是对蛋白质基因组学所存在问题的一个解决办法,然而,如果纳入过多的snp位点可能会造成蛋白质数据库的高度冗余性,导致结果中的假阳性过高;如果因此去掉部分难以检测到或不存在的蛋白质序列,尽管可能有一定的改善效果,但也可能有去除掉部分含量低的蛋白质而导致结果有偏差的风险。
技术实现要素:
7.本发明为了解决上述问题,提供了一种参考蛋白质数据库的构建方法,以及一种使用上述参考蛋白质数据库的构建方法得到的参考蛋白质数据库,基于蛋白质组学测定snp位点的方法。本发明通过提取样品中的蛋白质,经过质谱和生物信息学的分析,获取样品提供者尽可能多的snp位点信息,建立一个对于特定样品来说相对全面的参考蛋白质数据库,为减少特定参考蛋白质数据库的冗余度和尽可能多地鉴定样品的snp提供了新的解决方案。
8.本发明的第一方面提供了一种参考蛋白质数据库的构建方法,采用以下技术方案来实现:
9.s2、获取公共数据库中的snp位点信息,对snp位点进行筛选,得到导致编码氨基酸(amino acids,aa)改变的snp位点列表。
10.进一步的,该步骤的具体操作包括以下步骤:
11.s201、获取公共数据库的snp位点信息,下载对应的vcf文件和参考基因组。所述公共数据库可以包括表1中的一个或多个公共数据库。
12.表1
13.frequency,maf)的所有被研究过的突变位点。
18.因此,上述2个数据库具有数据权威性与准确性,且由于上述2个数据库中的数据可以供本发明在后续步骤中进行人群突变频率分析筛选,从而进一步提高了本法明检测查全率与查准率。
19.所述vcf文件的数据记录部分由以空格键分割的多列组成,前八列表示变异位点的相关信息,分别为:染色体名称、变异位点在染色体上的位置、变异位点在已有数据库中的id号(当不存在时可以用“.”表示)、参考碱基、变异碱基、质量得分、是否通过过滤标准、相关信息(例如测序深度);其后每一列表示某个样本在该位点的信息(例如突变频率)。
20.s202、对公共数据库的snp位点信息进行筛选,得到导致编码氨基酸改变的snp位点列表。
21.将s201步骤中获取的vcf文件中的snp位点信息和参考基因组比对分析,得到外显子区域的突变且突变后会导致编码氨基酸有所改变的snp位点列表。
22.更进一步的,挑选vcf文件中snp位点的具体操作为:
23.s2021、使用注释软件,对vcf文件基于参考基因组进行注释,以得到对应vcf文件中所有外显子区域的突变,输出每个snp对应的所属氨基酸原始序列和突变后的序列。
24.s2022、对原始氨基酸序列和突变后对应的氨基酸序列两两比较,挑选出氨基酸序列有改变的snp位点列表。
25.通过s202步骤从海量的snp信息中筛选出来了与氨基酸序列改变有关的snp位点,从而为后续步骤构建蛋白质数据库,以及进一步在蛋白质层面进行数据分析提供了基础。
26.更进一步的,在注释snp时,为了保障数据的可靠性以及权威性,应当使用具有可靠来源的当前最新参考基因组。本发明中针对1000genome的vcf文件使用hg19版本的参考基因组,针对dbsnp的vcf文件使用hg38版本的参考基因组。
27.优选地,为了在保障数据可靠性的前提下减少蛋白质数据库的冗余性而导致结果中的假阳性过高,本法发明还包括以下筛选步骤,即筛选出人群突变频率相对较高的高频突变。
28.优选地,本发明中筛选人群突变频率大于等于预设阈值的snp位点,人群突变频率包括maf或人群频率(allele frequency,af),本发明的预设阈值为0.005至0.5中的任意数。
29.进一步优选地,本发明的预设阈值为0.01,即筛选maf≥0.01或af≥0.01的snp位点。
30.具体而言,在s201获取公共数据库的snp位点信息的步骤中,选择人群突变频率大于预设阈值的数据信息。例如本发明中,以人群突变频率大于等于0.01为标准,优选地选择用dbsnp的common_all_20180418.vcf.gz文件。dbsnp数据库中提供了common_all_20180418.vcf.gz文件和all_20180418.vcf.gz,其中commoncommon_all_20180418.vcf.gz文件包含的snp范围是所有常见人的突变位点清单,即maf≥0.01。经过实验比较,发现在对同一数据使用all_20180418.vcf.gz来建库搜索时一方面会造成数据库的过度冗余,另一方面因为all_20180418.vcf.gz中大部分snp都是低频突变(maf<0.01),在对搜出的肽段进行fdr筛选时会导致fdr=0.01时对应的谱图分数过高。dbsnp因为已经选用了common_all_20180418.vcf.gz文件,无需再对s202步骤筛选出的snp位点列表进行频率的筛选。
31.或者,本发明在步骤s202筛选出的snp位点列表中进一步筛选出人群突变频率相对较高的高频突变。具体而言,例如在选用的1000genome数据库的数据中在vcf文件中添加对应的突变频率,并筛选出af≥0.01的snp,从而保证入选的每个snp有较高的概率被检测到,避免因为一些较低频率的snp导致数据库的过度冗余和假阳性过高。
32.筛选人群突变频率大于等于预设阈值的snp位点使得能够从海量的数据信息中获取蛋白质突变频率较高的信息,这样的高丰度蛋白质信息使得后续建立的gvp参考蛋白质数据库的精度大大提高,从而提升了运行效率以及检索精度。
33.s3、根据样品的蛋白丰度筛选出表达量高的蛋白的snp。
34.为了解决纳入过多的snp位点可能会造成蛋白质数据库的高度冗余性,但随意去掉部分可能难以检测到的蛋白质序列又会导致在蛋白质数据库数据缺失的难点,本发明采用基于样品对snp进行筛选的方式,即根据样品的蛋白丰度筛选出表达量高的蛋白的snp。
35.具体而言,包括步骤:
36.s301、对样品质谱数据进行常规蛋白质组学分析得到蛋白定量信息。首先需要对样品的蛋白进分析。蛋白丰度的挑选依赖于样品的蛋白质组成,先通过对样品的常规蛋白质组学分析得到样品中包含的蛋白列表及其含量。
37.s302、根据蛋白定量信息,计算每个蛋白的总表达量,按表达量进行筛选,以得到基于样品的易被检测到的蛋白列表。筛选可以按蛋白丰度降序排序,挑选排序前n(n为正整数)的蛋白,或者预设蛋白丰度阈值,剔除低于阈值的蛋白质,从而得到基于样品的易被检测到的蛋白列表。具体的n或阈值需要根据实际样品决定。
38.s303、根据所述蛋白列表对snp位点列表进行筛选,仅保留所述蛋白列表中对应蛋白的snp位点信息。
39.由于本法明基于样品进行蛋白质丰度筛选,因此针对特定样品保障了结果可靠性,同时也避免了数据库的过度冗余和假阳性过高。
40.s4、生成挑选出的snp位点列表对应的可遗传变异肽列表,根据公共蛋白质数据库生成基于样品的gvp参考蛋白质数据库。
41.进一步的,s4步骤包括以下步骤:
42.s401、对筛选出的snp位点列表,利用对样品处理所用的酶,对snp位点的原始蛋白质序列和突变后的蛋白质序列进行理论酶切,比较获得两者特异性的肽段,以生成可遗传变异肽(gvp)信息。
43.s402、下载公共蛋白质数据库的参考蛋白质序列,将不在参考蛋白质序列中的gvp添加到参考蛋白质序列中,以生成基于样品的gvp参考蛋白质数据库,所述gvp参考蛋白质数据库包括gvp列表中的所有gvp信息。
44.更进一步的,在判断gvp是否在公共蛋白质数据库中已经存在时,需要将异亮氨酸(氨基酸字母缩写i)替换为亮氨酸(氨基酸字母缩写l),因为两者分子量相等,搜库软件搜索时无法鉴别。
45.优选地,将所述可遗传变异肽汇集为可遗传变异肽列表,所述可遗传变异肽列表中包括所述可遗传变异肽对应的snp位点。
46.需要注意的是本发明的上述步骤的执行顺序并非固定,其中筛选人群突变频率大于等于预设阈值的snp位点的步骤以及步骤s3也可以在步骤s401后执行,即获得gvp列表,
再基于gvp列表进行进一步筛选。
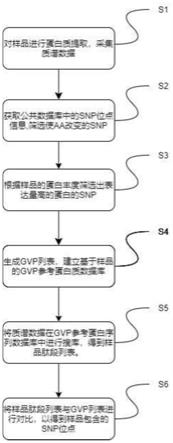
47.本发明的第二方面提供一种参考蛋白质数据库,参考蛋白质数据库通过第一方面提供的参考蛋白质数据库的构建方法构建得到。
48.本发明的第三方面提供一种基于蛋白质组学测定snp位点的方法,使用通过本发明第一方面的参考蛋白质数据库的构建方法构建得到参考蛋白质数据库以及可遗传变异肽列表。所述方法包括下述步骤:
49.s1、对样品进行蛋白质提取,用质谱仪进行蛋白质组学检测,采集质谱数据。
50.s5、将s1步骤获取的质谱数据在本发明第一方面得到的参考蛋白序列数据库中进行搜库,得到匹配的肽段图谱对应的样品肽段列表。
51.进一步的,s5步骤包括:将s4步骤得到的gvp参考蛋白质数据库添加到数据库搜索引擎中,设定参数并将s1得到的质谱文件在gvp参考蛋白质数据库中进行搜库,导出搜库结果,计算错误发现率(false discovery rates,fdr),fdr被用以校正多重比较所致的误差,在拒绝多个零假设时,fdr校正程序能够控制错误拒绝零假设(假阳性)的可能性,来找到合适的结果组合。设置fdr=0.01时对应的谱图的肽段谱图匹配得分为得分阈值,保留得分高于得分阈值的谱图,得到这些谱图对应的肽段列表即为样品肽段列表。
52.更进一步的,在将gvp参考蛋白质数据库添加到数据库搜索引擎之前,先将所有的异亮氨酸(氨基酸字母缩写i)替换为亮氨酸(氨基酸字母缩写l),两者分子量相等,数据库搜索引擎无法鉴别。
53.s6、将样品肽段列表与s4步骤生成的gvp列表进行对比,以所述可遗传变异肽得到所述样品包含的snp位点。
54.进一步的,s6步骤的具体操作为:将s5步骤得到样品肽段列表和s4步骤中的gvp列表进行对比,以得到样品的具体gvp信息,并通过gvp列表中对对应关系反推得到样品包含的snp位点信息。
55.本发明的第四方面提供一种电子设备,包括:处理器;以及存储器,用于存储所述处理器的可执行指令;其中,所述处理器配置为经由执行所述可执行指令来执行本发明的第一方面提供的参考蛋白质数据库的构建方法。
56.本发明的第五方面提供一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现本发明的第一方面提供的参考蛋白质数据库的构建方法。
57.本发明的有益效果是:
58.本发明用蛋白质组学检测样品的gvp反推snp位点的检测方法,可以适用于所有未经测序来基于蛋白质组学进行的snp检测。
59.本发明在无需对样品先测序分析的前提下利用蛋白质组学鉴定人的snp位点信息,节省了测序和分析成本,克服了部分场合中难以获取核酸做测序的情况,可以在法医辅助物证分析鉴定等方面发挥作用。
60.本发明以蛋白质作为snp位点的研究对象,具有灵敏度高、稳定性高、检测通量高的优点。
61.本发明通过样品蛋白质丰度筛选以及理论酶切的方式,基于样品构建gvp参考蛋白质数据库,从而得到基于样品的包含尽可能多的gvp信息的参考蛋白质数据库,避免错误去除蛋白质而产生的误差,提高有效数据完整性,也不会导致纳入过多的遗传变异信息造
成蛋白质数据库的高度冗余性。从而为后续通过参考蛋白质数据进行检索时提高结果准确性,并有效降低了结果假阳性。
62.本发明通过在得到导致编码氨基酸改变的snp位点的步骤中挑选出人群突变频率相对较高的高频突变,从而在保障数据可靠性的同时避免了数据库的过度冗余和假阳性过高,有效提高了运行效率。
63.应当理解的是,以上的一般描述和后文的细节描述仅是示例性和解释性的,并不能限制本公开以及本发明的保护范围。
附图说明
64.为了更清楚地说明本发明实施例中的技术方案,下面用附图/表对本发明的描述作简单地介绍。此处的附图被并入说明书中并构成本说明书的一部分,示出了符合本公开的实施例,并与说明书一起用于解释本公开的原理。显而易见地,下面描述中的附图仅仅是本公开的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
65.图1为实施例2的gvp参考蛋白质数据库的构建方法流程示意图;
66.图2为图1中步骤s2的详细方法流程示意图;
67.图3为图1中步骤s3的详细方法流程示意图;
68.图4为图1中步骤s4的详细方法流程示意图;
69.图5为实施例3的基于蛋白质组学测定snp的方法流程示意图;
70.图6a,6b,6c为实施例3中根据不同标准丰度蛋白进行筛选的snp数量结果示意图;
71.图7为实施例4的电子设备结构示意图;
72.图8为实施例5的计算机可读存储介质示意图。
具体实施方式
73.下面将结合说明书的附图和具体实施例对本发明的技术方案进行更清楚、完整地描述。显然,此处所描述的实施例仅仅是本发明的一部分实施例,而不是全部的实施例。基于本发明的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
74.除非特别说明,具体实施例中提到的试剂、设备均为本领域的常规试剂和设备,本领域技术人员根据现有技术可以得知上述试剂、设备的具体组成以及使用方法。
75.实施例1样品的制备
76.胰蛋白酶肽的制备
77.将105个左右的k562细胞加入含有100mm碳酸氢铵的8m尿素进行裂解,裂解的时间为30分钟。然后,采用600μl浓度为100mm的碳酸氢铵稀释细胞裂解物(将尿素降低至2m),使用dtt(终浓度10mm)还原。加入iaa(终浓度55mm)使蛋白烷基化,并加入100mg胰蛋白酶消化。使用seppak plus柱将肽脱盐,通过真空离心干燥后用0.1%乙酸稀释。每个实验中使用约200ng胰蛋白酶酶切产物肽进行分析。
78.本发明胰蛋白酶肽的制备过程,也可以采用本领域公知的方法进行制备。
79.对上述胰蛋白酶肽进行质谱分析,获取质谱数据文件。这里的质谱分析可以采用
现有的质谱分析的常规方法以及仪器。
80.实施例2基于样品的gvp参考蛋白质数据库的构建方法
81.本实施例基于实施例1中的样品进行gvp参考蛋白质数据库的构建。实施例2的gvp参考蛋白质数据库的构建方法流程示意图,图2为图1中步骤s2的详细方法流程示意图,图3为图1中步骤s3的详细方法流程示意图,图4为图1中步骤s4的详细方法流程示意图。如图1、图2、图3、图4所示,本实施例的具体步骤如下:
82.首先,执行步骤s2,获取公共数据库中的snp位点信息,对snp位点进行筛选,得到导致编码氨基酸改变的snp位点列表。
83.具体而言,如步骤s201,从1000genome的官方网站上下载所有染色体的vcf文件,从ncbi的官方网站上下载人的基于hg38的common_all.vcf.gz文件。
84.本实施例中优选地考虑到数据可靠性、准确性,以及后续步骤对于snp人群频率的筛选,以减少蛋白质数据库的冗余性,因此选择了1000genome和dbsnp两个数据库。
85.更进一步的,dbsnp数据库中提供了基于hg38的common_all_20180418.vcf.gz文件和all_20180418.vcf.gz文件,本实施例中以人群突变频率大于等于0.01为标准,优选地选择用dbsnp的common_all_20180418.vcf.gz文件,common_all_20180418.vcf.gz文件包含的snp范围是所有常见人的突变位点清单,即要求至少一个主要人群中次要等位基因频率(maf)≥0.01。经过实验比较,发现在对同一数据使用all_20180418.vcf.gz来建立基于样品的gvp参考蛋白质数据库,并使用样品进行搜索时一方面会造成gvp参考蛋白质数据库的过度冗余,另一方面因为其中大部分snp都是低频突变(maf<0.01),在对搜出的肽段进行fdr筛选时会导致fdr=0.01时对应的谱图分数(score)过高,所以本实施例优选地选择用dbsnp的common_all_20180418.vcf.gz。具体可参考表2。
86.表2 dbsnp选用不同人群突变频率文件搜索结果的比较
[0087][0088]
继续参考图2,如步骤s202,将vcf文件中的snp位点信息和参考基因组比对分析,得到外显子区域的突变且突变后会导致编码氨基酸有所改变的snp位点列表。
[0089]
步骤s202具体包括如下步骤:
[0090]
步骤s2021,使用注释软件,对vcf文件基于参考基因组进行注释,以得到对应vcf文件中所有外显子区域的突变,输出每个snp对应的所属蛋白原始序列和突变后的序列。
[0091]
本实施例中采用annovar注释软件对vcf文件进行注释,annovar是一个高效的注释工具,能够利用最新的数据来分析各种基因组中的遗传变异,由perl编写,支持包括vcf在内的多种输入和输出文件格式。首先先用convert2annovar.pl命令将vcf文件转换为
avinput格式,接着用annotate_variation.pl命令基于参考基因组,对avinput格式的文件进行注释,得到exonic_variant_function文件,该文件包括对应vcf文件中所有外显子区域的突变。用coding_change.pl命令输出exonic_variant_function文件中每个snp对应的所属蛋白原始氨基酸序列和突变后对应的氨基酸序列。
[0092]
进一步的,在注释snp时,针对1000genome的vcf文件应该使用hg19版本的参考基因组,针对dbsnp的vcf文件使用hg38版本的参考基因组,本实施例中参考基因组的下载通过annovar的annotate_variation.pl命令实现。
[0093]
在其他实施方式中,也可以使用snpeff或其他使用开源注释工具注释vcf文件,得到每个snp对应的所属蛋白原始氨基酸序列和突变后对应的氨基酸序列。
[0094]
步骤s2022,对原始氨基酸序列和突变后对应的氨基酸序列两两比较,挑选出导致氨基酸序列改变的snp位点列表。本实施例中采用python脚本对原始氨基酸序列和突变后对应的氨基酸序列两两比较,得到导致氨基酸序列改变的snp位点列表。
[0095]
在其他实施方式中,也可以使用其他编程语言或是程序对原始氨基酸序列和突变后对应的氨基酸序列进行比较,以得到导致氨基酸序列改变的snp位点,并建立snp位点列表。
[0096]
优选地,本实施例为了在保障数据可靠性的前提下减少蛋白质数据库的冗余性而导致结果中的假阳性过高,还包括筛选步骤s203。
[0097]
继续参考图2,如步骤s203,筛选出snp位点列表中人群突变频率相对较高的高频突变。从1000genome的vcf文件中提取出每个snp的人群频率,再按照eas_af(东亚人群中snp的人群频率)≥预设阈值对步骤s202中得到的snp位点列表进一步筛选。预设阈值的设置范围可以是0.005至1。本实施例中优选地设置预设阈值为0.01,根据实验当阈值设置为0.01时既能保证纳入列表里的snp有足够的概率被检测到,也能保障不会错误去除snp导致漏检,同时具有较佳的搜索效果。由于dbsnp产生的数据的maf≥0.01,因此不用再次进行筛选。
[0098]
参考图1,如步骤s3,在snp位点列表中根据样品的蛋白丰度筛选出表达量高的蛋白的snp位点。具体而言,如图3所示步骤s3包括下述步骤:
[0099]
首先如步骤s301,对实施例1中获取的样品质谱数据进行常规蛋白质组学分析得到蛋白定量信息,步骤s301具体包括下述操作步骤:
[0100]
步骤s3011,下载公共蛋白质数据库。本实施例使用swissprot数据库中人的参考蛋白数据库。swissprot数据库是经过注释的蛋白质数据库,由欧洲生物信息学研究所(ebi)维护。数据库由蛋白质序列条目构成,每个条目包含蛋白质序列、引用文献信息、分类学信息、注释等,注释中包括蛋白质的功能、转录后修饰、特殊位点和区域、二级结构、四级结构、与其它序列的相似性、序列残缺与疾病的关系、序列变异体和冲突等信息。swiss
‑
prot中尽可能减少了冗余序列,并与其它30多个数据建立了交叉引用,其中包括核酸序列库、蛋白质序列库和蛋白质结构库等。
[0101]
在其他实施例中,还可以使用国际蛋白质数据库(psd)、prosite数据库等公共蛋白数据库。
[0102]
步骤s3012,对实施例1中的样品的质谱文件导入搜库软件中进行检索,获取搜库结果中的蛋白定量信息。本次实施例所用为搜库软件为mascot,根据需要可以将质谱文件
的格式进行转换,例如将wiff文件转换为mgf文件,设定参数并将转换后的质谱文件导入mascot搜库软件中进行检索,检索使用的数据库为s3011步骤下载的公共蛋白质数据库。
[0103]
接着如步骤s302,根据蛋白定量信息,计算每个蛋白的总表达量,按表达量进行筛选,以得到基于样品的易被检测到的蛋白列表。具体而言,根据搜库结果中的蛋白定量信息,计算每个蛋白的总表达量,按照降序排列,挑选排序前n(n为正整数)的蛋白,或者预设蛋白丰度阈值,剔除低于阈值的蛋白质,从而得到基于样品的易被检测到的蛋白列表(参考表3)。具体的n或阈值需要根据实际样品决定。
[0104]
表3样品蛋白列表及表达量
[0105][0106][0107]
如步骤s303,根据步骤s302中得到的蛋白列表对snp位点列表再一次筛选,仅保留蛋白列表中对应蛋白的snp位点信息。本实施例中,为了进行实验效果的比较,分别保留snp所属蛋白在蛋白列表中的全部snp位点和表达丰度前1000、前500、前100、前50的蛋白列表的snp位点,即有五组对应的snp位点集和,前者对于后者是包含关系。所纳入的snp位点数量参考表4。
[0108]
表4各个筛选步骤得到的snp位点数量
[0109]
[0110]
由于本发明基于样品进行蛋白质丰度筛选,因此针对特定样品保障了结果可靠性,同时也避免了数据库的过度冗余和假阳性过高。
[0111]
继续参考图1,如步骤s4,生成筛选后的snp位点列表对应的可遗传变异肽,根据公共蛋白质数据库生成基于样品的gvp参考蛋白质数据库。
[0112]
具体而言如图4所示,如步骤s401,利用后续样品处理所用的酶对snp列表中的原始蛋白质序列和突变后的蛋白质序列进行理论酶切,本实例中后续实验用的酶为trypsin,所以这里理论酶切是以k/r作为酶切位点。比较得到的两个序列的肽段,挑选出其中不一样的且属于突变序列的肽段作为该snp对应的gvp,也就是说该snp出现会导致该肽段的出现。
[0113]
优选地,本实施例中将所有的gvp汇集为gvp列表,所述gvp列表中包括gvp信息,gvp对应的snp位点、蛋白质等信息,从而将gvp对应的snp位点、蛋白质等信息联系起来。本实例例中按蛋白丰度全部或前1000或前500或前100或前50分别筛选出182或117或58或27或6个snp位点。本实施例得到的gvp和对应蛋白质、snp位点信息参考表5,表5示例性地表示了蛋白丰度前100的gvp列表的部分。
[0114]
表5实施例1的可遗传变异肽列表
[0115]
[0116][0117]
如步骤s402,下载公共蛋白质数据库的参考蛋白质序列,将不在公共蛋白质数据库中的gvp添加到参考蛋白质序列中,以生成基于样品的gvp参考蛋白质数据库。本实施例的所述gvp参考蛋白质数据库包括gvp列表中的所有gvp信息。
[0118]
本实施例中的公共蛋白质数据库选用swissprot数据库中人的参考蛋白数据库。下载公共蛋白质数据库的参考蛋白质序列,本实施例中优选地将蛋白质序列中异亮氨酸(氨基酸字母缩写i)替换为亮氨酸(氨基酸字母缩写l),同样地,替换步骤s401中gvp列表中的i为l。
[0119]
将s401中gvp列表中的gvp信息逐条在参考蛋白质序列遍历检索,将不在参考蛋白质序列中的gvp添加到参考蛋白质序列中,以生成基于样品的gvp参考蛋白质数据库。优选地,本实施例中参考蛋白质序列以蛋白编号来区分。
[0120]
具体而言,在gvp信息逐条在参考蛋白质序列遍历检索时,首先判断该条gvp是否存在于参考蛋白质序列中,如否将该gvp添加到参考蛋白质序列中。为了简化数据库数量量,优选地,当判断对应肽段已在参考蛋白数据库中存在时,则进一步判断该gvp对应的数据序列是否以理论酶切位点为结尾,如是则将该gvp添加到数据序列后,如否则新增该gvp的数据序列并以理论酶切位点为结尾。例如,一个gvp所属蛋白为ktr33,且数据库中尚未包含krt33的gvp,则将该gvp添加入数据库中时,数据序列对应的数据头为
‘
krt33.1’,如果数据库中已有krt33对应的sap,则看该数据序列是否以酶切位点k/r结尾,如果是k/r结尾,可以直接将该gvp添加到序列后,如果不是k/r结尾,则需要将数据头的编号加1,新增一个条目,因为直接添加在非k/r结尾的序列后,在后续步骤中搜库软件无法知道要在这里断开,从而导致检索失败。
[0121]
本实施例公开了构建基于样品的gvp参考蛋白质数据库的方法,使得参考蛋白质数据库能够针对特定样品,从而在该gvp参考蛋白质数据库用于后续检索使用时,既避免了错误减少蛋白质范围而产生的误差,提高结果准确性,也不会导致纳入过多的遗传变异信息造成蛋白质数据库的高度冗余性,有效降低了结果假阳性。
[0122]
本实施例也公开了基于上述方法得到的gvp参考蛋白质数据库以及gvp列表。
[0123]
实施例3基于蛋白质组学测定snp的方法
[0124]
本实施例利用实施例2的基于样品的gvp参考蛋白质数据库的构建方法得到的参考蛋白质数据库以及gvp列表,对实施例1中的样品进行基于蛋白质组学测定snp。
[0125]
图5为基于蛋白质组学测定snp的方法流程示意图。如图5所示,首先如步骤s1,对样品进行蛋白质提取,用质谱仪进行蛋白质组学检测,采集质谱数据。
[0126]
接着如步骤s2至s4基于样品构建gvp参考蛋白质数据库以及gvp列表,包括步骤:
[0127]
步骤s2,获取公共数据库中的snp位点信息,对snp位点进行筛选,得到导致编码氨基酸改变的snp位点列表。
[0128]
步骤s3,在snp位点列表中根据样品的蛋白丰度筛选出表达量高的蛋白质的snp位点。
[0129]
步骤s4,生成筛选后的snp位点列表对应的可遗传变异肽,根据公共蛋白质数据库生成基于样品的gvp参考蛋白质数据库。
[0130]
步骤s2至s4的具体实施方法参照实施例2中介绍的方法,此处不再赘述。
[0131]
如步骤s5,将s1步骤获取的质谱数据在步骤s4得到的gvp参考蛋白序列数据库中进行搜库,得到匹配的肽段图谱对应的肽段列表。
[0132]
具体而言,设定参数并将步骤s4得到的包含gvp数据的gvp参考蛋白序列数据库以及步骤s1得到的质谱数据导入到mascot搜库软件中进行搜库。
[0133]
导出mascot搜库结果,计算fdr,通常认为fdr大于0.01的肽段是可信的,因此将fdr=0.01时对应的肽段谱图匹配得分作为得分阈值,取出得分高于得分阈值的谱图对应肽段。这些谱图对应的肽段列表即为样品肽段列表。
[0134]
如步骤s6,将样品肽段列表和步骤s4中得到的gvp列表进行比较,匹配到某个gvp,就认为该样品包含该gvp对应的snp位点,从而反推得到样品包含的snp位点信息。
[0135]
本实施例检测到的具体snp位点信息参考表6,本实施例共检测到27个gvp,对应27个snp。
[0136]
表6鉴定到的snp位点信息
[0137]
[0138][0139]
图6a,6b,6c为实施例3中根据不同标准丰度蛋白进行筛选的snp数量结果示意图,其中6a,6b,6c的横坐标均分别表示了不进行筛选(即全部)或按照蛋白质丰度前1000或前500或前100或前50的蛋白进行筛选,图6a纵坐标表示步骤s303筛选后纳入的snp位点数,图6b纵坐标表示步骤s6检测到的所有snp位点数,图6c纵坐标表示步骤s6检测到的大于fdr=0.01时score的对应的肽段的snp的位点数。根据图6a,6b,6c所示,筛选纳入的蛋白质丰度前1000的snp位点对于最终的检测结果数量没有影响,但却显著提高检索效率,而当筛选纳入的蛋白质丰度进一步降低至前50时,检测到的snp数量会有轻微降低。
[0140]
本实施例在尽量缩小蛋白质序列参考数据库保证非冗余性和结果准确性的同时尽量鉴定到更多的snp位点,给出了不依赖于个性化测序的挑选snp建库的方法,理论上可以针对每种样品类型自适应地构建最佳蛋白质数据库,为基于蛋白质组学的snp鉴定提供了解决方案。
[0141]
实施例4电子设备
[0142]
所属技术领域的技术人员能够理解,本发明的各个方面可以实现为系统、方法或程序产品。因此,本发明的各个方面可以具体实现为以下形式,即:完全的硬件实施方式、完全的软件实施方式(包括固件、微代码等),或硬件和软件方面结合的实施方式,这里可以统
称为“电路”、“模块”或“系统”。
[0143]
下面参照图7来描述根据本发明的这种实施方式的电子设备800。图7显示的电子设备800仅仅是一个示例,不应对本发明实施例的功能和使用范围带来任何限制。
[0144]
如图7所示,电子设备800以通用计算设备的形式表现。电子设备800的组件可以包括但不限于:上述至少一个处理单元810、上述至少一个存储单元820、连接不同系统组件(包括存储单元820和处理单元810)的总线830。
[0145]
其中,所述存储单元存储有程序代码,所述程序代码可以被所述处理单元810执行,使得所述处理单元810执行本说明书上述实施例2以及实施例3中描述的根据本发明各种示例性实施方式的步骤。例如,所述处理单元810可以执行如图1中所示的步骤s2,获取公共数据库中的snp位点信息,对snp位点进行筛选,得到导致编码氨基酸改变的snp位点列表。步骤s3,在snp位点列表中根据样品的蛋白丰度筛选出表达量高的蛋白的snp位点。步骤s4,生成筛选后的snp位点列表对应的可遗传变异肽,根据公共蛋白质数据库生成基于样品的gvp参考蛋白质数据库。
[0146]
存储单元820可以包括易失性存储单元形式的可读介质,例如随机存取存储单元(ram)8201和/或高速缓存存储单元8202,还可以进一步包括只读存储单元(rom)8203。存储单元820还可以包括具有一组(至少一个)程序模块8205的程序/实用工具8204,这样的程序模块8205包括但不限于:操作系统、一个或者多个应用程序、其它程序模块以及程序数据,这些示例中的每一个或某种组合中可能包括网络环境的实现。
[0147]
总线830可以为表示几类总线结构中的一种或多种,包括存储单元总线或者存储单元控制器、外围总线、图形加速端口、处理单元或者使用多种总线结构中的任意总线结构的局域总线。
[0148]
电子设备800也可以与一个或多个外部设备700(例如键盘、指向设备、蓝牙设备等)通信,还可与一个或者多个使得用户能与该电子设备600交互的设备通信,和/或与使得该电子设备800能与一个或多个其它计算设备进行通信的任何设备(例如路由器、调制解调器等等)通信。这种通信可以通过输入/输出(i/o)接口650进行。并且,电子设备800还可以通过网络适配器860与一个或者多个网络(例如局域网(lan),广域网(wan)和/或公共网络,例如因特网)通信。如图7所示,网络适配器860通过总线830与电子设备800的其它模块通信。应当明白,尽管图中未示出,可以结合电子设备600使用其它硬件和/或软件模块,包括但不限于:微代码、设备驱动器、冗余处理单元、外部磁盘驱动阵列、raid系统、磁带驱动器以及数据备份存储系统等。
[0149]
通过以上的实施方式的描述,本领域的技术人员易于理解,这里描述的示例实施方式可以通过软件实现,也可以通过软件结合必要的硬件的方式来实现。因此,根据本公开实施方式的技术方案可以以软件产品的形式体现出来,该软件产品可以存储在一个非易失性存储介质(可以是cd
‑
rom,u盘,移动硬盘等)中或网络上,包括若干指令以使得一台计算设备(可以是个人计算机、服务器、终端装置、或者网络设备等)执行根据本公开实施方式的方法。
[0150]
实施例5计算机可读存储介质
[0151]
在本公开的示例性实施例中,还提供了一种计算机可读存储介质,其上存储有能够实现实施例2、3的方法的程序产品。在一些可能的实施方式中,本发明的各个方面还可以
实现为一种程序产品的形式,其包括程序代码,当所述程序产品在终端设备上运行时,所述程序代码用于使所述终端设备执行本说明书上述实施例2、3的部分中描述的根据本发明各种示例性实施方式的步骤。
[0152]
参考图8所示,描述了根据本发明的实施方式的用于实现上述方法的程序产品900,其可以采用便携式紧凑盘只读存储器(cd
‑
rom)并包括程序代码,并可以在终端设备,例如个人电脑上运行。然而,本发明的程序产品不限于此,在本文件中,可读存储介质可以是任何包含或存储程序的有形介质,该程序可以被指令执行系统、装置或者器件使用或者与其结合使用。
[0153]
所述程序产品可以采用一个或多个可读介质的任意组合。可读介质可以是可读信号介质或者可读存储介质。可读存储介质例如可以为但不限于电、磁、光、电磁、红外线、或半导体的系统、装置或器件,或者任意以上的组合。可读存储介质的更具体的例子(非穷举的列表)包括:具有一个或多个导线的电连接、便携式盘、硬盘、随机存取存储器(ram)、只读存储器(rom)、可擦式可编程只读存储器(eprom或闪存)、光纤、便携式紧凑盘只读存储器(cd
‑
rom)、光存储器件、磁存储器件、或者上述的任意合适的组合。
[0154]
计算机可读信号介质可以包括在基带中或者作为载波一部分传播的数据信号,其中承载了可读程序代码。这种传播的数据信号可以采用多种形式,包括但不限于电磁信号、光信号或上述的任意合适的组合。可读信号介质还可以是可读存储介质以外的任何可读介质,该可读介质可以发送、传播或者传输用于由指令执行系统、装置或者器件使用或者与其结合使用的程序。
[0155]
可读介质上包含的程序代码可以用任何适当的介质传输,包括但不限于无线、有线、光缆、rf等等,或者上述的任意合适的组合。
[0156]
可以以一种或多种程序设计语言的任意组合来编写用于执行本发明操作的程序代码,所述程序设计语言包括面向对象的程序设计语言—诸如java、c++等,还包括常规的过程式程序设计语言—诸如“c”语言或类似的程序设计语言。程序代码可以完全地在用户计算设备上执行、部分地在用户设备上执行、作为一个独立的软件包执行、部分在用户计算设备上部分在远程计算设备上执行、或者完全在远程计算设备或服务器上执行。在涉及远程计算设备的情形中,远程计算设备可以通过任意种类的网络,包括局域网(lan)或广域网(wan),连接到用户计算设备,或者,可以连接到外部计算设备(例如利用因特网服务提供商来通过因特网连接)。
[0157]
应当注意,尽管在上文详细描述中提及了用于动作执行的设备的若干模块或者单元,但是这种划分并非强制性的。实际上,根据本公开的实施方式,上文描述的两个或更多模块或者单元的特征和功能可以在一个模块或者单元中具体化。反之,上文描述的一个模块或者单元的特征和功能可以进一步划分为由多个模块或者单元来具体化。
[0158]
此外,尽管在附图中以特定顺序描述了本公开中方法的各个步骤,但是,这并非要求或者暗示必须按照该特定顺序来执行这些步骤,或是必须执行全部所示的步骤才能实现期望的结果。附加的或备选的,可以省略某些步骤,将多个步骤合并为一个步骤执行,以及/或者将一个步骤分解为多个步骤执行等。
[0159]
通过以上的实施方式的描述,本领域的技术人员易于理解,这里描述的示例实施方式可以通过软件实现,也可以通过软件结合必要的硬件的方式来实现。因此,根据本公开
实施方式的技术方案可以以软件产品的形式体现出来,该软件产品可以存储在一个非易失性存储介质(可以是cd
‑
rom,u盘,移动硬盘等)中或网络上,包括若干指令以使得一台计算设备(可以是个人计算机、服务器、移动终端、或者网络设备等)执行根据本公开实施方式的方法。
[0160]
本领域技术人员在考虑说明书及实践这里公开的发明后,将容易想到本公开的其它实施方案。本技术旨在涵盖本公开的任何变型、用途或者适应性变化,这些变型、用途或者适应性变化遵循本公开的一般性原理并包括本公开未公开的本技术领域中的公知常识或惯用技术手段。说明书和实施例仅被视为示例性的,本公开的真正范围和精神由所附的权利要求指出。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1