一种对分裂位点进行筛选的方法及其应用与流程
1.本发明涉及蛋白分裂位点筛选领域,具体涉及一种对分裂位点进行筛选的方法及其应用。
背景技术:
2.蛋白内含肽能够将侧翼外蛋白(外部蛋白)连接成一个新的蛋白质片段,并将其本身切除出去,这一过程被称为蛋白质剪接。内含肽在许多自然生物中被发现,如细菌、真菌和低等植物,通常嵌入在重要的蛋白质中。在自然界中,蛋白质剪接产生两个独立的蛋白质(内含肽和外显蛋白)并在一个基因的控制下内含肽精确切除内部蛋白质片段(内含肽本身)和同时连接两边区域。自然界存在的蛋白内含肽存在几种形式,包括全长内含肽,迷你内含肽和自然产生的分裂内含肽。全长内含肽和迷你内含肽都是顺式剪接的内含肽,在其中存在或不存在一个内切酶结构域。分裂内含肽是反式剪接的内含肽,包含两个蛋白片段并且它们是由两个独立的基因转录和翻译而来。反式剪接需要两个分裂的内含肽片段共同表达,即n-蛋白(in,与n-外显蛋白的c端融合)和c-蛋白(ic,与c-外显蛋白的n端融合)。分裂的内含肽片段随后结合以恢复其活性,并催化n-外显蛋白和c-外显蛋白的连接。在顺式或反式剪接中,内含肽介导的剪接化学反应不需要任何酶或辅助因子的帮助,而只要求表达蛋白具有正确折叠结构。
3.现有科技文献(“a systematic approach to inserting split inteins for boolean logic gate engineering and basal activity reduction”,baojun wang等,nature communications,2021)记载了一种蛋白分裂位点筛选的方法,名称为intein-assisted bisection mapping(ibm)split site screening,也是基于噬菌体侵染机制介导的mini-mu transposon随机插入基因片段的方法,但其仅限于已经成功搜寻到分裂位点的一些荧光蛋白或抗生素抗性蛋白,这些蛋白本身可以激发荧光或是在抑制抗生素和在转录启动原件中有调控作用,使得它们具有可被高通量筛选检测出活性的特质。然而,大多数的重组蛋白,如il-2、ifn、egf、bfgf等这些具有高价值的蛋白药物,均没有可通过高通量筛选直接检测出来的特性。
技术实现要素:
4.鉴于上述现有技术中存在的问题,本发明的目的之一在于提供一种对分裂位点进行筛选的方法,先通过计算机编程构建蛋白数据库,再进行实验并通过质谱实现分裂位点的检测和证实;通过质谱实现最终的检测,代替高通量筛选,将其拓展到可以将任意活性蛋白进行分裂位点的寻找。
5.作为前提,本发明的实验原理(即关于噬菌体侵染机制介导的mini-mu transposon随机插入基因片段的方法)与上述科技文献“a systematic approach to inserting split inteins for boolean logic gate engineering and basal activity reduction”相同,若在本技术的文本中,对于相关作用原理的表述不够清楚,该文献也可作
为解释。
6.为实现上述目的,本发明第一方面提供了一种对分裂位点进行筛选的方法,其包括步骤:
7.s1、数据库建立:使用计算机语言编写程序,预测内含肽在氨基酸序列中的每相邻的两个氨基酸残基中嵌入后都发生了剪接反应将自身切除,相邻肽段相连后形成的氨基酸序列组成蛋白数据库;
8.s2、实验进行:在基因片段中通过分子克隆实验方法插入内含肽序列,翻译后得到肽段,将该肽段通过质谱检测是否含有标记氨基酸序列,并与所述蛋白数据库进行比对,实现对断裂位点的证实。
9.本发明中,计算机语言可采用能实现编程功能的所有语言,如使用python编写脚本,完成本发明步骤s1中数据库的建立。
10.本技术可以使用质谱进行检测的原理在于:由于分子克隆留下酶切位点在表达时也会被翻译成氨基酸,因此,是否产生的一段由marker sequence翻译而成的标记氨基酸序列,可作为剪接反应是否发生的标志,通过质谱检测翻译后得到的肽段中是否含有该标记氨基酸序列,可以实现将任意活性蛋白进行分裂位点的寻找,而不是目前仅限于已经成功搜寻到分裂位点的一些荧光蛋白或抗生素抗性蛋白。
11.在本技术的一些实施方式中,所述步骤s1的数据库建立包括:
12.s11、按顺序合并基因片段1、插入内含肽序列片段和基因片段2,得到新dna序列;
13.s12、将所述新dna序列翻译为新氨基酸序列;
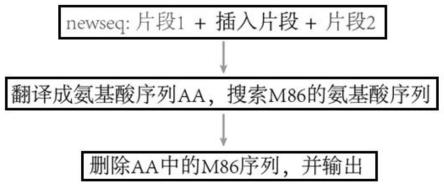
14.s13、搜索所述新氨基酸序列中的目标内含肽氨基酸序列(程序设定所述新氨基酸序列中含有所述目标内含肽氨基酸序列),删除所述目标内含肽氨基酸序列,得到输出氨基酸序列;
15.s14、预测将所述插入内含肽序列片段插入到基因片段1和基因片段2每个可能的位点,再重复步骤s11~s13,得到的所有所述输出氨基酸序列构成所述蛋白数据数据库。
16.根据本发明,步骤s13中,设定计算机编程自动删除所述新氨基酸序列中的目标内含肽氨基酸序列,从而得到输出氨基酸序列。
17.图1为本发明的一些实施方式中通过计算机编程进行数据库建立的流程示意图,可参照图1,图中的m86为蛋白内含肽ssp dnabm86,由在synechocystis spp.pcc6803中发现的ssp dnab内含肽改造而来。
18.在本技术的一些实施方式中,步骤s11中,在所述新dna序列中插入若干个碱基,以防止翻译时发生移码突变。在噬菌体转座之后,会将插入5’端的前若干个(如5个)碱基进行复制一遍,因此,为防止移码突变,在插入内含肽序列片段中,在复制的5个碱基的3’端(所述插入内含肽序列片段的5’端)插入若干个(如1个)碱基,以使得噬菌体转座之后复制的碱基与主动插入的碱基数相加满足3的倍数。图1为本发明的一些实施方式中插入片段的示意图,可参照图2,从i-5至i处的5个碱基即为噬菌体转座时复制的前5个碱基,在插入上述5’端和3’端分别带有酶切位点以及添加若干碱基的内含肽基因片段后在其下游添加这5个碱基,并将整段基因翻译为氨基酸序列再将内含肽序列删除,得到蛋白数据库中的其中一条氨基酸序列。将以上步骤编写为计算机程序语言(如python程序)对目的蛋白中每相邻两个氨基酸之间进行内含肽插入并进行推演,得到最终构建好的蛋白数据库(参见步骤s1的叙
述)。
19.根据本发明,噬菌体mu转座机制会将插入位置上游5个碱基在插入端下游处复制一次,由此综合酶切位点和转座机制最终留下的marker sequence。
20.本发明第二方面提供了一种如上述第一方面所述的方法在分裂位点筛选中的应用,尤其是实现对大肠杆菌抗原蛋白im7-6和/或cas9蛋白分裂位点筛选的应用。
21.本发明第三方面提供了一种如上述第一方面所述的方法在对il-2蛋白、ifn蛋白、egf蛋白和bfgf蛋白等的重组蛋白进行筛选的应用。
22.由于在上述科技文献中采用的是高通量筛选,这仅限于已经成功搜寻到分裂位点的一些荧光蛋白或抗生素抗性蛋白,这些蛋白本身可以激发荧光或是在抑制抗生素和在转录启动原件中有调控作用,使得它们具有可被高通量筛选检测出活性的特质。而本发明通过质谱实现最终的检测,将其拓展到可以将任意活性蛋白进行分裂位点的寻找。
23.本发明的有益效果包括以下内容的至少一项:
24.1)本发明提供的筛选方法合理的搭配计算机编程构建蛋白数据库;
25.2)本发明提供的筛选方法通过质谱实现最终的检测,创新性的拓展了现有的分裂位点筛选方案,能够拓展到将任意活性蛋白进行分裂位点的寻找;
26.3)本发明提供的筛选方法证实了断裂位点后,可接下来设计实验进行蛋白质组装,为之后的糖基化实验提供一种新的思路。
附图说明
27.图1示出了本发明的一些实施方式中通过计算机编程进行数据库建立的流程示意图;
28.图2示出了本发明的一些实施方式中插入片段的示意图;
29.图3示出了本发明实施例1的随机插入基因片段的操作步骤示意图;
30.图4示出了本发明实施例1中内含肽ssp dnabm86的核苷酸序列的示意图;
31.图5示出了本发明实施例1的筛选方法的示意图;
32.图6示出了本发明实施例1的质谱检测得到的多肽断裂位点结果;
33.图7示出了本发明实施例2的断裂内含肽split ssp dnabm86的核苷酸序列的示意图;
34.图8示出了本发明实施例2的质谱检测得到的多肽断裂位点结果一;
35.图9示出了本发明实施例2的质谱检测得到的多肽断裂位点结果二。
具体实施方式
36.为使本发明容易理解,下面将结合附图详细说明本发明。但在详细描述本发明前,应当理解本发明不限于描述的具体实施方式。还应当理解,本文中使用的术语仅为了描述具体实施方式,而并不表示限制性的。
37.在提供了数值范围的情况下,应当理解所述范围的上限和下限和所述规定范围中的任何其他规定或居间数值之间的每个居间数值均涵盖在本发明内。这些较小范围的上限和下限可以独立包括在较小的范围中,并且也涵盖在本发明内,服从规定范围中任何明确排除的限度。在规定的范围包含一个或两个限度的情况下,排除那些包括的限度之任一或
两者的范围也包含在本发明中。
38.除非另有定义,本文中使用的所有术语与本发明所属领域的普通技术人员的通常理解具有相同的意义。虽然与本文中描述的方法和材料类似或等同的任何方法和材料也可以在本发明的实施或测试中使用,但是现在描述了优选的方法和材料。
39.本发明采用的关于噬菌体侵染机制介导的mini-mu transposon随机插入基因片段的方法的相关实验原理可参见科技文献(“a systematic approach to inserting split inteins for boolean logic gate engineering and basal activity reduction”,baojun wang等,nature communications,2021),该篇文献全文可引为参考。
40.实施例1
41.可参照图3和图4,本实施例采用上述筛选方法对大肠杆菌抗原蛋白(im7-6)的多肽断裂位点进行验证。
42.如图3所示,图3即为上述科技文献“a systematic approach to inserting split inteins for boolean logic gate engineering and basal activity reduction”相同的实验原理,即噬菌体转座机制介导的mini-mu transposon随机插入基因片段的方法,具体操作如下:
43.1、使用转座酶mua对目的基因片段进行转座实验,将转座子随机插入目的片段中,并且其原理确保了每一个目的基因片段只插入一个转座子片段。转座子片段为一个完整的带有启动子,终止子等元件并且表达氯霉素抗性蛋白的表达系;
44.2、将转座好的基因片段通过无缝克隆方法连接到pet28a表达载体上,转化大肠杆菌top10扩增载体。使用氯霉素抗性平板对菌落进行筛选;
45.3、将上述平板中所有的菌落收集起来混合培养并提取其质粒,由于转座子上下游端各带有一个noti酶切位点,所以对其进行酶切实验将转座子片段替换为表达内含肽ssp dnabm86(对应图3中3.substitution中的cis-intein version方式),然后转化大肠杆菌top10感受态进行载体扩增,用kanamycin抗生素平板进行菌落筛选。然后再将所有菌落收集培养并提取质粒,转化到大肠杆菌bl21de3表达菌株中,并且再次收集平板所有菌落然后接种到lb培养基中进行蛋白表达。最终将混合表达出的蛋白进行纯化浓缩,得到样品。
46.如图4所示为内含肽ssp dnabm86的核苷酸序列的示意图,其带有酶切位点和为防止移码突变添加的额外碱基。图4中,两个框1中的gcggccgc为酶切位点的核苷酸序列,两个框2中的c碱基为为防止移码突变添加的额外碱基,框3中的xxxxx(x代表atcg中的某一碱基)即为噬菌体转座之后复制的5个碱基。需要说明的是,图4仅示出了部分核苷酸序列,尤其是框3中复制的5个碱基是非固定的。
47.如图5所示,为本发明与上述科技文献具有显著区别之处(该文献直接对样品进行高通量筛选),具体操作如下:
48.4、先使用python编写脚本,预测内含肽在氨基酸序列中的每相邻的两个氨基酸残基中嵌入后都发生了剪接反应将自身切除,相邻肽段相连后形成的氨基酸序列组成蛋白数据库;
49.5、将步骤3得到的样品使用质谱进行检测。
50.质谱检测结果如图6所示,由图中分析可得,对于大肠杆菌抗原蛋白(im7-6),aaalrply的氨基酸序列为发生酶切位点产生的marker sequence翻译而成的标记氨基酸序
列(噬菌体mu转座机制会将插入位置上游5个碱基在插入端下游处复制一次,由此综合酶切位点和转座机制最终留下的marker sequence即一个含有8个氨基酸残基的序列:aaalrpxx,其中,xx即为复制的5个碱基和为防止移码突变插入的一个碱基翻译而来);再通过步骤4得到的蛋白数据库进行搜索,当搜到全覆盖或部分覆盖marker sequence(部分覆盖在这里指至少搜到al两个氨基酸以证明内含肽剪接反应真实发生)的离子片段,即可确定大肠杆菌抗原蛋白(im7-6)的一个多肽断裂位点为y61-y62(如图5)。
51.实施例2
52.本实施例采用上述筛选方法对cas9蛋白的两个多肽断裂位点进行验证。
53.其步骤1-5大致与实施例1相同,不同之处在于,步骤3中“对其进行酶切实验将转座子片段替换为表达内含肽ssp dnabm86”替换为“对其进行酶切实验将转座子片段依次替换为断裂内含肽ssp dnabm86的n端、终止子及启动子转录元件和断裂内含肽ssp dnabm86的c端的基因片段(对应图3中3.substitution中的split intein version方式)”54.如图7所示为内含肽ssp dnabm86的核苷酸序列的示意图,其带有酶切位点和为防止移码突变添加的额外碱基。同上图4所述,图7中,两个框1中的gcggccgc为酶切位点的核苷酸序列,两个框2中的c碱基为为防止移码突变添加的额外碱基,框3中的xxxxx(x代表atcg中的某一碱基)即为噬菌体转座之后复制的5个碱基。相应的,图7也仅示出了部分核苷酸序列,尤其是框3中复制的5个碱基是非固定的。
55.质谱检测结果如图8和图9所示,由图中分析可得,对于cas9蛋白,aaalrppd和aaalrphv的氨基酸序列分别为发生酶切位点产生的marker sequence翻译而成的标记氨基酸序列(噬菌体mu转座机制会将插入位置上游5个碱基在插入端下游处复制一次,由此综合酶切位点和转座机制最终留下的marker sequence即一个含有8个氨基酸残基的序列:aaalrpxx,其中,xx即为复制的5个碱基和为防止移码突变插入的一个碱基翻译而来);再通过步骤4得到的蛋白数据库进行搜索,当搜到全覆盖或部分覆盖marker sequence(部分覆盖在这里指至少搜到al两个氨基酸以证明内含肽剪接反应真实发生)的离子片段,即可确定cas9蛋白的两个多肽断裂位点分别为d868-n869(图8)和181v-182d(图9)。
56.由上实施例可以看出,本技术提供的筛选方法合理搭配计算机编程构建蛋白数据库;再通过质谱实现最终的检测,创新性的拓展了现有的分裂位点筛选方案,能够拓展到将任意活性蛋白进行分裂位点的寻找;证实了断裂位点后,可接下来设计实验进行蛋白质组装,为之后的糖基化实验提供一种新的思路。
57.应当注意的是,以上所述的实施例仅用于解释本发明,并不构成对本发明的任何限制。通过参照典型实施例对本发明进行了描述,但应当理解为其中所用的词语为描述性和解释性词汇,而不是限定性词汇。可以按规定在本发明权利要求的范围内对本发明作出修改,以及在不背离本发明的范围和精神内对本发明进行修订。尽管其中描述的本发明涉及特定的方法、材料和实施例,但是并不意味着本发明限于其中公开的特定例,相反,本发明可扩展至其他所有具有相同功能的方法和应用。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1