一种基于DNA甲基化的生物学年龄预测模型的构建方法
一种基于dna甲基化的生物学年龄预测模型的构建方法
技术领域
1.本发明涉及一种适用于中国人群的基于dna甲基化的生物学年龄预测模型及构建方法。
背景技术:
2.人口老龄化是亟待解决的世界性问题。据who统计,全世界60岁及以上人口将从2015年的12%增加到2050年的22%。2000年我国进入老龄化社会,现已成为老龄人口最多,老龄化速度最快的国家。据统计,2017年我国居民60岁以上人口占17.9%,预测到2050年将占三分之一以上。人口老龄化导致衰老和衰老相关疾病的发病率不断上升,给社会和家庭带来沉重的负担,已成为当今重大的社会问题。衰老是指随着年龄的增加,机体在生理、心理、认知等方面的能力出现进行性下降和功能丧失,疾病易感性增加并最终导致生命终结的过程。衰老是生物体必然发生的一种现象,但是延缓衰老,降低衰老相关疾病的发生,实现健康老龄化是当今社会的重要目标,也是健康中国2030战略的核心内容。开展衰老防治研究已成为人口健康领域优先发展的内容,而衰老评估是衰老研究和防治的基础和关键环节。
3.目前衰老评价最常用的指标是日历年龄(chronological age),即按出生日期计算的年龄,也称时序年龄。日历年龄与人体器官功能的下降、慢性疾病的发生以及死亡风险有关。然而,相同日历年龄的个体衰老程度可能并不相同,即有些人在外表和功能上呈现年轻化,而有些人呈现较年老的特征。此外,相同日历年龄的个体发生衰老相关疾病的风险也存在很大差异,老年人健康结局的异质性很高。由此说明,日历年龄作为评价衰老的指标有局限性,不是理想的评价指标。
4.衰老是指在遗传和环境等因素的作用下,机体发生的遗传和表观遗传、生物化学及其它功能表型的变化,包括基因组不稳定性、端粒长度缩短、dna甲基化或去甲基化、蛋白质稳态改变、营养感应调节异常、线粒体功能紊乱、细胞凋亡、干细胞衰竭和细胞间通讯改变等。利用这些衰老相关分子生物学标志物建立的生物学年龄(biological age)可以更精确地反映机体的衰老程度、损伤修复和组织再生能力、功能状态,评价现在(或将来)的健康状况与寿命。相比日历年龄,生物学年龄是评价衰老更合适的指标。然而,生物学年龄的评价缺乏金标准,通常采用衰老相关的生物标志物来预测生物学年龄。常见的衰老标志物包括分子和表型标志物两类。前者如遗传易感性、特征性基因表达水平、小分子代谢物、dna甲基化、端粒长度等。表型标志物如血压、血脂、血糖等生理生化指标以及认知、记忆、握力等功能指标。这些标志物中,由于dna甲基化可以反映机体在遗传和环境因素作用下的分子改变,且与日历年龄有高度的相关性,在体内稳定,有高通量的检测方法等特点,被认为是目前最有前景的人群生物学年龄评价标志。
5.dna甲基化是指基因组dna序列中cpg岛的5
′
位胞嘧啶发生的甲基化共价修饰。cpg岛甲基化可以影响并调控基因的表达,与胚胎发育、恶性肿瘤、免疫缺陷等疾病的发生、发展密切相关。自上世纪60年代以来,众多研究已发现年龄与基因组dna甲基化的程度和分布
有关,某些cpg岛的甲基化程度会随着年龄的增长而增加。在人类基因组中,有两类年龄相关的cpg岛:一类是使基因活化或表达增强的低甲基化cpg岛,另一类是使基因表达抑制的高甲基化cpg岛。利用这些与年龄相关的甲基化cpg位点,采用多因素统计方法可以建立生物学年龄预测模型,称为甲基化年龄模型或表观遗传时钟。目前国际上最常用表观遗传时钟为horvath年龄时钟和hannum年龄时钟。horvath年龄时钟是利用illumina 27k甲基化芯片数据,以353个cpg位点作为标志建立年龄预测模型,而hannum年龄时钟利用illumina 450k芯片,以71个cpg位点作为标志建立年龄预测模型。两者预测的甲基化年龄与日历年龄均呈现较高的相关性(相关系数分别为0.97和0.96)。在调整日历年龄等混杂因素后,众多研究发现甲基化年龄与肥胖、胰岛素抵抗、代谢组分异常、衰老相关疾病(阿尔茨海默症、恶性肿瘤、心脑血管疾病、帕金森氏病等)的发病和死亡风险有关。此外,dna甲基化是一种可逆的化学修饰,近来研究发现,采用二甲双胍等综合治疗一年后,可以逆转甲基化年龄1.5岁,与对照组相比,可以减少甲基化年龄2.5岁。更令人振奋的是,目前研究发现,体外表达yamanaka因子(使体细胞转变成多能干细胞),可以完全重置表观遗传时钟。体内造血干细胞治疗或输注年轻供体的血液也可以逆转表观遗传时钟。这些研究结果说明以dna甲基化为基础的生物学年龄可用于衰老和抗衰老相关研究。
6.然而,现有的以dna甲基化为基础的生物学年龄预测模型仍然存在局限性。首先,无论是horvath还是hannum年龄时钟,所纳入的cpg位点数过多(分别为353和71个位点)。在目前条件下,只有全基因组甲基化芯片技术才能满足检测要求,因而对检测技术和检测成本的要求很高,限制了模型的临床应用。其次,现有模型的构建均以日历年龄为应变量,没有考虑日历年龄本身的异质性问题,没有按人群特征进行优化处理。从本质上,这些模型只能预测日历年龄或带有部分生物学年龄信息的“半生物学年龄”,并不能充分反映生物学年龄信息。第三,现有模型主要是以欧美人群数据为基础构建的,缺乏亚洲人群或中国人群数据,而不同种族之间的甲基化水平存在差异,因此这些模型可能不适用于中国人群。第四,部分cpg位点在不同血细胞间的甲基化水平存在差异,血细胞构成会影响全血标本总的dna甲基化水平。现有以全血标本构建的模型并未考虑血细胞异质性的影响,因此利用这些模型进行生物学年龄评估时还需要调整血细胞构成,从而增加模型的复杂性,不利于实际运用。
7.基于以上背景,目前急需建立一种适用于中国人群的基于dna甲基化的生物学年龄预测模型。在保证预测准确性的前提下,减少信息冗余,以最少的cpg位点数建立模型。建立该适合中国人群的、非侵入性和低检测成本的,非血细胞成分相关的实用生物学年龄预测模型将在中国人群的衰老评价和相关研究中具有非常重要的意义。
技术实现要素:
8.本发明的目的在于针对现有技术的不足,提供一种基于dna甲基化的生物学年龄预测模型的构建方法。本发明构建的预测模型包含较少的甲基化位点,适用于中国人群,且不受血细胞成分的影响,可以为中国人群的衰老评估和衰老相关疾病的研究和防治提供重要工具。
9.本发明的目的是通过以下技术方案来实现的:一种基于dna甲基化的生物学年龄预测模型的构建方法,包括以下步骤:
10.(1)dna甲基化样本数据的获取:从geo数据网站下载源自中国人群的、包含日历年龄资料的、全血样本450k甲基化芯片原始数据;
11.(2)dna甲基化样本数据的预处理:利用r语言软件包“champ”,对所获取的450k甲基化芯片原始数据进行预处理;
12.(3)甲基化年龄预测模型的位点筛选:以日历年龄作为因变量,235021个cpg位点作为自变量,性别作为协变量,采用弹性网络回归的方法对预测模型的cpg位点进行特征选择;弹性网络回归利用r语言软件包“glmnet”进行,其参数设置为:alpha=0.5,连接函数为“gaussian”,惩罚参数lambda采用10折交叉验证的方法确定,选择使均方根误差最小的lambda值;利用bootstrap自助重抽样的方法,对训练集样本进行多次重抽样;在每个重抽样的数据集中,利用上述弹性网络回归筛选得到一系列cpg位点,然后统计在多次重抽样中,每个cpg位点被筛选到的频率,选择筛选频率大于50%的cpg位点作为构建模型的候选甲基化位点;最终共得到31个候选cpg位点:cg16867657、cg07372824、cg07553761、cg14361627、cg24079702、cg13575925、cg21692159、cg03032497、cg20158366、cg18404041、cg06567855、cg23500537、cg07850154、cg21177396、cg06639320、cg17621438、cg11847992、cg06515235、cg15059474、cg01620164、cg11423680、cg18507365、cg18933331、cg19893664、cg15665792、cg17243289、cg03684893、cg16882373、cg25703552、cg00481951、cg27184585;
13.(4)甲基化年龄预测模型的构建与评价:采用多重线性回归、支持向量机、随机森林和梯度提升回归树进行模型的初步构建和评价,选择最优模型构建方式;然后,采用最优子集回归进一步筛选甲基化位点,计算模型中cpg位点数从1到31时各个最优模型的预测精度,比较不同cpg位点数下所能达到的预测效果,并结合贝叶斯信息准则,选择最佳cpg位点数的模型;最优子集回归利用r语言软件包“leaps”进行,从31个cpg位点中筛选出18个cpg位点:cg06567855、cg15059474、cg03684893、cg16867657、cg06639320、cg11423680、cg18404041、cg18507365、cg14361627、cg21177396、cg13575925、cg11847992、cg07372824、cg16882373、cg06515235、cg07850154、cg25703552、cg07553761;利用最优的多重线性回归方法对这18个cpg位点建立甲基化年龄预测模型;
14.(5)甲基化年龄预测模型的优化和生物学年龄预测模型的建立:利用任一省队列自然人群数据,对上述甲基化年龄预测模型进行优化;首先排除存在严重慢性病的人群和预测偏差最大的30%人群,剩余的样本为正常型人群,可认为其生物学年龄近似等于日历年龄;在正常型人群中,以日历年龄为应变量,采用多重线性回归的方法,对18个cpg位点的回归系数进行拟合调整,优化后得到的模型即为生物学年龄预测模型。
15.进一步地,所述步骤2包括以下子步骤:
16.(2.1)探针(cpg位点)的过滤,具体包括:
①
去除detection p值》0.01的探针,
②
去除non cpg探针,
③
去除含有snp(单核苷酸多态性)位点的探针,
④
去除多重反应位点的探针;
⑤
去除分布在性染色体上的探针;
17.(2.2)样本的过滤:利用r语言软件包“watermelon”进行主成分分析,排除离群的样本,即前两个主成分分布在2倍的四分位数间距以外的样本;此外,排除性别信息缺失的样本;
18.(2.3)探针信号的校准:对所有样本各个探针的信号值(β值)进行归一化处理,校正芯片设计中由于i型、ⅱ型探针数据分布不一致引起的偏倚;
19.(2.4)批次效应的校准:采用经验贝叶斯模型(empirical bayes),对来源于不同数据集的甲基化数据进行批次效应的校准;
20.(2.5)血细胞异质性的校准:剔除甲基化水平存在血细胞异质性的cpg位点。
21.本发明的有益效果是:本发明利用219例中国人群的甲基化数据,通过弹性网络结合bootstrap自助重抽样的方法,筛选得到31个建模候选的甲基化位点。采用多重线性回归、支持向量机、随机森林和梯度提升回归树进行模型的初步构建和评价,然后利用全子集回归进一步筛选甲基化位点,得到基于18个甲基化位点的甲基化年龄预测模型。然后,利用浙江队列自然人群数据,对甲基化年龄预测模型进行优化,最终得到基于18个甲基化位点的生物学年龄预测模型。该模型适用于中国人群,包含的甲基化位点数少,不受血细胞成分的影响,预测的精准性良好。该模型由于其简便性、经济性和准确性,可以在临床应用中广泛推广,为中国人群的衰老评估和衰老相关疾病的研究和防治提供重要工具。
附图说明
22.图1是发明的技术路线图;
23.图2是训练集最优子集回归中不同甲基化位点数模型的bic变化图。
具体实施方式
24.本发明涉及一种适用于中国人群的基于dna甲基化的生物学年龄预测模型,总体的技术路线见图1。本发明具体通过以下步骤构建得到:
25.步骤一、dna甲基化样本数据的获取
26.从geo数据网站(https://www.ncbi.nlm.nih.gov/geo/index.cgi)下载源自中国人群的、包含日历年龄资料的、血液来源的、450k dna甲基化芯片原始数据。根据搜索结果,共得到7个符合要求的独立数据库:gse104812,gse107737,gse116379,gse51388,gse53740,gse65638,gse84003。选择其中不含衰老相关疾病的健康样本数据,最终共得到227例450kdna甲基化芯片原始数据。
27.步骤二、dna甲基化样本数据的预处理
28.利用r语言软件包“champ”,按照以下步骤,对所获取的450k dna甲基化芯片原始数据进行预处理:
29.(1)探针(cpg位点)的过滤:
①
去除detection p值》0.01的探针,即信号强度低于阴性对照探针的探针;
②
去除non cpg探针,即非甲基化位点的质控探针;
③
去除含有snp(单核苷酸多态性)位点的探针;
④
去除多重反应位点的探针(multi-hit probes);
⑤
去除分布在性染色体上的探针。
30.(2)样本的过滤:
①
利用r语言软件包“watermelon”进行主成分分析,排除离群样本。离群样本定义为前两个主成分分布在2倍的四分位数间距以外的样本。
②
排除性别信息缺失的样本。
31.(3)探针信号的校准:采用“bmiq(beta-mixture quantile)”方法对所有样本各个探针的信号值(β值)进行归一化处理,校正芯片设计中由于i型、ⅱ型探针数据分布不一致引起的偏倚。
32.(4)批次效应的校准:采用经验贝叶斯模型(empirical bayes),对来源于不同数
据集的甲基化数据进行批次效应的校准。
33.(5)血细胞异质性的校准:多项研究表明,部分cpg位点的甲基化水平在不同血细胞之间存在差异,而全血中血细胞的构成会随着年龄增长而变化,为排除血细胞的构成对建模结果的影响,本发明通过参考当前有关甲基化位点血细胞异质性的研究结果,剔除甲基化水平存在血细胞异质性的cpg位点。
34.步骤三、甲基化年龄预测模型的位点筛选
35.经过上述的数据预处理,最终剩余样本219例,cpg位点数235021。对数据进行拆分,按照训练集70%,测试集30%的比例,采用年龄分层随机抽样(百分位数分5层)的方法,将数据划分为训练集(155例)和测试集(64例)。训练集和测试集样本的基本信息见表1。
36.表1:训练集和测试集样本的基本信息
[0037] 训练集(n=155)测试集(n=64)p值年龄,中位数(四分位数间距)32.0(22.0-47.2)32.0(21.8-47.2)0.700性别,人数(百分比)
ꢀꢀ
0.999男性76(49.0)32(50.0) 女性79(51.0)32(50.0) [0038]
注:年龄使用wilcoxon秩和检验比较组间差异,性别使用卡方检验比较组间差异。
[0039]
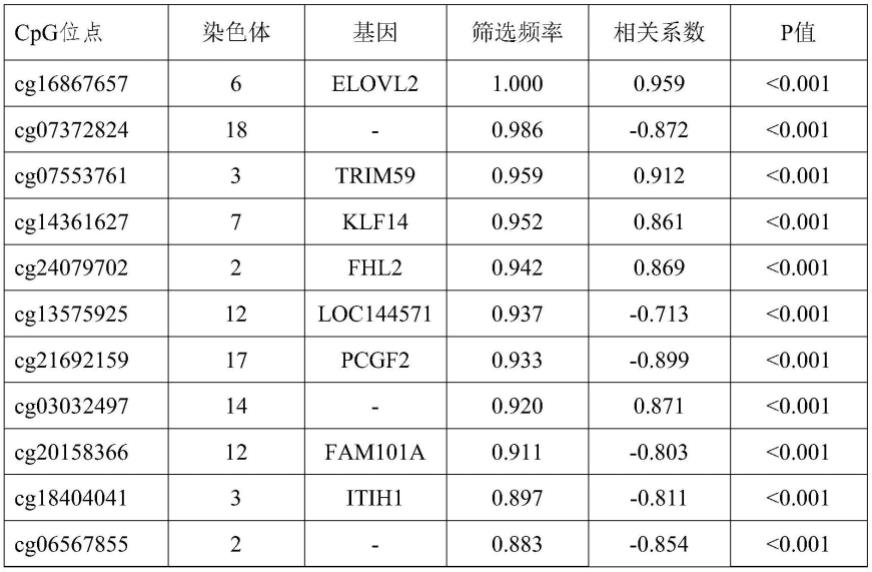
在训练集中,以日历年龄作为因变量,235021个cpg位点作为自变量,性别作为协变量,采用弹性网络(elastic net)回归的方法对预测模型的候选cpg位点进行筛选。弹性网络回归利用r语言软件包“glmnet”进行,其参数设置为:alpha=0.5,连接函数为“gaussian”,惩罚参数lambda采用10折交叉验证的方法确定,选择使均方根误差最小的lambda值。考虑到训练集样本人数较少,为增加筛选得到的cpg位点的稳定性,本发明利用bootstrap自助重抽样的方法,对训练集样本进行1000次重抽样。在每个重抽样的数据集中,利用上述弹性网络回归的方法筛选得到一系列cpg位点,然后统计在1000次重抽样中,每个cpg位点被筛选到的频率,选择频率大于50%的cpg位点作为构建预测模型的候选甲基化位点。该步骤共得到31个候选cpg位点,各个cpg位点的基本信息见表2。
[0040]
表2:31个候选cpg位点的基本信息
[0041]
[0042][0043]
注:筛选频率为在1000次重抽样中,通过弹性网络回归,各个cpg位点被筛选到的频率。
[0044]
相关系数为在训练集中,各个cpg位点与日历年龄之间的pearson相关系数。
[0045]
p值为各个相关系数对应的p值。
[0046]
步骤四、甲基化年龄预测模型的构建与评价
[0047]
基于上述筛选得到的31个cpg位点,在训练集中利用不同的建模方法构建模型。本发明选择多重线性回归、支持向量机、随机森林和梯度提升回归树这四种方法构建模型。模型评价方面,采用预测年龄和日历年龄的平均绝对误差(mean absolute error,mae)、均方根误差(root mean square error,rmse)以及二者之间的pearson相关系数(r)进行评价。mae和rmse越小,r越大,表示所建立的模型预测效果越好。本发明在训练集中使用bootstrap自助重抽样法(1000次重抽样)进行内部验证,并依据训练集构建好的模型在测试集中进行外部验证。四种建模方法的内部验证和外部验证的结果见表3。
[0048]
表3:31个cpg位点的四种建模方法评价结果
[0049][0050]
注:mae为预测年龄与日历年龄的平均绝对误差。
[0051]
rmse为预测年龄与日历年龄的均方根误差。
[0052]
r为预测年龄与日历年龄的pearson相关系数。
[0053]
由表3结果可知,利用31个cpg位点,各种建模方法均能对年龄进行较好的预测。在这之中,多重线性回归模型在内部验证结果较好的基础上,拥有最好的外部验证结果(最小的mae、rmse和最大的r)。此外,多重线性回归模型结构简单,拥有较好的可解释性,利于模型的实际推广应用。因此,本发明选择多重线性回归作为模型的构建方法。在多重线性回归模型的基础上,本发明利用最优子集回归,对候选的31个cpg位点进一步筛选压缩。最优子集回归利用r语言软件包“leaps”进行,计算cpg位点数从1到31时,所有可能模型的预测情况,选择在各个cpg位点数下最优的模型,共得到31个不同的模型。采用3个模型评价指标mae、rmse、r以及贝叶斯信息准则(bic)对这31个模型进行综合评判。不同cpg位点数下,其最优模型的评价结果见表4;不同cpg位点数下,其最优模型的bic值变化情况见图2.
[0054]
表4:全子集回归中不同cpg位点数下的最优模型评价结果
[0055]
[0056][0057]
注:mae为预测年龄与日历年龄的平均绝对误差。
[0058]
rmse为预测年龄与日历年龄的均方根误差。
[0059]
r为预测年龄与日历年龄的pearson相关系数。
[0060]
由表4结果可知,随着模型中cpg位点数的增加,模型内部验证和外部验证的mae、
rmse一开始快速下降,然后下降速度逐渐放缓;相关系数r一开始快速上升,然后上升速度逐渐放缓。当模型中cpg位点数达到18时,mae、rmse不会出现明显的下降,相关系数r也不会出现明显的上升。同时,由图2结果可知,随着模型中cpg位点数的增加,bic值呈现u型曲线的变化,当模型中cpg位点数为18时,bic达到最小值。因此,本发明认为,18个cpg位点是当前预测模型的最佳位点数,利用该18个cpg位点所建立的多重线性回归模型是当前最优的预测模型,可兼顾纳入较少的cpg位点数和模型预测的准确性。该多重线性回归模型中,18个cpg位点以及截距的具体回归系数见表5。考虑到目前甲基化检测有多种平台,为保证模型在不同检测平台间的可用性,减少因检测平台而造成的系统误差,本发明还计算了这18个cpg位点的标准化回归系数。
[0061]
表5:多重线性回归模型中18个cpg位点的回归系数和标准化回归系数
[0062][0063]
[0064]
注:利用标准化回归系数预测得到的是标准化预测年龄,需要进行去标准化才能得到原始的预测年龄。去标准化的方法为:标准化预测年龄
×
预测人群日历年龄的标准差+预测人群日历年龄的平均值。
[0065]
最后,对这18个cpg位点所建立的多重线性回归模型进行评价,其具体评价结果见表6。
[0066]
表6:18个cpg位点的多重线性回归模型评价结果
[0067][0068]
注:mae为预测年龄与日历年龄的平均绝对误差。
[0069]
rmse为预测年龄与日历年龄的均方根误差。
[0070]
r为预测年龄与日历年龄的pearson相关系数。
[0071]
由表6结果可知,该18个cpg位点的模型在训练集内部验证和测试集外部验证中的表现均良好。在内部验证中,该模型的预测效果优于horvath和hannum所建立的甲基化年龄预测模型(horvath:r=0.97,mae=2.9;hannum:r=0.963,rmse=3.88)。在外部验证中,该模型的预测效果同样优于horvath和hannum模型(horvath:r=0.96,mae=3.6;hannum:r=0.905,rmse=4.89)。
[0072]
步骤五、甲基化年龄预测模型的优化和生物学年龄预测模型的建立
[0073]
上述利用geo数据建立的甲基化年龄预测模型(geo模型)是利用日历年龄作为应变量进行拟合的,这样预测得到的甲基化年龄更多反映的是日历年龄,或者是带有部分生物学年龄信息的“半生物学年龄”。该预测年龄并不能充分反映生物学年龄信息,因此需要对上述甲基化年龄预测模型进行优化,建立生物学年龄预测模型。
[0074]
众多研究表明,在一个自然人群中,人群的衰老状况存在异质性。部分人群由于遗传、行为、环境等因素导致其生物学年龄发生偏离,高于或低于日历年龄(衰老型和年轻型),这部分人群的生物学年龄不等于日历年龄。中间剩余人群的生物学年龄偏离较小(正常型),可认为其生物学年龄等于日历年龄。因此,如果利用中间的正常型人群,以其日历年龄(等价于生物学年龄)作为应变量,对上述geo模型中的18个cpg位点的回归系数进行拟合调整,优化后得到的模型预测的就是生物学年龄,该模型即可认为是生物学年龄预测模型。在本发明中,利用人群的健康信息和geo模型预测的甲基化年龄与日历年龄的偏差来反映人群异质性的情况。基于相关生物学知识和统计学经验,本发明定义异质性人群为:(1)存在严重慢性病的人群;(2)预测偏差最大的30%人群。
[0075]
根据上述优化原理,本发明利用实验室已建立的浙江队列自然人群样本,采用随机抽样的方法,选取其中的1513人进行18个cpg位点的甲基化靶向测序(methyltarget法)。在这之中,排除甲基化靶向测序数据不合格以及年龄、性别信息存在缺失者,最终共得到1399例合格的样本。在这1399例样本中,排除衰老状况异质性的人群:(1)患有心脑血管疾
病、肿瘤、慢性肝病、慢性肾病者(294人);(2)geo模型预测的甲基化年龄与日历年龄的偏差最大的30%人群(332人)。由于甲基化靶向测序(methyltarget法)和geo模型(450k芯片)所用的检测平台不同,因此这里在计算偏差过程中,geo模型所用的回归系数为标准化回归系数。最后,在剩余的773例正常型样本中,以这些样本的日历年龄为应变量,采用多重线性回归的方法,对geo模型中的18个cpg位点的回归系数进行拟合调整,优化后得到的模型可认为是生物学年龄预测模型。该生物学年龄预测模型中18个cpg位点的回归系数见表7。同理,为保证生物学年龄预测模型在不同检测平台间的可用性,减少因检测平台而造成的系统误差,表7也呈现了生物学年龄预测模型中18个cpg位点的标准化回归系数。
[0076]
表7:生物学年龄预测模型中18个cpg位点的回归系数和标准化回归系数
[0077][0078][0079]
注:利用标准化回归系数预测得到的是标准化预测年龄,需要进行去标准化才能
得到原始的预测年龄。去标准化的方法为:标准化预测年龄
×
预测人群日历年龄的标准差+预测人群日历年龄的平均值。
[0080]
对生物学年龄预测模型进行评价,该生物学年龄预测模型在773例正常型样本和1399例总样本(自然人群)中的评价结果见表8。
[0081]
表8:18个cpg位点的生物学年龄预测模型评价结果
[0082][0083]
注:mae为预测年龄与日历年龄的平均绝对误差。
[0084]
rmse为预测年龄与日历年龄的均方根误差。
[0085]
r为预测年龄与日历年龄的pearson相关系数。
[0086]
由表8结果可知,该生物学年龄预测模型在773例正常型样本中的预测偏差相对较小,而在1399例总样本中的预测偏差相对较大。该项结果符合生物学年龄的理论特征,即在正常型人群中,生物学年龄近似等于日历年龄,两者之间偏差较小;而在自然人群中,由于部分人群存在异质性,生物学年龄不等于日历年龄,两者之间的偏差较大。此结果佐证了优化后的模型可以作为生物学年龄预测模型。
[0087]
综上所述,本发明认为,该包含18个cpg位点,经过优化后的模型可以作为一种适合中国人群的、非侵入性和低检测成本的,非血细胞成分相关的实用生物学年龄预测模型。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1