基于DNA存储的信息编码方法、装置、计算机设备及介质
基于dna存储的信息编码方法、装置、计算机设备及介质
技术领域
1.本发明涉及信息存储技术领域,尤其涉及一种基于dna存储的信息编码方法、装置、计算机设备及介质。
背景技术:
2.随着互联网和人工智能等信息技术和数字技术的快速发展,信息量呈指数级飞快增长,磁盘、硬盘、闪存等传统存储介质已经逐渐不能满足全世界范围内数据存储的需要。而将dna作为存储介质存在天然优势: 一是信息密度高,据微软研究院此前估计,1立方毫米的dna能够存储1个eb(exabyte,百亿亿字节)的数据;二是存储时间长、稳定性强,在合适的条件下,可以存储上万年;三是存储能耗很低。
3.dna是一种序列确定的生物大分子。dna存储技术就是将信息编码成碱基序列,从而存储在dna里。随后,dna可以复制、测序,接着经过解码读取里面的信息。在这几个过程中,具有长重复碱基或极端gc含量的dna在合成、复制以及测序时容易出错。如何快速、高效地避免信息编码后的dna序列出现长重复碱基或极端gc含量成为亟待解决的问题。
技术实现要素:
4.本发明实施例提供一种基于dna存储的信息编码方法、装置、计算机设备及介质,以解决如何避免信息编码后的dna序列出现长重复碱基或极端gc含量的问题。
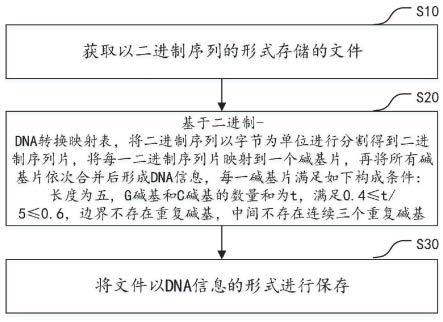
5.一种基于dna存储的信息编码方法,包括:获取以二进制序列的形式存储的文件;基于二进制-dna转换映射表,将二进制序列以字节为单位进行分割得到二进制序列片,将每一二进制序列片映射到一个碱基片,再将所有碱基片依次合并后形成dna信息,每一碱基片满足如下构成条件:长度为五,g碱基和c碱基的数量和为t,满足0.4≤t/5≤0.6,边界不存在重复碱基,中间不存在连续三个重复碱基;将文件以dna信息的形式进行保存。
6.进一步地,在将所有碱基片依次合并后形成dna信息之后,还包括:获取dna信息解码请求,基于dna信息解码请求,获取对应的dna信息;基于二进制-dna转换映射表,将dna信息转换为二进制序列,用于将dna信息转换为文件进行保存。
7.进一步地,在将文件以dna信息的形式进行保存之后,还包括:获取定时任务,当系统时间满足定时任务时,对二进制-dna转换映射表中的映射关系进行更新;或者,给映射关系建立基于公开文档的公开映射关系和基于私密文档的私密映射关系。
8.进一步地,在获取以二进制序列的形式存储的文件之前,还包括:基于碱基片的种类数量大于或者等于二进制序列片的种类数量以及转换便利的
原则,确定以携带八个比特的一个字节作为二进制序列片的分割单位;根据比特的个数8,确定28个二进制序列片分别对应的碱基片。
9.进一步地,确定28个二进制序列片分别对应的碱基片,包括:以四种碱基中的任一种碱基作为每一碱基片的第一位碱基,按照碱基片的构成条件继续合成第二位碱基直至最后一位碱基;以剩余三种碱基中的任一种碱基作为每一碱基片的第二位碱基,重复执行按照构成条件继续合成第二位碱基直至最后一位碱基的步骤,直至碱基片的种类数量等于二进制序列片的种类数量。
10.进一步地,将文件转换为dna碱基序列码的形式进行保存,包括:通过dna合成装置将dna碱基序列码合成为dna溶液或干粉并保存。
11.一种基于dna存储的信息编码装置,包括:获取二进制序列文件模块,用于获取以二进制序列的形式存储的文件;形成dna信息模块,用于基于二进制-dna转换映射表,将所述二进制序列以字节为单位进行分割得到二进制序列片,将每一所述二进制序列片映射到一个碱基片,再将所有所述碱基片依次合并后形成dna信息,每一所述碱基片满足如下构成条件:长度为五,g碱基和c碱基的数量和为t,满足0.4≤t/5≤0.6,边界不存在重复碱基,中间不存在连续三个重复碱基;保存dna信息模块,用于将所述文件以所述dna信息的形式进行保存。
12.一种计算机设备,包括存储器、处理器以及存储在所述存储器中并可在所述处理器上运行的计算机程序,所述处理器执行所述计算机程序时实现上述基于dna存储的信息编码方法。
13.一种计算机可读介质,所述计算机可读介质存储有计算机程序,所述计算机程序被处理器执行时实现上述基于dna存储的信息编码方法。
14.上述基于dna存储的信息编码方法、装置、计算机设备及介质,通过将文件通过具有线性复杂度的构成条件转换为dna信息进行保存,可严格保证编码后的全部dna信息中的连续重复碱基长度最长只有2、gc含量介于0.4和0.6之间,保障了dna信息具有线性复杂度,净信息密度(net information density, nid)为1.60,以有效保障dna的合成、复制和测序流程,提高dna的合成效率,提高信息存储的信息密度和延长信息稳定存储的时间,同时减低储存能耗。
附图说明
15.为了更清楚地说明本发明实施例的技术方案,下面将对本发明实施例的描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。
16.图1绘示本发明一实施例中基于dna存储的信息编码方法的应用环境示意图;图2绘示本发明一实施例中基于dna存储的信息编码方法的流程图;图3绘示本发明另一实施例中基于dna存储的信息编码方法的第一流程图;图4绘示本发明另一实施例中基于dna存储的信息编码方法的编码至解码全流程
的示意图;图5绘示本发明另一实施例中基于dna存储的信息编码方法的第二流程图;图6绘示本发明另一实施例中基于dna存储的信息编码方法的第三流程图;图7绘示本发明另一实施例中基于dna存储的信息编码方法的第四流程图;图8绘示本发明一实施例中基于dna存储的信息编码装置的示意图;图9绘示本发明一实施例中计算机设备的示意图。
具体实施方式
17.下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
18.本发明实施例提供的基于dna存储的信息编码方法,可应用在如图1的应用环境中,该基于dna存储的信息编码方法应用在基于dna存储的信息编码系统中,该基于dna存储的信息编码系统包括客户端和服务器,其中,客户端通过网络与服务器进行通信。客户端又称为用户端,是指与服务器相对应,为客户端提供本地服务的程序。进一步地,客户端为计算机端程序、智能设备的app程序或嵌入其他app的第三方小程序。该客户端可安装在但不限于各种个人计算机、笔记本电脑、智能手机、平板电脑和便携式可穿戴设备等计算机设备上。服务器可以用独立的服务器或者是多个服务器组成的服务器集群来实现。
19.传统的存储技术将信息以二进制序列(也即由0和1组成的序列)存到硬盘、光盘、u盘、cd等媒介中,比如音乐、图片、视频等文件在计算机的底层都是二进制序列。由于其机械特性,这些媒介经过读或写达到一定次数后会发生故障。另外,它们信息密度低、体积大,随着全球信息的爆炸式增长,已经难以满足存储需求。
20.在一实施例中,如图2所示,提供一种基于dna存储的信息编码方法,以该方法应用在图1中的服务器为例进行说明,具体包括如下步骤:s10.获取以二进制序列的形式存储的文件。
21.其中,二进制序列是文件以二进制形式保存的(只有0和1组成)的数字序列编码。
22.具体地,本实施例可对已二进制保存的各种形式的文档进行dna编码,各种形式的文档包括语音、文字、图像以及音乐等,此处不作具体限定。
23.s20.基于二进制-dna转换映射表,将二进制序列以字节为单位进行分割得到二进制序列片,将每一二进制序列片映射到一个碱基片,再将所有碱基片依次合并后形成dna信息,每一碱基片满足如下构成条件:长度为五,g碱基和c碱基的数量和为t,满足0.4≤t/5≤0.6,边界不存在重复碱基,中间不存在连续三个重复碱基。
24.其中,二进制-dna转换映射表是二进制编码和dna编码之间进行互相映射的表,比如,该二进制-dna转换映射表中的一组数据为:00000000 《
‑‑
》 atacg,表示二进制为00000000的数据可通过本实施例的映射后成为已dna编码形式atacg进行保存。
25.dna碱基序列码即为所有n个碱基为单位的碱基片合成后的编码。
26.具体地,构成dna的含氮碱基有四种:腺嘌呤(a)、鸟嘌呤(g)、胸腺嘧啶(t)和胞嘧啶(c)。本实施例通过二进制-dna转换映射表将以二进制序列保存的文件转换为以n个碱基
为单位的碱基片合成后的dna信息进行保存。
27.本实施例可采用8比特也即一个字节作为二进制序列的单位。举例说明,可将文件对应的连续二进制原始码按字节进行拆分后得到多个二进制序列片,每个序列片含8个比特,二进制序列即为多个8比特的二进制序列片合成的序列码。
28.举例说明,将文件的二进制码按一个字节(也就是有8个二进制位的二进制序列片,这样的二进制序列片一共有256个)一组分割,每一组映射到5个合适的碱基片(这样的碱基片共有256个)后进行合成,可得到二进制序列。
29.可以理解地,解码过程即为编码过程的逆序过程:将dna序列码按5个碱基一组分割,每组映射到一个有8个二进制位的二进制序列片。
30.s30.将文件以dna信息的形式进行保存。
31.具体地,将文件以dna信息的形式也即生物大分子的形式保存,可有效延长信息稳定存储的时间,同时减低储存能耗。
32.本实施例提供的基于dna存储的信息编码方法,通过将文件通过具有线性复杂度的构成条件转换为dna信息进行保存,可严格保证编码后的全部dna信息中的连续重复碱基长度最长只有2、gc含量介于0.4和0.6之间,保障了dna信息具有线性复杂度,净信息密度(net information density, nid)为1.60,以有效保障dna的合成、复制和测序流程,提高dna的合成效率,提高信息存储的信息密度和延长信息稳定存储的时间,同时减低储存能耗。
33.在一具体实施例中,如图3所示,在步骤s20之后,即在将所有碱基片依次合并后形成dna信息之后,还具体包括如下步骤:s201.获取dna信息解码请求,基于dna信息解码请求,获取对应的dna信息。
34.s202.基于二进制-dna转换映射表,将dna信息转换为二进制序列,用于将dna信息转换为文件进行保存。
35.其中,dna解码请求是将已dna信息存储的文件解码为以二进制序列存储的请求,也即解码是编码的逆向过程。
36.具体地,本实施例依旧采用二进制-dna转换映射表,将dna信息按碱基片进行分割后映射为二进制序列。优选地,二进制-dna转换映射表中包括至少一组碱基片和二进制序列片的映射关系,该映射关系也即每个二进制序列片和每个碱基片之间的二进制码和五个碱基的排列的具体对应关系。
37.进一步地,从编码到解码的全流程举例说明本实施例的实现流程:a编码过程:分割:将输入信息的二进制序列按8个二进制位一组分割。这样一定得到整数个组,因为8个二进制位是一个字节,而文件是以字节为单位存储,其大小一定是字节的整数倍。以图4中的输入信息为例,“01100011 10001101 01011011 10001110 10111011 00110111 01011000”一共分割成7组。
38.映射:按照二进制-dna转换映射表提供的映射关系,查询每一组二进制序列片对应的5-碱基片。比如“01100011”对应到“tcgta”,其他的类推。这样得到7组5-碱基片。
39.合并:将所得碱基片合并,就得到所需dna信息。该dna信息随后可合成dna作为信息记录载体。
40.b解码过程:分割:将要解码的dna信息按5个碱基一组分割。这样也一定会得到整数个组,因为前面编码时就是整数个组。比如“tcgta cactg tctct cacga cgtct agtgc tctac”分割为7组。
41.映射:按照二进制-dna转换映射表提供的映射关系,查询每一组碱基片对应的8-二进制序列片。比如“tcgta”对应到“01100011”,其余类推。这样得到7组8-二进制序列片。
42.合并:将所得二进制序列片合并,就得到解码后的信息。
43.本实施例用于实现从dna作为存储介质的载体中读取并解码出数据进行后续处理,是快速高效以及稳健的编解码方式。
44.在一具体实施例中,如图5所示,在步骤s30之后,即在将文件以dna信息的形式进行保存之后,还具体包括如下步骤:s3011.获取定时任务,当系统时间满足定时任务时,对二进制-dna转换映射表中的映射关系进行更新;或者,s3012给映射关系建立基于公开文档的公开映射关系和基于私密文档的私密映射关系。
45.具体地,为了加强文件的保存安全性和可靠性,本实施例提供的方法可定期更新二进制-dna转换映射表中的映射关系,也即将每个二进制序列片和每个碱基片中的五个碱基的排列顺序进行变更。或者,给可公开文档建立一套可公开的映射关系给大众使用,同时,建立私密的私密映射关系给具有安全性或私密性的文档来使用。
46.在一具体实施例中,如图6所示,在步骤s10之前,即在获取以二进制序列的形式存储的文件之前,还具体包括如下步骤:s101.基于碱基片的种类数量大于或者等于二进制序列片的种类数量以及转换便利的原则,确定以携带八个比特的一个字节作为二进制序列片的分割单位。
47.s102.根据比特的个数8,确定28个二进制序列片分别对应的碱基片。
48.具体地,为了使算法的复杂度达到线性,本技术将文件的二进制序列切割成一定长度的二进制序列片,再将每个二进制序列片映射到合适的dna碱基片。碱基片的种类数量大于或者等于二进制序列片的种类数量的原则,即为4n≥28,并可推导出2n≥8。其中,28是二进制序列片的总数。为了节省碱基片资源,本实施例中可取n的最小值作为碱基片的碱基个数。
49.举例说明,由于计算机上的文件以字节为单位存储,每个字节是8位,二进制序列片的长度可以固定为8,这样二进制序列片的总数共有28=256种。接下来需确定256个合适的碱基片。
50.当即一字节数为二进制序列片的单元时,n至少为4。然而长度为4的碱基片中很大
一部分具有很长的连续重复碱基(比如aaat有3个连续重复碱基)或极端gc含量(比如gcct的gc含量是75%)。这样的碱基片组合起来得到的碱基序列的连续重复碱基更长,gc含量也无法控制。所以x不能取4,至少应该取5。
51.本实施例提供的方法将文件转换为多个二进制序列片合成的二进制序列,利于后续通过二进制序列片和碱基片之间的映射关系实现保存格式的迅速转换。优选地,过二进制序列片和碱基片之间的映射关系记录与二进制-dna转换映射表,该表包括至少一组碱基片和二进制序列片的映射关系。本实施例可基于二进制序列片的总数适配出对应的碱基片的个数,节省碱基片的存储资源。
52.在一具体实施例中,如图7所示,在步骤s102中,即确定28个二进制序列片分别对应的碱基片,具体包括如下步骤:s1021.以四种碱基中的任一种碱基作为每一碱基片的第一位碱基,按照碱基片的构成条件继续合成第二位碱基直至最后一位碱基。
53.s1022.以剩余三种碱基中的任一种碱基作为每一碱基片的第二位碱基,重复执行按照构成条件继续合成第二位碱基直至最后一位碱基的步骤,直至碱基片的种类数量等于二进制序列片的种类数量。
54.具体地,继续以n=5举例进行说明。从长度为5的所有碱基片中筛选出256个合适的可作为存储介质的碱基片。
55.令abcde为长度为5的碱基片,其中a、b、c、d、e的取值都在集合{a,t,c,g}中。考虑到连续重复碱基的长度,它们应该满足这样的条件:条件1:a≠b,d≠e;条件2:b、c、d三个不全部相同。
56.条件1是说在碱基片的边界上不能有重复碱基,以避免组合后出现3或3个以上连续重复碱基(比如aatcg和tcgga组合就会出现3个连续重复碱基aaa)。条件2是说碱基片内部不能出现3个连续重复碱基,这也是为了避免连续重复碱基过长。另外,令t为碱基片中g和c的数量之和。为了控制编码后的碱基序列中gc含量,t应当满足如下条件:条件3:0.4≤t/5≤0.6也就是说,每个碱基片的gc含量都介于0.4和0.6之间,从而编码后的碱基序列的gc含量也位于这个区间。解出条件3得到2≤t≤3,意味着碱基片中g和c的数量之和只能是2或3。
57.根据以上条件,就能筛选出合适的碱基片。
58.从a开始,可以取{a,t,c,g}中任何一个,有四种可能。由于b 不能跟a相同,b只有3种可能。也就是说,假如a取a,b就不能取a,只能取t、c、g。c的取值有四种,因为它处于碱基片的中间,前面只有两个碱基。d 的取值要谨慎,必须保证不能使b、c、d三个全部相同,而且g和c的总数不能少于1或超过3(此时可以是1、2或3,因为后面还有一个碱基e)。比如,假若前面是gtt,那d不能再取t,否则违背条件2,d可以取a、c或g。接下来,e的取值也要具体看前面几个。e 不能跟d相同,而且要保证g和c的总数为2或3.比如,假若前面是gtta,那e不能取a,否则违背条件1;进一步地,e只能取g或c来满足条件3。这样,就筛选除了合适的碱基片,如下文所示的二进制-dna转换映射表中的映射关系,其中符号“《
‑‑
》”表示映射,该符号左
侧是长度为8的二进制序列片(称为8-二进制序列片),右侧是长度为5的碱基片(称为5-碱基片):00000000 《
‑‑
》 atacg 00000001 《
‑‑
》 atagc 00000010 《
‑‑
》 atcac00000011 《
‑‑
》 atcag 00000100 《
‑‑
》 atctc 00000101 《
‑‑
》 atctg00000110 《
‑‑
》 atcga 00000111 《
‑‑
》 atcgt 00001000 《
‑‑
》 atcgc00001001 《
‑‑
》 atgac 00001010 《
‑‑
》 atgag 00001011 《
‑‑
》 atgtc00001100 《
‑‑
》 atgtg 00001101 《
‑‑
》 atgca 00001110 《
‑‑
》 atgct00001111 《
‑‑
》 atgcg 00010000 《
‑‑
》 acatc 00010001 《
‑‑
》 acatg00010010 《
‑‑
》 acaca 00010011 《
‑‑
》 acact 00010100 《
‑‑
》 acacg00010101 《
‑‑
》 acaga 00010110 《
‑‑
》 acagt 00010111 《
‑‑
》 acagc00011000 《
‑‑
》 actac 00011001 《
‑‑
》 actag 00011010 《
‑‑
》 actca00011011 《
‑‑
》 actct 00011100 《
‑‑
》 actcg 00011101 《
‑‑
》 actga00011110 《
‑‑
》 actgt 00011111 《
‑‑
》 actgc 00100000 《
‑‑
》 acgat00100001 《
‑‑
》 acgac 00100010 《
‑‑
》 acgag 00100011 《
‑‑
》 acgta00100100 《
‑‑
》 acgtc 00100101 《
‑‑
》 acgtg 00100110 《
‑‑
》 acgca00100111 《
‑‑
》 acgct 00101000 《
‑‑
》 agatc 00101001 《
‑‑
》 agatg00101010 《
‑‑
》 agaca 00101011 《
‑‑
》 agact 00101100 《
‑‑
》 agacg00101101 《
‑‑
》 agaga 00101110 《
‑‑
》 agagt 00101111 《
‑‑
》 agagc00110000 《
‑‑
》 agtac 00110001 《
‑‑
》 agtag 00110010 《
‑‑
》 agtca00110011 《
‑‑
》 agtct 00110100 《
‑‑
》 agtcg 00110101 《
‑‑
》 agtga00110110 《
‑‑
》 agtgt 00110111 《
‑‑
》 agtgc 00111000 《
‑‑
》 agcat00111001 《
‑‑
》 agcac 00111010 《
‑‑
》 agcag 00111011 《
‑‑
》 agcta00111100 《
‑‑
》 agctc 00111101 《
‑‑
》 agctg 00111110 《
‑‑
》 agcga00111111 《
‑‑
》 agcgt 01000000 《
‑‑
》 tatcg 01000001 《
‑‑
》 tatgc01000010 《
‑‑
》 tacac 01000011 《
‑‑
》 tacag 01000100 《
‑‑
》 tactc01000101 《
‑‑
》 tactg 01000110 《
‑‑
》 tacga 01000111 《
‑‑
》 tacgt01001000 《
‑‑
》 tacgc 01001001 《
‑‑
》 tagac 01001010 《
‑‑
》 tagag01001011 《
‑‑
》 tagtc 01001100 《
‑‑
》 tagtg 01001101 《
‑‑
》 tagca01001110 《
‑‑
》 tagct 01001111 《
‑‑
》 tagcg 01010000 《
‑‑
》 tcatc01010001 《
‑‑
》 tcatg 01010010 《
‑‑
》 tcaca 01010011 《
‑‑
》 tcact01010100 《
‑‑
》 tcacg 01010101 《
‑‑
》 tcaga 01010110 《
‑‑
》 tcagt01010111 《
‑‑
》 tcagc 01011000 《
‑‑
》 tctac 01011001 《
‑‑
》 tctag01011010 《
‑‑
》 tctca 01011011 《
‑‑
》 tctct 01011100 《
‑‑
》 tctcg01011101 《
‑‑
》 tctga 01011110 《
‑‑
》 tctgt 01011111 《
‑‑
》 tctgc01100000 《
‑‑
》 tcgat 01100001 《
‑‑
》 tcgac 01100010 《
‑‑
》 tcgag01100011 《
‑‑
》 tcgta 01100100 《
‑‑
》 tcgtc 01100101 《
‑‑
》 tcgtg01100110 《
‑‑
》 tcgca 01100111 《
‑‑
》 tcgct 01101000 《
‑‑
》 tgatc01101001 《
‑‑
》 tgatg 01101010 《
‑‑
》 tgaca 01101011 《
‑‑
》 tgact01101100 《
‑‑
》 tgacg 01101101 《
‑‑
》 tgaga 01101110 《
‑‑
》 tgagt
01101111 《
‑‑
》 tgagc 01110000 《
‑‑
》 tgtac 01110001 《
‑‑
》 tgtag01110010 《
‑‑
》 tgtca 01110011 《
‑‑
》 tgtct 01110100 《
‑‑
》 tgtcg01110101 《
‑‑
》 tgtga 01110110 《
‑‑
》 tgtgt 01110111 《
‑‑
》 tgtgc01111000 《
‑‑
》 tgcat 01111001 《
‑‑
》 tgcac 01111010 《
‑‑
》 tgcag01111011 《
‑‑
》 tgcta 01111100 《
‑‑
》 tgctc 01111101 《
‑‑
》 tgctg01111110 《
‑‑
》 tgcga 01111111 《
‑‑
》 tgcgt 10000000 《
‑‑
》 catac10000001 《
‑‑
》 catag 10000010 《
‑‑
》 catca 10000011 《
‑‑
》 catct10000100 《
‑‑
》 catcg 10000101 《
‑‑
》 catga 10000110 《
‑‑
》 catgt10000111 《
‑‑
》 catgc 10001000 《
‑‑
》 cacat 10001001 《
‑‑
》 cacac10001010 《
‑‑
》 cacag 10001011 《
‑‑
》 cacta 10001100 《
‑‑
》 cactc10001101 《
‑‑
》 cactg 10001110 《
‑‑
》 cacga 10001111 《
‑‑
》 cacgt10010000 《
‑‑
》 cagat 10010001 《
‑‑
》 cagac 10010010 《
‑‑
》 cagag10010011 《
‑‑
》 cagta 10010100 《
‑‑
》 cagtc 10010101 《
‑‑
》 cagtg10010110 《
‑‑
》 cagca 10010111 《
‑‑
》 cagct 10011000 《
‑‑
》 ctatc10011001 《
‑‑
》 ctatg 10011010 《
‑‑
》 ctaca 10011011 《
‑‑
》 ctact10011100 《
‑‑
》 ctacg 10011101 《
‑‑
》 ctaga 10011110 《
‑‑
》 ctagt10011111 《
‑‑
》 ctagc 10100000 《
‑‑
》 ctcat 10100001 《
‑‑
》 ctcac10100010 《
‑‑
》 ctcag 10100011 《
‑‑
》 ctcta 10100100 《
‑‑
》 ctctc10100101 《
‑‑
》 ctctg 10100110 《
‑‑
》 ctcga 10100111 《
‑‑
》 ctcgt10101000 《
‑‑
》 ctgat 10101001 《
‑‑
》 ctgac 10101010 《
‑‑
》 ctgag10101011 《
‑‑
》 ctgta 10101100 《
‑‑
》 ctgtc 10101101 《
‑‑
》 ctgtg10101110 《
‑‑
》 ctgca 10101111 《
‑‑
》 ctgct 10110000 《
‑‑
》 cgata10110001 《
‑‑
》 cgatc 10110010 《
‑‑
》 cgatg 10110011 《
‑‑
》 cgaca10110100 《
‑‑
》 cgact 10110101 《
‑‑
》 cgaga 10110110 《
‑‑
》 cgagt10110111 《
‑‑
》 cgtat 10111000 《
‑‑
》 cgtac 10111001 《
‑‑
》 cgtag10111010 《
‑‑
》 cgtca 10111011 《
‑‑
》 cgtct 10111100 《
‑‑
》 cgtga10111101 《
‑‑
》 cgtgt 10111110 《
‑‑
》 cgcat 10111111 《
‑‑
》 cgcta11000000 《
‑‑
》 gatac 11000001 《
‑‑
》 gatag 11000010 《
‑‑
》 gatca11000011 《
‑‑
》 gatct 11000100 《
‑‑
》 gatcg 11000101 《
‑‑
》 gatga11000110 《
‑‑
》 gatgt 11000111 《
‑‑
》 gatgc 11001000 《
‑‑
》 gacat11001001 《
‑‑
》 gacac 11001010 《
‑‑
》 gacag 11001011 《
‑‑
》 gacta11001100 《
‑‑
》 gactc 11001101 《
‑‑
》 gactg 11001110 《
‑‑
》 gacga11001111 《
‑‑
》 gacgt 11010000 《
‑‑
》 gagat 11010001 《
‑‑
》 gagac11010010 《
‑‑
》 gagag 11010011 《
‑‑
》 gagta 11010100 《
‑‑
》 gagtc11010101 《
‑‑
》 gagtg 11010110 《
‑‑
》 gagca 11010111 《
‑‑
》 gagct11011000 《
‑‑
》 gtatc 11011001 《
‑‑
》 gtatg 11011010 《
‑‑
》 gtaca11011011 《
‑‑
》 gtact 11011100 《
‑‑
》 gtacg 11011101 《
‑‑
》 gtaga11011110 《
‑‑
》 gtagt 11011111 《
‑‑
》 gtagc 11100000 《
‑‑
》 gtcat11100001 《
‑‑
》 gtcac 11100010 《
‑‑
》 gtcag 11100011 《
‑‑
》 gtcta
11100100 《
‑‑
》 gtctc 11100101 《
‑‑
》 gtctg 11100110 《
‑‑
》 gtcga11100111 《
‑‑
》 gtcgt 11101000 《
‑‑
》 gtgat 11101001 《
‑‑
》 gtgac11101010 《
‑‑
》 gtgag 11101011 《
‑‑
》 gtgta 11101100 《
‑‑
》 gtgtc11101101 《
‑‑
》 gtgtg 11101110 《
‑‑
》 gtgca 11101111 《
‑‑
》 gtgct11110000 《
‑‑
》 gcata 11110001 《
‑‑
》 gcatc 11110010 《
‑‑
》 gcatg11110011 《
‑‑
》 gcaca 11110100 《
‑‑
》 gcact 11110101 《
‑‑
》 gcaga11110110 《
‑‑
》 gcagt 11110111 《
‑‑
》 gctat 11111000 《
‑‑
》 gctac11111001 《
‑‑
》 gctag 11111010 《
‑‑
》 gctca 11111011 《
‑‑
》 gctct11111100 《
‑‑
》 gctga 11111101 《
‑‑
》 gctgt 11111110 《
‑‑
》 gcgat11111111 《
‑‑
》 gctta在一具体实施例中,在步骤s20中,即将文件转换为dna碱基序列码的形式进行保存,具体包括如下步骤:s21.通过dna合成装置将dna碱基序列码合成为dna溶液或干粉并保存。
59.具体地,通过寡链核苷酸的拼接,现有的多种技术已经可以人工合成特定的dna序列,其中化学法已经成熟,酶促合成法正在发展。化学法分为去保护、偶联、加帽(可选)及氧化四个步骤,特点是出现时间早,需要使用有毒试剂。酶促法相对温和,较少损伤dna,准确性较高,副产物较少。
60.dna与生物息息相关,不会像其它存储介质一样被时代淘汰。dna的存储密度非常高,目前世界上最紧凑的硬盘存储密度仅仅是它的千分之一。利用dna存储数据,可在一粒盐的体积中储存10部完整的高清电影。dna是生物学研究的核心,随着时间的推移和技术的成熟,在dna上存取数据会越来越方便。
61.分析本技术提供的方法的复杂度。8为输入文件的二进制序列的长度,则实施编码或解码的映射所需的步数是关于8的线性函数,因而编码和解码都是线性复杂度。本发明的方法将每8个二进制位映射到5个四进制碎片,净信息密度为8/5=1.60。
62.进一步地,本实施例提出的基于dna存储的信息编码方法可通过快速、高编码效率和稳健的构成条件将原存储为二进制的各种信息存储到dna信息中,净信息密度(net information density, nid)可达1.60,且编码后的全部dna序列中的连续重复碱基(homopolymer)长度最长只有2个碱基,gc含量严格控制在40%至60%之间。
63.应理解,上述实施例中各步骤的序号的大小并不意味着执行顺序的先后,各过程的执行顺序应以其功能和内在逻辑确定,而不应对本发明实施例的实施过程构成任何限定。
64.在一实施例中,提供一种基于dna存储的信息编码装置,该基于dna存储的信息编码装置与上述实施例中基于dna存储的信息编码方法一一对应。如图8所示,该基于dna存储的信息编码装置包括获取二进制序列文件模块10、形成dna信息模块20和保存dna信息模块30。各功能模块详细说明如下:获取二进制序列文件模块10,用于获取以二进制序列的形式存储的文件;形成dna信息模块20,用于基于二进制-dna转换映射表,将所述二进制序列以字节为单位进行分割得到二进制序列片,将每一所述二进制序列片映射到一个碱基片,再将所有所述碱基片依次合并后形成dna信息,每一所述碱基片满足如下构成条件:长度为五,g碱
基和c碱基的数量和为t,满足0.4≤t/5≤0.6,边界不存在重复碱基,中间不存在连续三个重复碱基;保存dna信息模块30,用于将所述文件以所述dna信息的形式进行保存。
65.关于基于dna存储的信息编码装置的具体限定可以参见上文中对于基于dna存储的信息编码方法的限定,在此不再赘述。上述基于dna存储的信息编码装置中的各个模块可全部或部分通过软件、硬件及其组合来实现。上述各模块可以硬件形式内嵌于或独立于计算机设备中的处理器中,也可以以软件形式存储于计算机设备中的存储器中,以便于处理器调用执行以上各个模块对应的操作。
66.在一实施例中,提供了一种计算机设备,该计算机设备可以是服务器,其内部结构图可以如图9所示。该计算机设备包括通过系统总线连接的处理器、存储器、网络接口和数据库。其中,该计算机设备的处理器用于提供计算和控制能力。该计算机设备的存储器包括非易失性介质、内存储器。该非易失性介质存储有操作系统、计算机程序和数据库。该内存储器为非易失性介质中的操作系统和计算机程序的运行提供环境。该计算机设备的数据库用于基于dna存储的信息编码方法相关的数据。该计算机设备的网络接口用于与外部的终端通过网络连接通信。该计算机程序被处理器执行时以实现一种基于dna存储的信息编码方法。
67.在一实施例中,提供一种计算机设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,处理器执行计算机程序时实现上述实施例基于dna存储的信息编码方法,例如图2所示s10至步骤s20。或者,处理器执行计算机程序时实现上述实施例中基于dna存储的信息编码装置的各模块/单元的功能,例如图8所示模块10至模块20的功能。为避免重复,此处不再赘述。
68.在一实施例中,提供一种计算机可读介质,其上存储有计算机程序,计算机程序被处理器执行时实现上述实施例基于dna存储的信息编码方法,例如图2所示s10至步骤s20。或者,该计算机程序被处理器执行时实现上述装置实施例中基于dna存储的信息编码装置中各模块/单元的功能,例如图8所示模块10至模块20的功能。为避免重复,此处不再赘述。
69.本领域普通技术人员可以理解实现上述实施例方法中的全部或部分流程,是可以通过计算机程序来指令相关的硬件来完成,该计算机程序可存储于一非易失性计算机可读取介质中,该计算机程序在执行时,可包括如上述各方法的实施例的流程。其中,本技术各实施例中所使用的对存储器、存储、数据库或其它介质的任何引用,均可包括非易失性和/或易失性存储器。非易失性存储器可包括只读存储器(rom)、可编程rom(prom)、电可编程rom(eprom)、电可擦除可编程rom(eeprom)或闪存。易失性存储器可包括随机存取存储器(ram)或者外部高速缓冲存储器。作为说明而非局限,ram以多种形式可得,诸如静态ram(sram)、动态ram(dram)、同步dram(sdram)、双数据率sdram(ddrsdram)、增强型sdram(esdram)、同步链路(synchlink) dram(sldram)、存储器总线(rambus)直接ram(rdram)、直接存储器总线动态ram(drdram)、以及存储器总线动态ram(rdram)等。
70.所属领域的技术人员可以清楚地了解到,为了描述的方便和简洁,仅以上述各功能单元、模块的划分进行举例说明,实际应用中,可以根据需要而将上述功能分配由不同的功能单元、模块完成,即将所述装置的内部结构划分成不同的功能单元或模块,以完成以上描述的全部或者部分功能。
71.以上实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述实施例对本发明进行了详细的说明,本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明各实施例技术方案的精神和范围,均应包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1