基于GRNN的肿瘤基因点突变特征图谱提取与分类方法
基于grnn的肿瘤基因点突变特征图谱提取与分类方法
技术领域
1.本发明属于基因点突变特征图谱提取与分类领域,尤其是一种基于grnn的肿瘤基因点突变特征图谱提取与分类方法。
背景技术:
2.dna微阵列也叫基因芯片,是近几年发展起来的一种能快速、高效检测dna片段序列、基因表达水平的新技术,通过dna微阵列芯片实验人们可以得到基因表达谱数据,通过分析这些数据,人们可以挖掘出具有生物学意义的信息和知识。当前生物信息学研究的重要领域是如何从基因表达谱数据中选取包含样本分类信息的特征基因,建立分类器,实现肿瘤的分型诊断。但由于肿瘤基因表达谱数据样本呈现样本少、维数高、信息基因少和高噪声等特点,阻碍了研究者高效地从基因微阵列中选取肿瘤相关基因,即信息基因。
3.近年来,研究学者提出多种肿瘤数据挖掘方法,主要有:聚类分析、主成分分析(pca) 、独立分量分析(ica)、k-近邻(k-nn)、非负矩阵分解 (nmf)、自组织映射(som)、支持向量机(svm)、人工神经网络(ann)、贝叶斯神经网络等经典常用的分类方法,取得了较好的分类效果。但由于生物信息学中基因特征表达谱数据维度的急剧增加,导致“维数灾难”,且产生的大量无关信息或冗余信息,增加计算复杂度,造成肿瘤特征基因选择算法普遍存在泛化能力差和运行效率较低的问题。
4.广义回归神经网络(grnn)是径向基网络的另外一种变形形式,具有良好的非线性逼近性能,训练方便,在许多学科和工程领域得到了广泛的应用,但目前并未见将grnn应用于肿瘤基因点突变的特征图谱提取与分类的报导。
技术实现要素:
5.本发明所要解决的技术问题是提供一种基于grnn的肿瘤基因点突变特征图谱提取与分类方法,应用grnn对特征提取的基因进行分类,提高运行效率的同时提高基因表达谱数据的样本分类正确率。
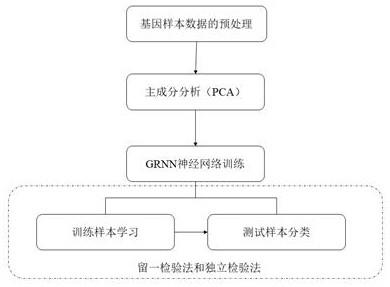
6.为解决上述问题,本发明采用的技术方案为:基于grnn的肿瘤基因点突变特征图谱提取与分类方法,包括以下步骤:s1、预处理:对基因样本数据进行预处理;s2、降维处理:采用主成分分析方法对预处理后的基因样本数据进行降维处理,得到基因样本数据的主成分分量;s3、分类模型训练和最优基因组合筛选:采用grnn对主成分分量进行学习,训练网络权值,得到网络模型和最优基因组合;s4、采用步骤s3得到的网络模型对肿瘤样本基因进行分类,并采用留一检验法和独立检验法评估分类准确性。
7.进一步地,步骤s1中,预处理方法为:基因样本数据组成矩阵,对矩阵中的数据进行标准化处理,得到矩阵,
矩阵的均值为0,方差为1。
8.进一步地,步骤s2包括:s21、根据公式计算矩阵的相关系数矩阵,其中,i为行数,j为列数;s22、对于相关系数矩阵,采用雅可比方法求特征方程的个特征值、
……
,且,的特征向量为,并且满足;s23、选择k个主成分分量,使得k个主成分的方差和占全部总方差的比例并使所选的这个主成分尽可能多地保留原来个基因的信息,得到的主成分矩阵记为。
9.进一步地,步骤s3包括:s31、假设是两个随机变量,其联合概率密度为,若已知的观测值为相对的回归为:的回归为:即在输入为的条件下,的预测输出;s32、应用非参数估计,由步骤s23所得的主成分矩阵数据集按以下公式估算密度函数以下公式估算密度函数以下公式估算密度函数其中,为样本容量,为随机变量的维数,为光滑因子;s33、将步骤s32的公式代入步骤s31的公式,可得
。
10.进一步地,步骤s4包括:s41、从步骤s3得到的最优基因组合中选取最优基因作为1样本;s42、构建训练集和测试集,肿瘤样本基因作为0样本,将1样本和0样本随机分配到训练集和测试集中;s43、采用步骤s3得到的网络模型对训练集和测试集进行分类,如果平均收敛曲线成功收敛,则得到分类结果;若得到分类结果,如果平均收敛曲线未收敛,则重复步骤s41至s43,直到平均收敛曲线成功收敛。
11.进一步地,步骤s42中,1样本和0样本的比例为2:1。
12.本发明的有益效果是:本发明利用grnn对特征提取的基因进行分类,构建更具解释性的混合肿瘤基因分类模型,提高了基因表达谱数据的样本分类正确率。实验结果表明本发明构建grnn能够有效的提取与肿瘤相关的信息基因,且分类性能较稳定,尤其是在分类样本较少,类别不平衡时具有明显的优势。
附图说明
13.图1是本发明的流程图;图2是本发明对结肠癌肿瘤(colorectal cancer)进行基因降维分类结果的示意图;图3是本发明对支气管肺癌(lung cancer)进行基因降维分类结果的示意图。
具体实施方式
14.下面结合附图和实施例对本发明进一步说明。
15.本发明的基于grnn的肿瘤基因点突变特征图谱提取与分类方法,对结肠癌肿瘤(colorectal cancer)进行基因降维分类,如图1所示,包括以下步骤:s1、预处理:对基因样本数据进行预处理。
16.预处理的具体方法为:基因样本数据组成矩阵,对矩阵中的数据进行标准化处理,得到矩阵,矩阵的均值为0,方差为1。标准化处理可消除量纲对评价结果的影响。
17.s2、降维处理:采用主成分分析方法对预处理后的基因样本数据进行降维处理,得到基因样本数据的主成分分量。具体包括s21、根据公式计算矩阵的相关系数矩阵,其中,i为行数,j为列数;
s22、对于相关系数矩阵,采用雅可比方法求特征方程的个特征值、
……
,且,的特征向量为,并且满足;s23、选择k个主成分分量,使得k个主成分的方差和占全部总方差的比例并使所选的这个主成分尽可能多地保留原来个基因的信息,得到的主成分矩阵记为。
18.采用上述数据处理措施后选取的特征基因子集中含15个基因:x53799、m29273、u21914、l00352、d14520、x90858、r80427、x75208、d29808、m59807、d13627、m22760、r56070、y00062、r50158。
19.s3、分类模型训练和最优基因组合筛选:采用grnn对主成分分量(特征提取后的基因数据形成的个候选基因子集)进行学习,训练网络权值,得到网络模型和最优基因组合。具体包括s31、假设是两个随机变量,其联合概率密度为,若已知的观测值为相对的回归为:的回归为:即在输入为的条件下,的预测输出;s32、应用非参数估计,由步骤s23所得的主成分矩阵数据集按以下公式估算密度函数以下公式估算密度函数以下公式估算密度函数其中,为样本容量,为随机变量的维数,为光滑因子;s33、将步骤s32的公式代入步骤s31的公式,并交换积分与求和的顺序,有
由于所以最终可得。
20.s4、采用步骤s3得到的网络模型对肿瘤样本基因进行分类,并采用留一检验法和独立检验法评估分类准确性。具体过程为:s41、从步骤s3得到的最优基因组合中选取最优基因作为1样本;s42、构建训练集和测试集:肿瘤样本基因作为0样本,将1样本和0样本随机分配到训练集和测试集中,1样本和0样本的比例为2:1左右。
21.s43、采用步骤s3得到的网络模型对训练集和测试集进行分类,如果平均收敛曲线成功收敛,则得到分类结果;若得到分类结果,如果平均收敛曲线未收敛,则重复步骤s41至s43,直到平均收敛曲线成功收敛。
22.采用上述方法对结肠癌肿瘤(colorectal cancer)进行基因降维分类的结果如图2所示,在10次实验中,迭代次数200左右,随着训练次数增加,平均损失数函数值基本保持不变,并趋于稳定,构建验证集并对验证集运用留一交叉校准,准确率平均为97.5%,对测试集运用留一交叉校准准,准确率平均为96.7%。
23.此外,采用本发明对结肠癌数据集进行分类,结果如图3所示,在10次实验中,迭代次数200左右,随着训练次数增加,平均损失数函数值基本保持不变,并趋于稳定,构建验证集并对验证集运用留一交叉校准,准确率平均为97.2%,对测试集运用留一交叉校准,准确率平均为96.9%。
24.可见,本发明可以在迭代次数较低的情况下保证基因表达谱数据的样本分类的准确率,提高分类效率,尤其是在分类样本较少,类别不平衡时具有明显的优势。
25.以上所述仅为本发明的优选实施例而已,并不用于限制本发明,对于本领域的技术人员来说,本发明可以有各种更改和变化。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1