一种基因编辑点突变方案自动设计方法及系统与流程
1.本发明涉及基因编辑的技术领域,特别是一种基因编辑点突变方案自动设计方法及系统。
背景技术:
2.在现有技术中,crispr/cas9通过设计特异性向导rna识别靶序列引导cas9核酸内切酶对靶序列的pam上游进行切割,使靶位点dna双链断裂;dna双链断裂后,利用细胞的非同源末端连接(nhej)或同源重组(hdr)的方式对切割位点进行修复,实现dna水平的基因敲除、敲入或点突变。crispr/cas9以其操作简单、成本低且编辑效率等优势,成为生命科学领域最炙手可热的技术之一,已经广泛应用于诸多模式生物的基因编辑相关功能研究中,如哺乳动物(大鼠、小鼠、猪、兔、猴等)、斑马鱼、干细胞、肿瘤细胞系及细菌真菌等。
3.基于crispr/cas9的点突变基因编辑方案的设计看似不难,但对于基因编辑初学者来说,需要几天时间,效果不一定理想;对于有基因编辑经验的研究者,也可能因为信息来源不全面及人的知识有限,较难获取最优的解决方案。因此,人工设计方案繁杂费时,现如今研究出一种自动设计基因编辑方案的计算机智能方法成为亟需解决的问题。
技术实现要素:
4.针对上述缺陷,本发明的目的在于提出一种基因编辑点突变方案自动设计方法及系统,能够综合分析目标突变位点附近的序列和grna,自动设计基因编辑方案的方法,实现基因上的点突变基因编辑,提高了选择方案的效率及准确性。
5.为达此目的,本发明采用以下技术方案:
6.一种基因编辑点突变方案自动设计方法,包括以下步骤:
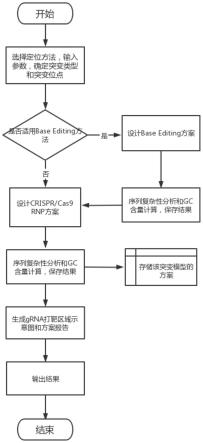
7.步骤a:根据定位方法确定突变类型和突变位点,其中定位方法包括amino acid方法、snp方法、chromosomal location方法和nucleotide sequence方法;
8.步骤b:判断突变位点是否适用base editing方法进行点突变基因编辑,若判断为是,则执行步骤c,若判断为否,则执行步骤d;
9.步骤c:根据突变类型设计base editing方案,进行grna序列的筛选,其中grna序列需包含突变位点;
10.步骤d:根据突变类型设计基于crispr和cas9的rnp方案,进行grna序列的筛选、oligo序列选择和突变位点引入,其中grna序列需包含突变位点;
11.步骤e:对突变位点前后各500bp范围内的基因序列进行序列复杂性分析和gc含量计算,并保存分析结果;
12.步骤f:根据分析结果生成和输出grna打靶区域示意图和方案报告。
13.优选的,在步骤a中,所述amino acid方法包括以下步骤:选择物种,输入基因,查询ncbi数据库和ensembl数据库并获取该基因的转录本、基因序列和编码序列数据;选取其中一个转录本和其中一种突变类型,并输入相关参数得到最终的突变位点;
14.所述snp方法包括以下步骤:输入snp位点编号,查询ncbi数据库获取该位点所在基因的数据、该位点在染色体上的位置以及该snp位点的等位碱基,通过基因和snp位点在染色体上的位置计算出该snp位点在该基因上的位置;选取其中一种等位碱基,根据原序列的碱基和等位碱基自动计算出突变类型;
15.所述chromosomal location方法包括以下步骤:选择物种和基因组版本,选择染色体号,查询自定义数据库获取该染色体的基因序列;选择其中一种突变类型,输入参数“起始位置”和“结束位置”,查询ensembl数据库以获取该位置段对应的碱基和包含这个位置段的基因,计算得出该突变位置段在基因上的位置;
16.所述nucleotide sequence方法包括以下步骤:选择物种,输入基因,查询ncbi数据库和ensembl数据库并获取该基因的转录本、基因序列和编码序列数据;选取其中一个转录本,输入参数“野生型序列”和“突变后序列”,根据两段序列的差异和野生型序列,在基因上的位置自动计算得出突变类型和突变位点。
17.优选的,在所述amino acid方法中,选取其中一种突变类型,并输入相关参数得到最终的突变位点,分别对应以下步骤:
18.当选择突变类型为“突变”时:输入参数“氨基酸序号”和“突变后的氨基酸”,根据氨基酸序号反推计算得出该突变位点位于基因上的准确位置;
19.当选择突变类型为“删除”时:输入参数“起始位置”和“结束位置”,根据这两个位置就能从编码序列上获取到要删除的序列,再根据基因上编码区域的分布位置计算得出删除的序列片段在基因上的准确位置;
20.当选择突变类型为“插入”时:输入参数“起始位置”和“插入的序列”,根据基因上编码区域的分布位置计算得出在基因上插入序列的位置;
21.当选择突变类型为“删除+插入”时:输入参数“起始位置”、“结束位置”和“插入的序列”,根据起始位置和结束位置从编码序列上获取到要删除的序列片段,以及计算出删除的序列片段在基因上的准确位置。
22.优选的,所述步骤c:判断突变位点是否适用base editing方法进行点突变基因编辑,包括判断条件,所述判断条件为:单个碱基突变,且该碱基是由c变成t或g变成a。
23.优选的,所述步骤d:根据突变类型设计基于crispr和cas9的rnp方案,进行grna的筛选、oligo序列选择和突变位点引入,具体包括以下步骤:
24.步骤d1:根据突变类型筛选grna序列,并对筛选得出的grna进行等级划分;
25.步骤d2:选出grna序列中特异性分数最高的grna1和grna2;
26.步骤d3:分别为grna1和grna2设计oligo序列;
27.步骤d4:在oligo序列中引入突变位点。
28.优选的,所述步骤d1:根据突变类型筛选grna序列,并对筛选得出的grna进行等级划分;其中,突变类型包括“突变、插入、删除、删除+插入”四种类型,针对不同的突变类型具有对应的操作步骤;
29.当突变类型为“突变”时,具体包括以下步骤:
30.步骤a1:在突变位点上下游各50bp范围内筛选出所有grna序列,并通过crispor在线软件获取它们的特异性得分、脱靶情况和切割得分等信息;
31.步骤a2:根据grna的特异性分数、切割分数、脱靶情况、gc含量等条件筛选出特异
性分数大于80、切割效率大于0.5、无脱靶、gc含量在40%-60%之间的grna;
32.步骤a3:根据突变位点和grna切割位点的距离对grna进行等级划分:
33.a:n≤5,b:5《n≤10,c:10《n≤23,d:n》23(a为最高级);
34.步骤a4:按照等级由高到低先选出一条相对位置n最小的grna作为grna1,若同一等级中有两条及以上的grna序列则另外选出特异性分数最高的grna作为grna2,否则从下一等级中选择相对位置n最小的一条作为grna2;
35.步骤a5:分别为grna1和grna2设计oligo序列,以切割位点和突变位点距切割位点最远的一端为起始分别向外延伸60bp,该范围内的序列即为oligo序列;
36.步骤a6:在oligo中引入突变位点;
37.步骤a7:分析突变位点前后各500bp范围内的序列的复杂性和gc含量;
38.当突变类型为“插入、删除、删除+插入”时,具体包括以下步骤:
39.步骤b1:在删除/插入位点上下游35bp范围内筛选出所有grna序列,并通过在线软件获取它们的特异性得分、脱靶情况和切割得分等信息;
40.步骤b2:根据grna特异性分数、切割分数、脱靶情况、gc含量等条件筛选出特异性分数大于80、切割效率大于0.5、无脱靶、gc含量在40%-60%之间的grna;
41.步骤b3:根据grna切割位点和编辑区域的相对位置对grna进行等级划分:
42.a:n=0,其中切割位点在编辑区域内部,b:n≤5,c:5《n≤10,d:10《n《23;
43.步骤b4:若a级里有两条及以上的grna,则选择特异性分数最高的两条作为grna1和grna2,否则将从下一等级中选择相对位置n最小的两条作为grna1和grna2;
44.步骤b5:设计oligo:若切割位置在编辑区域内部,则以编辑区域为中心前后各延伸60bp作为oligo序列;若切割位置不在编辑区域内部,则分别以切割位置和编辑区域的最远端为起始向外延伸60bp;
45.步骤b6:在oligo中引入突变位点;
46.步骤b7:分析突变位点前后各500bp范围内的序列的复杂性和gc含量。
47.更进一步的说明,所述步骤d4:在oligo序列中引入突变位点,具体包括以下步骤:
48.步骤d41:输入oligo序列,判断oligo序列上grna的pam结构是否发生变化,若是,则输出oligo序列,若否,则执行步骤d42;
49.步骤d42:判断grna是否在基因编辑区内,若是,则执行步骤d43;若否,则在grna引入随机突变:ngg
→
ngc,再执行步骤d46;
50.步骤d43:grna从3’端到5’端依次引入同义突变,判断是否在pam结构ngg上,若是,则执行步骤d44,若否,则执行步骤d45;
51.步骤d44:判断是否改变了pam结构,若否,返回步骤d43;若是,则执行步骤d46;
52.步骤d45:判断是否和目标突变点重叠,若是,则执行步骤d46,若否,返回步骤d43;;
53.步骤d46:修改oligo序列并输出。
54.一种基因编辑点突变方案自动设计系统,采用如上文所述的一种基因编辑点突变方案自动设计方法的步骤,包括突变位点解读模块、判断模块、第一方案设计模块、第二方案设计模块、计算模块和结果生成模块;
55.所述突变位点解读模块用于根据定位方法确定突变类型和突变位点;
56.所述判断模块用于判断突变位点是否适用base editing方法进行点突变基因编辑;
57.所述第一方案设计模块用于设计base editing方案,进行grna的评估和筛选;
58.所述第二方案设计模块用于设计基于crispr和cas9的rnp方案,进行grna的筛选、oligo序列选择和突变位点引入;
59.所述计算模块用于对突变位点前后各500bp范围内的序列进行序列复杂性分析和gc含量计算,并保存分析结果;
60.所述结果生成模块用于根据分析结果生成和输出grna打靶区域示意图和方案报告。
61.一种电子设备,包括存储器、处理器及存储在所述存储器上并可在所述处理器上运行的计算机程序,所述处理器执行所述程序时实现如上文所述的一种基因编辑点突变方案自动设计方法的步骤。
62.一种非暂态计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现如上文所述的一种基因编辑点突变方案自动设计方法的步骤。
63.上述技术方案中的一个技术方案具有以下有益效果:针对人工设计方案繁杂费时、对实验技术人员经验依赖较高等问题,提出一种能够综合分析目标突变位点附近的序列和grna,自动设计基因编辑方案的方法,实现基因上的点突变基因编辑:选择突变位点的定位方法amino acid(氨基酸)、snp(单核苷酸多态性位点)、chromosomal location(染色体位置)和nucleotide sequence(核苷酸序列)中的其中一种,根据不同的定位方法确定突变类型和突变位点,综合分析突变位点前后的序列,设计两种类型的点突变方案,分别为base editing和rnp点突变方案,其中,base editing方案包括grna评估和筛选,rnp点突变方案包括grna的筛选、oligo选择和突变位点引入。通过线上设计点突变方案替代繁杂耗时的人工方案,只需选择位点信息的参数,经过grna筛选和在oligo上额外引入突变,即可达得到一份最优方案,为科研用户节省大量时间。
附图说明
64.图1是本发明的一个实施例的方法流程图;
65.图2是本发明中在oligo中引入突变位点的流程示意图;
66.图3是本发明的一个实施例的输出结果图;
具体实施方式
67.下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,仅用于解释本发明,而不能理解为对本发明的限制。
68.下面结合图1至图2描述本发明实施例的一种基因编辑点突变方案自动设计方法,包括以下步骤:
69.步骤a:根据定位方法确定突变类型和突变位点,其中定位方法包括amino acid方法、snp方法、chromosomal location方法和nucleotide sequence方法;
70.步骤b:判断突变位点是否适用base editing方法进行点突变基因编辑,若判断为
是,则执行步骤c,若判断为否,则执行步骤d;
71.步骤c:根据突变类型设计base editing方案,进行grna序列的筛选,其中grna序列需包含突变位点;
72.步骤d:根据突变类型设计基于crispr和cas9的rnp方案,进行grna序列的筛选、oligo序列选择和突变位点引入,其中grna序列需包含突变位点;
73.步骤e:对突变位点前后各500bp范围内的基因序列进行序列复杂性分析和gc含量计算,并保存分析结果;
74.步骤f:根据分析结果生成和输出grna打靶区域示意图和方案报告。
75.在现有技术中,crispr/cas9通过设计特异性向导rna识别靶序列引导cas9核酸内切酶对靶序列的pam上游进行切割,使靶位点dna双链断裂;dna双链断裂后,利用细胞的非同源末端连接(nhej)或同源重组(hdr)的方式对切割位点进行修复,实现dna水平的基因敲除、敲入或点突变。crispr/cas9以其操作简单、成本低且编辑效率等优势,成为生命科学领域最炙手可热的技术之一,已经广泛应用于诸多模式生物的基因编辑相关功能研究中,如哺乳动物(大鼠、小鼠、猪、兔、猴等)、斑马鱼、干细胞、肿瘤细胞系及细菌真菌等。
76.基于crispr/cas9的点突变基因编辑方案的设计看似不难,但对于基因编辑初学者来说,需要几天时间,效果不一定理想;对于有基因编辑经验的研究者,也可能因为信息来源不全面,而没有选到最优的解决方案。
77.在本发明实施例中,针对人工设计方案繁杂费时、对实验技术人员经验依赖较高等问题,提出一种能够综合分析目标突变位点附近的序列和grna,自动设计基因编辑方案的方法,实现基因上的点突变基因编辑:选择突变位点的定位方法amino acid(氨基酸)、snp(单核苷酸多态性位点)、chromosomal location(染色体位置)和nucleotide sequence(核苷酸序列)中的其中一种,根据不同的定位方法确定突变类型和突变位点,综合分析突变位点前后的序列,设计两种类型的点突变方案,分别为base editing和rnp点突变方案,其中,base editing方案包括grna评估和筛选,rnp点突变方案包括grna的筛选、oligo选择和突变位点引入。通过线上设计点突变方案替代繁杂耗时的人工方案,只需选择位点信息的参数,经过grna筛选和在oligo上额外引入突变,即可达得到一份最优方案,为科研用户节省大量时间。
78.更进一步的说明,在步骤a中,所述amino acid方法包括以下步骤:选择物种,输入基因,查询ncbi数据库和ensembl数据库并获取该基因的转录本、基因序列和编码序列数据;选取其中一个转录本和其中一种突变类型,并输入相关参数得到最终的突变位点;
79.所述snp方法包括以下步骤:输入snp位点编号,查询ncbi数据库获取该位点所在基因的数据、该位点在染色体上的位置以及该snp位点的等位碱基,通过基因和snp位点在染色体上的位置计算出该snp位点在该基因上的位置;选取其中一种等位碱基,根据原序列的碱基和等位碱基自动计算出突变类型;
80.所述chromosomal location方法包括以下步骤:选择物种和基因组版本,选择染色体号,查询自定义数据库获取该染色体的基因序列;选择其中一种突变类型,输入参数“起始位置”和“结束位置”,查询ensembl数据库以获取该位置段对应的碱基和包含这个位置段的基因,计算得出该突变位置段在基因上的位置;需要说明的是,染色体的基因序列源自ncbi数据库,由于一条染色体的序列有几亿个字符串,因此经过处理后将其简化并存储
在服务器上的自定义数据库里,提高查询速度。
81.所述nucleotide sequence方法包括以下步骤:选择物种,输入基因,查询ncbi数据库和ensembl数据库并获取该基因的转录本、基因序列和编码序列数据;选取其中一个转录本,输入参数“野生型序列”和“突变后序列”,根据两段序列的差异和野生型序列,在基因上的位置自动计算得出突变类型和突变位点。
82.更进一步的说明,在所述amino acid方法中,选取其中一种突变类型,并输入相关参数得到最终的突变位点,分别对应以下步骤:
83.当选择突变类型为“突变”时:输入参数“氨基酸序号”和“突变后的氨基酸”,根据氨基酸序号反推计算得出该突变位点位于基因上的准确位置;例如:氨基酸序号为50,原氨基酸为ser(序列agc)突变成gly(序列ggc),即a
→
g,就能根据第50个氨基酸在基因上的位置,得出a
→
g该突变在基因上的准确位置;
84.当选择突变类型为“删除”时:输入参数“起始位置”和“结束位置”,根据这两个位置就能从编码序列上获取到要删除的序列,再根据基因上编码区域的分布位置计算得出删除的序列片段在基因上的准确位置;
85.当选择突变类型为“插入”时:输入参数“起始位置”和“插入的序列”,根据基因上编码区域的分布位置计算得出在基因上插入序列的位置;
86.当选择突变类型为“删除+插入”时:输入参数“起始位置”、“结束位置”和“插入的序列”,根据起始位置和结束位置从编码序列上获取到要删除的序列片段,以及计算出删除的序列片段在基因上的准确位置。
87.更进一步的说明,所述步骤c:判断突变位点是否适用base editing方法进行点突变基因编辑,包括判断条件,所述判断条件为:单个碱基突变,且该碱基是由c变成t或g变成a。
88.更进一步的说明,所述步骤d:根据突变类型设计基于crispr和cas9的rnp方案,进行grna的筛选、oligo序列选择和突变位点引入,具体包括以下步骤:
89.步骤d1:根据突变类型筛选grna序列,并对筛选得出的grna进行等级划分;
90.步骤d2:选出grna序列中特异性分数最高的grna1和grna2;
91.步骤d3:分别为grna1和grna2设计oligo序列;
92.步骤d4:在oligo序列中引入突变位点。
93.更进一步的说明,所述步骤d1:根据突变类型筛选grna序列,并对筛选得出的grna进行等级划分;其中,突变类型包括“突变、插入、删除、删除+插入”四种类型,针对不同的突变类型具有对应的操作步骤;
94.当突变类型为“突变”时,具体包括以下步骤:
95.步骤a1:在突变位点上下游各50bp范围内筛选出所有grna序列,并通过crispor在线软件获取它们的特异性得分、脱靶情况和切割得分等信息;需要说明的是,crispor在线软件是一款现有的生物数据库与在线工具;
96.步骤a2:根据grna的特异性分数、切割分数、脱靶情况、gc含量等条件筛选出特异性分数大于80、切割效率大于0.5、无脱靶、gc含量在40%-60%之间的grna;
97.步骤a3:根据突变位点和grna切割位点的距离对grna进行等级划分:
98.a:n≤5,b:5《n≤10,c:10《n≤23,d:n》23(a为最高级);
99.步骤a4:按照等级由高到低先选出一条相对位置n最小的grna作为grna1,若同一等级中有两条及以上的grna序列则另外选出特异性分数最高的grna作为grna2,否则从下一等级中选择相对位置n最小的一条作为grna2;
100.步骤a5:分别为grna1和grna2设计oligo序列,以切割位点和突变位点距切割位点最远的一端为起始分别向外延伸60bp,该范围内的序列即为oligo序列;
101.步骤a6:在oligo中引入突变位点;
102.步骤a7:分析突变位点前后各500bp范围内的序列的复杂性和gc含量;
103.当突变类型为“插入、删除、删除+插入”时,具体包括以下步骤:
104.步骤b1:在删除/插入位点上下游各35bp范围内筛选出所有grna序列,并通过在线软件获取它们的特异性得分、脱靶情况和切割得分等信息;
105.步骤b2:根据grna特异性分数、切割分数、脱靶情况、gc含量等条件筛选出特异性分数大于80、切割效率大于0.5、无脱靶、gc含量在40%-60%之间的grna;
106.步骤b3:根据grna切割位点和编辑区域的相对位置对grna进行等级划分:
107.a:n=0,其中切割位点在编辑区域内部,b:n≤5,c:5《n≤10,d:10《n《23;
108.步骤b4:若a级里有两条及以上的grna,则选择特异性分数最高的两条作为grna1和grna2,否则将从下一等级中选择相对位置n最小的两条作为grna1和grna2;
109.步骤b5:设计oligo:若切割位置在编辑区域内部,则以编辑区域为中心前后各延伸60bp作为oligo序列;若切割位置不在编辑区域内部,则分别以切割位置和编辑区域的最远端为起始向外延伸60bp;
110.步骤b6:在oligo中引入突变位点;
111.步骤b7:分析突变位点前后各500bp范围内的序列的复杂性和gc含量。
112.更进一步的说明,所述步骤d4:在oligo序列中引入突变位点,具体包括以下步骤:
113.步骤d41:输入oligo序列,判断oligo序列上grna的pam结构是否发生变化,若是,则输出oligo序列,若否,则执行步骤d42;
114.步骤d42:判断grna是否在基因编辑区内,若是,则执行步骤d43;若否,则在grna引入随机突变:ngg
→
ngc,再执行步骤d46;
115.步骤d43:grna从3’端到5’端依次引入同义突变,判断是否在pam结构ngg上,若是,则执行步骤d44,若否,则执行步骤d45;
116.步骤d44:判断是否改变了pam结构,若否,返回步骤d43;若是,则执行步骤d46;
117.步骤d45:判断是否和目标突变点重叠,若是,则执行步骤d46,若否,返回步骤d43;;
118.步骤d46:修改oligo序列并输出。
119.本实施例还公开一种基因编辑点突变方案自动设计系统,采用如上文所述的一种基因编辑点突变方案自动设计方法的步骤,包括突变位点解读模块、判断模块、第一方案设计模块、第二方案设计模块、计算模块和结果生成模块;
120.所述突变位点解读模块用于根据定位方法确定突变类型和突变位点;
121.所述判断模块用于判断突变位点是否适用base editing方法进行点突变基因编辑;
122.所述第一方案设计模块用于设计base editing方案,进行grna的评估和筛选;
123.所述第二方案设计模块用于设计基于crispr和cas9的rnp方案,进行grna的筛选、oligo序列选择和突变位点引入;
124.所述计算模块用于对突变位点前后各500bp范围内的序列进行序列复杂性分析和gc含量计算,并保存分析结果;
125.所述结果生成模块用于根据分析结果生成和输出grna打靶区域示意图和方案报告。
126.本实施例还公开一种电子设备,包括存储器、处理器及存储在所述存储器上并可在所述处理器上运行的计算机程序,所述处理器执行所述程序时实现如上文所述的一种基因编辑点突变方案自动设计方法的步骤。
127.本实施例还公开一种非暂态计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现如上文所述的一种基因编辑点突变方案自动设计方法的步骤。
128.根据本发明实施例的一种基因编辑点突变方案自动设计方法及系统的其他构成等以及操作对于本领域普通技术人员而言都是已知的,这里不再详细描述。
129.上述一种基因编辑点突变方案自动设计系统中的各模块可全部或部分通过软件、硬件及其组合来实现。上述各模块可以硬件形式内嵌于或独立于电子设备中的处理器中,也可以以软件形式存储于电子设备的存储器中,以便于处理器调用执行以上各模块对应的操作。
130.本领域普通技术人员可以理解实现上述实施例方法中的全部或部分流程,是可以通过计算机程序来指令相关的硬件来完成,计算机程序可存储于一非易失性计算机可读取存储介质中,该计算机程序在执行时,可包括如上述各方法的实施例的流程。其中,本技术所提供的各实施例中所使用的对存储器、存储、数据库或其它介质的任何引用,均可包括非易失性和/或易失性存储器。非易失性存储器可包括只读存储器(rom)、可编程rom(prom)、电可编程rom(eprom)、电可擦除可编程rom(eeprom)或闪存。易失性存储器可包括随机存取存储器(ram)或者外部高速缓冲存储器。作为说明而非局限,ram以多种形式可得,诸如静态ram(sram)、动态ram(dram)、同步dram(sdram)、双数据率sdram(ddrsdram)、增强型sdram(esdram)、同步链路(synchlink)dram(sldram)、存储器总线(rambus)直接ram(rdram)、直接存储器总线动态ram(drdram)、以及存储器总线动态ram(rdram)等。
131.所属领域的技术人员可以清楚地了解到,为了描述的方便和简洁,仅以上述各功能单元、模块的划分进行举例说明,实际应用中,可以根据需要而将上述功能分配由不同的功能单元、模块完成,即将装置的内部结构划分成不同的功能单元或模块,以完成以上描述的全部或者部分功能。
132.以上对本发明的具体实施例进行的描述只是为了说明本发明的技术路线和特点,其目的在于让本领域内的技术人员能够了解本发明的内容并据以实施,但本发明并不限于上述特定实施方式。凡是在本发明权利要求的范围内做出的各种变化或修饰,都应涵盖在本发明的保护范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1