用于分辨生物分子质谱成像的系统及其使用方法与流程
1.本发明涉及分辨生物分子质谱成像的系统,特别涉及用于分辨生物分子质谱成像的系统及其使用方法,属于生物分子质谱成像分辨技术领域。
背景技术:
2.质谱成像是以质谱技术为基础的成像方法,该方法通过质谱直接扫描生物样品成像,可以在同一张组织切片或组织芯片上同时分析数百种分子的空间分布特征。简单而言,质谱成像技术就是借助于质谱的方法,再配套上专门的质谱成像软件控制下,使用一台通过测定质荷比来分析生物分子的标准分子量的质谱仪来成像的方法。
3.根据中国专利号为“cn113376127a”公开了一种适于生物分子多重检测的成像系统,所述成像系统至少包括第一光学路径和第二光学路径;所述第一光学路径为从激发光源至物镜,且所述激发光源和所述物镜之间设置有滤光片组,所述滤光片组被配置和布置为将从所述激发光源发出的激发光引导至所述物镜;所述第二光学路径为从所述物镜至图像采集装置,且所述物镜和所述图像采集装置之间设置有反光镜,所述反光镜被配置和布置为将由所述物镜接收到的荧光投射到所述图像采集装置;所述激发光源至少包括第一激发光源和第二激发光源,所述第一激发光源用于荧光信号的激发,所述第二激发光源用于光激化学发光信号的激发。所述成像系统能够同时实现荧光和光激化学发光成像。
4.但是上述技术方案在实际对生物分子分辨的过程中,变量较多,检测复杂,在检测的环节中,一个变量的变化,容易造成生物分子分辨错位,影响分辨的准确度,为此我们设计了用于分辨生物分子质谱成像的系统及其使用方法。
技术实现要素:
5.本发明的目的在于提供用于分辨生物分子质谱成像的系统及其使用方法,以解决上述背景技术中提出的问题。
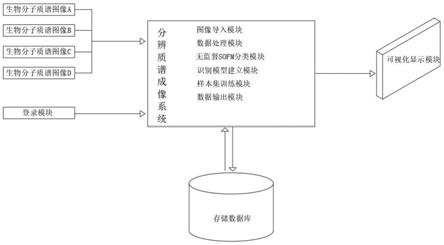
6.为实现上述目的,本发明提供如下技术方案:用于分辨生物分子质谱成像的系统,包括分布质谱成像系统、登录模块、可视化显示模块和存储数据库,所述分布质谱成像系统与登录模块信号连接,所述分布质谱成像系统与可视化显示模块和存储数据库电性连接,所述分布质谱成像系统包括图像导入模块、数据处理模块、无监督sofm分类模块、识别模型建立模块、样本集训练模块和数据输出模块;
7.图像导入模块:用于将生物分子质谱图像导入至分布质谱成像系统中;
8.数据处理模块:对导入的图像进行处理;
9.无监督sofm分类模块:据每个样品点的分子组成和含量的差异与相似度分类,分为样品区和非样品区两类;
10.识别模型建立模块:建立分辨生物分子质谱图像的人工神经网络函数;
11.样本集训练模块:对一直的样品进行分辨训练;
12.数据输出模块:将分辨处理的数据发送至可视化显示模块进行显示。
13.作为本发明的一种优选技术方案,所述图像导入模块具体为:首先将原始图像格式数据转换,将jpg图像转换为imzxml格式,将imzxml格式数据进行重构成质谱图像数据。
14.作为本发明的一种优选技术方案,所述数据处理模块具体为:将重构成的质谱图像数据进行质谱成像数据进行基线扣除,剔除出现机率小的质谱峰信号,以排除化学噪声的干扰和增强样品区域与非样品区的对比度。
15.作为本发明的一种优选技术方案,所述无监督sofm分类模块:具体为采用质谱绝对峰强作为输入变量,应用matlab的人工神经网络函数开发的sofm分类软件实现样品区与非样品区的区分,在具体应用sofm分类时,选用1
×
2神经元结构将整个分析区域分为组织样品区与非组织样品区两类。
16.作为本发明的一种优选技术方案,所述样本集训练模块:应用学习向量量化网络lvq对样品区的生物组织进行有监督的学习训练建立模型,应用模型对其它未知样品进行类别识别,对已知样品进行验证,再对未知样品进行预测。
17.作为本发明的一种优选技术方案,所述数据输出模块具体为:将分辨的生物分子质谱成像数据进行分类整理,建立数字图表,并将数字图表发送至可视化显示模块进行显示。
18.作为本发明的一种优选技术方案,应用于权利要求1-6任意一项所述的用于分辨生物分子质谱成像的系统,包括以下步骤:
19.s1:用户通过登录模块登录,登录入分辨质谱成像系统,并通过图像导入模块将生物分子质谱图像导入至分辨质谱成像系统;
20.s2:将质谱图像数据格式为标准通用的imzxml格式,对原始质谱数据重构成质谱图像数据;
21.s3:通过数据处理模块对质谱成像数据进行基线扣除,信号剔除操作,以排除化学噪声的干扰和增强样品区域与非样品区的对比度;
22.s4:对处理后的质谱成像数据进行无监督sofm分类,根据每个样品点的分子组成和含量的差异与相似度分类,分为样品区和非样品区两类;
23.s5:对样品区内的质谱成像数据进行有监督的lvq训练建立识别模型;
24.s6:采用一对或多对已知样品的数据进行识别训练,建立模型,建立好的识别模型由在相同实验条件获得的已知样品进行验证再对未知样品进行预测。
25.与现有技术相比,本发明的有益效果是:
26.本发明用于分辨生物分子质谱成像的系统及其使用方法,通过组合应用人工神经网络技术实现生物组织质谱成像的分辨,应用无监督sofm方法把非样品区排除在外等措施,减少无关变量和无关样品采样点的干扰,增加可靠性获得高准确度,可以精确到具体单个采样点的识别具有高精度的优点,在经过大量样品验证后拥有作为常规工具应用于基于生物分子成像临床医学研究和生命科学研究的潜能。
附图说明
27.图1为本发明的系统框图;
28.图2为本发明的方法流程图。
具体实施方式
29.下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
30.请参阅图1-2,本发明提供了用于分辨生物分子质谱成像的系统及其使用方法的技术方案:包括分布质谱成像系统、登录模块、可视化显示模块和存储数据库,所述分布质谱成像系统与登录模块信号连接,所述分布质谱成像系统与可视化显示模块和存储数据库电性连接,所述分布质谱成像系统包括图像导入模块、数据处理模块、无监督sofm分类模块、识别模型建立模块、样本集训练模块和数据输出模块;
31.图像导入模块:用于将生物分子质谱图像导入至分布质谱成像系统中;
32.数据处理模块:对导入的图像进行处理;
33.无监督sofm分类模块:据每个样品点的分子组成和含量的差异与相似度分类,分为样品区和非样品区两类;
34.识别模型建立模块:建立分辨生物分子质谱图像的人工神经网络函数;
35.样本集训练模块:对一直的样品进行分辨训练;
36.数据输出模块:将分辨处理的数据发送至可视化显示模块进行显示。
37.图像导入模块具体为:首先将原始图像格式数据转换,将jpg图像转换为imzxml格式,将imzxml格式数据进行重构成质谱图像数据。
38.数据处理模块具体为:将重构成的质谱图像数据进行质谱成像数据进行基线扣除,剔除出现机率小的质谱峰信号,以排除化学噪声的干扰和增强样品区域与非样品区的对比度。
39.无监督sofm分类模块:具体为采用质谱绝对峰强作为输入变量,应用matlab的人工神经网络函数开发的sofm分类软件实现样品区与非样品区的区分,在具体应用sofm分类时,选用1
×
2神经元结构将整个分析区域分为组织样品区与非组织样品区两类。
40.样本集训练模块:应用学习向量量化网络lvq对样品区的生物组织进行有监督的学习训练建立模型,应用模型对其它未知样品进行类别识别,对已知样品进行验证,再对未知样品进行预测。
41.数据输出模块具体为:将分辨的生物分子质谱成像数据进行分类整理,建立数字图表,并将数字图表发送至可视化显示模块进行显示。
42.用于分辨生物分子质谱成像的系统,包括以下步骤:
43.s1:用户通过登录模块登录,登录入分辨质谱成像系统,并通过图像导入模块将生物分子质谱图像导入至分辨质谱成像系统;
44.s2:将质谱图像数据格式为标准通用的imzxml格式,对原始质谱数据重构成质谱图像数据;
45.s3:通过数据处理模块对质谱成像数据进行基线扣除,信号剔除操作,以排除化学噪声的干扰和增强样品区域与非样品区的对比度;
46.s4:对处理后的质谱成像数据进行无监督sofm分类,根据每个样品点的分子组成和含量的差异与相似度分类,分为样品区和非样品区两类;
47.s5:对样品区内的质谱成像数据进行有监督的lvq训练建立识别模型;
48.s6:采用一对或多对已知样品的数据进行识别训练,建立模型,建立好的识别模型由在相同实验条件获得的已知样品进行验证再对未知样品进行预测。
49.在本发明的描述中,需要理解的是,指示的方位或位置关系为基于附图所示的方位或位置关系,仅是为了便于描述本发明和简化描述,而不是指示或暗示所指的装置或元件必须具有特定的方位、以特定的方位构造和操作,因此不能理解为对本发明的限制。
50.在本发明中,除非另有明确的规定和限定,例如,可以是固定连接,也可以是可拆卸连接,或成一体;可以是机械连接,也可以是电连接;可以是直接相连,也可以通过中间媒介间接相连,可以是两个元件内部的连通或两个元件的相互作用关系,除非另有明确的限定,对于本领域的普通技术人员而言,可以根据具体情况理解上述术语在本发明中的具体含义。
51.尽管已经示出和描述了本发明的实施例,对于本领域的普通技术人员而言,可以理解在不脱离本发明的原理和精神的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由所附权利要求及其等同物限定。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1