生成包含自动标注文件的基准病理数据集的方法及系统
1.本发明属于医学肿瘤识别领域,特别是涉及生成包含自动标注文件的基准病理数据集的方法及系统。
背景技术:
2.在人工智能(ai)的大趋势下,实现人工智能与数字病理学的结合可能是该领域的趋势。对于病理学家来说,许多癌症的检测和分析越来越依赖于数字病理学,越来越多的深度学习模型被提出,用来评估和预测肿瘤。然而,这些智能算法都需要大量的带有标注的高质量数据集。
3.目前数据集都需要专业人员手工标注,成本高,准确率低,可获取量很少,识别分析结果的准确率和算法鲁棒性不足以满足实际应用中计算机辅助病理诊断的要求。虽然已有学者意识到了带有标注信息数据集地重要性,一些数据集也被制作出来。但是,所有这些已经得到的数据集仍然需要专业人员手工进行标注,即使是专业病理医生,也容易遗漏小区域的肿瘤,收集带有大量注释信息的数据集来训练深度学习模型变得不切实际。
技术实现要素:
4.本发明的目的是提供一种生成包含自动标注文件的基准病理数据集的方法,以解决上述现有技术存在的问题。
5.为实现上述目的,本发明提供了一种生成包含自动标注文件的基准病理数据集的方法,包括以下步骤:
6.获取病理图像,其中所述病理图像包括:目标图像和高光谱图像;对所述高光谱图像提取伪彩图;
7.对所述高光谱图像中的像素进行识别标注,得到病理数据集的标签部分;
8.对所述目标图像和所述伪彩图进行聚类,基于所述聚类结果对所述目标图像和所述伪彩图进行染色归一化处理,得到病理数据集的图像部分;基于所述标签部分和所述图像部分,得到病理数据集。
9.优先地,得到病理数据集的标签部分的过程包括:
10.通过病理识别模型对所述高光谱图像中的像素进行识别标注,得到病理数据集的标签部分。
11.优先地,对所述高光谱图像中的像素进行识别标注之前还包括:
12.提取所述高光谱图像的特征像素,基于所述特征像素的通道值及标签构建病理识别模型,通过决策树算法对所述病理识别模型进行训练,直到输出的误差减小到期望值,得到训练好的病理识别模型,通过训练好的病理识别模型对所述高光谱图像中的像素进行识别标注。
13.优先地,对所述目标图像和所述伪彩图进行聚类的过程包括:
14.基于所述目标图像和所述伪彩图中的像素值,对所述目标图像和所述伪彩图分别
进行聚类,得到聚类结果。
15.优先地,得到病理数据集的图像部分的过程包括:
16.基于所述聚类结果,分别计算第一亮度值和第二亮度值,将所述第一亮度值和所述第二亮度值进行比较,若所述第一亮度值小于所述第二亮度值,则将所述伪彩图中像素点的红绿蓝三通道的值替换成所述目标图像中像素点的红绿蓝三通道的值;否则不替换;基于比较结果得到病理数据集的图像部分;
17.其中所述第一亮度值为所述伪彩图中像素点的亮度值,所述第二亮度值为所述目标图像中像素点亮度值。
18.另一方面,为了实现上述技术目的,本发明提供了一种生成包含自动标注文件的基准病理数据集的系统,包括:病理图像获取模块、数据集标签获取模块及数据集图像获取模块;
19.所述病理图像获取模块,用于获取病理图像,其中所述病理图像包括:目标图像和高光谱图像;对所述高光谱图像提取伪彩图;
20.所述数据集标签获取模块,用于对所述高光谱图像中的像素进行识别标注,得到病理数据集的标签部分;
21.所述数据集图像获取模块,用于对所述目标图像和所述伪彩图进行聚类,基于所述聚类结果对所述目标图像和所述伪彩图进行染色归一化处理,得到病理数据集的图像部分。
22.优选地,所述数据集标签获取模块包括模型构建单元;
23.所述模型构建单元,用于提取所述高光谱图像的特征像素,基于所述特征像素的通道值及标签构建病理识别模型,通过决策树算法对所述病理识别模型进行训练,直到输出的误差减小到期望值,得到训练好的病理识别模型。
24.优选地,所述数据集标签获取模块还包括标签获取单元;
25.所述标签获取单元,用于通过所述训练好的病理识别模型对所述高光谱图像中的像素进行识别标注,得到病理数据集的标签部分。
26.优选地,所述数据集图像获取模块包括图像处理单元;
27.所述图像处理单元,基于所述目标图像和所述伪彩图中的像素值,对所述目标图像和所述伪彩图分别进行聚类,得到聚类结果。
28.优选地,所述数据集图像获取模块还包括亮度值比较单元;
29.所述亮度值比较单元,基于所述聚类结果,分别计算第一亮度值和第二亮度值,将所述第一亮度值和所述第二亮度值进行比较,若所述第一亮度值小于所述第二亮度值,则将所述伪彩图中像素点的红绿蓝三通道的值替换成所述目标图像中像素点的红绿蓝三通道的值;否则不替换;基于比较结果得到病理数据集的图像部分;其中所述第一亮度值为所述伪彩图中像素点的亮度值,所述第二亮度值为所述目标图像中像素点亮度值。
30.本发明的技术效果为:本发明获取目标图像、原图像及高光谱图像;利用高光谱图像实现了病理影像的自动标注,得到数据集的标签部分;通过对目标图像和原图像进行聚类计算,进一步实现染色体归一化,得到数据集的图片部分,不需要大量数据提前训练网络,本发明充分利用高光谱图像的光谱信息,生成了包含自动标注文件的基准病理数据集,缓解了计算机辅助诊断方面研究数据集紧缺的情况。
附图说明
31.构成本技术的一部分的附图用来提供对本技术的进一步理解,本技术的示意性实施例及其说明用于解释本技术,并不构成对本技术的不当限定。在附图中:
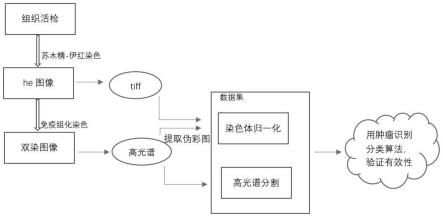
32.图1为本发明实施例中的方法流程图;
33.图2为本发明实施例中的数据集中标签变化过程示意图;
34.图3为本发明实施例中的染色标准化流程图;
35.图4为本发明实施例中的数据集中图像变化过程示意图;
36.图5为本发明实施例中的系统示意图。
具体实施方式
37.需要说明的是,在不冲突的情况下,本技术中的实施例及实施例中的特征可以相互组合。下面将参考附图并结合实施例来详细说明本技术。
38.需要说明的是,在附图的流程图示出的步骤可以在诸如一组计算机可执行指令的计算机系统中执行,并且,虽然在流程图中示出了逻辑顺序,但是在某些情况下,可以以不同于此处的顺序执行所示出或描述的步骤。
39.实施例一
40.如图1所示,本实施例中提供一种生成包含自动标注文件的基准病理数据集的方法,包括以下步骤:
41.获取病理图像,其中病理图像包括:目标图像和高光谱图像;对高光谱图像提取伪彩图;
42.对高光谱图像中的像素进行识别标注,得到病理数据集的标签部分;
43.对目标图像和伪彩图进行聚类,基于聚类结果对目标图像和伪彩图进行染色归一化处理,得到病理数据集的图像部分;基于标签部分和图像部分,得到病理数据集。
44.在一些实施例中,得到病理数据集的标签部分的过程包括:
45.通过病理识别模型对高光谱图像中的像素进行识别标注,得到病理数据集的标签部分。
46.在一些实施例中,对高光谱图像中的像素进行识别标注之前还包括:
47.提取高光谱图像的代表性像素,基于代表性像素的通道值及标签构建病理识别模型,通过决策树算法对病理识别模型进行训练,直到输出的误差减小到期望值,得到训练好的病理识别模型,通过训练好的病理识别模型对高光谱图像中的像素进行识别标注。
48.在一些实施例中,对目标图像和伪彩图进行聚类的过程包括:
49.基于目标图像和伪彩图中的像素值,对目标图像和伪彩图分别进行聚类,得到聚类结果。
50.在一些实施例中,得到病理数据集的图像部分的过程包括:
51.基于聚类结果,分别计算第一亮度值和第二亮度值,将第一亮度值和第二亮度值进行比较,若第一亮度值小于第二亮度值,则将伪彩图中像素点的r、g、b的值替换成目标图像中像素点的r、g、b的值;否则不替换;基于比较结果得到病理数据集的图像部分;
52.其中第一亮度值为伪彩图中像素点的亮度值,第二亮度值为目标图像中像素点亮度值。
53.生成病理数据集的具体实施步骤包括:
54.(1)组织活检并制备病理切片,得到单染,双染和高光谱图像;
55.该步骤具体为:
56.①
、组织活检,制备苏木精-伊红染色(h&e)的病理切片以及h&e与免疫组化染色(cam5.2)的双染病理切片;
57.②
、使用全玻片彩色扫描仪得到单染图其放大20倍后的全玻片彩色图像;
58.③
、用显微高光谱成像平台得到双染图的高光谱图像;
59.④
、从高光谱图像中抽三个近似r、g、b波段得到伪彩图;
60.(2)利用xgboost的改进集成算法lightgbm训练分类器,通过高光谱图像多个通道的光谱信息,区分出两个感兴趣区域:病变区域非病变区域。得到数据集的标签部分;
61.该步骤具体为:
62.①
、利用envi软件挑选部分具有代表性的像素,将这些像素所有通道的值及其标签(癌症区域为0,正常区域为1)生成excel表;
63.②
、将生成的excel表输入lightgbm进行训练,lightgbm采用了基于直方图(histogram)的决策树算法,基本思想是:把连续的浮点特征值离散化成k个整数;遍历数据,根据离散化后的值作为索引在直方图中累积统计量;然后根据直方图的离散值,遍历寻找最优的分割点。相比基于预排序(pre-sorting)的xgboost,直方图算法有占用内存小和时间复杂度低的优点。
64.在histogram算法之上,lightgbm进行进一步的优化。首先它抛弃了大多数gbdt工具使用的按层生长(level-wise)的决策树生长策略,而使用了带有深度限制的按叶子生长(leaf-wise)算法,降低了更多误差,提升了精度。但可能会长出比较深的决策树,产生过拟合,所以叶子节点数是其中最重要的参数;
65.③
、调整参数,叶子节点数的值越大准确率越高,但是太大会出现过拟合,将其从30调为60后,准确率从0.97上升为0.98;
66.④
、用训练好的模型对整幅图中每个像素进行预测,》0.5作黑色正常区域,《0.5作白色癌症区域;
67.⑤
、对得到的图像进行中值滤波(窗口大小为36)优化结果。数据集中标签过程示意图,如图2所示。
68.(3)提出了一种结合k-means聚类和wasserstein距离的染色标准化算法,得到数据集的图像部分。
69.该步骤具体为:
70.①
、对原图像(双染图像)和目标图像(h&e单染图像)分别进行kmeans聚类,各聚20类;
71.②
、根据原图像和目标图像灰度图中每个像素的值所属的区间(0到255分成255个区间),得到上述40类的分布;
72.③
、由上述的分布利用wasserstein距离为原图中的每一类找到目标图中最相近的一类,公式如下:
73.74.其中,γ为每一个可能的联合分布,
75.x和y为从γ中采样得的样本,
76.||x-y||为这对样本的距离,
77.wasserstein距离就是在所有可能的联合分布中样本对距离的期望值能够取到的下界。
78.④
、比较原图中每个像素点的亮度(0.299*r+0.587*g+0.114*b)和该像素点所属类别对应目标图中类别中心的亮度,如果小于,则将该点处r、g、b的值换为对应目标图中类别中心处r、g、b的值;否则将该点处r、g、b的值不变。具体染色标准化流程图,如图3所示;数据集中图像过程示意图,如图4所示。
79.本实施例有益效果:
80.本实施例提出的生成包含自动标注文件的基准病理数据集的方法,通过制备多标记的病理切片,利用高光谱图像多出的一维光谱信息实现了病理影像的自动标注,得到数据集的标签部分;使用kmeans等无监督的方法实现染色体归一化,得到数据集的图片部分,不需要大量数据提前训练网络。本发明充分利用高光谱图像的光谱信息,生成了包含自动标注文件的基准病理数据集,缓解了计算机辅助诊断方面研究数据集紧缺的情况。
81.实施例二
82.如图5所示,本发明提供了一种生成包含自动标注文件的基准病理数据集的系统,包括:病理图像获取模块、数据集标签获取模块及数据集图像获取模块;
83.病理图像获取模块,用于获取病理图像,其中病理图像包括:目标图像和高光谱图像;对高光谱图像提取伪彩图;
84.数据集标签获取模块,用于对高光谱图像中的像素进行识别标注,得到病理数据集的标签部分;
85.数据集图像获取模块,用于对目标图像和伪彩图进行聚类,基于聚类结果对目标图像和伪彩图进行染色归一化处理,得到病理数据集的图像部分。
86.在一些实施例中,数据集标签获取模块包括模型构建单元;
87.模型构建单元,用于提取高光谱图像的代表性像素,基于代表性像素的通道值及标签构建病理识别模型,通过决策树算法对病理识别模型进行训练,直到输出的误差减小到期望值,得到训练好的病理识别模型。
88.在一些实施例中,数据集标签获取模块还包括标签获取单元;
89.标签获取单元,用于通过训练好的病理识别模型对高光谱图像中的像素进行识别标注,得到病理数据集的标签部分。
90.在一些实施例中,数据集图像获取模块包括图像处理单元;
91.图像处理单元,基于目标图像和伪彩图中的像素值,对目标图像和伪彩图分别进行聚类,得到聚类结果。
92.在一些实施例中,数据集图像获取模块还包括亮度值比较单元;
93.亮度值比较单元,基于聚类结果,分别计算第一亮度值和第二亮度值,将第一亮度值和第二亮度值进行比较,若第一亮度值小于第二亮度值,则将伪彩图中像素点的r、g、b的值替换成目标图像中像素点的r、g、b的值;否则不替换;基于比较结果得到病理数据集的图像部分;其中第一亮度值为伪彩图中像素点的亮度值,第二亮度值为目标图像中像素点亮
度值。
94.以上所述,仅为本技术较佳的具体实施方式,但本技术的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本技术揭露的技术范围内,可轻易想到的变化或替换,都应涵盖在本技术的保护范围之内。因此,本技术的保护范围应该以权利要求的保护范围为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1