基于深度迁移学习模型的药物分子骨架替换和筛选方法
1.本发明涉及计算机辅助药物研发领域,尤其涉及一种基于深度迁移学习模型的药物分子骨架替换和筛选方法。
背景技术:
2.药物研发的总体过程耗时长、成本高、成功率低。从获得临床批件到药物上市过程中的临床失败率超过90%,而推进一个新的治疗药物到上市审批的相关成本预计高达20亿美元以上,且平均需要15年。早期的计算机辅助药物设计方法主要有定量构效关系(qsar)模型,即利用回归模型来寻找分子描述符合生物活性之间的联系。与此同时,机器学习方法(如支持向量机、随机森林和决策树)已被应用于药物发现任务中。但由于特征提取较少,整体预测效果不佳。此外,传统的“骨架跃迁”概念强调骨架跃迁的两个关键组成部分:新化合物相对于模板化合物具有不同的骨架结构,但有相似的生物活性。骨架跃迁的原理是结构相似的分子其活性也相似。但是,传统的骨架跃迁方法存在结构相似性与活性结果的相关性不足、新骨架合成难度和成本较高、骨架新颖性较低等问题。
3.随着人工智能技术的蓬勃进步,人工智能赋能药物研发可以极大缩短药物研发周期,大幅降低企业时间成本和研发成本,提高药物研发成功率,因此人工智能+药物研发的企业新赛道正在逐渐产生和发展。近年来,深度学习这一新兴的人工智能技术的进步,加速和改善了药物发现过程,并在分子性质预测、虚拟筛选、逆合成分析和分子生成模型等应用中取得了令人瞩目的成果。与传统的“浅层”机器学习方法相比,深度学习采用的是具有多个隐藏层的深度神经网络,可以表征和学习更复杂的知识。目前,将基于消息传递神经网络的深度学习方法引入骨架活性预测是解决传统骨架跃迁方法存在问题的有效方法。申请号为201910818097x的中国专利文件公开了一种基于深度学习的药物分子生成方法,该方法学习已知类药分子的语义和特征后得到预训练模型,然后使用该预训练模型在迁移训练阶段学习某个具体靶点的药物分子的特征得到迁移训练模型,生成分子数据。该方法深度学习的性能很大程度上仍取决于训练数据的大小。在有限的假设类中,较大的样本量会产生更准确的模型。然而,在药物发现过程的每个阶段,由于标签数据的产生需要耗时且昂贵的实验,标签数据的规模都很小,通常从十到几万不等。
技术实现要素:
4.针对上述存在的问题,本发明提供了一种基于深度迁移学习模型的药物分子骨架替换和筛选方法,通过结合药物靶点上下游和迁移学习任务上下游的双上下游策略,解决了基于小样本数据集训练深度学习模型局限性带来的准确性不足、收敛度不高、训练较难的问题。
5.本发明为解决上述技术问题采用的技术方案如下:
6.基于深度迁移学习模型的药物分子骨架替换和筛选方法,包括如下步骤:
7.步骤一,获取源域数据集和目标域数据集;
8.步骤二,将待筛选化合物以及步骤一所获得的源域数据集和目标域数据集的对应化合物分别转变为化合物骨架;
9.步骤三,输入步骤二所获得的源域数据集对应化合物骨架的简化分子线性输入规范格式文本和生物活性数据到图神经网络模型中预训练得到网络模型;
10.步骤四,将步骤二所获得的目标域数据集对应化合物骨架的简化分子线性输入规范格式文本和生物活性数据输入到步骤三获得的预训练好的网络模型中,并对该网络模型中参数进行微调,得到新的参数及模型;
11.步骤五,基于步骤四得到的针对目标域数据集的深度学习网络模型对待筛选化合物进行骨架综合筛选,获得分子骨架综合得分,筛选出分子骨架;
12.步骤六,将上述筛选出的分子骨架替换到目标小分子化合物中,获得系列骨架替换后的全新分子。
13.进一步的,所述目标域数据集是小样本数据集,源域数据集是目标域关联任务的大样本数据集;所述源域数据集和目标域数据集可以从pubchem、chembl、bindingdb等公开数据库和公开文献中获得。
14.进一步的,所述步骤二具体包括:使用开源包rdkit中基于的murcko骨架聚类化合物库对源域化合物、目标域化合物和待筛选化合物进行骨架提取。
15.进一步的,所述图神经网络模型包括定向消息传递神经网络模块、前馈神经网络模块,所述定向消息传递神经网络模块用于提取分子特征;所述前馈神经网络模块用于完成分子性质的分类和预测。
16.进一步的,在将源域数据集对应化合物骨架的简化分子线性输入规范格式文本,即smiles文本输入到图神经网络模型中前,使用开源包rdkit对输入的源域数据集对应化合物smiles文本进行计算,获得对应的小分子化合物的原子特征xv和化学键特征e
vw
,作为图神经网络模型的最初输入特征,;
17.所述定向消息传递神经网络模块中,在进行消息传递前,先对其初始隐藏层进行初始化计算:
[0018][0019]
其中,τ为relu激活函数,wi为可学习的矩阵参数,cat(xv,e
vw
)是将原子特征与化学键特征合并为相应矩阵;
[0020]
然后在定向消息传递的每一步骤t上,对化合物原子特征的隐藏层和传递信息以及化学键特征的隐藏层和传递信息进行更新;
[0021][0022][0023][0024]hv
=τ(w
a cat(xv,mv))
[0025]
然后首先对原子的隐藏层进行求和,获得分子的特征向量h:
[0026][0027]
最终通过进行化合物性质预测,其中f(.)是一个前馈神经网络模块,进而得到预训练好的图神经网络模型。
[0028]
进一步的,所述步骤四中对预训练好的网络模型中参数进行微调得到新的参数及模型的具体内容包括:将预训练好的图神经网络模型的输出层随机初始化,保持其他层的框架不变,输入目标域数据集对应化合物骨架的简化分子线性输入规范格式文本和生物活性数据,对网络模型各层参数进行微调,得到新的针对目标域小样本关联任务的预测模型。
[0029]
进一步的,所述对网络模型各层参数进行微调包括创建一个新的神经网络,即目标模型,然后将基于源域数据集所训练的模型中除最终的前馈神经网络模块以外的所有模型设计及其参数迁移到目标模型上;然后向所述目标模型增加输出层,该输出层结构与基于源域数据集所训练的模型的输出层结构相同但初始参数随机更新;然后使用步骤二所获得的目标域数据集化合物对应骨架对所述目标模型进行训练;输出层从头开始训练,对其它层参数进行微调,最终获得针对目标域小样本关联任务的预测模型。
[0030]
进一步的,步骤五中所述获得分子骨架综合得分的具体内容包括:首先基于步骤四得到的针对目标域小样本关联任务的深度学习网络预测模型对待筛选化合物库进行活性预测,获得预测生物活性值;对预测生物活性值排序前20%小分子化合物骨架取归一化的分子对接打分、类药性打分和合成难度打分的平均值作为最终的分子骨架综合得分;然后基于所述分子骨架综合得分获得前10%小分子骨架。
[0031]
进一步的,所述待筛选化合物库为drugbank数据库。
[0032]
进一步的,所述取归一化的分子对接打分、类药性打分和合成难度打分的平均值作为最终的分子骨架综合得分的具体内容包括:
[0033]
使用autodock4.2.6软件对分子骨架及目标域靶点蛋白进行结构能量优化,然后通过全局分子对接技术获得多个评分最高的构象,然后取前5个优势构象di(i=1,2,3,4,5),计算平均评分,作为分子对接打分
[0034]
通过开源包rdkit计算分子骨架的类药性打分q
score
∈[0,1];
[0035]
通过开源包rdkit计算分子骨架的合成难度打分sa
score
∈[1,10];
[0036]
通过归一化算法使得(d
score
,q
score
,sa
score
)∈[0,1];
[0037]
所述归一化算法为:
[0038][0039]
其中,x=(d
score
,q
score
,sa
score
),max(x)和min(x)分别是预测生物活性值排序前20%小分子化合物骨架中对应指标打分最高的打分和最低的打分;
[0040]
所述分子骨架综合得分为:
[0041][0042]
其中,y是分子骨架综合得分,且y∈[0,1];x'd是归一化的分子对接打分,x'q是归
一化的类药性打分,x'
sa
是归一化的合成难度打分。
[0043]
本发明的技术方案能产生以下的技术效果:
[0044]
本发明所提供的基于深度迁移学习模型的药物分子骨架筛选和替换方法通过结合药物靶点上下游和迁移学习任务上下游的双上下游策略,帮助小样本量数据的上下游靶点建立全新分子骨架筛选和替换模型,解决了小样本数据集的分子性质预测效果差的问题,通过引入迁移学习思路,提取预训练网络特征值并微调,达到对小样本数据集有更好的训练效果,为新骨架合成难度及成本较高、骨架新颖性较低等问题的解决提供了应用思路。本发明训练出来的模型可以广泛应用于抗肿瘤信号通路靶点、抗病毒靶点等靶向药物分子骨架筛选和替换任务,帮助数据稀缺靶点分子设计提供全新药物分子骨架。
附图说明
[0045]
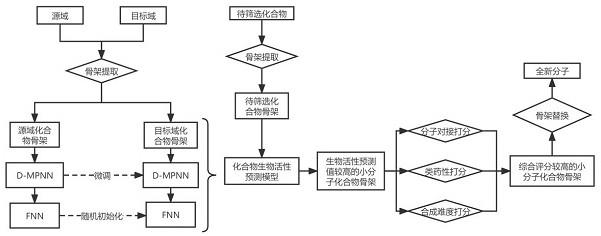
图1为本发明的基于深度迁移学习模型的药物分子骨架替换和筛选方法的算法框架和计算流程。
[0046]
图2为本发明的实施例中筛选出的部分潜在分子骨架。
具体实施方式
[0047]
为使本发明实施例的目的、技术方案和优点更加清楚,下面将结合本技术具体实施例及对应的附图对本发明的技术方案进行清楚、完整地描述。显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。
[0048]
如图1所示,本发明所述的基于深度迁移学习模型的药物分子骨架替换和筛选方法的整体流程如下:
[0049]
s1、获取源域数据集和目标域数据集的对应化合物
[0050]
所述目标域数据集为小样本数据集,源域数据集为大样本数据集;其中,源域是目标域关联任务的数据集。源域和目标域数据从pubchem、chembl、bindingdb等公开数据库和公开文献中获得。以二分类变量(active=1,inactive=0)定义药物分子生物活性标签,其中ic
50
》10μm定义为inactive,ic
50
《10μm定义为active。
[0051]
s2、将待筛选化合物及所获得的源域数据集和目标域数据集的对应化合物转变为化合物骨架
[0052]
使用开源包rdkit中基于的murcko骨架聚类化合物库对源域数据集和目标域数据集的对应化合物以及待筛选化合物进行骨架提取,获得后续计算所需要的源域、目标域以及待筛选的化合物骨架。通过此方法可以去除化合物多余的侧链并仅表达化合物的骨架结构,便于从骨架层面对化合物进行表达。所获得的源域和目标域对应分子骨架数据集即为训练集用于训练模型。
[0053]
s3、输入步骤二所获得的源域数据集对应化合物骨架的简化分子线性输入规范格式(即smiles)文本和生物活性数据到图神经网络模型中预训练得到网络模型
[0054]
建立图神经网络模型,所述图神经网络模型包括定向消息传递神经网络(d-mpnn)模块和前馈神经网络(fnn)模块,其中d-mpnn模块被用来提取和学习分子特征,fnn模块用于完成分子性质的分类和预测;当消息传递神经网络将训练集分子的smiles字符串作为输入文本,然后通过开源包rdkit在内部将其转换为图形进行分子表征。与此同时,d-mpnn模
块在分子中提取和学习分子特征化后,fnn模块使用其学习到的分子特征来构建分类器并预测分子特性,最终得到预训练好的网络模型。
[0055]
具体地,通过使用开源包rdkit对输入的小分子化合物smiles文本进行计算,获得对应的小分子化合物的原子特征xv和化学键特征e
vw
,并作为模型的最初输入特征。
[0056]
所述定向消息传递神经网络模块中,在进行消息传递前,先对其初始隐藏层进行初始化计算:
[0057][0058]
其中,τ为relu激活函数,wi为可学习的矩阵参数,cat(xv,e
vw
)是将原子特征与化学键特征合并为相应矩阵;
[0059]
然后在定向消息传递的每一步骤t上,对化合物原子特征的隐藏层和传递信息以及化学键特征的隐藏层和传递信息进行更新。
[0060][0061][0062][0063]hv
=τ(w
a cat(xv,mv))
[0064]
然后首先对原子的隐藏层进行求和,获得分子的特征向量h:
[0065][0066]
最终通过进行化合物性质预测,其中f(.)是一个前馈神经网络模块,进而得到预训练好的图神经网络模型。
[0067]
s4,将目标域数据集对应化合物骨架的smiles文本和对应靶点的生物活性数据输入到上述获得的预训练好的网络模型中,并对该网络模型中参数进行微调,得到新的参数及模型
[0068]
所述对该网络模型中参数进行微调是指创建一个新的神经网络,即目标模型;在目标模型中,除最终用于预测化合物性质的fnn模块以外,将上述基于源域数据集所训练得到的模型中的所有模型设计及其参数迁移到该目标模型上;进一步地,向目标模型增加输出层,其结构与基于源域所训练的模型的输出层结构相同但初始参数随机更新。然后使用目标域化合物对应骨架对目标模型进行训练;输出层从头开始训练,而其他层参数进行微调。最终,获得针对目标域数据集的深度学习网络模型,即为针对目标域小样本关联任务数据集的预测模型。
[0069]
s5,基于步骤s4得到的针对目标域数据集的深度学习网络模型对待筛选化合物进行骨架综合筛选,获得分子骨架综合得分,进而筛选出分子骨架;
[0070]
首先基于上述针对目标域数据集的深度学习网络预测模型对drugbank数据库进行活性预测,获得预测生物活性值;所述预测方法优选分类变量,得到预测生物活性值是一
个0《score《1的数值,表示该骨架有活性(label=1)的概率值。
[0071]
然后对预测生物活性值排序前20%小分子化合物骨架取归一化的分子对接打分、类药性打分和合成难度打分的平均值作为最终的分子骨架综合得分;然后基于所述分子骨架综合得分获得前10%小分子骨架。
[0072]
其中,所述取得分子骨架综合得分的具体内容包括:
[0073]
使用autodock4.2.6软件对分子骨架及目标域靶点蛋白进行结构能量优化,然后通过全局分子对接技术获得多个评分最高的构象,然后取前5个优势构象di(i=1,2,3,4,5),计算平均评分,作为分子对接打分
[0074]
通过开源包rdkit计算分子骨架的类药性打分q
score
∈[0,1];
[0075]
通过开源包rdkit计算分子骨架的合成难度打分sa
score
∈[1,10];
[0076]
通过归一化算法使得(d
score
,q
score
,sa
score
)∈[0,1];
[0077]
所述归一化算法为:
[0078][0079]
其中,x=(d
score
,q
score
,sa
score
),max(x)和min(x)分别是预测生物活性值排序前20%小分子化合物骨架中对应指标打分最高的打分和最低的打分;
[0080]
所述分子骨架综合得分为:
[0081][0082]
其中,y是分子骨架综合得分,且y∈[0,1];x'd是归一化的分子对接打分,x'q是归一化的类药性打分,x'
sa
是归一化的合成难度打分。本实施例中部分筛选出的骨架如图2所示。
[0083]
s6,将筛选出的分子骨架替换到目标小分子化合物中,获得系列骨架替换后的全新分子。
[0084]
为了验证本发明的有效性,本实施例采用目标域靶点是b1,源域靶点a1是抗肿瘤信号通路上目标域靶点b1下游大样本数据集靶点;和目标域靶点是b2,源域靶点a2是抗肿瘤信号通路上目标域靶点b2下游大样本数据集靶点这两组关联任务数据集作为输入,分别作为抗肿瘤信号通路靶点和抗病毒靶点进行了验证;其中各靶点集数据输入信息如表1所示:
[0085]
表1各靶点集数据输入信息
[0086][0087]
结合本实施例所提供的基于深度迁移学习模型的药物分子骨架筛选和替换方法和上述两组关联任务数据集信息,得到本方法在两组数据集与不同算法的分类性能比较,比较结果如表2所示。
[0088]
表2本实施例方法在两组数据集与不同算法的分类性能比较
[0089][0090][0091]
如表2,本实施例的预测模型性能结果显示,本发明所提供的基于深度迁移学习模型的药物分子骨架筛选和替换方法通过结合药物靶点上下游和迁移学习任务上下游的双上下游策略,显著提高了小样本量数据的上/下游靶点药物分子骨架筛选模型的预测性能,解决了小样本数据集的分子性质预测效果差的问题。并且,本实施例说明了本发明训练出来的模型在抗肿瘤信号通路靶点、抗病毒靶点等靶向药物分子骨架筛选和替换任务上都具
有极好的预测表现,帮助数据稀缺靶点分子设计提供全新药物分子骨架。
[0092]
以上详细描述了本发明的较佳具体实施例,并不对本发明起到任何限制作用。任何所属技术领域的技术人员,在不脱离本发明的技术方案的范围内,对本发明揭露的技术方案和技术内容做任何形式的等同替换或修改等变动,均属未脱离本发明的技术方案的内容,仍属于本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1