通过低深度全基因组测序技术预测髓母细胞瘤分型的计算机可读存储介质和装置及其应用的制作方法
本发明涉及诊断领域中的通过低深度全基因组测序技术预测髓母细胞瘤分型的计算机可读存储介质和装置及其应用。
背景技术:
1、髓母细胞瘤是儿童最常见的脑肿瘤,死亡率高。2010年,在波士顿召开的共识会议上,科学界就其分子亚型(wnt、shh、group 3和group 4)达成了共识。研究表明,不同的分子亚型表现出不同的基因型特征和预后。目前,通过nanostring技术检测肿瘤样本的转录组数据,并利用该转录组rna表达数据对髓母细胞瘤进行分子分型的方法已被广泛接受。但肿瘤样本在长时间的存储过程中,rna降解会增加分析的失败率,并且nanostring技术通量较低,检测费用昂贵。
技术实现思路
1、本发明所要解决的技术问题是如何基于dna全基因组低深度测序数据对髓母细胞瘤进行分子分型。
2、为了解决上述技术问题,本发明首先提供了预测髓母细胞瘤患者分子亚型的计算机可读存储介质,所述计算机可读存储介质存储有计算机程序,所述计算机程序使计算机执行如下步骤:
3、a1)获得已知分子亚型髓母细胞瘤样本的测序数据、年龄特征和性别特征;
4、a2)对所述已知分子亚型髓母细胞瘤样本的测序数据进行cnv检测获得已知分子亚型髓母细胞瘤样本的cnv结果,对所述已知分子亚型髓母细胞瘤样本的cnv结果进行高频cnv检测获得已知分子亚型髓母细胞瘤样本的arm-level scna和focal scna结果;
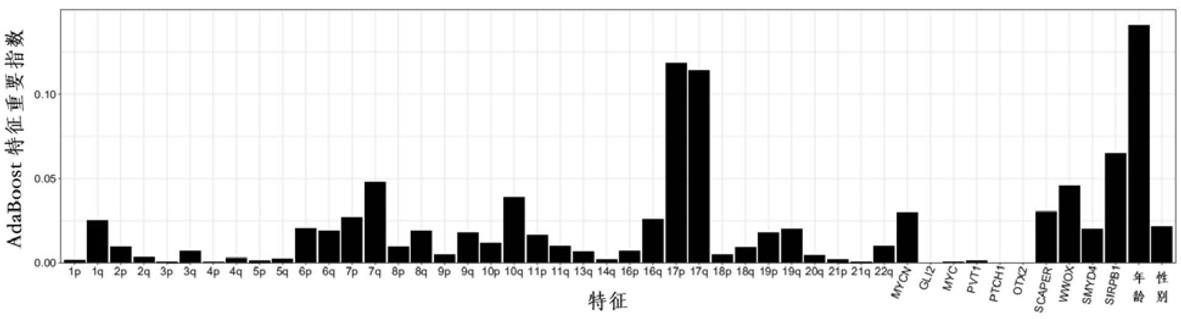
5、a3)基于48个特征,使用机器学习算法构建髓母细胞瘤分子分型模型;所述48个特征包括从所述arm-level scna和focal scna结果中获取的36个染色体臂水平拷贝数变异特征和10个基因水平拷贝数变异特征,以及年龄特征和性别特征;所述36个染色体臂包括:1p,1q,2p,2q,3p,3q,4p,4q,5p,5q,6p,6q,7p,7q,8p,8q,9p,9q,10p,10q,11p,11q,13q,14q,16p,16q,17p,17q,18p,18q,19p,19q,20q,21p,21q和22q;所述p代表染色体短臂,所述q代表染色体长臂;所述10个基因包括:mycn,gli2,myc,pvt1,otx2,scaper,wwox,sirpb1,ptch1和smyd4;
6、a4)获取待测髓母细胞瘤患者的测序数据;将所述测序数据比对到参考基因组获得比对结果文件;
7、a5)对所述比对结果文件进行cnv检测获得待测髓母细胞瘤患者cnv结果;将所述待测髓母细胞瘤患者的cnv结果和所述已知分子亚型髓母细胞瘤样本的cnv结果进行合并获得合并cnv结果;检测所述合并cnv结果,获得高频cnv结果;基于所述高频cnv结果提取待测髓母细胞瘤患者的arm-level scna和focal scna结果;
8、a6)基于所述待测髓母细胞瘤患者的arm-level scna和focal scna结果以及年龄和性别信息提取待测髓母细胞瘤患者的所述48个特征,基于所述待测髓母细胞瘤患者的所述48个特征使用所述髓母细胞瘤分子分型模型预测待测髓母细胞瘤患者分子亚型分型。
9、上述计算机可读存储介质中,所述1p具体可指1号染色体短臂。所述1q具体可指1号染色体长臂。所述染色体臂水平拷贝数变异特征可包括染色体臂缺失特征或染色体臂扩增特征。所述10个基因具体可为mycn(nc_000002.12,feb 3, 2014),gli2(nc_000002.12,feb 3, 2014),myc(ng_007161.2, sep 20, 2017),pvt1(nc_000008.11,feb 3, 2014),otx2(nc_000014.9,feb 3, 2014),scaper(nc_000015.10,feb 3, 2014),wwox(nc_000016.10, feb 3, 2014),sirpb1(nc_000020.11, feb 3, 2014),ptch1(nc_000009.12,feb 3, 2014)和smyd4(nc_000017.11, feb 3, 2014)。所述基因水平拷贝数变异特征可包括基因缺失特征或基因扩增特征。
10、上述计算机可读存储介质中,所述待测髓母细胞瘤患者的测序数据可为低深度全基因组测序数据。所述低深度可为所述测序数据的测序深度大于等于2。
11、上述计算机可读存储介质中,所述机器学习算法可为朴素贝叶斯、随机森林、adaboost迭代算法、逻辑回归或支持向量机。
12、上述计算机可读存储介质中,所述机器学习算法可为支持向量机。所述支持向量机的核函数可为线型核函数kernel='linear'。所述支持向量机的其余参数可为默认参数。所述朴素贝叶斯、随机森林、adaboost迭代算法或逻辑回归的参数可为默认参数。
13、上述计算机可读存储介质中,所述已知分子亚型髓母细胞瘤样本的测序数据可为芯片测序数据。所述cnv检测可为使用dnacopy和/或readdepth软件;所述高频cnv检测可为使用gistic2软件进行检测。
14、为了解决上述技术问题,本发明还提供了预测髓母细胞瘤患者分子亚型的装置,所述装置可包括如下模块:
15、b1)已知髓母细胞瘤样本数据获得模块:用于获得已知分子亚型髓母细胞瘤样本的测序数据、年龄特征和性别特征;
16、b2)已知髓母细胞瘤样本的高频cnv检测模块:用于基于所述已知分子亚型髓母细胞瘤样本的测序数据检测获得已知分子亚型髓母细胞瘤样本的cnv结果,基于所述已知分子亚型髓母细胞瘤样本的cnv结果进行高频cnv检测获得已知分子亚型髓母细胞瘤样本的arm-level scna和focal scna结果;
17、b3)髓母细胞瘤分类模型构建模块:用于基于48个特征,使用机器学习算法构建髓母细胞瘤分子分型模型;所述48个特征包括从所述arm-level scna和focal scna结果中获取的36个染色体臂水平拷贝数变异特征和10个基因水平拷贝数变异特征,以及年龄特征和性别特征;所述36个染色体臂包括:1p,1q,2p,2q,3p,3q,4p,4q,5p,5q,6p,6q,7p,7q,8p,8q,9p,9q,10p,10q,11p,11q,13q,14q,16p,16q,17p,17q,18p,18q,19p,19q,20q,21p,21q和22q;所述p代表染色体短臂,所述q代表染色体长臂;所述10个基因包括:mycn,gli2,myc,pvt1,otx2,scaper,wwox,sirpb1,ptch1和smyd4;
18、b4)待测髓母细胞瘤患者cnv检测模块:用于获取待测髓母细胞瘤患者的测序数据;基于所述测序数据与参考基因组的比对结果获得待测髓母细胞瘤患者的比对结果文件;基于所述比对结果文件检测待测髓母细胞瘤患者cnv结果;
19、b5)高频cnv检测模块:用于将所述待测髓母细胞瘤患者的cnv结果和所述已知分子亚型髓母细胞瘤样本的cnv结果进行合并获得合并cnv结果;检测所述合并cnv的高频cnv结果;基于所述高频cnv结果提取待测髓母细胞瘤患者的arm-level scna和focal scna结果;
20、b6)待测髓母细胞瘤患者分子亚型预测模块:用于基于所述待测髓母细胞瘤患者的arm-level scna和focal scna结果提取待测髓母细胞瘤患者的所述48个特征,基于所述待测髓母细胞瘤患者的所述48个特征使用所述髓母细胞瘤分子分型模型预测待测髓母细胞瘤患者分子亚型分型。
21、上述装置中,所述1p具体可指1号染色体短臂。所述1q具体可指1号染色体长臂。所述染色体臂水平拷贝数变异特征可包括染色体臂缺失特征或染色体臂扩增特征。所述10个基因具体可为mycn(nc_000002.12,feb 3, 2014),gli2(nc_000002.12,feb 3, 2014),myc(ng_007161.2, sep 20, 2017),pvt1(nc_000008.11,feb 3, 2014),otx2(nc_000014.9,feb 3, 2014),scaper(nc_000015.10,feb 3, 2014),wwox(nc_000016.10, feb 3,2014),sirpb1(nc_000020.11, feb 3, 2014),ptch1(nc_000009.12, feb 3, 2014)和smyd4(nc_000017.11, feb 3, 2014)。所述基因水平拷贝数变异特征可包括基因缺失特征或基因扩增特征。
22、上述装置中,所述待测髓母细胞瘤患者的测序数据可为低深度全基因组测序数据;所述低深度可为所述测序数据的测序深度大于等于2。
23、上述装置中,所述机器学习算法可为朴素贝叶斯、随机森林、adaboost迭代算法、逻辑回归或支持向量机。
24、上述装置中,所述机器学习算法具体可为支持向量机;所述支持向量机的核函数可为线型核函数kernel='linear'。所述支持向量机的其余参数可为默认参数。所述朴素贝叶斯、随机森林、adaboost迭代算法或逻辑回归的参数可为默认参数。
25、所述已知分子亚型髓母细胞瘤样本的测序数据可为芯片测序数据。所述cnv检测可为使用dnacopy和/或readdepth软件;所述高频cnv检测可为使用gistic2软件进行检测。
26、上文所述的计算机可读存储介质的下述任一种应用也属于本发明的保护范围:
27、c1)在制备预测髓母细胞瘤患者分子亚型分型的产品中的应用;
28、c2)在开发或制备治疗或缓解髓母细胞瘤的药物中的应用;
29、c3)在开发或制备髓母细胞瘤指导药物的产品中的应用;
30、c4)在制备预测髓母细胞瘤患者分子亚型分型的预后的产品中的应用。
31、上文所述的装置的下述任一种应用也属于本发明的保护范围:
32、d1)在制备预测髓母细胞瘤患者分子亚型分型的产品中的应用;
33、d2)在开发或制备治疗或缓解髓母细胞瘤的药物中的应用;
34、d3)在开发或制备髓母细胞瘤指导药物的产品中的应用;
35、d4)在制备预测髓母细胞瘤患者分子亚型分型的预后的产品中的应用。
36、本发明提供了一种基于dna 全基因组低深度(大于等于2×深度)测序技术,使用机器学习算法实现髓母细胞瘤的分子分型。
37、本发明的技术方案中包括模型训练和样本检测两部分,模型训练部分使用公共数据进行,样本检测部分使用临床收集到的髓母细胞瘤样本。
38、以下是模型训练部分:本发明从geo(gene expression omnibus)中检索到gse37385(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=gse37385)数据集,共1097例髓母细胞瘤snp阵列cel数据和临床信息。经过一系列的质量评估后,保留800个样本,作为机器学习模型的训练集(640个样本)和测试集(160个样本)。此外,本发明从robinson g (robinson, g., parker, m., kranenburg, t. et al. novel mutationstarget distinct subgroups of medulloblastoma. nature 488, 43–48 (2012). )等人的研究中收集了32个髓母细胞瘤样本作为验证集。
39、对于以上训练集、测试集、验证集的样本,使用penncnv软件(wang k, li m,hadley d. et al. penncnv: an integrated hidden markov model designed for high-resolution copy number variation detection in whole-genome snp genotypingdata genome research 17:1665-1674, 2007)计算每个样本的log r ratio(lrr)和b等位基因频率(baf),作为 dnacopy软件(seshan ve, olshen a.dnacopy: dna copy numberdata analysis. r package version 1.72.3.)的输入数据,分析cnv(copy numbervariant拷贝数变化),获得每个样本的cnv结果。
40、使用gistic2软件(mermel ch, schumacher se, hill b, meyerson ml. et al.gistic2.0 facilitates sensitive and confident localization of the targetsof focal somatic copy-number alteration in human cancers. genome biol. 2011;12(4):r41.)分析上述样本的cnv数据,获取每个样品的高频cnv结果:即arm-level scna(arm-level somatic copy number alteration染色体臂水平体细胞拷贝数变化,定义为发生拷贝数变化的长度大于等于染色体臂长度的50%)和focal scna(focal somatic copynumber alteration局部体细胞拷贝数变化,定义为发生拷贝数变化的长度小于染色体臂长度的50%,本发明focal scna限定在基因水平的长度)。使用gistic2软件分析高频cnv需要大量样本队列构建背景突变频率,训练集640例样本作为一个队列进行分析,测试集以及验证集的每一个样本和训练集640样本组成一个641个样本队列进行gistic2分析,获得每个样本的arm-level scna和focal scna。
41、以gistic2得到的高频cnv以及年龄和性别作为输入特征数据,采用五倍交叉验证(训练集640例样本,测试集160例样本),使用adaboost选择出重要的48个特征。基于这48个特征分别使用naive bayes、random forest、adaboost、logistic regression和supportvector machine(svm,支持向量机)五种机器学习分类算法模型,将样本分成wnt,shh,group3,group4 四个亚型,并用auroc评估每个算法性能。结果显示,svm机器学习分类算法模型综合性能优于其他四种分类算法,因此最终选择svm作为髓母细胞瘤样本分子分型模型的算法。
42、以下是样本检测部分:对于临床待测髓母细胞瘤组织样本,获得测序数据,数据质控合格后,首先分析得到cnv数据,将该样本和训练集的640例样本组成一个队列,使用gistic2分析,获取该待测样本的arm-level scna和focal scna特征,加入该样本的年龄和性别信息,使用svm分类算法模型对样本进行分子分型,wnt,shh,group3,group4每个分子亚型都得到一个概率,如果最大两个亚型概率之差大于等于0.1,那么最终结果是概率最大的亚型,如果概率之差小于0.1,则无法确定分子分型结果。
43、本发明由于采取以上技术方案,其具有以下优点:
44、1、只需dna 低深度wgs测序数据,对于样本要求较低,容易获取样本;
45、2、使用五种机器学习模型中性能最优的svm,预测准确性高。
46、上文所述dna测序数据可为测序深度大于等于2×的测序数据。
- 还没有人留言评论。精彩留言会获得点赞!