病原体耐药单碱基突变检测方法、装置、设备及存储介质与流程
本技术涉及基因检测,特别是涉及一种病原体耐药单碱基突变检测方法、装置、设备及存储介质。
背景技术:
1、微生物耐药性是指病原体微生物为了生存获得了抵抗药物效果能力。耐药性一旦发生意味着病原体将不能被完全杀死并且病原体的增殖将不可被抑制。由耐药性病原体感染造成的疾病将很难甚至不能被治愈。病原体耐药性严重影响着世界公共健康,中国是世界上耐药性发生率较高的国家,这与抗生素的过度使用有关,在中国70%的住院治疗和20%的院外治疗的病人使用抗生素治疗,这个比例是who推荐抗生素使用比例的两倍。儿童尤其是新生儿由于自身免疫力较差,因此受耐药性病原体的影响更加严重。对耐药性病原体进行研究、统计和预测能帮助我们更好的理解耐药性发生的机制,进而能够指导我们更好的对抗耐药病原体对人类的侵害。
2、目前关于对细菌耐药性的检测主要有以下几种方法:细菌耐药表型的检测(包括耐药筛选试验、折点敏感试验等),β-内酰胺酶检测,特殊耐药菌检测和耐药基因检测等。其中最为直观的检测方法是以体外培养的药物检测,其缺陷是培养时间长。随着耐药机理的研究逐步深入,分子生物学方法检测细菌耐药逐渐被临床接受。常见检测细菌耐药基因的方法主要有:聚合酶链式反应(pcr)、pcr-限制性片段长度多态性分析(pcr-rflp)、pcr-单链构象多态性分析(pcr-sscp)、生物芯片技术、dna测序等。传统的基因鉴定方法为pcr法和dna测序法,其中传统的pcr法检测周期较长,通常需要3-4个小时;sanger测序法对引物特异性要求较高,且价格较昂贵,后期数据分析较复杂,不适合临床推广使用。另外,随着测序技术的发展,基于二代测序及三代测序病原体耐药性分析算法也被开发出来。三代测序由于成本偏高,目前并不适合进行大样本量的分析。二代宏基因组测序由于其高效性及成本优势,已经越来越多的被应用于病原体检测领域。多种基于二代宏基因组测序的耐药同源基因分析方法也相继被开发出来。
3、而单碱基变异位点也是病原体产生耐药性的原因之一。目前基于二代测序的微生物snps分析方法主要都是基于单个物种的测序数据。二代宏基因组测序中,单个物种的数据往往偏少,这往往会造成测序数据的覆盖度的不足,从而影响基于mngs数据对单个物种进行snp检测的准确性,导致无法有效鉴定单碱基突变位点。
技术实现思路
1、有鉴于此,本技术提供一种病原体耐药单碱基突变检测方法、装置、设备及存储介质,以解决现有二代宏基因组测序方式无法有效鉴定单碱基突变位点的问题。
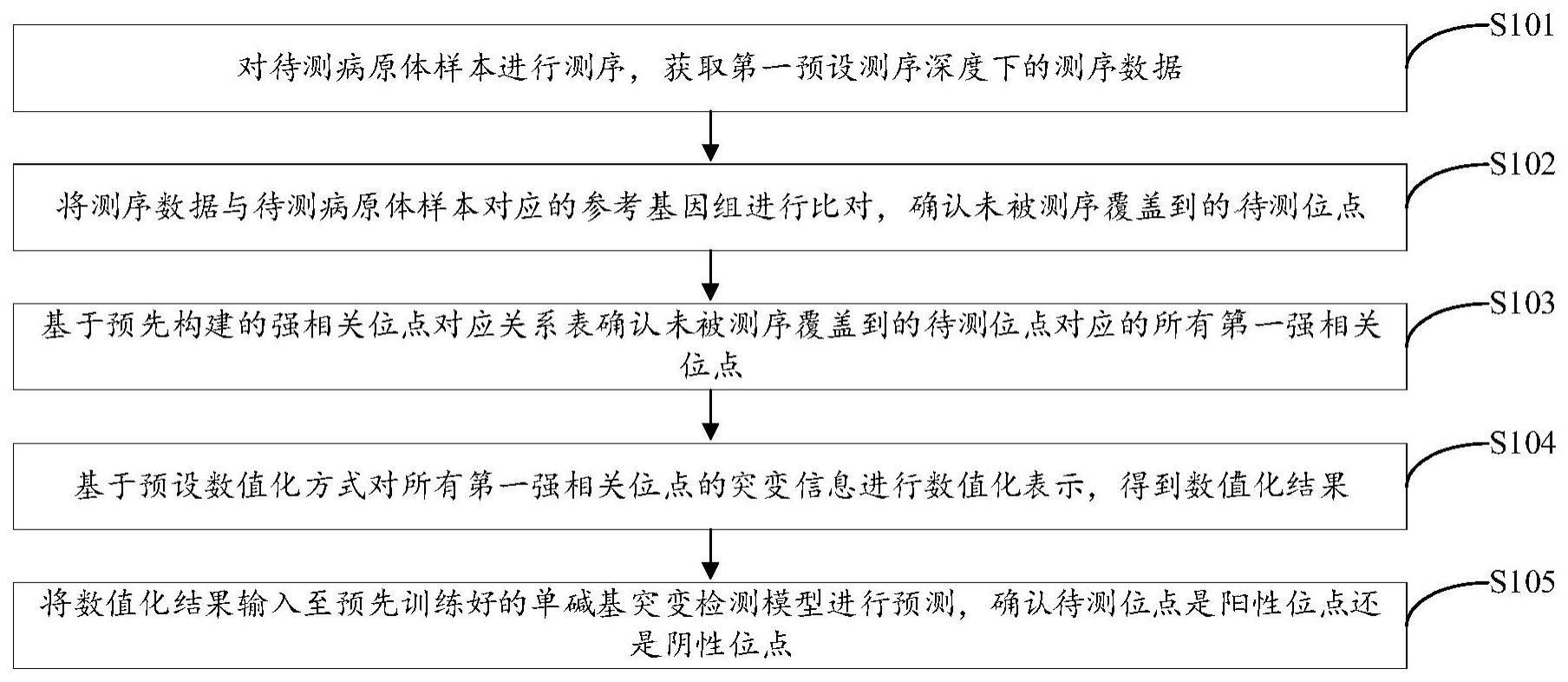
2、为解决上述技术问题,本技术采用的一个技术方案是:提供一种病原体耐药单碱基突变检测方法,其包括:对待测病原体样本进行测序,获取第一预设测序深度下的测序数据;将测序数据与待测病原体样本对应的参考基因组进行比对,确认未被测序覆盖到的待测位点;基于预先构建的强相关位点对应关系表确认未被测序覆盖到的待测位点对应的所有第一强相关位点;基于预设数值化方式对所有第一强相关位点的突变信息进行数值化表示,得到数值化结果;将数值化结果输入至预先训练好的单碱基突变检测模型进行预测,确认待测位点是阳性位点还是阴性位点。
3、作为本技术的进一步改进,基于预设数值化方式对所有第一强相关位点的突变信息进行数值化表示,得到数值化结果,包括:根据测序数据与待测病原体样本对应的参考基因组的比对结果确认每个第一强相关位点是否被测序覆盖到且是否发生突变;构建第一矩阵作为数值化结果,第一矩阵的行表示待测病原体样本的所有菌株,第一矩阵的列表示第一强相关位点,其中,被测序覆盖到且发生突变的第一强相关位点赋值为1,未被测序覆盖到或未发生突变第一强相关位点赋值为0。
4、作为本技术的进一步改进,第一预设测序深度为3层测序深度或3层以上测序深度。
5、作为本技术的进一步改进,单碱基突变检测模型表示强相关位点与相关耐药单碱基突变的映射关系,单碱基突变检测模型基于耐药单碱基位点和耐药单碱基位点对应的强相关位点变异结果的数值化表示训练得到。
6、作为本技术的进一步改进,预先训练单碱基突变检测模型的步骤,包括:从数据库中获取目标菌的种分类水平下所有细菌株的基因组数据,并在第二预设测序深度下对每个细菌株进行模拟测序,得到模拟测序数据;将模拟测序数据与病原体的参考基因组进行比对,确认模拟测序结果中所有位点的变异结果;将变异结果数值化,包括:构建第二矩阵,第二矩阵的行表示病原体样本的所有菌株,第二矩阵的列表示氨基酸位点,其中,发生突变的氨基酸位点赋值为1,未发生突变的氨基酸位点赋值为0;统计数据库中记录的目标菌的耐药单碱基位点,利用第二矩阵计算每个耐药单碱基位点与其他任意一个位点的相关性,定义相关性阈值,相关性小于或等于阈值的位点定义为耐药单碱基位点的第二强相关位点;构建每个耐药单碱基位点与该耐药单碱基位点对应的第二强相关位点之间的映射关系,得到待训练的单碱基突变检测模型;根据第二强相关位点的变异结果进行数值化,包括:构建第三矩阵,第三矩阵的行表示细菌株,第三矩阵的列表示第二强相关位点,其中,发生突变的第二强相关位点赋值为1,未发生突变的第二强相关位点赋值为0;利用第三矩阵和耐药单碱基位点训练单碱基突变检测模型。
7、作为本技术的进一步改进,计算每个耐药单碱基位点与其他任意一个位点的相关性,定义相关性阈值,相关性小于或等于阈值的位点定义为耐药位点的第二强相关位点,包括:基于皮尔逊方法循环计算第二矩阵中每任意两列数组之间的皮尔逊系数,且利用预设阈值获取方式获取相关性阈值;根据皮尔逊系数和相关性阈值确认两列数组之间强相关时,确认两列数组之间对应位置的位点互为强相关位点的对应关系;根据强相关位点的对应关系确认耐药单碱基位点对应的所有第二强相关位点。
8、作为本技术的进一步改进,确认两列数组之间对应位置的位点互为强相关位点之后,还包括:将互为强相关位点的对应关系记录至强相关位点对应关系表。
9、为解决上述技术问题,本技术采用的又一个技术方案是:提供一种病原体耐药单碱基突变检测装置,其包括:测序模块,用于对待测病原体样本进行测序,获取第一预设测序深度下的测序数据;比对模块,用于将测序数据与待测病原体样本对应的参考基因组进行比对,确认未被测序覆盖到的待测位点;确认模块,用于基于预先构建的强相关位点对应关系表确认未被测序覆盖到的待测位点对应的所有第一强相关位点;数值化模块,用于基于预设数值化方式对所有第一强相关位点的突变信息进行数值化表示,得到数值化结果;预测模块,用于将数值化结果输入至预先训练好的单碱基突变检测模型进行预测,确认待测位点是阳性位点还是阴性位点。
10、为解决上述技术问题,本技术采用的再一个技术方案是:提供一种计算机设备,所述计算机设备包括处理器、与所述处理器耦接的存储器,所述存储器中存储有程序指令,所述程序指令被所述处理器执行时,使得所述处理器执行如上述任一项的病原体耐药单碱基突变检测方法的步骤。
11、为解决上述技术问题,本技术采用的再一个技术方案是:提供一种存储介质,存储有能够实现上述任一项的病原体耐药单碱基突变检测方法的程序指令。
12、本技术的有益效果是:本技术的病原体耐药单碱基突变检测方法通过获取待测病原体样本在第一预设测序深度下的测序数据,并将测序数据与参考基因组进行比对,确认其中未被测序覆盖的待测位点,再通过预先构建的强相关位点对应关系表确认该待测位点对应的所有强相关位点,根据测序数据与参考基因组之间的比对结果,结合预设数值化方式对所有强相关位点进行数值化,再将数值化的结果输入至单碱基突变检测模型进行检测,确认待测位点是阳性位点还是阴性位点,其基于病原体基因组进化过程中出现的基因连锁不平衡的现象,利用位点之间的强相关性,通过将待测位点对应的所有强相关位点的突变信息进行数值化表示,再利用数值化表示的结果来预测待测位点是否发生突变,从而对未被测序覆盖的位点的突变信息进行准确预测。
- 还没有人留言评论。精彩留言会获得点赞!