基于转换器和图同构网络的抗癌药物响应预测方法
本发明涉及一种生物信息学分析技术,特别是涉及一种抗癌药物响应预测技术。
背景技术:
1、得益于新一代基因测序技术和高通量分析技术的发展,以及人工智能、大数据等技术的应用,精准医疗已逐渐地成为新兴的概念。如何根据癌症患者的个体特征实施个性化的治疗方案是精准医疗的主要挑战之一,同时准确地预测抗癌药物响应是抗癌药物设计和精准医疗的一个关键和具有挑战性的任务。随着大量基因组学和药理学数据的积累以及高效的药物特征提取方法和组织学数据的结合,使得利用计算模型辅助药物响应预测成为可能,这大大促进癌症基因组学研究。然而,由于基因组学数据具有很大的复杂性和差异性,因此许多研究人员在研究癌细胞的基因组学数据和药物响应之间的关系上投入大量的时间和精力。一个迫切的研究任务是开发出更有效的方法,从这些复杂的数据集中提取有用的信息,并找到癌症信息与药物响应之间的联系,为精准医疗提供有价值的帮助。目前很多基于传统网络或矩阵分解的预测方法泛化能力不强,如何引进更多的特征信息、构建更优的方法和提升模型的鲁棒性有着重要的意义。
技术实现思路
1、发明目的:针对本领域目前预测方法的性能低和鲁棒性差的局限性,提出一种基于转换器和图同构网络的抗癌药物预测方法。
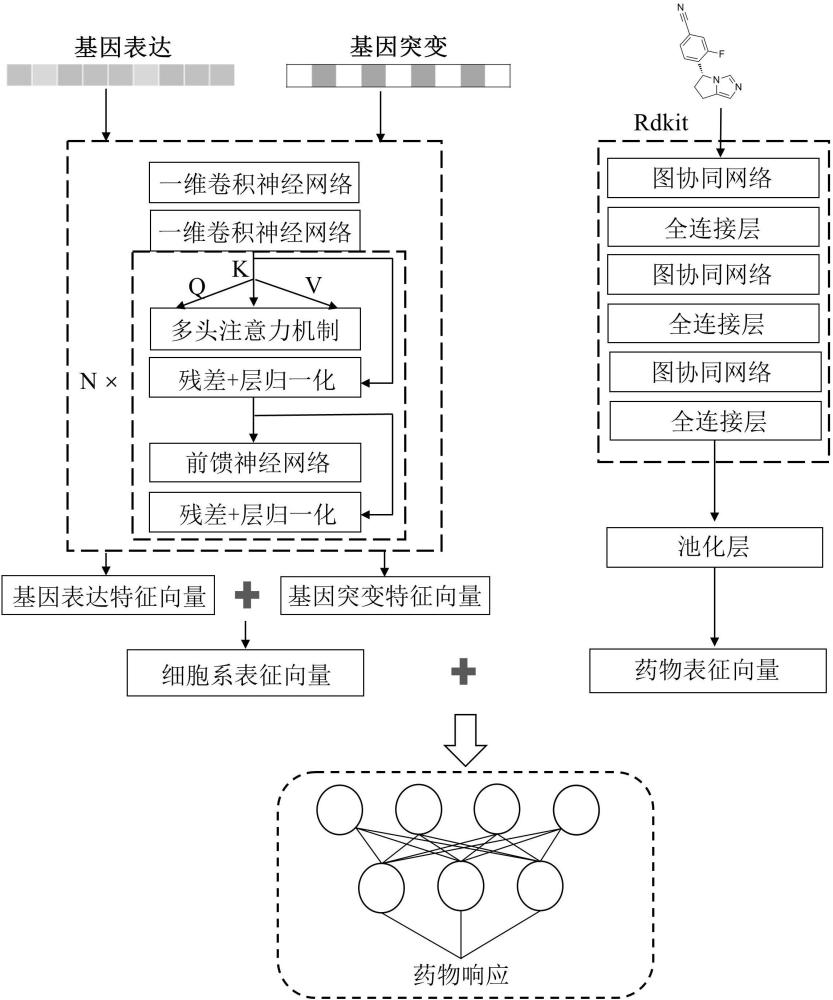
2、基于转换器和图同构网络的抗癌药物响应预测方法,其特征在于,包括以下步骤:
3、步骤1:数据收集:从癌症药物敏感性基因组学数据库获取 m个细胞系的基因表达和基因突变数据、 n个药物的标识符号和 p个已知的药物响应值,利用药物的标识符从pubchem数据库得到药物的三维化学结构表示;
4、步骤2:药物特征数据预处理:利用rdkit这一开源化学信息学软件,从药物的三维化学结构中得到药物分子的原子特征矩阵和邻接矩阵,通过填充0来使所有药物分子的原子特征矩阵具有相同的维度;
5、步骤3:细胞系特征数据预处理:首先,利用癌症与基因相关性矩阵 a,其描述癌症与基因的相关性,每行表示一个肿瘤细胞系,每列表示一个基因,矩阵元素值表示癌细胞与基因相关度大小,筛选出相关性大于0.5的基因,将筛选后的向量作为癌细胞系的特征向量;其次,利用细胞系的标志基因子集筛选出原基因特征向量中的标志基因,将由标志基因组成的向量作为癌细胞系的特征向量;再次,将两个基因向量不重合的基因进行拼接,再将其与两个基因向量重合的部分合并,通过在特征向量缺少元素的位置填充0来使所有细胞系的基因表达和突变特征向量具有相同的维度;最后,可得一个长度为 q维的基因表达和突变的向量作为最终癌细胞系的特征向量;
6、步骤4:癌细胞系的多组学特征提取:利用一维卷积神经网络和转换器编码器模块提取癌细胞系的多组学特征信息,具体操作过程为:
7、(1)利用公式(1)—(3),将细胞系的输入特征矩阵 x经过线性变换分别得到查询矩阵 q、键值矩阵 k和值矩阵 v,
8、 q=x×w q, (1)
9、 k=x×w k, (2)
10、 v=x×w v, (3)
11、其中, w q、 w k和 w v分别是其对应的线性变换矩阵;
12、(2)为防止内积过大而导致的梯度消失,利用公式(4),可得到放缩点积注意力的输出 head,
13、 head=attention( q,k,v) =softmax( qk t /sqrt( d k)) v, (4)
14、其中, d k是矩阵 q和 k的行维度, softmax(•)为激活函数, attention(•)是放缩点积注意力的输出, sqrt(•)是开平方函数;
15、(3)利用公式(5)—(7),对 q、 k和 v进行线性变换,可产生最终的注意力输出矩阵 z,
16、 z=mutilhead( q,k,v) , (5)
17、 mutilhead( q,k,v) =contact( head1, head2,..., head n) w z, (6)
18、 head i= attention( qw q i, kw k i, vw v i) , (7)
19、其中, w z是输出对应的线性变换矩阵, head i是第 i次放缩点积注意力的输出, n是多头注意力的头数, mutilhead(•)是多头注意力的输出, contact(•)是拼接操作 ;
20、(4)为解决深度网络训练过程中的梯度消失和梯度爆炸问题,利用公式(8),将 z作为残差连接和层归一化模块的输入,可得到该子模块的输出矩阵 t,
21、 t=layernorm( x+z) , (8)
22、其中, layernorm(•)是层归一化;
23、(5)将公式(8)的输出 t作为前馈神经网络的输入,利用公式(9),可得到对应的输出矩阵 ft,
24、 ft=max(0 ,tw1 +b1) w2 +b2, (9)
25、其中, w1是第一个全连接层的权重矩阵, b1是第一个全连接层的偏置矩阵, w2是第二个全连接层的权重矩阵, b2是第二个全连接层的偏置矩阵, max(•)是激活函数;
26、(6)将公式(9)的输出 ft作为残差连接和层归一化模块的输入,利用公式(10),可得到最终的特征表示矩阵 o,
27、 o=layernorm( t+ft), (10)
28、其中, o是最终的细胞系的基因表达特征矩阵或者基因突变特征矩阵,将这两个特征矩阵相加就可以得到细胞系的表征矩阵;
29、步骤5:药物的化学结构特征提取:利用由图同构网络和全连接层组成的子模块,对药物的化学结构特征进行处理,具体操作过程为:
30、(1)图同构网络的聚合方式为:
31、 h k v =mlp(( 1+ε) h h-1 v+ sum( h k-1 u), (11)
32、其中, h k v是目标节点的第 k层的表示向量, h k-1 u是目标节点的第 k-1层的表示向量, mlp(•)是全连接层;
33、(2)将经过图同构网络和全连接层组成的子模块,对药物的化学结构特征进行处理,再将处理后的特征矩阵作为药物的表征矩阵;
34、步骤6:特征拼接和预测:将长度为 k的细胞系和药物表征向量进行拼接,输入全连接层预测药物响应。
35、有益效果:本发明为克服本领域目前方法的预测性能低和鲁棒性差的局限性,提出一种基于转换器和图同构网络的抗癌药物响应预测方法。主要贡献有:(1)在细胞系特征向量中应用转换器编码器中的多头自注意力模块,可以对不同基因进行加权处理。这种加权处理能够捕捉基因之间的关联性和重要性,从而生成更有代表性和蕴含丰富信息的特征向量。(2)在图同构网络模型的每一层节点特征的更新都考虑全局的药物图结构信息,这意味着图同构网络模型能够综合考虑整个药物图的模式和特征,而不仅仅局限于局部邻居节点的信息。这种全局性的特征更新能够提高特征向量的表示能力,并有助于捕捉药物图的整体模式。
- 还没有人留言评论。精彩留言会获得点赞!